食管鳞癌淋巴结转移关键基因鉴定及预后分析
2022-01-08李孟祥程维刚冯笑山高社干齐义军
李孟祥,程维刚,陈 攀,冯笑山,高社干,齐义军
(河南科技大学 a.信息工程学院,b.临床医学院第一附属医院,中国 洛阳 471023)
食管癌是全球最常见的恶性肿瘤之一,发病率和死亡率分别位居所有恶性肿瘤的第七位和第六位[1]。2015年,中国食管癌的新发病例和死亡病例分别为24.6万例和18.8万例[2],占全球新发病例和死亡病例的43%和36.9%。从组织学上看,中国食管癌90%以上为食管鳞状细胞癌(Esophageal squamous cell carcinoma,ESCC)[3]。ESCC具有高度的侵袭和转移能力,导致大部分ESCC患者初次确诊时已发生肿瘤转移,而肿瘤转移是ESCC患者高死亡率、预后极差的主要原因之一。
食管黏膜及黏膜下层具有丰富的淋巴管网,并具有独特的淋巴引流方式,极易发生食管内广泛或跳跃性转移及颈、胸、腹三野淋巴结转移。淋巴结转移与肿瘤浸润深度和肿瘤分化程度密切相关,术中淋巴结清扫数目、淋巴结转移阳性数目、淋巴结转移阳性率等是判定ESCC预后的重要独立危险因素[4-6]。此外,淋巴结转移也是ESCC TNM分期中重要的病理特征之一,直接影响ESCC患者术后治疗方案的选择[7]。因此,通过基因差异表达谱分析,确定与淋巴结转移高度相关的分子变异,对于ESCC个体化精准治疗极其重要。
极端梯度提升(Extreme gradient boosting,XGBoost)是基于Boosting集成的一种机器学习算法,适用于大规模数据的分布式并行运算。该算法具有高维数据处理、分析缺失值、运算效率高及可迁移性强等优点,近年来被广泛应用于数据挖掘。本研究利用XGBoost算法分析基因表达数据库(Gene expression omnibus,GEO)中ESCC mRNA转录组数据,鉴定ESCC淋巴结转移关键mRNA分子谱,构建淋巴结转移分类模型。
1 材料与方法
1.1 ESCC mRNA表达谱数据
从GEO下载数据集GSE53624和GSE53622,这两个数据集分别包括119例和60例ESCC和配对癌旁组织基因表达谱数据,基因芯片平台为GPL18109(Agilent-038314 CBC Homo sapiens lncRNA + mRNA microarray V2.0)。从https://www.agilent.com/下载平台GPL18109的探针组序列,利用GENCODE和SeqMap进行序列比对,重新注释数据,提取mRNA表达谱数据。
1.2 ESCC淋巴结转移相关mRNA鉴定
根据文献报道方法[8],为减少不同样本之间异质性的影响,将ESCC与配对癌旁组织表达值的差值作为ESCC mRNA表达谱数据。根据淋巴结转移与否,将ESCC样本分为淋巴结转移阴性组(N0组)和淋巴结转移阳性组(N1组,包括临床分期中N1,N2和N3),筛选淋巴结转移相关mRNA分子。将GSE53624中119例ESCC样本随机分为训练集(60例)和测试集(59例),GSE53622中60例ESCC样本作为独立验证组。表1显示了3个数据集中ESCC样本的人口统计学和临床病理特征,包括年龄、性别、肿瘤部位、肿瘤分级、T stage及TNM stage等。在60例训练集中,以student’s T检验P<0.05和淋巴结转移阳性组与阴性组间表达值之差大于0.5为筛选标准,鉴定ESCC淋巴结转移相关的差异表达mRNA分子。
1.3 算法简介
XGBoost算法以CART分类树为基学习器,来源于Boosting方法。在迭代过程中,后一个模型对前一个模型的误差进行校正,通过拟合残差优化目标函数,提高预测分类准确率。XGBoost对损失函数进行二阶泰勒展开,并在损失函数中加入正则项以控制目标函数的下降和模型复杂度,防止模型过拟合[9]。逻辑回归(Logistic regression,LR)是一种广义的线性模型,是在线性回归的基础上外加一层Sigmoid函数映射。支持向量机(Support vector machine,SVM)算法通过非线性映射将输入空间映射到一个高维空间,进而在高维空间中构造最优分类超平面,利用支持向量最大化几何间隔,降低分类误差。本研究分别使用R 语言中bestglm,e1701和xgboost等程序包实现LR,SVM和XGBoost模型构建。
1.4 模型预测效能评价
本文应用R 3.63进行统计学分析。使用受试者操作特征(Receiver operating characteristic,ROC)曲线比较每个预测模型的敏感性和特异性,并以ROC曲线下面积(Area under ROC curve,AUC)值评价作为预测结果,AUC值反映分类模型预测的精准度。所有ROC曲线应用pROC函数包进行计算。K-S(Kolmogorov-Smirnov)值是另一种分类模型效能的评价指标,K-S曲线将选定的阈值作为横轴,分类模型的真阳性率(True positive rate,TPR)和假阳性率(False positive rate,FPR)均绘制到纵轴,K-S值为所有TPR和FPR差值的绝对值中的最大值,K-S值的大小与分类模型区分特征的准确性呈正相关。
1.5 生物学功能及通路富集分析
对ESCC淋巴结转移关键基因进行Gene Ontology(GO)富集分析,包括细胞成分(Cellular component,CC)、生物学过程(Biological process,BP)和分子功能(Molecular function,MF),根据超几何分布检验的错误发现率(False Discovery Rate,FDR),确定关键基因的细胞定位、分子功能及参与的生物学过程。使用ClusterProfiler程序包进行富集分析及相关绘图[10]。
1.6 生存分析
采用Kaplan-Meier法绘制生存曲线,对数秩和检验(Log-Rank test)进行生存期差异显著性检验。连续变量用survminer包中函数surv_cutpoint确定最佳截断值,将连续变量简化为二分类变量。再用单因素和多因素Cox比例风险回归模型确定预后的影响因素,似然比检验(Likelihood ratio test)确定模型显著性,并根据多变量分析结果绘制森林图。应用survival函数包进行生存分析。
2 结果
2.1 ESCC淋巴结转移相关差异表达基因
分析GSE53624训练集中60例ESCC样本mRNA表达谱数据,根据t检验P<0.05且两组之间mRNA表达值均数之差>0.5为筛选条件,鉴定了ESCC淋巴结转移相关的159个差异表达基因,包括淋巴结转移阳性组中31个高表达和128个低表达的mRNA分子(图1a)。
2.2 ESCC淋巴结转移关键分子
以上述的159个ESCC淋巴结转移相关mRNA分子作为XGBoost模型的初始特征集合,网格搜索和5倍交叉验证方法确定XGBoost模型超参数,包括最大迭代次数(nrounds=200)、学习率(eta=0.1)、单棵树最大深度(max_depth=4)、最小减损函数下降值(gamma=0.2)、随机采样特征比率(colsample_bytree=0.3)以及叶子节点最小权重(min_child_weight=0.7)等,用GSE53624训练集60例ESCC样本建立ESCC淋巴结转移预测模型。根据Gain值评价159个mRNA分子重要性,图1b显示了159个差异表达mRNA的重要性分布,其中18个mRNA分子的重要性分值大于0.02,其余mRNA分子的重要性分值小于0.02。因此,本研究将重要性分值>0.02的18个mRNA分子作为ESCC淋巴结转移的关键mRNA分子,将其纳入分类模型。18个mRNA分子及其Gain值分别为:MASP1(0.068 5),ANOS1(0.063 2),CENPP(0.058 5),ABCG2(0.043 8),GALNT12(0.035 1),IP6K3(0.034 5),SLC16A5(0.032 6),MMP27(0.030 7),C6orf15(0.030 5),KRT6C(0.028 6),CXCL10(0.028 1),RIMS2(0.027 3),RPTN(0.026),LIMA1(0.025 1),KRT6B(0.024 9),TNC(0.022 6),LCE3D(0.021 8)和APLF(0.021 5)。
2.3 构建ESCC淋巴结转移分类模型
为构建ESCC淋巴结转移分类器,从重要性分值最大的两个mRNA分子开始依次增加mRNA分子,构成逐渐增大的mRNA分子集合,用XGBoost,LR和SVM算法计算2~20个mRNA组合在测试集中预测ESCC淋巴结转移的AUC值。在XGBoost算法中,重要性分值最高的两个mRNA分子模型在测试集中诊断ESCC淋巴结转移的AUC值为0.618 3,随着特征性mRNA分子增加,分类模型的诊断效能逐渐升高,18个mRNA分子模型的AUC值最大(0.793 7,图1c);而LR与SVM算法在特征分子增加过程中,最大的AUC值分别为0.713和0.714,预测结果不稳定(图1c)。
为比较本研究建立的18个mRNA分子预测ESCC淋巴结转移的效能(XGB-18 mRNA),应用同样的18个mRNA分子,建立了LR-18 mRNA和SVM-18 mRNA的分类模型。LR-18 mRNA和SVM-18 mRNA在同一的训练集上进行超参数及核函数选择。XGB-18 mRNA,LR-18 mRNA和SVM-18 mRNA 3种模型在测试集中诊断ESCC淋巴结转移的AUC值分别为0.793 7,0.676 0和0.695 8,XGB-18 mRNA模型预测ESCC淋巴结转移的效能显著高于LR模型(Z=2.35,P=0.018)和SVM模型(Z=1.60,P=0.11),其ROC曲线见图1d。在外部验证集(GSE53622)中,XGB-18 mRNA,LR-18 mRNA和SVM-18 mRNA 3个模型的ROC曲线如图1e所示,AUC值分别为0.711,0.669和0.673,可见XGB-18 mRNA模型的AUC值最高。此外,利用模型区分度评价指标K-S值对XGB-18 mRNA,LR-18 mRNA和SVM-18 mRNA 3个模型进行评估(图1f-h),其K-S值分别为0.468,0.242和0.300,表明XGB-18 mRNA模型预测ESCC淋巴结转移的准确性最高。
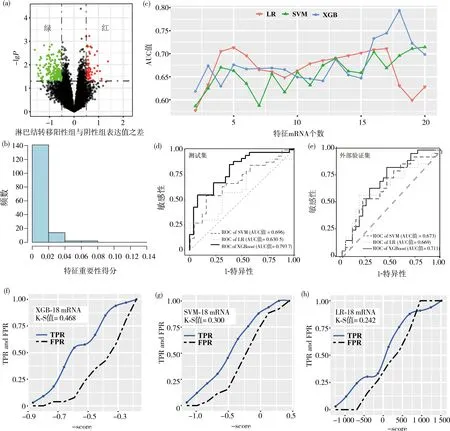
注:红色和绿色分别代表在淋巴结转移阳性组中表达量高于和低于淋巴结转移阴性组的mRNA。图1 特征mRNA选择、模型建立与模型效果评价(a)训练集中差异表达mRNA火山图;(b)XGBoost模型中159个mRNA的重要性评分;(c)不同mRNA特征子集的预测性能;(d)3种预测模型在测试集上的ROC曲线比较;(e)3种模型在外部验证集中的ROC曲线比较;(f-h)3个模型的K-S(Kolmogorov-Smirnov)值Fig. 1 Feature selection,model construction and prediction efficiency (a)Volcano plot for the differential expression of mRNAs in the training set,(b)the importance scores of 159 mRNAs assessed by the XGBoost algorithm,(c)prediction performances of different mRNA feature subsets,(d and e)receiver operating characteristic curves of three prediction models in the testing set and the external validation set,and (f-h)Kolmogorov Smirnov values of the three models
2.4 生存分析
为明确XGB-18 mRNA分类模型的临床意义,本实验分析了XGB-18 mRNA分类模型在ESCC生存预后中的作用。在59例测试集和60例外部验证集中,以XGB-18 mRNA预测值的最佳界值将样本分为高风险组和低风险组。XGB-18 mRNA高低风险组ESCC患者生存曲线表明,XGB-18 mRNA高风险组ESCC患者的总体生存时间低于低风险组,GSE53624测试集中XGB-18 mRNA高、低风险组ESCC患者的中位生存时间分别为12.6和56.2月,生存分析显示该模型预测值为预后危险因素(HR 3.91,95%CI 1.95~7.84;P<0.000 1,图2a)。进一步做亚组分析,淋巴结转移阴性患者中XGB-18 mRNA高风险组ESCC患者生存时间也明显低于低风险组(HR 6.75,95%CI 1.59~28.76;P=0.003 3,图2b),淋巴结转移阳性患者中,XGB-18 mRNA高风险组ESCC患者生存时间明显较低风险组短(HR 3.11,95%CI 1.24~7.82;P=0.012,图2c)。在外部验证集中,高、低风险组中位生存时间分别为10.2和39.8月,生存分析显示模型预测值为预后风险因素(HR 2.27,95%CI 0.99~5.25;P=0.048 9,图2d);亚组分析结果与测试集中类似。
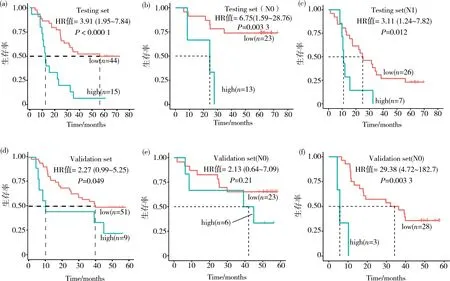
图2 测试集和验证集ESCC患者的生存分析(a)测试集59例ESCC患者中,高和低XGB-18 mRNA score组的生存曲线;(b-c)测试集中分别以淋巴结转移阴性、阳性分组做亚组分析,高和低XGB-18 mRNA score组的生存曲线;(d)外部验证集60例ESCC中,高和低XGB-18 mRNA score组的生存曲线;(e-f)验证集中分别以淋巴结转移阳性、阴性分组做亚组分析,高和低XGB-18 mRNA score组的生存曲线Fig. 2 Survival analysis of ESCC in the testing set and validation set (Survival curves of 59 ESCC patients with high-and low-XGB-18 mRNA scores in the testing set (a),and the subsets of N0 (b)and N1 (c);survival curves of 60 ESCC patients with high-and low-XGB-18 mRNA scores in the validation set (d),and the subsets of N0 (e)and N1 (f))
2.5 单因素和多因素Cox回归模型分析
将测试集和验证集中ESCC患者的人口学、临床病理特征及XGB-18 mRNA模型预测值score进行单因素Cox回归模型分析,发现N stage、TNM stage和XGB-18 mRNA预测值是ESCC预后影响因子(P<0.05,表2)。多因素Cox回归模型分析结果表明,XGB-18 mRNA预测值score(HR 3.3,95%CI 1.48~7.6;P=0.004,表3)是影响ESCC预后的独立因子。在GSE53622验证集中,多因素Cox回归模型分析结果与测试数据集类似(HR 4.0,95%CI 1.52~10.7;P=0.005,表3)。
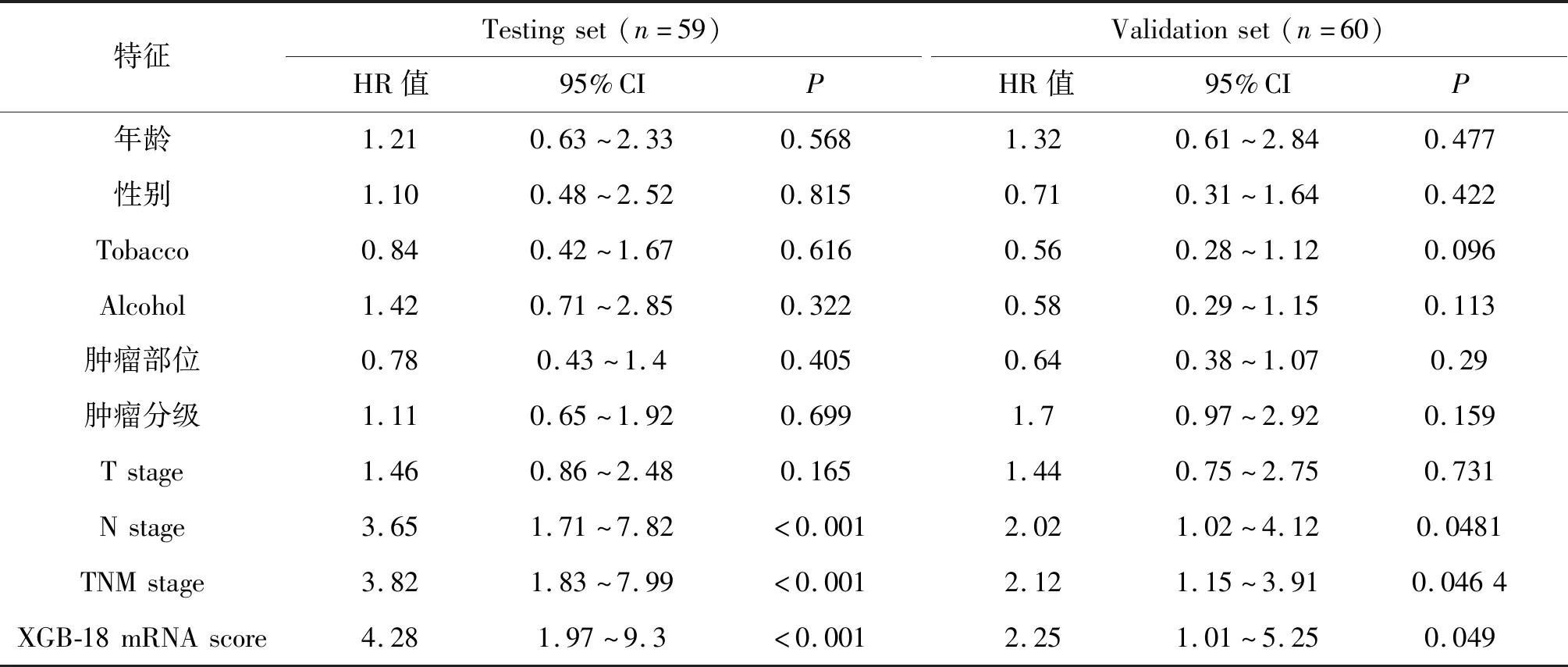
表2 测试集和验证集ESCC患者的人口学特征、临床病理特征和XGB-18 mRNA预测值score的单因素Cox回归模型分析Tab. 2 Univariate Cox regression analyses of the demographics characteristics,clinicopathological characteristics and XGBoost-18 mRNA scores of ESCC from the test set and the validation set
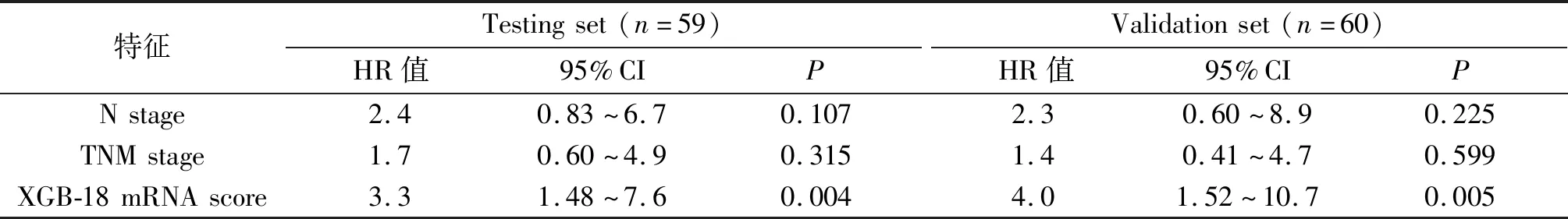
表3 测试集和验证集上ESCC患者的多因素Cox回归模型分析Tab. 3 Multivariate Cox regression analyses of ESCC from the test set and the validation set
2.6 GO富集分析
对XGB-18 mRNA模型纳入的18个mRNA 进行GO富集分析,在生物学过程中,上皮角化过程显著富集,参与该生物学过程的有4个mRNA分子,分别是KRT6B,KRT6C,LCE3D和RPTN等(图3a)。这4个mRNA分子在ESCC中均为低表达,并且在淋巴结转移阳性ESCC中的表达进一步降低,提示淋巴结转移阳性的ESCC癌细胞角化过程受阻,使癌细胞不同程度地失去上皮特征性分子表达,可能发生了上皮间质转化(图3d-g)。细胞定位富集分析结果表明,18个mRNA分子主要位于细胞内不溶性膜、角质微丝、突触周围和突触相关细胞骨架(图3b)。分子功能富集分析结果表明,18个mRNA分子的功能主要包括己烷基磷酸肌醇6,1,3,5等激酶活性、CXCR3受体结合、异生型跨膜ATP酶活性和cAMP依赖的蛋白激酶调节活性(图3c)。
3 讨论
由于食管黏膜及黏膜下层具有丰富的淋巴管网,因此淋巴结转移是食管癌转移的主要方式,浸润至黏膜下层的食管癌细胞发生淋巴结转移阳性率可达20%~30%,而局限于黏膜层的食管癌较少发生淋巴结转移。淋巴结转移是食管癌进展期的主要特征,与食管癌患者的术后生存预后密切相关,多学科综合治疗是进展期食管癌最佳治疗方案,而预后预测及分型是食管癌患者进行个体化治疗的关键,故而精准诊断ESCC淋巴结转移是ESCC临床治疗的关键[11,12]。
本研究基于ESCC mRNA表达谱数据,通过差异分析确定了与ESCC淋巴结转移相关的159个mRNA分子,并将其作为输入特征结合机器学习算法建立模型。根据这些mRNA分子特征在模型中的重要性,最终建立了由18个mRNA分子组成的XGB-18 mRNA ESCC淋巴结转移诊断模型。18个mRNA分子的生物学功能富集分析表明(图3a-c),发生淋巴结转移的ESCC细胞角质化过程受阻,KRT6B,KRT6C,LCE3D及RPTN等与细胞角质化相关的4个基因在淋巴结转移阳性ESCC中表达均下调。正常食管鳞状上皮细胞表达上皮细胞粘附分子(Ep-CAM)、钙粘附蛋白(E-cadherin)、紧密连接蛋白(Occludin,ZO-1)和细胞骨架相关(Keratin,ezrin),维系上皮组织结构完整性,防止物理、化学、微生物、炎症分子等有害因子的损伤作用。在食管上皮癌变过程中,癌变细胞失去这些结构性分子,使细胞间粘附力降低,获得迁移和侵袭能力。由此可见,参与细胞角质化过程的基因可能作为淋巴结转移阳性ESCC的诊断和临床治疗的靶分子。
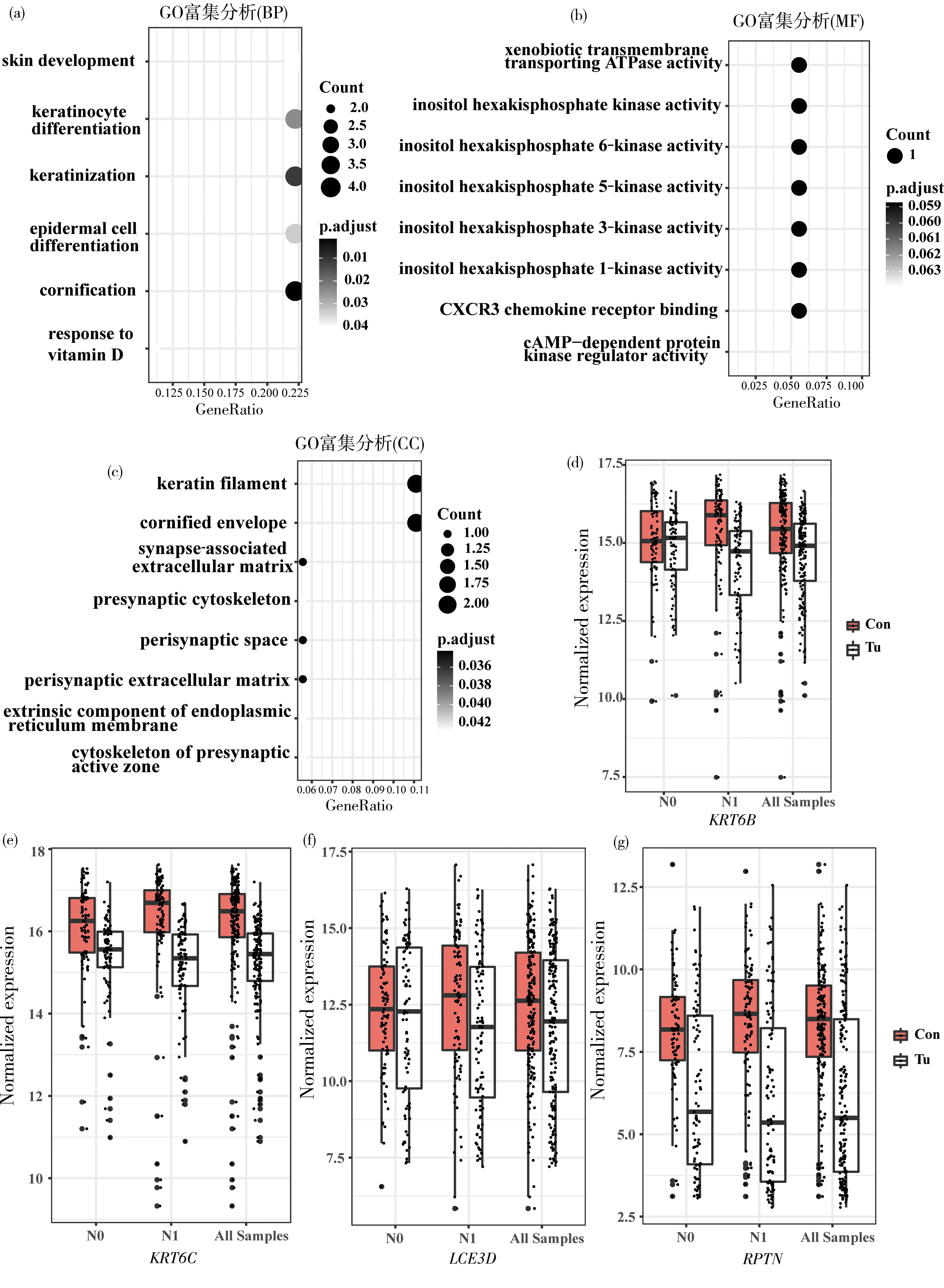
图3 18个ESCC淋巴结转移关键分子的GO富集分析与4个角质化相关分子在ESCC和癌旁组织中的表达(a)生物过程(BP)富集分析结果;(b)细胞组成(CC)富集分析结果;(c)分子功能(MF)富集分析结果;(d-g)分子KRT6B,KRT6C,LCE3D和RPTN在淋巴结转移阴性组(N0)、淋巴结转移阳性组(N1)以及所有179例样本中的表达量
由于病变部位、手术难度等因素影响,不同ESCC患者在手术过程中淋巴结清扫数目和区域存在较大差异。癌症基因图谱数据库(The Cancer genome atlas,TCGA)中,92例ESCC中包括53例ESCC患者进行了手术淋巴结清扫,淋巴结清扫数目从1到83个不等。2015年郑州大学附属肿瘤医院胸外科的一项研究表明[13],2010—2014年治疗的313例胸段ESCC患者中有122例(38.97%)发生了淋巴结转移,313例患者共清扫淋巴结4 461枚(平均14.2枚/人),其中癌转移的淋巴结有294枚,淋巴结转移率为6.59%。其中,喉返神经旁淋巴结转移率最高(25.5%),贲门胃左动脉旁次之(18.2%)。另一项南京医科大学一附院肿瘤科分析了1 791例食管癌样本淋巴结转移情况[14],其中1 693例ESCC(占94.5%)样本中发生淋巴结转移的患者有586例(34.61%),术中共清扫淋巴结17 674枚(平均10.4枚/人),淋巴结转移阳性为1 409枚,淋巴结转移率为7.97%。以上研究表明,ESCC术中淋巴结清扫数目和清扫区域具有较大的异质性,淋巴结清扫数目过少、阳性淋巴结未被检测出等导致淋巴结转移阴性患者可能存在假阴性。2017年关于食管癌根治术胸部淋巴结清扫的中国专家共识中指出,2016版NCCN建议的二野或三野淋巴结清扫数目须达到11~15枚[15],才能提供较为准确的N分期和TNM分期判断,正确指导术后治疗。此外,本研究包含的病例资料中未提供淋巴结转移区域信息,可能导致预后预测结果存在误差。
目前,XGBoost算法在生物医学领域应用广泛,据此建立的生物学模型的诊断效果较为理想[16-18]。有研究表明[16],经过数据归一化、超参数选优后,用XGBoost算法以肝功能、血脂、肾功能、乙肝、血常规指标等为特征建立血糖值的回归模型,以均方根误差和平均绝对百分比误差作为模型评价指标,结果表明基于XGBoost算法的模型具有精度高、运行快、稳定性强等优势,较基于SVM的模型和随机森林模型预测精确。另一研究[17]利用XGBoost算法挖掘与N2-3期淋巴结转移相关的因素,通过交叉验证和多次随机化实验得到最佳变量组合,该组合包括肿瘤大小、组织学类型、多灶性、淋巴管浸润、ER阳性细胞百分比和前哨淋巴结阳性数目等,结果表明,基于XGBoost算法的预测模型优于逻辑回归算法模型,达到0.80 (95%CI 0.65~0.92)。
总之,本文利用XGBoost算法鉴定了18个ESCC淋巴结转移关键基因,并建立了XGB-18 mRNA分类模型,其预测ESCC淋巴结转移的准确性高于LR模型和SVM模型,并且XGB-18 mRNA模型预测的风险值,是ESCC预后独立危险因素,为ESCC患者临床个体化治疗策略及方案制定提供理论依据和指导。
