利用COI基因鉴别长三角地区常见嗜尸性蝇类
2022-01-07徐旺王禹王嫚王颖慧王江峰
徐旺,王禹,王嫚,王颖慧,王江峰
苏州大学法医学系,江苏 苏州215000
近年来,法医昆虫学的相关研究进展十分迅速,研究者通过实验已获得了大量嗜尸性蝇类的生长发育数据。现场勘验人员通常利用形态学方法对案发现场出现的嗜尸性蝇类进行种属鉴别,结合相应的蝇类生长发育数据,即可有效地估计死亡时间(postmortem interval,PMI)[1]。
然而在实际工作中,形态学鉴别方法并不总是有效的。一方面,案发现场收集的蝇类样本可能为低龄幼虫或蛹,有些样本是不完整的,增加了利用形态学方法进行种属鉴别的难度。另一方面,大部分现场勘验人员缺乏昆虫学知识,难以通过形态学方法对现场的蝇类样本进行鉴定。
随着DNA 检测和分析技术的飞速发展,法医昆虫学领域逐渐尝试应用各种分子生物学方法对法医学昆虫(尤其是嗜尸性蝇类)进行种属鉴定,如限制性片段长度多态性(restriction fragment length polymorphism,RFLP)[2]、扩增片段长度多态性(amplified fragment length polymorphism,AFL)[3]等。然而大量研究[4-6]表明,DNA 条形码技术(DNA barcoding)可以更有效地对嗜尸性蝇类进行种属鉴别。陈庆等[4]通过对线粒体DNA 上的细胞色素c 氧化酶亚基Ⅰ(cytochrome c oxidase subunit Ⅰ,COⅠ)基因1 076 bp序列的测定和比较分析,成功地将不同地点采集的7个嗜尸性蝇类物种进行了区分,序列信息的聚集结果和形态学鉴定的种类归属完全一致。GUO等[5]利用700bp的COⅠ基因序列和678 bp的period基因序列对9个麻蝇物种进行了鉴定。MÁRQUEZ-ACERO 等[6]通过分析599 bp 的COⅠ基因序列,鉴定了哥伦比亚地区的6 种丽蝇。在众多基因中,线粒体DNA 的COⅠ基因被证实能够对大部分嗜尸性蝇类物种进行高效的区分,因此也是法医昆虫学分子鉴定研究中最常用的基因[4]。
虽然此前对于蝇类鉴定的研究很多,但是,研究所采用的蝇类样本多为使用小块肉或者小型动物尸体放置于自然环境所采集的,且主要采集的是成虫,这就存在以下问题:(1)采集的蝇类可能只是在尸体环境停留而并不取食,并非嗜尸性蝇类。(2)样本数量较少,多数蝇类物种只有1 只个体,难以保证所获得序列的准确性。(3)研究所采用的基因及其引物没有统一的指标。因此想要解决某一地区的嗜尸性蝇类的种属鉴别问题时不知道使用哪种引物和指标,导致嗜尸性蝇类分子鉴定研究的推广遇到阻碍。
鉴于以上原因,本研究的虫源全部来自本实验室已经研究过生长发育规律的种类,均来自长三角地区,且在本实验室饲养繁殖多代,并被证明是嗜尸性昆虫,拟通过基于COⅠ基因片段序列对长三角地区常见嗜尸性蝇类的种属进行分子鉴定,以期为推断死亡时间提供技术支持。
1 材料与方法
1.1 虫源
依据《中国常见蝇类检索表》进行形态学种类鉴定,本研究中的7 个蝇类物种分别为大头金蝇Chrysomya megacephala、绯颜裸金蝇Chrysomya rufifacies、巨尾阿丽蝇Aldrichina grahami、厩腐蝇Muscina stabulans、棕尾别麻蝇Boettcherisca peregrina、丝光绿蝇Lucilia sericata和肥躯金蝇Chrysomya pinguis,分别在2015—2019 年采自于野外动物尸体上,并经过实验室多代培养,遗传稳定(表1)。

表1 嗜尸性蝇类的生物学分类Tab.1 Biological classification of necrophagous flies
1.2 DNA 提取、PCR 扩增和测序
使用HighPrep™ Insect DNA 试剂盒(美国Mag-Bio Genomics 公司),根据说明书操作提取全基因组DNA。本研究的各个物种均来自该物种的同一母体,所以不进行种内遗传差异检测,每个蝇类物种取2~5只进行DNA 提取,每个个体提取得到20 μL 的DNA样本。
通过COⅠ基因的通用引物进行PCR 扩增,引物序列为:LCO-1490(5'-GGTCAACAAATCATAAAGATA TTGG-3')和HCO-2198(5'-TAAACTTCAGGGTGACCA AAAAATCA-3')。PCR 反应体系为25 μL,包括2 μL(50~200 ng/μL)的模板DNA、12.5 μL 的2×GoTaq®绿色Master Mix(美国Promega 公司)、各1 μL 的上下游引物(50 μmol/L),添加无核酸酶水至反应体系为25 μL。PCR 扩增循环参数:95 ℃2 min;95 ℃30 s,45 ℃30 s,73 ℃1 min,共30 个循环;73 ℃5 min,于4 ℃保存。
扩增产物由生工生物工程(上海)股份有限公司按Sanger 测序方法完成测序。
1.3 序列分析
使用SnapGene Viewer 4.3.11 软件(美国Insightful Science 公司)查看测序结果,对比正反向测序结果拼接获得目的序列。将序列上传至GenBank(https://www.ncbi.nlm.nih.gov/genbank/GenBank),经BLAST(Basic Local Alignment Search Tool,https://blast.ncbi.nlm.nih.gov/Blast.cgi)查询与数据库中所有已知核酸序列进行序列同源性搜索比对。通过MEGA 7.0.26 软件(https://www.megasoftware.net/)以Kimura 2-parameter模型计算碱基组成和种间进化分歧率,以邻接法(neighbor joining method,NJ)和非加权组平均法(unweighted pair-group method with arithmetic means,UPGMA)构建系统发育树,并以鳞翅目的小地老虎Agrotis ipsilon作为外群,Bootstrap 值为500。
2 结果
2.1 种间进化分歧率分析
种间进化分歧率(表2)结果显示,种间进化分歧率最大的是厩腐蝇与棕尾别麻蝇,达到了15.3%;而种间进化分歧率最低的是大头金蝇与肥躯金蝇,为2.2%。

表2 种间进化分歧率Tab.2 Sequence divergence between species(%)
2.2 碱基组成分析
将正反向序列拼接后,获得658 bp 的目的序列(表3)。本研究中蝇类物种的平均碱基组成为A(30.14%)、T(38.23%)、C(15.98%)、G(15.65%),其中A+T(68.37%)远高于C+G(31.63%)。
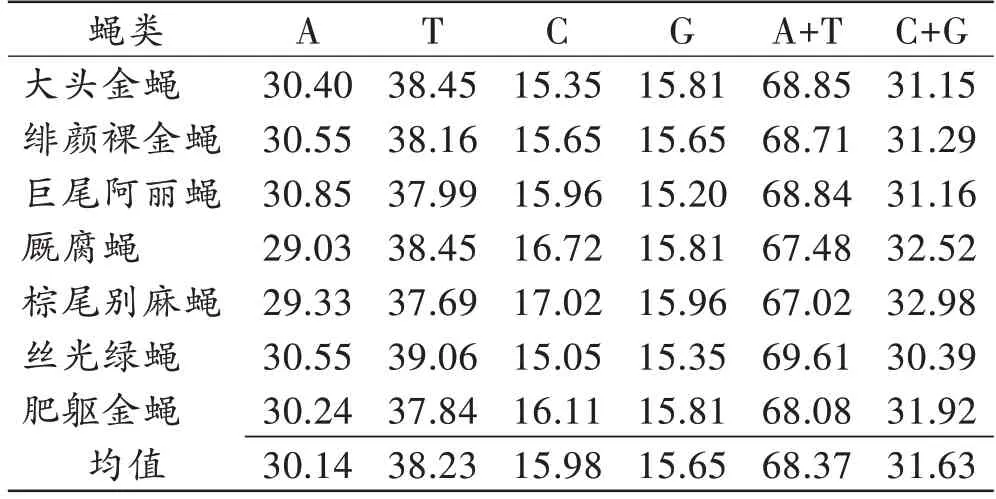
表3 COI基因序列的核苷酸频率分布Tab.3 Summary of nucleotide frequency distribution for COI gene sequences(%)
2.3 系统发育树分析
本研究中的蝇类物种系统发育树如图1所示,7个双翅目物种聚集在一起,且均与作为外群的鳞翅目的小地老虎距离较远。在两种系统发育树中,大头金蝇、肥躯金蝇和绯颜裸金蝇3 个物种均聚集在一簇,且支持度较高(>95%)。在NJ 树中,多个分支的支持度极低(<50%),且与生物学分类(表1)并不一致,如蝇科的厩腐蝇与麻蝇科的棕尾别麻蝇聚集成了一簇;而在UPGMA 法构建的系统发育树中,5 个丽蝇科的物种聚集成簇,其与厩腐蝇(蝇科)间的距离相较于棕尾别麻蝇(麻蝇科)间更近,且各分支支持度均较高(>85%),与生物学分类(表1)相符。故倾向于接受UPGMA 法构建的系统发育树结果。
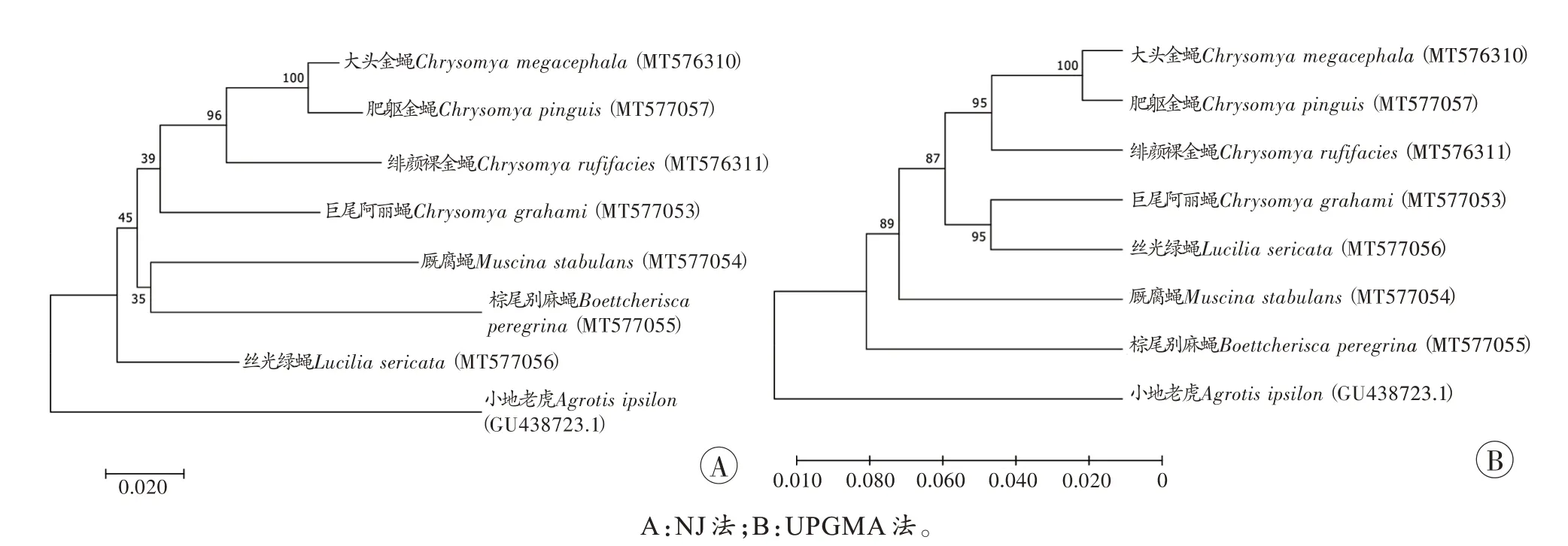
图1 COI系统发育树Fig.1 Phylogenetic tree for COI gene sequences
3 讨论
本研究从3 个科的蝇类(丽蝇科、麻蝇科和蝇科)中提取线粒体COⅠ基因,采用的蝇类样本均来自经过实验室多代培养的种群。本研究的主要目的是验证通过COⅠ基因对长三角地区的常见嗜尸性蝇类进行分子鉴定的可靠性。
利用UPGMA 对双翅目3 科7 种常见嗜尸性蝇类样本构建系统发育树,近缘种属形成姊妹簇或树形的分支部分,再构成不同科的进化树分支。聚类效果较好,且与形态学鉴定结果一致。整体上可以将7 种蝇类分丽蝇科、蝇科、麻蝇科三大分支,所有样本均位于某一特定的分支上,尽管不能对所有物种进行准确鉴别,但对物种的鉴定起到了很好的验证和补充。
线粒体DNA 的进化和遗传速度快,不需要基因重组。因此,在评估昆虫类群间的系统发育关系方面,线粒体DNA 比核DNA 更具优势[7]。其中,COⅠ基因已广泛应用于法医学上重要的蝇类鉴定[8]。然而,基因条形码技术对近源物种进行鉴定时的效果并不十分理想。例如,BHARTI 等[9]通过使用一段1 423 bp的COⅠ基因片段对印度地区的常见嗜尸性丽蝇进行分子鉴定,结果表明当使用该基因片段时,星岛金蝇Chrysomya chani和大头金蝇2 个物种的种间进化分歧率极低(0.11%),无法将这两个物种进行区分。因此,DNA 条形码技术并不能解决所有的种属鉴别的问题。有研究[5]发现,同时使用多个基因进行分子鉴定是有必要的,这样可以增加鉴定的准确率。
为了可靠地进行物种的分子鉴定,目标种群具有较高的种间遗传多样性(>3%)是非常重要的[10-11]。在本研究中,除了大头金蝇和肥躯金蝇2 个物种之间的种间进化分歧率(2.2%)小于3%以外,其他物种间均大于3%,可以很好地区分这些物种。大头金蝇和肥躯金蝇之间的进化分歧率较低是可以解释的,因为两者是近源物种,并且先前也有研究[12]表明,近源的金蝇物种有非常低的种间进化分歧率。因此,可能需要增加其他基因片段,如核基因ITS2或cytb等[13],有待进一步研究。
对所得样本的COⅠ基因进行核苷酸频率分析,结果显示碱基组成具有明显的AT 偏倚,这与此前的大多数研究一致[14]。根据本研究中UPGMA 法构建的系统进化树,丽蝇科与蝇科物种间的距离相较于丽蝇科与麻蝇科更近,这说明丽蝇科与蝇科的亲缘关系可能更为密切,该结果也与卓荦等[15]的研究结果相符合。不同科的蝇类物种分别位于进化树上的不同分支,且各分支的Bootstrap 检验可信度均大于85,说明聚类结果较可靠,能够有效区分。此外,系统进化树中位于同一分支的两对物种间的种间进化分歧率为各物种间进化分歧率中最低,证实了结果的可靠性。
总而言之,本研究验证了分子鉴定技术在长三角地区常见嗜尸性蝇类种属鉴别中的可行性,同时证实了COⅠ基因片段序列能够较为准确地对不同蝇种的种属进行鉴别。今后的研究,既要对嗜尸性蝇类物种进行全面的研究,也要对其他嗜尸性昆虫进行分子鉴定的探究,从而为PMI 推断提供有力的支撑。
