毛竹NLP 转录因子鉴定及其响应氮素的表达模式
2021-12-29袁婷婷朱成磊李紫阳宋新章高志民
袁婷婷,朱成磊,李紫阳,宋新章,高志民*
(1.国际竹藤中心竹藤资源基因科学与基因产业化研究所,国家林业和草原局/北京市共建竹藤科学与技术重点实验室,北京 100102;2.浙江农林大学,浙江 杭州 311300)
氮素是植物生长发育必需的大量营养元素之一[1-2]。近些年,氮的利用效率倍受关注,涉及氮的吸收、运输与同化等方面,其中,氮的吸收转运已成为研究的热点。研究表明,RWP-PK 家族中的NLP (NIN-like protein)转录因子在响应氮饥饿、氮信号调节过程中处于中心地位[3]。NLP 家族成员都具有RWP-RK 和PB1 两个典型的结构域,RWPRK 结构域具有与DNA 结合的功能,PB1 结构域具有蛋白相互作用的功能[4]。拟南芥(Arabidopsis thaliana)中NLPs 与硝酸盐顺式作用元件NRE(Nitrate-responsive elements)结合调控硝酸盐响应相关基因的表达,进而影响拟南芥的生长发育[5],如过表达拟南芥NLP7能够通过增强氮和碳的同化,促进植物生长[6],NLP8 则对于硝酸盐促进种子萌发至关重要[7]。过表达OsNLP1促进水稻(Oryza sativaL.)生长,提高水稻氮素利用率和产量[8]。另外,在植物Nitrate-CPK-NLP (硝酸盐-Ca2+传感蛋白激酶-NLP)信号通路中,NLPs 协调植物对硝酸盐信号的感知和对下游转录因子、转运蛋白、碳氮代谢过程的调控[9],在氮饥饿、氮磷互作以及根系生长过程中发挥着重要作用[10]。
随着测序技术的快速发展,已经对包括水稻[11]、拟南芥[7]、小麦(Triticum aestivumL.)[12]和玉米(Zea maysL.)[13]等多种植物中NLPs 进行全基因组鉴定和功能研究。竹子作为禾本科(Gramineae)的特殊类群,尤其是木本竹,因其生长快、材性好,已成为缓解木材供给不足的重要替代材料。毛竹(Phyllostachys edulis(Carrière) J.Houz.)林占我国竹林总面积的72.96%[14],是最具代表性的木本竹种。氮肥显著影响毛竹幼苗的生长[15]、笋的品质和产量及其叶片生理特性[16],施用氮肥已成为竹林增产的重要手段。未施氮肥的情况下,氮沉降在很大程度上补充了土壤氮,提高了毛竹光合作用效率[17-18]、增加生物量、增强抗性[19]。然而,毛竹吸收转运氮的机制尚未见报道,其中,NLP 家族成员的调控作用更不清楚。本文以毛竹为研究对象,在全基因组水平系统分析NLP 分子特征的基础上,研究在氮饥饿过程以及复氮后NLP基因的表达模式,以期为深入研究其功能提供参考。
1 材料与方法
1.1 植物材料培养与处理
将毛竹种子播种于基质(泥炭∶蛭石=7∶3)中,置于25℃的温室中培养,待实生苗生长至2 个月时,选择株高和长势一致的植株,将种子去掉,根系用去离子水洗净,置于全营养液中培养2 周,然后转入缺氮木村B 营养液[20]中进行氮饥饿处理,并于氮饥饿72 h 后,用6 mmol·L−1的硝酸盐(KNO3和KCl 配置而成)进行复氮处理。分别于氮饥饿处理以及复氮处理0、0.5、1、2、4、8、12 和24 h 取植株的根系,用液氮处理后保存于−80℃,用于RNA 提取,进行定量PCR (qPCR)分析,氮饥饿0 h 和72 h 的植株分别作为对照,试验采用3 次生物学重复。
1.2 毛竹NLP 全基因组鉴定
分别从Rice Genome Annotation Project (http://rice.plantbiology.msu.edu/)和Tair (https://www.arabidopsis.org/index.jsp)数据库下载水稻和拟南芥的NLP转录因子的CDS 和氨基酸序列[11,21],作为诱饵序列在毛竹基因组数据库BambooGDB (http://bamboo.bamboogdb.org/)中 进 行BlastP、BlastN (e-value=1 e−10)比对,获取毛竹NLP的候选基因,对获得的候选序列在PFAM 数据库(http://pfam.xfam.org)逐条进行蛋白保守结构域的鉴定和分析,仅保留具有编码完整保守结构域的序列,并进行基因命名。
1.3 毛竹NLP 基因序列及其编码蛋白序列分析
使用TBtools 工具绘制毛竹NLP的基因结构图;使用ProtParam (http://web.expasy.org/prot-param/)和Plant-mPLoc 算法(http://www.csbio.sjtu.edu.cn)分析PeNLPs 编码蛋白的基本理化特性及亚细胞定位;使 用MEME Version 5.1.0 (http://meme-suite.org/tools/meme)分析毛竹NLP 蛋白的保守基序。
1.4 毛竹NLP 家族成员系统进化分析与功能预测
使用MEGA 7.0 软件的Clustal W 对毛竹、水稻、拟南芥、玉米、毛果杨(Populus trichocarpa(Torr.&Gray))、二穗短柄草 (Brachypodium distachyon(L.) Beauv.)和蒺藜苜蓿 (Medicago truncatulaGaertn.)的NLP 蛋白序列[22]进行同源比对分析,用邻接法(Neighbor-joining method)、bootstrap 试验重复1000 次、其它参数设置为默认值构建系统进化树。
1.5 毛竹NLP 的表达分析
利用本实验室的毛竹26 个不同组织转录组测序(RNA-Seq)数据(NCBI 的SRA 号为:SRS 1847073、SRS1847072、SRS1847071、SRS1847070、SRS1847069、SRS1847068、SRS1847067、SRS 1847066、SRS1847065、SRS1847064、SRS1847063、SRS1847062、SRS1847061、SRS1847060、SRS 1847059、SRS1847058、SRS1847057、SRS1847056、SRS1847055、SRS1847054、SRS1847053、SRS 1847052、SRS1847051、SRS1847050、SRS1847049、SRS1847048)[23],构建毛竹NLP基因的组织表达谱,以Log2(FPKM+1)的值进行表达量热图绘制[24]。
根据毛竹NLP基因序列,使用Primer Premier 5.0 软件设计特异定量引物,由北京博迈德基因技术有限公司合成(表1)。用于qPCR 分析的cDNA是由1.1 节中经过氮饥饿以及复氮处理不同时间的毛竹根系,通过TRIzol (Invitrogen)法从中提取RNA,用Promega 反转录试剂盒合成cDNA。qPCR 实验在qTOWER2.2 系统上进行,体系参照Roche Light Cycler®480 SYBR Green I Master kit 试剂盒,程序为95℃预变性10 min;95℃变性10 s,60℃解链10 s,40 个循环。选择PeTIP41作为内参基因[25],相对表达量的数据处理用2−ΔΔCT法[26]。
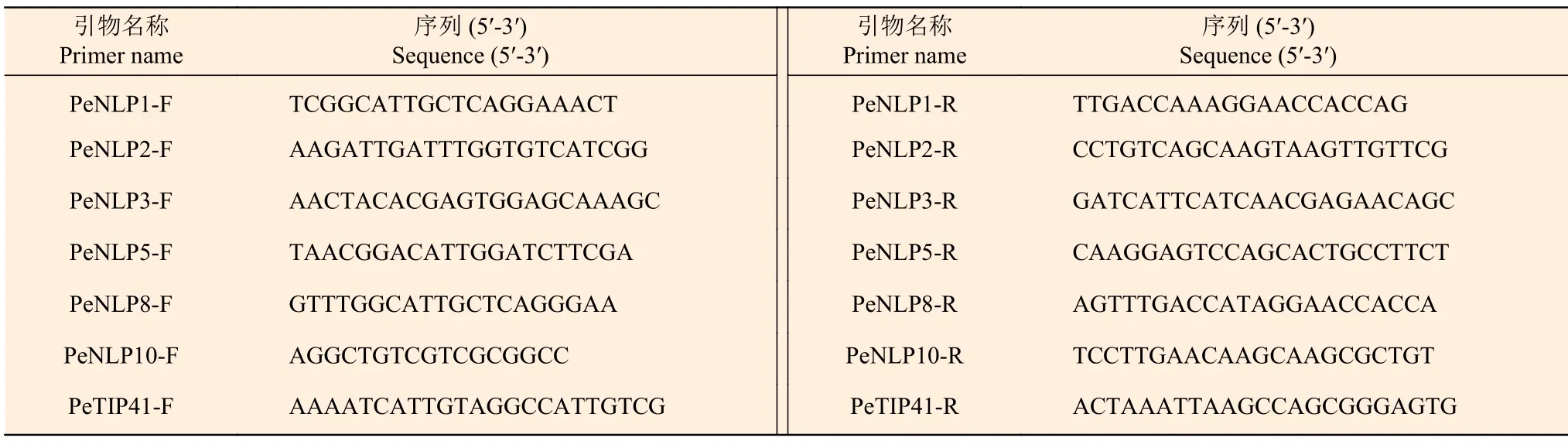
表1 qPCR 所用引物Table 1 Primers used in qPCR
2 结果与分析
2.1 毛竹NLP 家族基因的鉴定
在毛竹新基因组数据[23]中共鉴定出10 个编码完整NLP 蛋白的基因,命名为PeNLP1~PeNLP10。PeNLPs 的编码区长度为2145~2892 bp,编码蛋白长度为714~963 aa,分子量(Molecular weight,MW)为 77.41~105.08 kDa,理论等电点(Theoretical isoelectric point,pI)为5.36~6.25,亚细胞定位预测除PeNLP9 位于叶绿体外,其余成员均位于细胞核内(表2)。

表2 PeNLPs 编码蛋白的基本特征Table 2 Basic characteristics of the proteins encoded by PeNLPs
2.2 PeNLPs 基因结构及其编码蛋白保守基序分析
基因结构分析发现,PeNLPs 均包含4 个内含子,但不同基因内含子的大小和位置存在一定的差异。依据系统进化关系,PeNLPs 可分为3 个组,同一个组内的基因成员具有相似的结构以及内含子长度和位置,如II 组中PeNLP2和PeNLP3的基因结构高度相似,PeNLP2的最短内含子和最长内含子分别为134 bp 和740 bp,PeNLP3的最短内含子和最长内含子分别为133 bp 和730 bp,二者非常接近,组与组之间的基因结构差异明显(图1A)。
蛋白保守基序分析表明:PeNLPs 共有20 个保守基序(图1B),其中,Motif 1~Motif 7、Motif 9、Motif 12、Motif 13为共有基序,Motif 19、Motif 14 和Motif 16 分别为Ⅰ、Ⅱ和Ⅲ组成员所特有。另外,Ⅰ组成员缺少Motif 11,Ⅱ组成员缺少Motif 8 和Motif 17,Ⅲ组成员缺少Motif 15、Motif 18 和Motif 20。

图1 PeNLPs 基因结构(A)及其编码蛋白保守基序(B)分析Fig.1 Gene structure (A) and the conserved motifs (B) of the proteins encoded by PeNLPs
2.3 PeNLPs 的进化分析
为了解不同物种间NLP 的进化关系,预测PeNLPs 的潜在功能,构建了基于毛竹、水稻、拟南芥、玉米、毛果杨、二穗短柄草和苜蓿NLP 蛋白氨基酸序列的系统进化树。结果表明:60 个NLPs 分成了3 个组(Ⅰ、Ⅱ和Ⅲ),双子叶植物NLPs 主要分布在Ⅰ组中,而单子叶植物NLPs 则在3 个组中均有分布(图2),这与前人的研究一致[22]。PeNLPs 分布在Ⅰ、Ⅱ和Ⅲ的成员个数分别为4、2、4 个,这和其它单子叶植物成员在3 个组的个数分布情况相似。在每个组中,多数PeNLPs与水稻NLPs 聚类在较近的分支,其次是二穗短柄草的NLPs,这说明毛竹与水稻、二穗短柄草在进化上的亲缘关系较近,而与拟南芥、毛果杨等双子叶植物的亲缘关系较远。

图2 NLP 家族成员的系统发育分析Fig.2 Phylogenetic analysis of NLP family members
共线性分析发现,9 个PeNLPs 存在共线关系,并组成6 个共线性基因对,即PeNLP1和PeNLP8、PeNLP2和PeNLP3、PeNLP4和PeNLP7、PeNLP4和PeNLP10、PeNLP5和PeNLP6、PeNLP7和PeNLP10(图3A),且共线性基因的Ka/Ks均小于1 (图3B),说明这些基因在进化中经历了纯化选择。此外,有9 个PeNLPs 与5 个OsNLPs 之间存在共线性,具有共线关系的PeNLPs 数量多于OsNLPs 数量,这可能与毛竹进化过程中发生过基因组加倍事件有关[27]。

图3 毛竹与水稻的NLP 家族基因共线性分析Fig.3 Collinearity analysis of NLP family genes between Ph.edulis and O.sativa
2.4 PeNLPs 的组织特异性表达
利用毛竹转录组数据,对PeNLPs 的组织表达特异性进行分析。结果表明:PeNLPs 部分成员的表达具有组织特异性(图4),如PeNLP2在叶片、叶鞘和箨片中特异表达,PeNLP5和PeNLP9分别在叶鞘和0.1 cm 根中特异表达,推测它们是在特定的部位发挥功能;还有部分成员呈组成型表达,如PeNLP10在26 个组织中均检测到表达,且在各个组织中的表达量变化不大,推测它在毛竹各个部位及各个部位的不同生长阶段均能发挥作用。然而,有些成员在26 个组织中检测不到表达,如PeNLP6和PeNLP7,推测它们可能为诱导表达型,或是存在功能冗余基因。
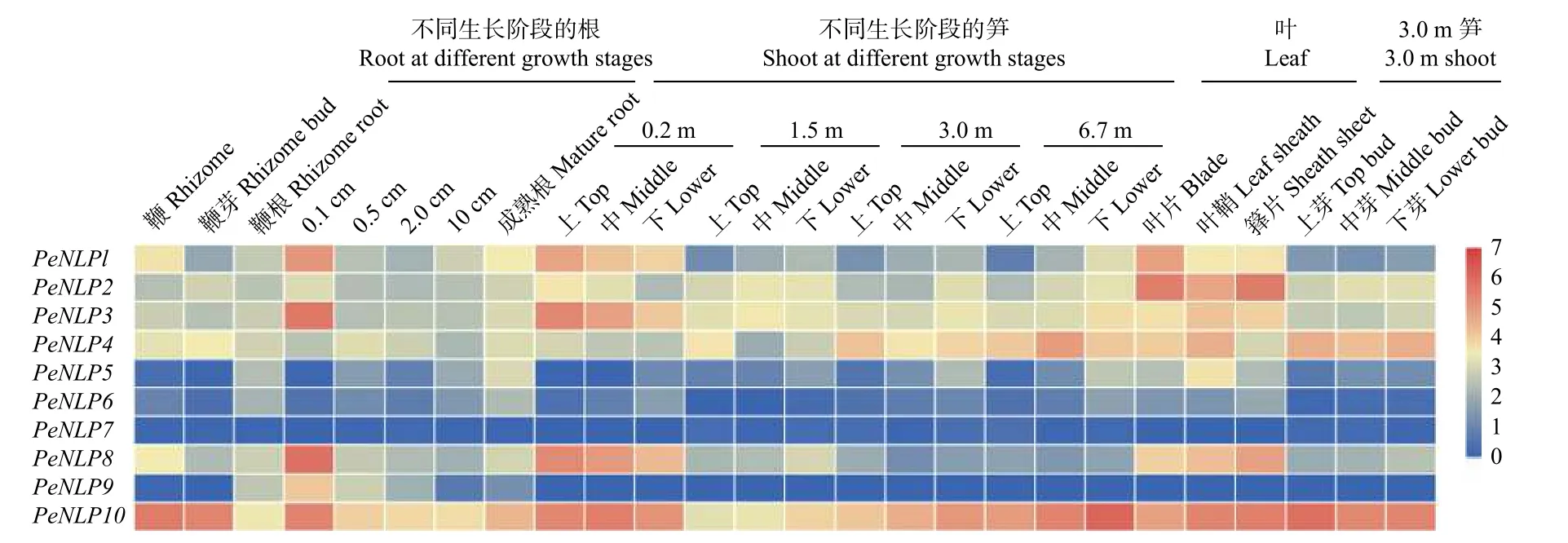
图4 毛竹不同组织中PeNLPs 的表达分析Fig.4 Expression analysis of PeNLPs in different tissues of moso bamboo
此外,PeNLPs 在同一组织的不同生长阶段的表达呈现一定的差异性。在根中,部分PeNLPs 的表达呈现出在生长早期表达高于生长后期的规律,如PeNLP1、PeNLP3、PeNLP8在0.1 cm 根中的表达量高于0.5 cm、成熟根等其它生长阶段的根中的表达量,在0.2 m 笋中的表达量高于1.5、6.7 m 等其它高度的笋中的表达量,这可能是由于这3 个成员主要在根和笋的生长早期发挥作用;但PeNLP4却呈相反的表达趋势,其在3.0 m 和6.7 m 笋中的表达量高于在0.2 m 和1.5 m 笋中的表达量。由此表明,这些PeNLPs 可能在根或笋的不同生长阶段具有不同的调控作用。
2.5 氮饥饿及复氮过程中PeNLPs 的表达模式
氮饥饿及复氮过程中PeNLPs 的表达模式分析表明:在氮饥饿处理过程中,PeNLPs 的表达呈现不同程度的波动变化,这说明不同时间PeNLPs 对氮饥饿的响应程度不同,且各个成员之间存在一定的差异(图5)。在施加氮饥饿1 h 内,PeNLP1的表达量迅速上升,且达到极显著水平,PeNLP2、PeNLP3、PeNLP5、PeNLP8和PeNLP10则极显著下降(p<0.01),表明PeNLPs 对氮饥饿的响应均较迅速。随着氮饥饿的施加,在1~24 h 过程中,PeNLP1的表达为先下降、后上升、再下降,至氮饥饿24 h 时的表达量与开始氮饥饿时(0 h)的表达量接近;PeNLP2、PeNLP5和PeNLP10的表达为先上升、后下降、再上升,至氮饥饿24 h 时,PeNLP5 极显著高于0 h,PeNLP2和PeNLP10则极显著低于0 h (p<0.01);PeNLP3和PeNLP8呈先上升、后下降、再上升、再下降、再上升的波动表达模式,至氮饥饿24 h 时,PeNLP3的表达极显著低于0 h,PeNLP8的表达极显著高于0 h (p<0.01)。由此推测,毛竹对氮饥饿过程的响应可能存在不同的信号通路,PeNLPs 各成员通过不同的表达来参与毛竹对氮饥饿的响应。

图5 氮饥饿过程中PeNLPs 的表达分析Fig.5 Expression analysis of PeNLPs during nitrogen starvation
对氮饥饿72 h 后的毛竹进行复氮处理,在复氮过程中PeNLPs 的表达量均显著或极显著上调(p<0.05 或p<0.01),说明复氮促进了PeNLPs 的表达,但呈现不同的波动变化(图6)。如PeNLP1、PeNLP3、PeNLP8和PeNLP10呈现相似的变化趋势,即先上升、后下降,再上升、再下降;PeNLP2和PeNLP5呈现相似的变化趋势,即先上升、后下降,再上升、再下降,再上升、后下降。PeNLP1、PeNLP2、PeNLP3、PeNLP5和PeNLP10的表达量基本是在复氮处理后12 h 达到峰值,在处理后24 h 较12 h都显著下降,而PeNLP8则是在处理后24 h 达到峰值,较处理后12 h 显著上升,说明它们的表达可能存在昼夜节律,各个成员响应硝酸盐表达的波动差异,表明不同PeNLPs 对硝酸盐的响应可能具有不同的信号通路。

图6 复氮过程中PeNLPs 的表达分析Fig.6 Expression analysis of PeNLPs during nitrogen resupply
3 讨论
本研究基于毛竹新基因组数据,共鉴定到10 个毛竹NLP基因家族成员,比水稻(6 个)、玉米(9 个)、二穗短柄草(7 个)和苜蓿(5 个)等单子叶植物中成员数量多,而少于双子叶植物毛果杨(14 个)中的成员,这可能与不同物种在进化过程中经历的基因组加倍或基因丢失有关[28]。亚细胞定位预测10 个PeNLPs 有9 个定位在细胞核中,这符合转录因子的特性,而PeNLP9 定位在叶绿体中,这可能是NLP 家族的核穿梭现象[29],与拟南芥、水稻NLP 的核穿梭特征[20]类似。系统进化分析表明,60 个NLPs 分为3 个组,各组成员功能相似但也出现了一定的功能分化,如Ⅰ组中拟南芥和水稻成员主要参与硝酸盐信号转导和氮素营养调控植物生长发育[30],Ⅱ组成员中AtNLP8呈组成型表达,当硝酸盐存在时,AtNLP8 可以直接结合到编码ABA 分解代谢酶基因CYP707A2的启动子区,降低种子中ABA 含量并促进种子萌发[7];Ⅲ组中成员个数相对较多,AtNLP6/7 能够结合硝酸盐响应基因ANR1、CIPK8的启动子,调控氮吸收、同化和信号转导过程[31]。虽然可以根据已知功能的同源蛋白预测PeNLPs 的功能,但要对PeNLPs 在氮素利用率(NUE)中的作用给出明确的结论,尚有待于实验验证。
氮素利用效率受氮感知、吸收、转运、同化和再利用效率等因素的影响,其调控是一个复杂的网络,其中,NLP 转录因子是硝态氮吸收利用的核心转录因子[32]。拟南芥中AtNLP6/7 通过感知硝酸盐信号后活性被诱导,进而结合到高亲和力硝酸盐转运基因蛋白(NRT2.1)、亚硝酸还原酶(NIR1)等基因的启动子区和硝酸还原酶(NIA1)基因的3′侧翼区的氮应答DNA 元件(NRE)上[33]。AtNLP6/7还可以和TCP20 相互作用形成异源二聚体,正向调控氮同化及氮信号途径中的基因NRT1.1、NIA1和NIA2的表达,参与硝酸盐的同化和信号传递[34],此外,NLPs 还能靶向LBD37-39 和NIGT1 两类转录因子基因,通过触发二级转录事件协同响应硝酸盐过程[35]。蒺藜苜蓿MtNLP1 在细胞核中与MtNIN互作,阻碍MtNIN 激活CRE1和NF-YA1,进而抑制了根瘤菌感染以及节的形成[36]。另外,作为转录因子,NLP 也受到其它转录因子的调控,如拟南芥AtNLP3 受到bZIP1 的“非绑定”的调控方式来快速响应硝酸盐[37]。
NLPs 除与上游的转录因子互作、下游基因结合来参与调控响应硝酸盐外,还通过自身表达的变化来响应氮饥饿及复氮诱导过程。在毛竹根中PeNLPs 的表达对氮饥饿作出快速反应,如在氮饥饿1 h 时,PeNLP1的表达量上升至对照的30 倍,而PeNLP3的表达量则降低至对照的0.2 倍,这与硝酸盐处理玉米时ZmNLPs 的表达变化趋势相类似[38]。而在氮饥饿后复氮处理过程中,所有PeNLPs的表达均显著上调,说明硝酸盐诱导PeNLPs 的表达,这与前人在水稻、拟南芥和玉米等物种中的研究结果一致[39]。此外,依据拟南芥中的NRE 元件以及水稻中鉴定的NRE-like 元件,对PeNLPs 的下游靶基因进行预测,结果表明:毛竹NPF 家族的PeNPF2.4、PeNPF2.5、PeNPF2.7、PeNPF4.1、PeNPF4.6、PeNPF6.2、PeNPF6.5、PeNPF6.8和PeNPF8.19的启动子区有TGACCC…N…AAGAG序列[8,40],推测它们可能是PeNLPs 的靶基因,但还有待于进一步验证。目前,对于NLPs 的认知主要是基于拟南芥、水稻和豆类[41]等草本植物,而对于木本植物NLPs 的功能验证鲜有报道,木本植物的生长需要大量的氮[42],且多年生长处于复杂的环境中,它们可能比草本植物具有更加复杂的调控网络,在氮饥饿及复氮处理过程中,PeNLPs 表达的波动变化表明其参与调控的复杂性。因此,要揭示非草非木的竹子中NLPs 的功能,尚需更广泛的深入研究。
4 结论
本研究从毛竹中鉴定出10 个NLP 家族成员(PeNLP1~ PeNLP10),依据蛋白序列差异它们可分成3 个组,分别具有4、2、4 个成员。PeNLPs 在毛竹各个组织的生长早期呈现高表达,这可能与各组织早期快速生长需要大量的氮素有关。氮素诱导PeNLPs 的表达,在氮饥饿和复氮过程中呈现波动变化模式,表明毛竹对氮饥饿过程的响应可能存在不同的信号通路,PeNLPs 各成员通过不同的表达来参与毛竹对氮饥饿的响应。研究结果为深入探索PeNLPs 的功能,解析其调控分子机制奠定了基础。
