珠海市不同时期新型冠状病毒基因组测序及溯源分析
2021-12-15龙冬玲黄辉涛魏泉德黄文燕
龙冬玲,黄辉涛,魏泉德,黄文燕
2019年12月,新型冠状病毒(SARS-CoV-2)在武汉被首次报道,目前该病毒在全球范围内已造成大流行[1]。截至北京时间2021年5月6日6时30分,全球累计确诊新冠肺炎病例155 790 320例,累计死亡3 254 439例,全球单日新增确诊病例857 854例[2]。新型冠状病毒属于β属冠状病毒,是迄今为止被发现的冠状病毒中第7种可以使人类致病的冠状病毒,其全基因组包含近30 000个核苷酸碱基,由14个蛋白编码区域构成。目前研究显示,新型冠状病毒与蝙蝠类SARS样冠状病毒基因组相似性最高,与导致人类感染的其他冠状病毒差异较大。新型冠状病毒全基因组测序可提示病毒的变异情况和揭示传染病的传播途径,对疾病防控策略的制定具有重要意义。珠海市于2020年1月19日检出首例新型冠状病毒肺炎病例,截止至2021年5月5日24时,全市累计确证病例113例,其中15例为境外输入病例[3]。为了解珠海市新型冠状病毒的变异和溯源情况,为后期及时准确追踪溯源病毒,我们选取了4份不同时期确诊为新型冠状肺炎病例的呼吸道标本进行测序分析,现将结果分析如下。
1 材料与方法
1.1 样本来源 4份上呼吸道样本均采集于珠海市2020年1月至12月确诊为SARS-CoV-2病例患者标本,其中咽拭子样本3份,鼻咽拭子1份。采用中山达安基因股份有限公司的新型冠状病毒2019-nCoV核酸检测试剂盒检测,确诊病例疾病分型参照新型冠状病毒肺炎诊疗方案(试行第七版)[4],具体样本信息见表1。
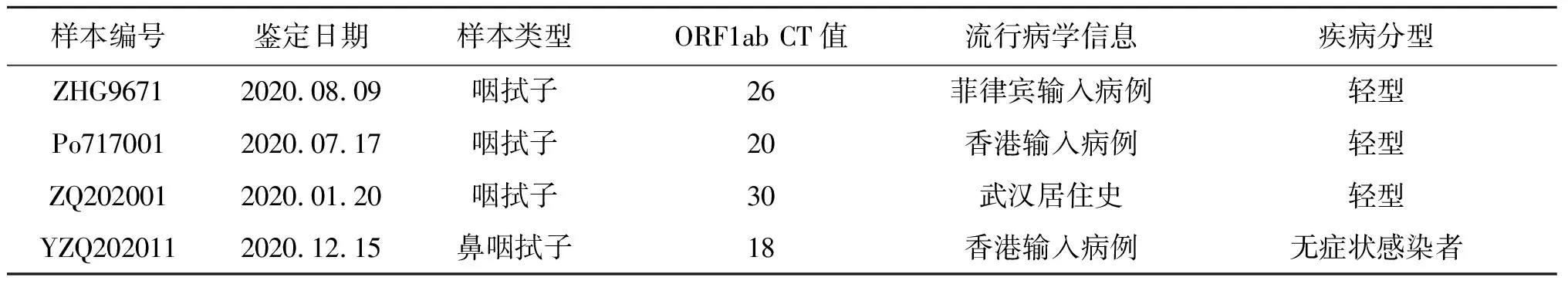
表1 标本信息Tab.1 Sample information
1.2 全基因组文库构建和测定 采用病毒核酸提取试剂盒(Qiagen RNeasy Mini Kit,货号:74104)提取200 μL样本的总RNA;应用随机引物逆转录方法,采用逆转录试剂盒(LunaScriptTMRT SuperMix Kit,货号:E3010L)生成cDNA。根据ARTIC Network公布的实验方案合成扩增引物(COVID-19 primers V3版)。采用多重PCR扩增试剂盒(Q5○RHot Start High-Fidelity 2X Master Mix,货号:M0494S)获得多重扩增产物。反应条件:98 ℃ 30 s(1 cycle);98 ℃ 15 s,65 ℃ 5 min(35 cycle);65 ℃ 5 min(1 cycle)。扩增产物回收采用核酸磁珠纯化试剂盒(AMPure XP beads,货号:A63880)等比例纯化回收。使用核酸定量试剂(Qubit dsDNA HS ASSAY kit,货号:1646715)对纯化核酸进行定量。采用产物末端修复试剂盒(NEB Next End repair/dA-tailing Module,货号:E7546S)对扩增产物末端修复,接头连接采用试剂盒(NEB Next Quick Ligation Module,货号E6056S;Native Barcoding Kit,EXP-NBD104/EXP-NBD114)对样本进行无扩增条形码标记与接头连接。上机测序采用试剂盒(Flow Cell Primming Kit,货号:EXP-FLP002)进行高通量测序。
1.3 数据分析处理 使用RAMPART软件对下机数据进行序列修剪和质量评估,使用minimap2和medaka组装软件生成一致性序列。参考基因组为早期公布的2019-nCoV病毒株Wuhan-Hu-1的基因组(GenBank Accession No.NC045512.2)。采用IQ-TREE软件构建序列分子进化树,进化树构建所纳入的参考序列以在GISAID数据库中筛选出来与所测序列相似性最高、同时期的国际上公布的序列作为同源性分析的参考基因组,每条目标序列选取250条相似序列进行分析,相同的参考基因组只选择一次,相似性序列查找完成后使用mega7.0软件线下分析,最大似然法构建系统进化树,步长值选择1 000,纳入线下分析的参考基因组分别为hCoV-19/Wuhan/WH01/2019、hCoV-19/Colombia/AMA-INS-VG-1193/2021、 hCoV-19/England/20134017404/2020、 hCoV-19/England/CAMC-137AA76/2021、hCoV-19/England/QEUH-C5B84E/2020、 hCoV-19/Guangdong/20SF028/2020、hCoV-19/Guangdong/20SF040/2020、hCoV-19/HongKong/HKPU-01524/2020、 hCoV-19/HongKong/HKPU-01574/2020、hCoV-19/HongKong/HKPU-02039/2020、hCoV-19/HongKong/HKPU-02290/2020、hCoV-19/HongKong/HKPU-06284/2020、hCoV-19/HongKong/HKPU-07782/2020、hCoV-19/HongKong/HKPU-08506/2020、hCoV-19/HongKong/HKPU-5161/2020、 hCoV-19/HongKong/HKPU-04778/2020、hCoV-19/HongKong/HKPU-5183/2020、hCoV-19/HongKong/HKU-201216-036/2020、hCoV-19/HongKong/HKU-201216-037/2020、hCoV-19/HongKong/HKU-201216-060/2020、hCoV-19/HongKong/HKU-201216-172/2020、hCoV-19/HongKong/HKU-201216-232/2020、hCoV-19/HongKong/HKU-201216-260/2020、hCoV-19/HongKong/HKU-201216-346/2020、hCoV-19/HongKong/HKU-201216-367/2020、hCoV-19/HongKong/HKU-201216-372/2020、hCoV-19/HongKong/VM20040795/2020、hCoV-19/Spain/NC-IBV-001418/2020、hCoV-19/Thailand/Chon-buri-SQ-NP0073/2020、hCoV-19/USA/NC-CDC-STM-000022772/2021。
2 结 果
2.1 基因组测序基本情况 4份样本经nanopore三代基因组测序结果见表2,基因组平均测序深度3 468×,基因组覆盖度在94.6%~99.7%。
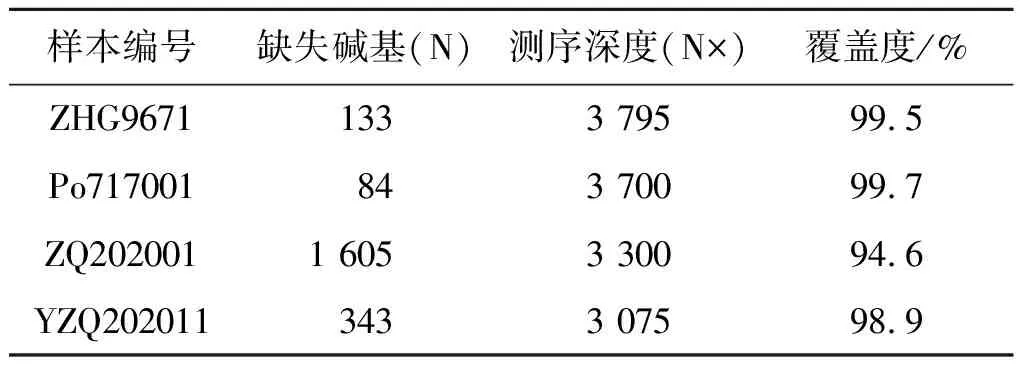
表2 基因组测序结果Tab.2 Results of the genome sequencing
2.2 核苷酸及氨基酸变异情况 以SARS-CoV-2 Wuhan-Hu-1株作为参考基因组,测序结果显示4份样本共检测到46个碱基位点突变,其中涉及氨基酸性质改变的非同义突变有27个,涉及5个编码区域,其中导致ORF1编码区氨基酸变异有12个、N基因编码区6个、S基因编码区6个、ORF3基因编码区2个和ORF9b基因编码区1个。除早期武汉居住史的病例ZQ202001,其余样本在S基因编码区均出现D614G的变异。位点变异情况详见表3。
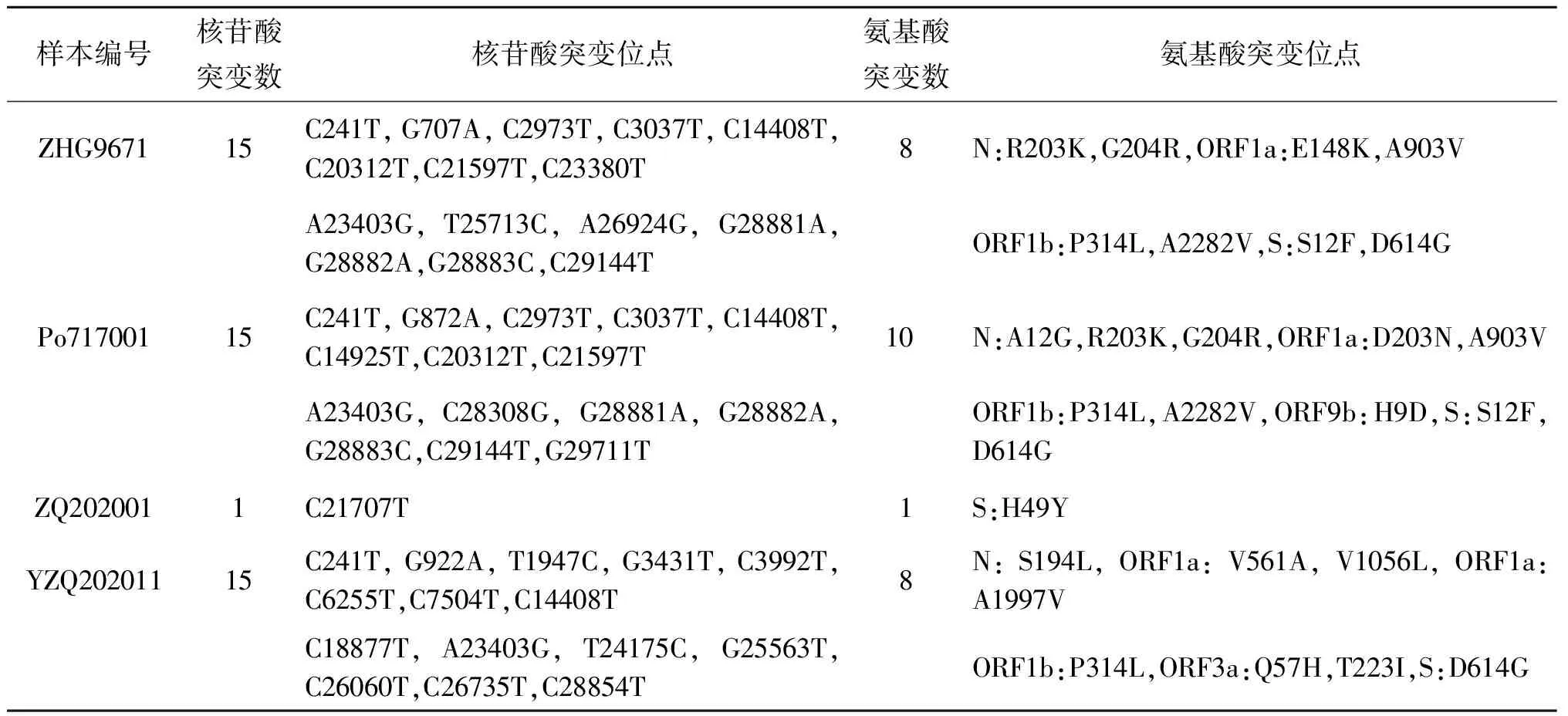
表3 位点变异情况Tab.3 Mutations of nucleotide and amino acid
2.3 系统进化树分析 全基因组测序分析结果显示,珠海市4份不同时期SARS-CoV-2病例来源于不同的进化分支,早期武汉输入病例ZQ202001分属于B,其核苷酸突变只与参考基因组SARS-CoV-2 Wuhan-Hu-1株相差1个碱基数,而2020年7、8月鉴定的来自菲律宾的境外输入病例ZHG9671与香港输入病例Po717001同属于B.1.1.63分支,与同时期香港公布的序列核苷酸相似性较高,遗传距离相近。2020年12月鉴定的香港输入病例YZQ202011分属于B.1.36分支。见图1。

图1 珠海市不同时期SARS-CoV-2全基因组系统发育进化图Fig.1 Phylogenetic tree based on SARS-CoV-2 complete genomes in different decades in Zhuhai city
3 讨 论
2020年1月底,SARS-CoV-2病毒刺突蛋白出现D614G突变,使得突变株陆续成为了后期的全球流行株[5]。目前,SARS-CoV-2经过在全球不同人群间突变进化与新冠疫苗选择压力的双重选择下,已演变成800多种不同的亚型或分支[6],一些突变株如501Y.V2南非突变株、B.1.617印度流行变异株的出现导致了新一轮疫情的暴发。本次研究选取的主要为2020年不同时期COVID-19病例的标本,基因组测序结果显示珠海市检测的疫情早期样本ZQ2020014与参考株SARS-CoV-2 Wuhan-Hu-1株的基因组高度相近,提示COVID-19大流行早期基因组保持着较低的进化水平,多数碱基突变导致氨基酸保守置换,对病毒的表型并未发生潜在的影响,与相关文献报道结论一致[7]。其余3份样本为全球疫情大流行期间检出的境外输入病例,共检测到45个碱基位点突变,其中导致氨基酸性质改变的突变有26个,以ORF1编码区氨基酸突变频率最高,不同于已报道的基因组变异均匀分布于各编码区[8-9],推测可能是选取例数太少所导致的偏差。
2020年5月以后,国内疫情逐渐趋于稳定,珠海市的COVID-19病例以境外输入为主,基因组测序分结果显示,来自菲律宾的境外输入病例ZHG9671与香港输入病例Po717001同属于B.1.1.63分支,2020年12月鉴定的香港输入病例YZQ202011分属于B.1.36分支,3份输入病例标本均与同时期香港公布的序列核苷酸相似性较高,遗传距离相近。从全基因组系统发育分析中可发现,各个国家上传的病毒基因组不仅在本国有聚集性的出现,并且在多个分支中可发现有许多不同国家的散发株,这说明SARS-CoV-2在全球范围内稳定持续传播着,世界范围内的高密度人口流动可使病毒在短时间内扩散,此外目前研究已报道世界范围内有大量的新冠感染者症状轻微或无症状[10],无症状感染者在COVID-19流行期间是潜在的强传染源,具有隐匿性,临床上不易被发现。亟需在目前筛查密切接触者、高风险地区人员筛查以及感染源调查的基础加强大数据的追踪管理,加入定期核酸监测来管控。
目前,对COVID-19临床样本的基因组测序多采用二代测序、三代测序平台完成。第三代Nanopore纳米孔测序技术属于单分子测序,它根据将一个纳米孔蛋白固定在电阻膜上,通过物理手段使DNA双链解链成单链,利用马达蛋白牵引DNA单链传过纳米孔,因不同碱基携带不同电荷在通过纳米孔时引起电阻膜上电流的变化识别碱基,实现高通量测序。三代测序具有测序读长长(超过150 kb)、速度快、测序数据实时监控、机器方便携带等特点。Lu等[11]曾采用不同测序平台对广东省疫情早期的临床病例样本进行测序,结果显示Nanopore混样建库PCR测序表现良好,尤其适用于病毒载量低的样本。本次研究所选取的4份样本均可获得27 493~29 810 bp长度的基因组序列数,基因组覆盖度94.6%~99.7%不等,但样本ZQ2020001和YZQ202011基因组序列缺失较多,测序质量与病毒载量、文库构建情况、原始标本核酸完整性密切相关,CT<30的临床标本均能获得较好的测序结果,本研究中的ZQ2020001和YZQ202011初次检测CT值虽均在30以下,但基因组缺失较多,可能是在检测过程中操作不当导致的核酸降解。对于COVID-19临床标本,应在检测阳性时及时提取全基因组进行疫源追踪,核酸反复冻融对核酸的稳定性及完整性均有较大影响,不利于后续基因组测序。
综上所述,本次研究对本市COVID-19部分临床标本进行全基因组测序,溯源分析显示基因组之间核苷酸突变具有多态性,4份临床标本均具有外地旅行史,均为输入病例,未出现本地感染。三代测序技术可有效用于原始样本中新型冠状病毒基因组序列的溯源分析,在地市级疾控应用前景较好。
利益冲突:无
引用本文格式:龙冬玲,黄辉涛,魏泉德,等.珠海市不同时期新型冠状病毒基因组测序及溯源分析[J].中国人兽共患病学报,2021,37(11):1003-1007. DOI:10.3969/j.issn.1002-2694.2021.00.138
