基于线粒体COI基因的3种草蛉的系统发育关系分析*
2021-12-09胡红岩张职显卢珍华任相亮马小艳马亚杰宋贤鹏
胡红岩,张职显,卢珍华,任相亮,马小艳,马亚杰,宋贤鹏,马 艳
(中国农业科学院 棉花研究所,棉花生物学国家重点实验室,河南 安阳 455000)
草蛉为昆虫纲(Insecta)脉翅目(Neuroptera)草蛉科(Chrysopidae)昆虫,能捕食蚜虫、叶螨和粉虱以及鳞翅目卵和幼虫等多种农林害虫,是生物防治上重要的天敌类群。草蛉资源丰富,种类繁多,在世界范围内广泛分布,据统计,目前全世界已知草蛉种类约1 415种[1]。草蛉科分为网蛉亚科(Apochrysinae)、幻草蛉亚科(Nothochrysinae)和草蛉亚科(Chrysopinae),其中草蛉亚科是最大的亚科,大部分的草蛉隶属于该亚科。由于存在地理亚种和生态型复杂等问题,部分草蛉在形态学上极为相似,难以鉴定,分类位置变动频繁,使得草蛉科的分类及系统发育关系变得较为复杂[2]。目前,草蛉科部分属种的命名及分类地位仍存在分歧,仅依赖外部形态特征进行草蛉物种鉴定存在一定的困难[3]。传统形态学分类能在较高级的分类阶元内确定物种的分类地位,但在较低级的分类阶元中,有时则难以确定物种的分类。分子生物学在昆虫分类中的应用和发展,为解决草蛉属种的鉴定问题提供了新思路。细胞色素氧化酶亚基I基因(COI)是动物线粒体DNA(mtDNA)中最保守的编码蛋白基因,其结构相对保守,同属物种的COI基因序列片段又存在足够的变异,可用于区分亲缘关系很近的物种[4]。因此,基于COI基因片段的DNA条形码技术被广泛应用于物种鉴定及系统进化研究[5-8]。
国内外已有关于草蛉线粒体基因序列的研究。如WINTERTON等[9]利用16S rRNA和COI基因序列片段分析了33种草蛉的系统发育关系。HARUYAMA等[10]通过COI基因片段序列分析和系统发育构建,探讨日本通草蛉(Chrysoperla nipponensis) A型和B型隐种与德国普通草蛉(C.carnea)的分子标记方法,并对日本通草蛉A型隐种的起源进行分析。PALOMARES-PÉREZ等[11]利用COI、COII、ND2和ND5分子标记方法,对墨西哥普通草蛉(C.carnea)的9个地理种群进行系统发育分析。目前,国内对草蛉科的研究主要集中在形态学的分类鉴定及生物防治等方面,而对草蛉的分子生物学及重要基因序列研究较少。聂瑞娥等[12]构建了日本通草蛉的cDNA文库,并基于16S rRNA序列对草蛉科7种昆虫的系统发育关系进行分析。部分研究者对草蛉线粒体基因组进行测序和注释,并依据基因组数据对草蛉亚科不同种进行系统发育分析[13-14]。YI等[3]利用线粒体COI基因片段对北京地区49种草蛉进行条形码分析,对草蛉科的18个种进行分子鉴定,发现其中的2个隐存种,认为基于COI基因的DNA条形码技术在草蛉分子鉴定方面具有很好的应用前景。
本研究以中国华北棉区3种常见草蛉的4个不同地理种群为研究对象,对其线粒体COI基因片段进行测序,统计分析不同序列的碱基组成及变异位点信息,并从GeneBank中下载其余9种草蛉昆虫的11条COI基因序列片段,构建草蛉的系统发育树,分析不同草蛉的亲缘关系,探讨DNA条形码技术在草蛉物种鉴定中应用的可能性,为草蛉分类、资源保护及应用研究提供理论依据。
1 材料与方法
1.1 试验材料
供试草蛉于2019年6—7月分别在山西运城、河北邱县、河南鹿邑和河南安阳等地棉田采集,标本采集信息见表1。室内采用形态学分类方法鉴定为中华通草蛉(Chrysoperla sinica)、丽草蛉(Chrysopa formosa)和大草蛉(Chrysopa pal-
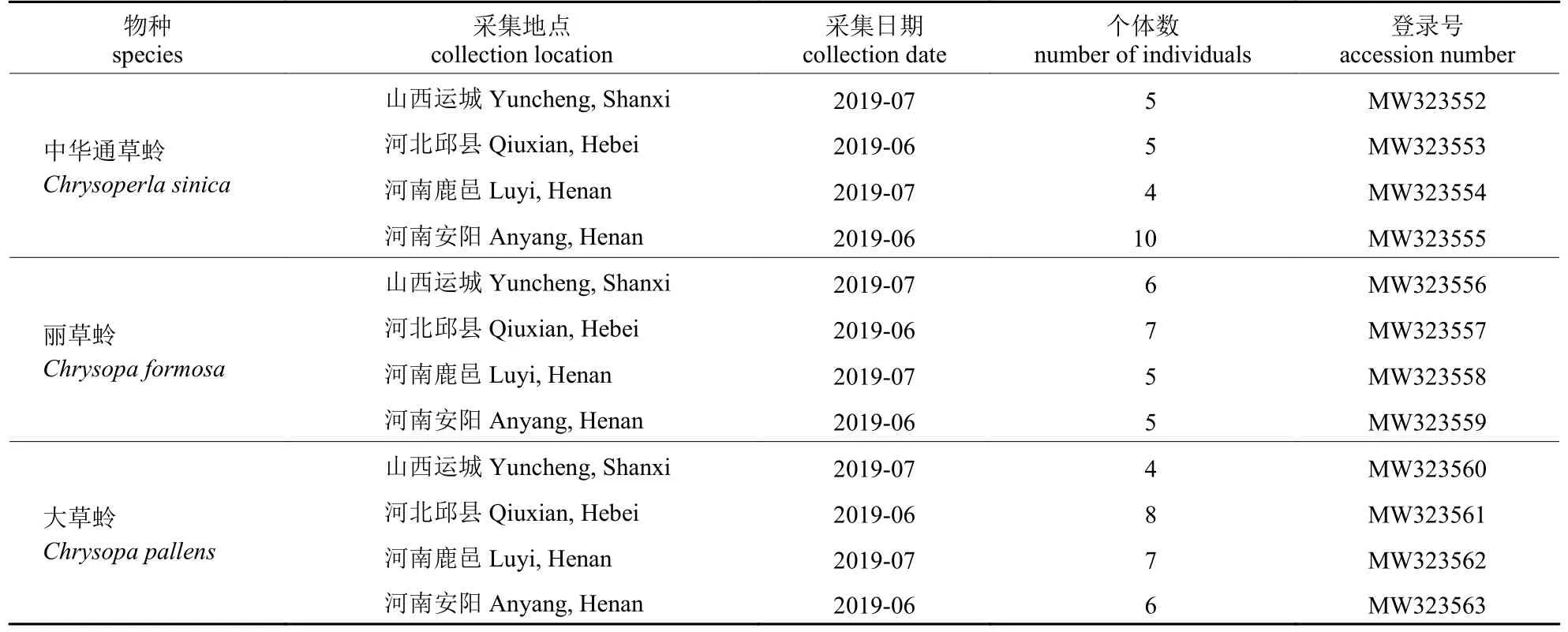
表1 3种草蛉的采集信息Tab.1 The sampling information of three green lacewing species
lens),试验用虫均为成虫,采集后的昆虫浸泡于95%酒精中,保存于中国农业科学院棉花研究所植保室,在-20 ℃冰箱中低温保存备用。从Gen-Bank基因库中下载9种草蛉昆虫共11条COI基因序列用于序列的对比分析,详细信息见表2。
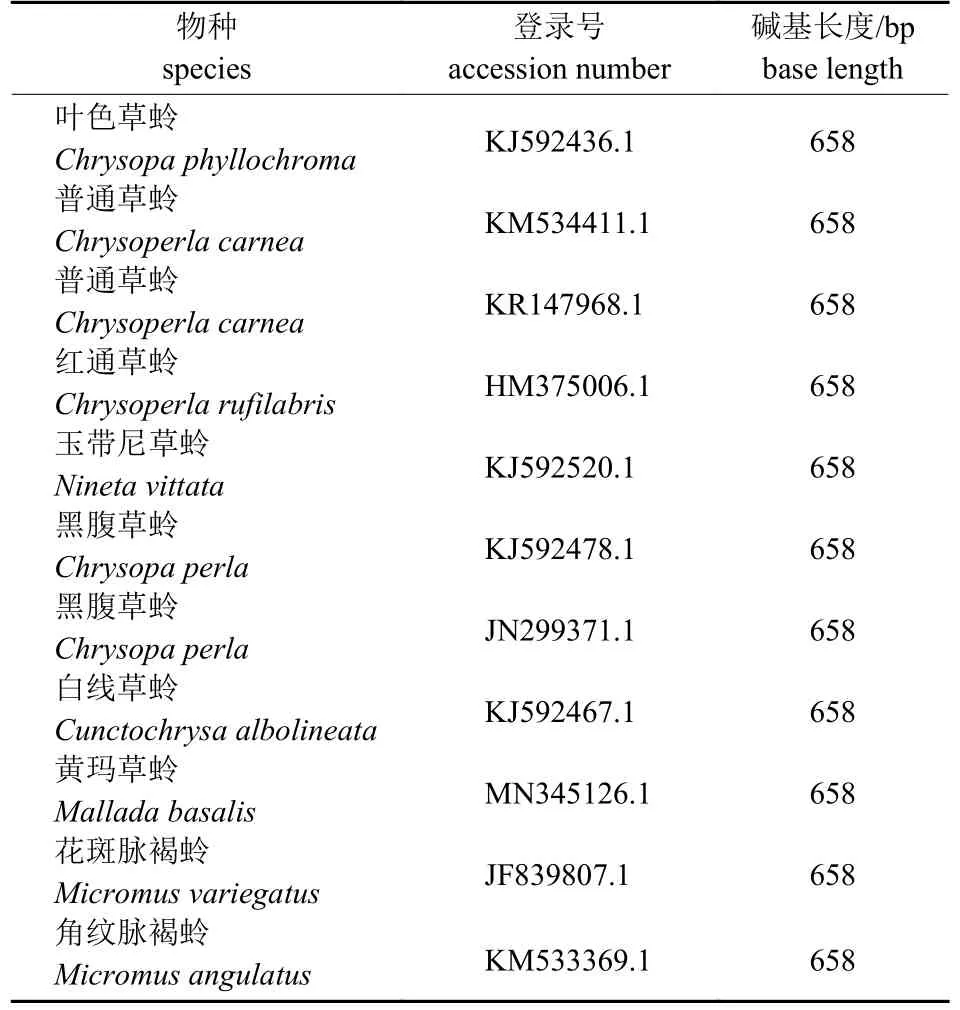
表2 NCBI下载的9种草蛉线粒体COI基因片段信息Tab.2 The information of mitochondrial COI gene sequence of nine green lacewing species downloaded from NCBI
1.2 试验方法
1.2.1 材料预处理
参考张德华等[15]的方法,将保存的标本用0.9%的生理盐水浸泡24 h,随后用蒸馏水冲洗干净,放在干净的滤纸上待用。
1.2.2 基因组DNA提取
挑取单头草蛉成虫置于1.5 mL离心管中,根据上海生工生物工程股份有限公司Ezup动物组织基因组DNA抽提试剂盒说明书的操作步骤提取草蛉DNA模板,DNA样品于-20 ℃冰箱保存备用。
1.2.3 mtDNA-COI基因扩增与检测
对3种草蛉总DNA中的线粒体细胞色素氧化酶(mtDNA-COI)基因进行PCR检测。所用引物为通用引物COIL1490:5′-GGTCAACAAATCATAAAGATATTG-3′和COIH2198:5′-TAAACTTCAGGGTGACCAAAAAATCA-3′[16]。PCR扩增反应体系为25 μL,包括10×PCR Buffer 2.5 μL、dNTPs混合液2.5 μL、5 U/μL的Taq聚合酶0.2 μL、10 μmol/L的上下游引物各1 μL和DNA模板1 μL和ddH2O 16.8 μL。反应条件:94 ℃预变性4 min,94 ℃变性30 s,53 ℃退火45 s,72 ℃延伸1 min,35个循环,最后72 ℃延伸10 min。PCR反应结束后,取5 μL mtDNA-COI基因扩增产物于1%琼脂糖凝胶电泳检测。选取目标条带清晰的样品委托上海生工生物工程股份有限公司对扩增产物进行双向测序。
1.3 数据分析
采用DNAMAN软件查看序列质量。对所测的3种草蛉COI基因序列分别进行比对和拼接。拼接结果在NCBI网站进行BLAST检索,与GeneBank基因库中同源序列对比,将获得的草蛉COI基因序列提交至GeneBank数据库,获得GeneBank登录号。在GeneBank数据库中下载11条草蛉的COI基因序列(表2),采用MEGA 7.0软件分析23条序列的碱基组成、位点信息、碱基转换与颠换值及其比值(R)等。以褐蛉科的花斑脉褐蛉(Micromus variegatus)和角纹脉褐蛉(M.angulatus)为外群,基于Kimura 2-parameter遗传距离模型分析不同草蛉的遗传距离。采用最大似然法(ML)和邻接法(NJ)构建系统发育树,采用的模型分别为GTR+I和p-distance模型。自展检验(bootstrap test)评估所建系统树的可靠性,重复1 000次评估各分支的置信值。
2 结果与分析
2.1 COI基因序列组成与变异
对4个地理种群的3种草蛉COI基因序列测序后去除上下游引物,得到658 bp的COI基因片段。经过比对分析发现:同种草蛉同一地理种群个体间COI基因片段序列没有差异。因此,将获得的4个地理种群的3种草蛉的12条草蛉COI基因序列提交至GeneBank数据库,获得GeneBank登录号(表1)。12条序列与NCBI上下载的9种草蛉序列进行对比分析,去掉前面1个碱基得到657 bp的有效长度。片段中未发现碱基插入或缺失,其中保守位点469个,变异信息位点188个,简约信息位点150个,自裔位点38个。序列结构中A、T、G和C的含量分别为28.6%、41.1%、15.4%和14.9%,A+T平均含量为69.7%,G+C平均含量为30.3%,呈现出明显的A+T碱基偏好。NCBI数据库中拟果蝇(Drosophia simulans) (JF872266.1、JF872338.1和JF-872410.1)线粒体COI基因片段序列的A+T平均含量为67.2%,G+C平均含量为32.8%,黑腹果蝇(D.melanogaster) (JF871600.1、KT117503.1和JF868423.1)线粒体COI基因片段序列的A+T平均含量为69.3%,G+C平均含量为30.7%。3种草蛉与拟果蝇和黑腹果蝇及其他昆虫的线粒体基因特征[13-14]类似。同种草蛉同一地理种群个体间碱基没有差异,不同地理种群之间碱基差异很小。其中,中华通草蛉4个地理种群间有3个变异位点,与同属的普通草蛉间有7个变异位点;丽草蛉4个地理种群间有1个变异位点,与同属的叶色草蛉间有60个变异位点;大草蛉4个地理种群间有1个变异位点,与同属的黑腹草蛉间有62个变异位点。表明草蛉COI基因序列比较保守,种内变异位点数小于种间。
2.2 碱基替换
由12种草蛉 COI 基因序列的转换数和颠换数以及位点变异速率 (表3) 可知:核苷酸的转换位点为25,颠换位点为40,R为0.6,转换主要发生在A和T之间,颠换主要发生在G和C之间。其中,第1位点替换数为8,R为7.4;第2位点替换数为1,R为2.7;第3位点替换数为56,R值为0.4。可见,碱基替换主要发生在第3位点,其次为第1位点,第2位点的碱基在基因序列中相对比较保守。
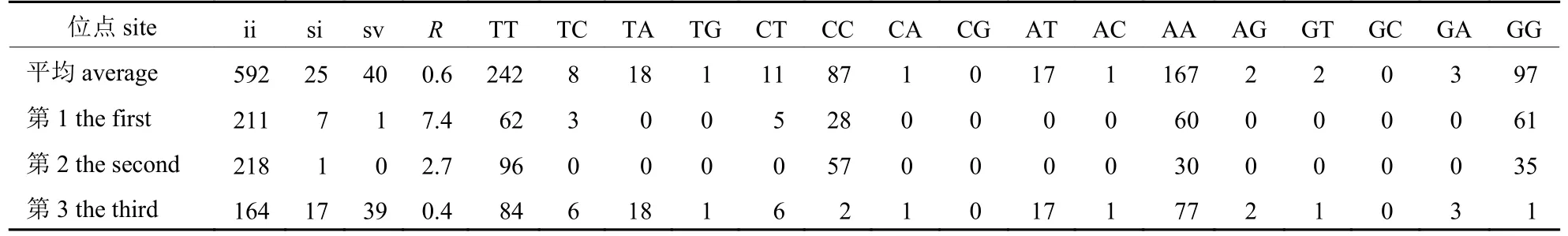
表3 12种草蛉COI基因的核苷酸碱基替换值Tab.3 The nucleotide base substitution value of COI gene in 12 lacewing species
2.3 遗传距离分析
由表4可知:同物种间的遗传距离介于0~0.005,其中,中华通草蛉4个地理种群的种内遗传距离介于0~0.005,丽草蛉4个地理种群的种内遗传距离介于0~0.002,大草蛉不同地理种群的种内遗传距离也介于0~0.002,黑腹草蛉2个地理种群的种内遗传距离介于0~0.002,普通草蛉2个地理种群的种内遗传距离为0。不同种间的遗传距离介于0.022~0.184,其中,外群角纹脉褐蛉与草蛉亚科的中华通草蛉和普通草蛉的遗传距离最大(0.184),普通草蛉和中华通草蛉的遗传距离最小(0.022)。遗传距离分析结果表明:草蛉COI基因序列相对保守,种内遗传距离小于种间。

表4 基于COI基因的Kimura 2-parameter模型的12种草蛉种群遗传距离Tab.4 The genetic distance among 12 lacewing species populations based on Kimura 2-parameter model of COI gene
2.4 系统发育树分析
以褐蛉科的花斑脉褐蛉(M.variegatus)和角纹脉褐蛉(M.angulatus)为外群,分别构建ML树和NJ树,所显示的拓扑结构相同,仅在各分支的置信度上略有差异。由图1可知:2个外群位于树的基部位置,与草蛉亚科的其他种类分开,说明花斑脉褐蛉和角纹脉褐蛉亲缘关系较近且较为原始,这与形态学分类一致。同一物种以较高的置信度聚为一小支,如大草蛉的4条序列以100%的置信度聚在一起,黑腹草蛉的2条序列聚为一支,丽草蛉的4条序列聚为一支,且分支自展值均为100%。近缘种以较高的置信度聚在一起,如普通草蛉2条序列和中华通草蛉4条序列聚在一起,分支自展值为100%。同属的不同种类聚在一起,如大草蛉、黑腹草蛉、叶色草蛉和丽草蛉,这与形态学分类相一致。
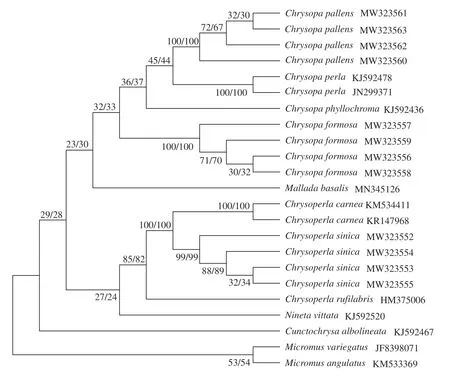
图1 基于COI基因序列的最大似然法(ML)/邻接法(NJ)系统发育树Fig.1 Maximum likelihood (ML)/neighbor-joining (NJ) tree based on the analysis of COI gene sequence
3 讨论
中国草蛉资源丰富,草蛉的分类及鉴定对天敌资源保护及优势草蛉天敌物种的开发和利用具有重要意义[17]。长期以来草蛉主要依赖传统形态学的方法进行鉴定,分类方法的局限性使一些形态特征相似或相近的种鉴定比较困难,关于草蛉科的区系研究及分类目前仍存在很多争议[2,18-19]。本研究运用DNA条形码技术,对草蛉亚科部分昆虫进行系统发育分析,进而从分子水平上明确不同草蛉的分类地位。运用无脊椎动物线粒体COI基因序列通用引物,对棉田常见的3种草蛉COI基因进行扩增和测序,序列拼接后在基因库中进行BLAST搜索。结果显示:中华通草蛉、大草蛉和丽草蛉COI基因序列与NCBI上登录的草蛉类线粒体COI基因序列同源性在95%以上,说明所测序列准确性强、可信度高。COI基因序列相对保守,同种草蛉不同地理种群之间序列差异很小。与NCBI下载的其他草蛉序列比较分析发现:核苷酸在第3位点替换数为56,远大于第1位点和第2位点的替换数。转换和颠换的比值R可以用于评估序列的饱和度,R值越低物种的分歧时间越长,进化速率越快[20]。密码子位点分析表明:第3位点的R值仅为0.4,低于第1位点和第2位点。可见,第3位点的进化速率最快。COI基因结构稳定,很少有插入和缺失基因,密码子第3位点碱基能够自由变异,用以区分近缘物种,因此,线粒体COI基因被广泛应用于物种区分[21-23]。
在DNA条形码分析中,种内、种间遗传距离间的差异程度是物种鉴定的判断标准之一。HEBERT等[24]对动物线粒体COI基因条形码进行了大量研究,认为98%的物种种内遗传距离小于0.02,种间平均遗传距离差异为11.3%。本研究结果表明:地理来源对同种草蛉COI基因片段序列的影响不大,草蛉种内平均遗传距离为0.001,明显低于遗传距离为0.02的阈值,种间的平均遗传距离为0.116,种间遗传距离是种内遗传距离的116倍,种内遗传距离明显小于种间,符合DNA条形码鉴定物种的标准,表明COI基因序列适用于草蛉的分类鉴定。
基于COI基因的草蛉ML/NJ系统发育树的拓扑结构分析发现:来自不同地理来源的同种草蛉物种聚为同一小支,如大草蛉和丽草蛉等,且分支自展值均为100%;草蛉物种间的近缘种聚集在一起,如普通草蛉、中华通草蛉和红通草蛉(分支自展值≥82%);亲缘关系较远的物种间差异表现较为明显,如白线草蛉和黄玛草蛉等。遗传距离和系统进化树综合分析结果表明:同一属内不同种间的遗传距离较小,亲缘关系较近;不同属间的遗传距离较大,亲缘关系较远;外群褐蛉科与草蛉科的10个种之间的亲缘关系较远,遗传距离也较大,与草蛉形态学分类结果基本一致。
4 结论
草蛉线粒体COI基因序列能够区分不同物种,证实基于COI基因的DNA条形码技术可以作为草蛉物种鉴定的辅助工具。研究结果为草蛉科昆虫DNA条形码数据库的构建提供了理论依据。
