基于微卫星和线粒体的四川白水河国家级自然保护区林麝遗传多样性研究
2021-12-01胡大明侯真真邓承敏陈旭吴杰陈磊陈勤岳碧松张修月
胡大明,侯真真,邓承敏,陈旭,吴杰,陈磊,陈勤,岳碧松,张修月*
(1.四川白水河国家级自然保护区,四川彭州611930;2.四川大学生命科学学院,濒危动物繁殖与保护遗传四川省重点实验室,成都610065;3.四川省养麝研究所,四川都江堰610016)
林麝Moschusberezovskii隶属鲸偶蹄目Cetartiodactyla麝科Moschidae麝属,国家一级重点保护野生动物,CITES附录Ⅰ物种(Smith,解焱,2009;魏辅文等,2021)。雄性林麝分泌的麝香是名贵中药材,也是高级香水和香料的原材料,具有极高的药用和经济价值(王海燕等,2006;王淯等,2006)。受经济利益和市场需求驱使,野生林麝一度受到大量猎杀,加上栖息地丧失等,我国野生林麝种群数量急剧下降,由20世纪60年代的250余万头下降到 20世纪90年代的10余万头(盛和林,1996)。尽管我国从20世纪50年代已开始林麝的人工驯养与繁殖研究,但由于疾病、遗传等因素影响,圈养林麝种群数量增加缓慢、规模养殖困难(袁阳等,2020)。因此,精确了解野生资源和遗传状况对林麝保护和资源利用具有重要意义。
近年来,濒危动物种群及其栖息地得到了较好的保护,但野生林麝资源的研究多是基于粪便、毛发等痕迹开展的种群密度及数量估算(胡忠军等,2007;姚刚,2014;姜海瑞等,2015;胡大明等,2019;王霞等,2020),仅有2个对野生种群遗传多样性的研究:冯慧等(2014)基于mtDNA D-Loop和姚刚(2014)基于主要组织相容性复合体对野生种群和圈养种群的比较,但受选用的分子标记限制,没有基于个体识别的遗传多样性调查。
微卫星是目前应用得最广泛、最优良的分子标记,能够进行精确的个体识别和遗传多样性估算。白水河国家级自然保护区内林麝具有一定的种群数量(胡大明等,2019;彭科等,2021;温平等,2021),但林麝种群遗传多样性未有报道。本研究基于微卫星和mtDNA D-Loop序列对保护区林麝进行遗传多样性研究和种群数量估计,了解保护区林麝种群数量和遗传状况,为野生林麝保护提供基础资料。
1 研究地概况
四川白水河国家级自然保护区位于四川省彭州市龙门山镇和小鱼洞镇(103°41′~103°57′E,31°10′~31°29′N),总面积301.50 km2,野生动植物资源丰富,生态环境良好(胡大明等,2019)。
2 材料与方法
2.1 样品采集
1份圈养林麝肝脏样本来自四川省米亚罗养殖场,10份圈养林麝粪便样本来自四川养麝研究所都江堰养麝场,46份野生林麝粪便样本来自白水河国家级自然保护区(表1),所有标本保存于四川大学生命科学学院生物资源与生态环境教育部重点实验室。野外粪便样品采用样线采集,样线设置在20条保护区日常监测线路内(根据粪便颜色和状况采集5 d以内的粪便),每条线路选择 3~7条样线,每条样线长3 km。
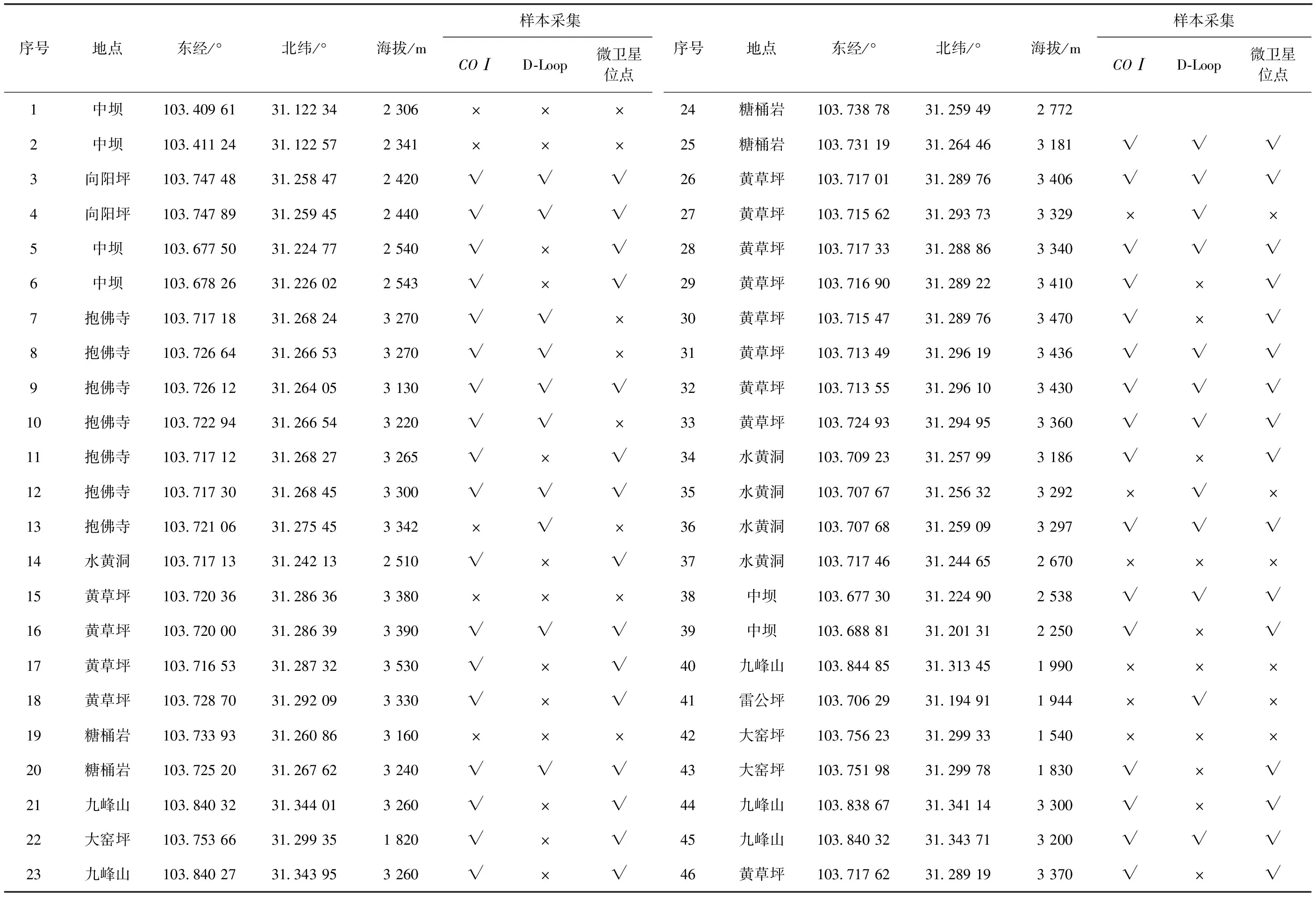
表1 46份野生林麝粪便样本采样信息
2.2 DNA提取及PCR扩增检测
分别使用M5 HiPer Universal DNA Mini Kit超强通用型DNA提取试剂盒MF033-plus-04(北京聚合美生物科技有限公司)和土壤/粪便DNA提取试剂盒TD601-50(北京天漠科技开发有限公司)提取林麝肝脏以及粪便DNA。提取的DNA-20 ℃保存备用。
PCR扩增体系总体积为25 μL,其中,正、反引物各1 μL、2×PCR Mix 12.5 μL、肝脏0.5 μL、粪便DNA 2 μL,用ddH2O补齐。S1000TM Thermal Cycler进行PCR扩增,程序为:94 ℃ 5 min;94 ℃ 30 s,56 ℃ 30 s,72 ℃ 30 s,35个循环;72 ℃ 10 min;4 ℃保存。
2.3 适于粪便DNA扩增的微卫星位点筛选
林麝微卫星位点已有较多报道,但有稳定扩增特性的四碱基微卫星位点较少,且用于粪便DNA扩增的报道更少。粪便等非损伤性取样的DNA一般量少且降解严重,其质量比其他组织DNA差,位点扩增能力也较差,因此本研究从林麝基因组序列以及卢婷(2017)已筛选的四碱基微卫星中重新挑选了部分四碱基微卫星位点进行优化。使用Krait v1.0.3(Duetal.,2017)从林麝基因组中预测潜在的多态性四碱基微卫星标记,使用Primer Premier 5.0设计引物,送北京擎科梓熙生物技术有限公司合成备用。根据引物参考Tm值设置温度梯度、使用肝脏DNA进行PCR条件优化。使用圈养林麝粪便DNA筛选优化后具有稳定扩增的引物对,选出能稳定扩增的引物对用于野生林麝粪便DNA的扩增。
PCR扩增体系和程序如2.2,扩增产物经2.5%琼脂糖凝胶电泳检测后,取5 μL送北京擎科梓熙生物技术有限公司基因分型。基因分型在ABI3730 DNA Analyzer上进行,使用GeneMapper version 4.0决定样本的等位基因数,等位基因大小相对于分子内标GS500LIZ决定。
2.4 野生林麝粪便鉴定及个体识别
野生林麝粪便使用COⅠ基因进行分子鉴定,引物序列:COⅠ-F:5’-ATAGCATTTCCCCGGA-3’,COⅠ-R:5’-TGTTCAGGTTTCGGTCTGT-3’,产物长度300 bp。PCR扩增后经电泳检测,产物条带清晰单一、长度与预期一致,送北京擎科生物科技有限公司测序,测序后使用BLAST在线比对鉴定。
利用MICRO-CHECKER(Oosterhoutetal.,2010)评估基因分型数据库数据的准确性;利用Cervus(Marshalletal.,1998),以各位点的期望杂合度(HE)、观察杂合度(HO)以及多态信息含量(PIC)选择最优位点排列顺序,进行基于微卫星分型数据的林麝个体鉴定,并计算PID和PID(sib)。PID为一个群体中随机抽取的2个不相关个体有完全相同基因型的概率,PID(sib)为一个群体中随机抽取2个同父同母的个体有完全相同基因型的概率(Kalinowskietal.,2010)。当PID<0.001且PID(sib)<0.01时,则得到个体识别所需最少数量的微卫星位点(Waits,2001)。
2.5 遗传多样性评估
通过MEGA 5.2(Tamuraetal.,2011)对线粒体D-Loop成功测序的序列进行比对,比对无误的序列利用DnaSP v5(Rozas,1995)计算遗传多样性指标参数:单倍型多样性(h)、核苷酸多样性(π)。
基因分型结果利用MICRO-CHECKER校正和评估,检测基因分型数据结果的可信度;利用Cervus 3.0.7计算等位基因数(A)、HE、HO以及PIC等参数。衡量微卫星位点变异程度的重要指标是PIC:PIC>0.5为高度多态性,0.5>PIC>0.25为中度多态性,PIC<0.25为低度多态性。哈迪-温伯格平衡(HW平衡)检验利用Genpop1.2(Raymond & Rousset,1995)进行,种群内Wright近交系数(Fis)使用PopGene1.31计算(Yehetal.,1999)。
2.6 种群数量及密度估算
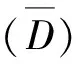
式中,n为样线数,xi为第i条样线的林麝个体数,Li为第i条样线面积。
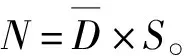
式中,S为保护区内林麝适宜栖息地总面积。
3 结果
3.1 样本采集及林麝粪便样品的鉴定
经粪便形态初步鉴定,在20条样线上采集到林麝粪便样品49个,经COⅠ鉴定为 46份林麝样品。
3.2 微卫星位点的筛选
利用Krait v1.0.3从林麝基因组中预测获得33个四碱基微卫星位点,加上卢婷等(2017)筛选的8个位点,共41个位点。以肝脏DNA和圈养林麝粪便DNA为模板,筛选出能稳定扩增的引物 7对。对筛选出的7对引物进行双色荧光标记(FAM或HEX)(表2)。

表2 适于林麝粪便DNA扩增的7个微卫星位点信息
3.3 林麝个体识别
按照LS-22、LS19、LS-21、LS-33、LS-17、LS-34、LS-23的顺序逐个增加用于个体识别的微卫星位点,对使用6个及以上微卫星位点能成功扩增的粪便样品进行微卫星分型,得到分型数据库。利用Cervus3.0.7按照位点的HO对分型数据进行个体识别模拟分析,当使用7个微卫星位点进行个体识别时,在46份样本中识别出30只林麝,PID(sib)<0.01(表3;图1),这符合种群大小精确评估的要求。
3.4 遗传多样性分析
3.4.1 D-Loop序列特征及遗传多样性评估对46份林麝粪便DNA进行线粒体D-Loop的PCR扩增,23份能够成功扩增并测序,序列长584 bp,碱基含量为A(32.34%)、G(14.17%)、T(31.65%)和C(21.84%),23个序列共39个多态性位点,占比6.68%。使用DnaSP定义了6个单倍型,Hap2拥有最多的个体数,为10个(表4)。遗传多样性指标:h=0.721 3,π=0.023 44。
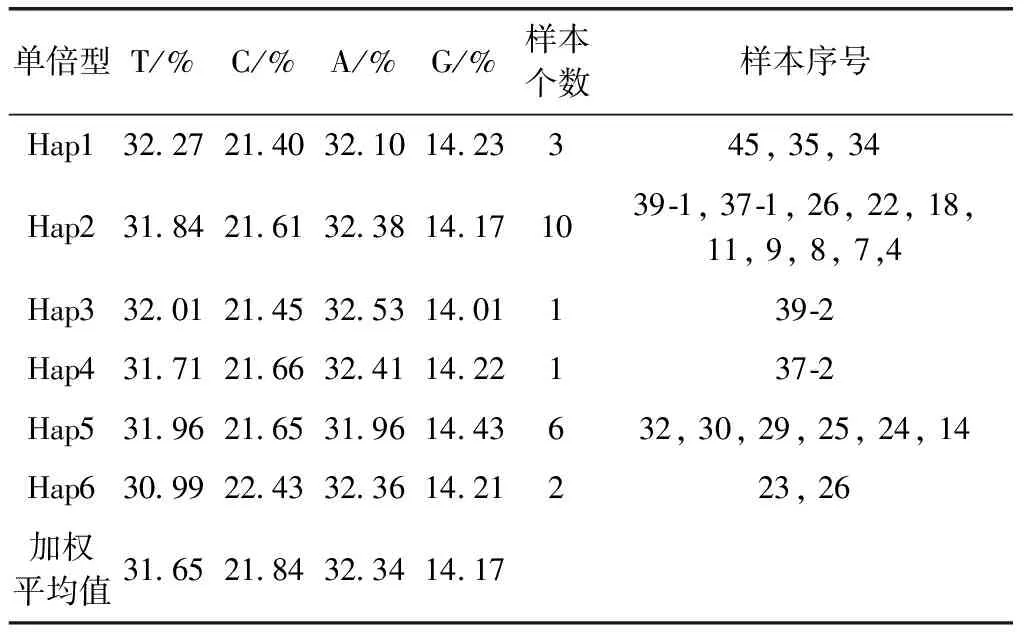
表4 林麝23个(6个单倍型)D-Loop序列(584 bp)的碱基组成
3.4.2 微卫星标记的遗传多态性评估7个位点的等位基因数为3~7,共35个。其中,等位基因数≥6的2个,占28.57%;等位基因数=5的3个;位点LS-34的等位基因数量最多(7个),位点LS-33的最少(3个)。保护区野生林麝种群的Ho为0.211~0.800,平均0.563;HE为0.475~0.798,平均0.602;PIC为0.419~0.750,平均0.536;种群内的Fis为-0.143 7~0.555 6;3个位点符合HW平衡(P>0.05),2个显著偏离HW平衡(0.01 表5 基于微卫星标记的白水河国家级自然保护区林麝种群遗传多样性分析 利用林麝个体识别结果,计算出样线内林麝的平均密度为4.39头·km-2,按照保护区林麝适宜栖息地面积约248.31 km2(胡大明等,2019)计算,保护区内林麝的种群数量约为1 090头。 物种遗传多样性水平与物种进化潜力密切相关(Frankham,1995),因此濒危物种遗传资源保护是物种保护的核心内容。了解濒危物种遗传多样性状况,是制定有效保护措施的基础。在濒危动物保护遗传研究中,通常采用粪便、毛发等非损伤性取样,获得的DNA不但量少且质量较差,因此获得适于非损伤性取样样本DNA扩增的遗传标记尤为重要。本研究基于基因组和发表的微卫星位点,筛选到7个四碱基的多态性微卫星位点,在圈养林麝 1 d 的粪便中能稳定扩增,对白水河国家级自然保护区获得的46个林麝粪便DNA样本扩增结果显示,在大部分样本中能成功扩增,并且基于 7个位点在46个样本中鉴定到30只林麝个体,表明这些位点能够应用于基于粪便DNA的野生林麝保护遗传学研究。 与微卫星位点的等位基因数量相比,杂合度不受样本数量的影响,是种群遗传多样性评估的较好参数。黄杰等(2013)利用7个微卫星位点对米亚罗圈养林麝进行遗传评估,平均HE和HO分别为0.854和0.782,王豆等(2019)使用13对微卫星检测巴山、秦岭和川西林麝3个圈养群体,平均HE和HO分别为0.830 2和0.389 7,表明这些圈养种群保存了较高的遗传多样性水平。野生种群微卫星多样性水平还未见报道,本研究基于微卫星检测到白水河保护区林麝种群的平均HE和HO为0.602和0.563,处于中等水平。因使用的位点不同,种群间遗传多样性比较受限,因此在林麝的保护工作中,应联合相关部门开展标准化微卫星标记系统的研究,建立共享平台,方便种群间比较研究。 HW平衡是评价种群遗传平衡的参数,处于遗传平衡的种群不容易受到近交及外来种群的干扰(杨建宝等,2012),种群相对稳定。濒危物种常常表现HW平衡偏离(Ardrenetal.,1999),造成偏离的原因很多,如近亲交配、种群迁移和遗传漂变等。本研究中有4个位点偏离HW平衡,均为杂合不足,且表现为近亲繁殖。保护区林麝种群表现这些遗传特征的原因有待进一步研究。 线粒体D-Loop是遗传多样性评估的常用分子标记。保护区野生林麝种群线粒体控制区与先前报道的线粒体控制区富含AT的结论一致(Brownetal.,1986),且G含量明显低于其他碱基,表现出较明显的碱基偏倚。Peng等(2008)对四川3个圈养林麝种群进行了D-Loop多样性评估,结果发现米亚罗种群的h和π分别为0.752和0.029 9、金凤山种群的为0.830和 0.028 2、马尔康种群的为0.836和0.056 8,冯慧等(2014)把陕西1个圈养种群和3个野生种群混合进行D-Loop遗传多样性分析,发现了较高的单倍型多样性(h=0.929)和核苷酸多样性(π=0.044 24),但这种混合种群研究可能高估遗传多样性水平。本研究在鉴定的30只个体中,成功扩增了21只个体,鉴定了6个单倍型,h和π分别为0.721 3和0.023 44,比圈养种群低,可能因为圈养种群建群者来源于多个野生种群。 目前对于林麝野生种群遗传多样性评估报道较少,无法评估各野生种群间遗传多样性差异,为了更好保护林麝,今后应大力开展类似研究工作,以全面了解我国林麝遗传资源状况。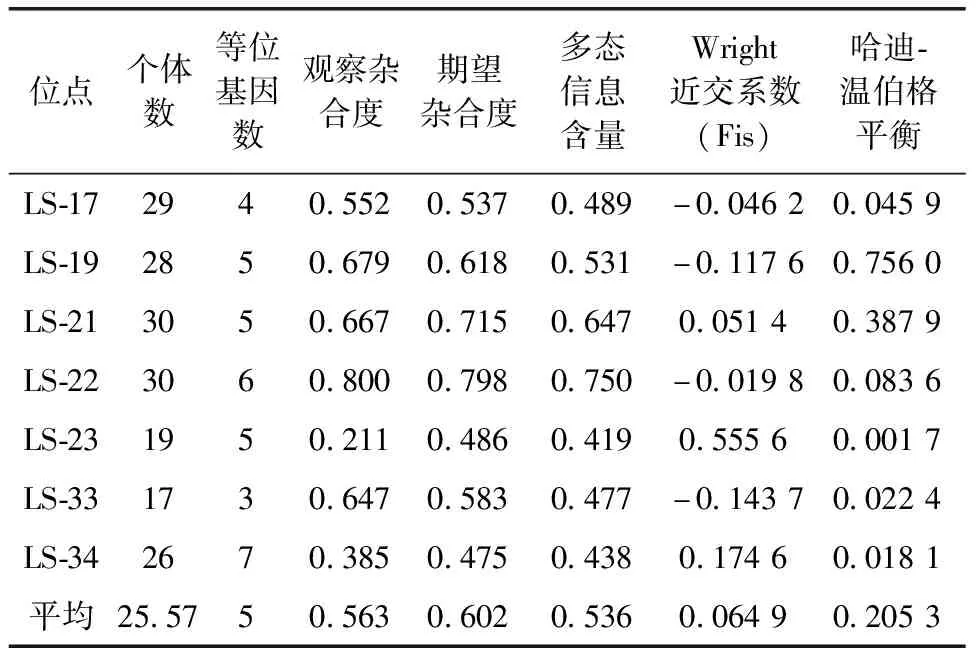
3.5 保护区林麝种群数量估计
4 讨论
