深度学习生物医学实体关系抽取研究综述
2021-11-12张益嘉鲁明羽
隗 昊,周 爱,张益嘉,陈 飞,屈 雯,鲁明羽
大连海事大学 信息科学技术学院,辽宁 大连116026
随着生物医学和互联网技术的发展,近年来,生物医学领域相关资料、文献、数据等数字化文本信息呈现出指数级增长趋势[1]。海量的生物医学文献中蕴含着丰富的、前沿的生物医学知识,是相关从业人员重要的知识来源,要从中迅速准确地获取真正需要的特定知识,必须借助于智能化的文本挖掘等有效技术手段和工具来应对信息爆炸时代的挑战。作为文本挖掘的关键基础和重要内容,信息抽取的相关任务日渐成为人们关注的热点。
信息抽取[2](Information Extraction,IE)肇始于20世纪60年代中期,是指从非结构化或半结构化文本中提取关键信息并整合为结构化信息的一项综合技术,目前已被广泛应用于信息检索、问答系统、舆情分析等多个重要任务。其中,命名实体识别(Named Entity Recognition,NER)和关系抽取(Relation Extraction,RE)作为信息抽取的核心任务和基础工作,也顺理成章地成为研究重点之一。在生物医学领域,信息抽取工作包括从医学文献中识别疾病名、药品名、化合物名等实体,并抽取实体间的交互作用关系等(如图1所示),这对于生物医学数据挖掘和知识发现研究的展开有着非常重要的意义和应用价值。例如,识别并抽取蛋白质之间的相互作用关系可以构建蛋白质复杂网络,以此进行复合物发现工作;提取药物和药物以及药物和疾病之间的相互作用关系可以开发“药物-疾病”知识图谱,并由此进行“老药新用”研究;药物之间的相互作用关系可以发现药物之间的不良反应,可应用于指导病人科学服药的在线诊疗系统。因此,以命名实体识别和关系抽取为代表的生物医学信息抽取工作在领域知识图谱构建、药物重定位、智能医疗等研究中具有非常重要的意义和作用。

图1 生物医学实体关系抽取Fig.1 Biomedical entity relation extraction
近年来,随着计算机硬件性能和计算能力的提升,由Hinton等人[3]提出的深度学习方法迅速成为研究热点,因其几乎不需要任何人工参与模型的训练过程且性能优异、效果稳定而被广泛应用于图像处理、自然语言处理(Natural Language Processing,NLP)等领域。常见的深度神经网络模型有卷积神经网络[4](Convolutional Neural Network,CNN)、Transformer网络[5]、循环神经网络[6](Recurrent Neural Network,RNN)及其变种的长短时记忆网络[7](Long Short-Term Memory,LSTM)和门控循环单元网络[8](Gated Recurrent Unit,GRU)等。随着深度学习时代的来临,神经网络模型为生物医学领域的命名实体识别和关系抽取工作也带来了新的突破。本文将对基于深度学习方法的生物医学命名实体识别和关系抽取的发展历程和研究进展分别进行归纳和概括。
1 命名实体识别
1.1 生物医学命名实体识别简介
在生物医学领域,识别领域文本中的实体是整个生物医学文本挖掘工作的基础和关键,命名实体识别任务主要包括识别生物医学文本中的基因、蛋白质、疾病、药物、化合物等名称,一直是NLP领域中的研究热点。由于生物医学文本的自身存在复杂性高、缩写词多、领域性强等特点,与通用领域相比,生物医学领域实体识别任务存在以下诸多难点,如:领域专有实体数量多且识别困难;短语类实体较多且边界难以划分;领域实体缺少统一的命名规则,产生许多存在歧义的命名实体;存在大量缩写、嵌套、含特殊字符的命名实体等。示例如图2所示。

图2 生物医学命名实体识别难点示例Fig.2 Difficulties of biomedical named entity recognition
1.2 生物医学命名实体识别主要评估指标
生物医学命名实体识别的评价标准为当模型对基于BIO或IOBES等方案标注的实体边界及预先定义的实体类型均正确识别时,方可判定预测结果为正确。评估模型性能常用的指标为准确率(P)、召回率(R)和F1值,具体的计算方法如下:

其中,TP表示模型预测为正例的真实正例个数,即正确预测的样本个数;FP表示模型预测为正例的真实负例个数;FN表示模型预测为负例的真实正例个数。FP+FN为模型预测错误的样本总数。
1.3 生物医学命名实体识别研究进展
早期生物医学的实体识别工作均是由人工构造词典或规则的方法完成[9-13]。这类方法均需要由领域专家参与构建领域词典或规则模板,费时费力且对领域知识依赖性强,扩展性和可移植性差,后期多被用于数据清洗,并与机器学习方法结合以提升模型性能。随着计算机技术的快速发展,传统机器学习方法被用于生物医学领域实体识别工作[14-15]。这类模型通过数据预处理进行特征选择,由机器代替部分人工完成对输入样本的学习和训练,这相对降低了人工负担和工作成本,在一定程度上提升了工作效率,但该类方法依旧依赖于特征工程,无法完全摆脱人工参与。
在基于深度学习方法的生物医学NER研究方面,Yao等人[16]利用神经网络对大量生物医学文本训练生成词向量,然后构建多层CNN进行命名实体识别。Li等人[17]采用双向长短期记忆网络(BiLSTM)方法构建NER模型。Zhao等人[18]在疾病名和化合物识别任务中提出了多标签CNN方法,将实体识别任务作为分类任务处理,使用多标签机制获取相邻输出标签间的关系。随着研究的不断深入,神经网络模型被发现无法处理序列标注任务中的标签间强依赖问题,即预测标签不仅与当前时刻的输入特征相关,还与先前时刻的预测标签相关。为了解决上述问题,研究者们考虑到条件随机场模型(Conditional Random Field,CRF)在序列标注问题中的优势,借鉴其主要思想,将标签转移得分加入到了目标函数中进行标签预测,提出了BiLSTM-CRF架构,如图3所示。Zeng等人[19]在药物名识别任务中构建了BiLSTMCRF模型,将双向LSTM与CRF结合,并使用了词向量和字符向量两种特征表示,在2011和2013DDI实体识别任务中均取得了较好的效果。Lyu等人[20]使用基于大量生物医学未标注数据预训练的词向量和字符向量作为特征表示输入,构建了BiLSTM-RNN神经网络模型,最后通过CRF层解析句子标签,在JNLPBA和BC2GM数据集上F1值达到73.79%和86.55%。Li等人[21]提出CNN-BiLSTM-CRF模型,利用CNN获取字符级特征表示用于捕获生物医学领域词汇的内部结构特征,与词向量结合后通过BiLSTM-CRF模型完成实体识别,在不依赖任何特征工程的前提下在JNLPBA和BC2GM数据集上F1值达到74.40%和89.09%。
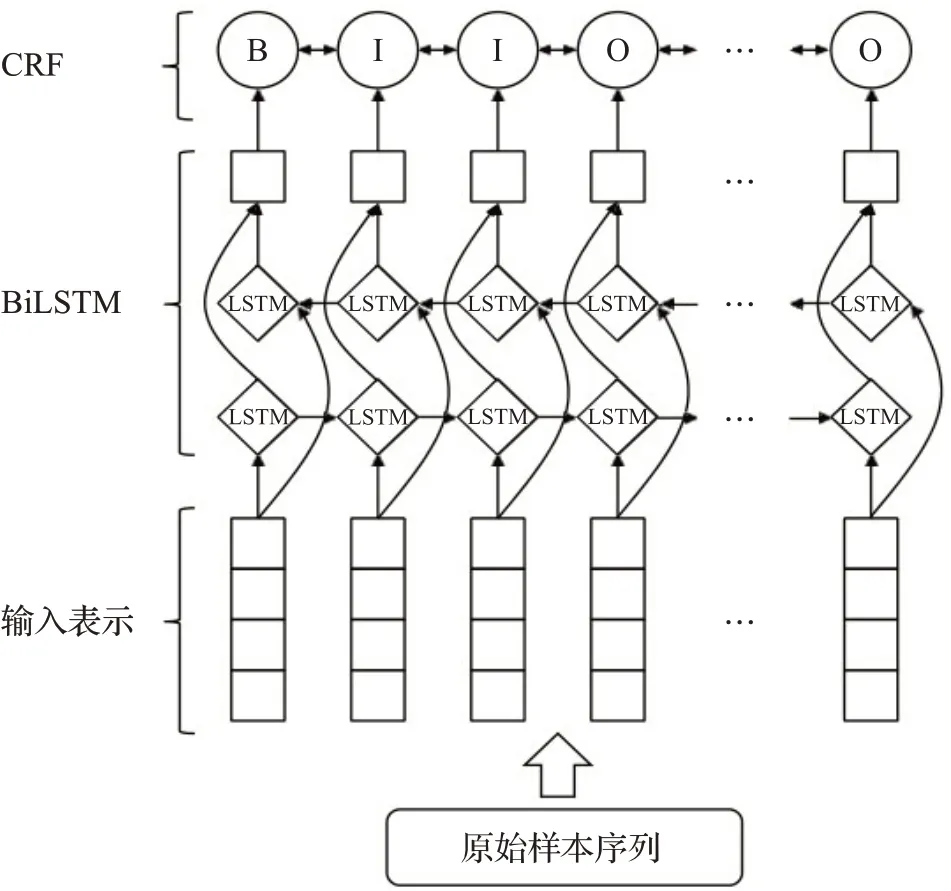
图3 BiLSTM-CRF模型基本框架Fig.3 Framework of BiLSTM-CRF model
对于生物医学实体识别,文本序列中的不同字符携带信息的重要性和影响程度不同,通常存在大量的冗余信息,如何在神经网络特征学习中突出局部关键信息的作用成为提高NER模型性能的关键。Rei等人[22]基于BiLSTM-CRF的基础架构,提出将词级向量和字符级向量的拼接方式改为基于注意力机制的权重重分配求和,用于突出在特定的生物医学领域下关键信息的重要程度,模型在四个生物医学领域公共数据集上均取得了较好的效果。Pandey等人[23]基于BiLSTM结合注意力机制构建了Encoder-Decoder模型,最后使用Skip-Chain CRF获取标签序列,在根据Medline数据库和电子健康记录(EHR)构建的两个数据集中的实体识别效果较Baseline均有明显提升。由于生物医学领域实体存在命名规则不统一以及大量缩写形式,Luo等人[24]在面向文档级化合物命名实体识别中,构建了BiLSTM-CRF与注意力机制相结合的NER模型,通过引入注意力机制获取全局信息以保障文档级数据中相同实体标记的一致性,并提供了四种注意力权重的计算方法,在CHEMDNER和CDR语料中分别取得了91.14%和92.57%的F1值,证明了注意力机制的有效性。近期,随着ELMo、BERT等预训练语言模型的出现,基于情境化向量表示的神经网络模型也被广泛地应用于生物医学领域。Lee等人[25]基于大量的生物医学未标注语料训练了BioBERT语言模型,Jin等人[26]基于PubMed摘要训练了BioELMo语言模型,Hakala等人[27]针对生物医学实体识别任务构建了多语种BERT。这类生物医学预训练语言模型在各项主流NLP任务上均取得了先进的性能。Yu等人[28]将BERT引入了BiLSTM-CRF模型,基于上下文语境动态更新的向量表示辅助模型在电子病历实体识别任务上实现了最佳性能。而Naseem等人[29]则整合了词向量、字符向量、BioELMo和BioBERT多种特征表示,并构建了基于注意力机制的BiLSTM-CRF,该模型在多种生物医学NER数据集上均取得了较好的性能。这类方法需要依赖高性能设备和大规模运算,但是大大提升了模型的特征捕获和编码能力。
深度学习方法虽然避免了对领域专家、专业知识和特征工程的依赖,但一般需要大量预先标注的训练语料,所以在基于深度学习方法的命名实体识别任务中如何在获取更多训练样本的同时降低噪声数据的影响也是最近研究的重点。除了上述基于单任务学习的深度学习方法外,由于特定领域训练样本不充分,许多学者采用迁移学习、多任务学习的方式训练模型,将与目标任务关联密切的辅助任务联合起来协同训练,在减轻对带标注的训练数据依赖的同时获取更多额外信息。Wang等人[30]基于BiLSTM-CRF构建了三种多任务学习框架,并通过参数共享的方式建立全局模型,分别学习不同生物医学实体类型的特征表示,通过对不同类型实体间词级和字符级的信息共享,模型性能得到明显提升,在包含四种生物医学实体类型的五个标准数据集上取得了先进的实验结果。由于生物医学领域缺少充足的标注训练样本。Wei等人[31]提出了基于多任务学习的多通道BiGRU-CRF模型,首先分别利用生物医学领域预训练词向量、字符向量和ELMo拼接后生成丰富的输入特征,并且使用单独的双向门控循环单元网络(BiGRU)对其进行训练,然后引入了类别标签相似的额外生物医学语料库辅助训练,该方法在JNLPBA和NCBI-Disease数据集上F1值达到76.0%和88.7%。Giorgi等人[32]从提升模型的泛化性出发,以当前主流的实体识别模型BiLSTM-CRF为基线,从多任务学习、迁移学习和变分Dropout三个方面进行改进,最终模型性能在多种生物医学数据集上均有提升,该项评估也验证了迁移学习、多任务学习等策略应用在训练样本较少的生物医学领域中的有效性。
表1 列举了近年来研究所采用的部分代表性数据集,包括名称、实体类型以及下载链接。表2归纳和总结了基于深度学习的生物医学命名实体识别方法的类型、代表工作和优缺点。

表1 生物医学领域命名实体识别部分代表性数据集Table 1 Mainstream corpora of biomedical named entity recognition

表2 生物医学命名实体识别方法总结及优缺点概述Table 2 Summary and limitations of biomedical named entity recognition methods
2 关系抽取
2.1 生物医学关系抽取简介
生物医学领域关系抽取任务在于判断文本中两个实体之间的关系,比如在药物文本中判断两个药物之间是相互促进、减弱还是会产生副作用等。生物医学关系抽取揭示了医学领域疾病、药物、基因等重要实体之间的语义关系[33],在医学知识图谱构建、可视化关系网络生成、老药新用等研究中具有重要作用。典型的生物医学关系抽取任务有蛋白质交互关系抽取[34](Protein-Protein Interaction,PPI)、药物相互作用抽取[35](Drug-Drug Interaction,DDI)、化合物疾病交互关系抽取[36](Chemical-Disease Relation,CDR)等。
生物医学文本资源丰富,但其中蕴含的信息错综复杂,存在大量领域性词汇,对专业知识积累要求较高。与通用领域相比,生物医学领域关系抽取任务存在以下诸多难点,如:文本句式冗长复杂、存在关系的实体对分布密集以及存在大量重叠关系等。相关研究表明,生物医学语料中所包含的实体数量为通用领域的2~3倍,存在关系的实体对更为通用领域4~6倍[37]。示例如图4所示,例中复杂的长句下不仅且包含大量生物医学领域特定词汇和缩写形式,而且存在密集分布的重叠关系实体对,此类情况在生物医学文本中非常常见,大大增加了关系抽取工作的难度。

图4 生物医学关系抽取难点示例Fig.4 Difficulties of biomedical relation extraction
2.2 生物医学关系抽取主要评估指标
生物医学关系抽取的评价标准为当模型能够正确识别测试样本的预定义关系类型时,方可判定预测结果为正确。与命名实体识别任务类似的,评估关系抽取模型性能常用的指标为准确率、召回率和F1值,依据模型提供的TP、FP和FN计算相应的指标。
2.3 生物医学关系抽取研究进展
早期的生物医学领域关系抽取工作均是基于词典和规则层面,即领域专家组织专业研究团队根据生物医学领域相关的词典、知识库、本体库等通过自然语言处理工具预处理后由人工设计规则模板完成医学实体间关系的抽取[38-40]。上述基于生物医学词典和规则的关系抽取方法在词典规模大、模板设计准确的情况下具有较高的准确性,但是可移植性和可扩展性差且要求工作人员具备专业的生物医学领域知识,无法识别模板外的生物医学实体关系,在海量的数字化医学资源中效果不佳,而且在实际研究过程中,生物医学领域词典的构建和规则模板的设计耗费了大量精力和人力。随着计算机技术的发展,传统机器学习方法在一定程度上减轻了研究者们的人工负担,降低了生物医学领域关系抽取任务的专业性壁垒。
传统机器学习方法将生物医学关系抽取作为文本n元分类问题处理,通过模型从标注好的语料中抽取丰富特征训练后得到n元分类器[41-42]。另外,通过自行设计适应生物医学领域特性的核函数用于实体间关系的抽取也是传统机器学习方法的主要研究内容之一,通过向高维特征空间的映射实现对分类样本的线性可分,是一种计算高维空间内积的方法[43-45]。基于传统机器学习方法的生物医学关系抽取与基于词典和规则的方法相比虽然一定程度上减少了人工负担,避免了构建大规模词典和设计大量规则模板,但仍需要大量的特征工程,且在处理语料方面产生了较多的资源消耗。
近年来,随着深度学习方法在各个领域的迅速风靡,研究者们开始将其应用于生物医学关系抽取工作。深度神经网络仅需少量甚至无需任何特征工程,即可自发地从领域文本中提取特征训练模型,且依旧保持较高的准确性和稳定性。在生物医学领域,CNN、LSTM和Transformer成为了当前关系抽取的三大主流神经网络架构。Liu等人[46]结合生物医学领域预训练词向量和位置向量表示构建了Text-CNN模型用于药物间相互作用提取,位置向量可以反映出输入样本中每个单词与目标实体对间的距离,适用于实体对分布密集的生物医学长句,模型在DDI2013数据集上F1值达到了69.75%。Hua等人[47]提出基于最短依存路径(Shortest Dependency Path,SDP)的CNN模型用于蛋白质相互作用提取,SDP可以直接提取出层次结构复杂的生物医学长句式中的核心依存结构,将其与词向量组合作为特征输入,在AIMed和BioInfer数据集上F1值分别达到66.6%和75.3%。Zhao等人[48]提出一种两阶段句法CNN,将词向量与句法信息、位置、词性等多种外部特征结合,使用Enju和Word2vec生成句法词向量,最后使用卷积操作提取特征完成关系分类。Lim等人[49]提出一种改进的二叉树LSTM,将词向量结合了位置、句法信息等特征,并为药物相互作用关系的检测和分类提供了多种模式,在DDI2013评测数据中关系检测F1值达到83.8%,关系分类F1值达到73.5%。Asada等人[50]将注意力机制与CNN结合用于药物相互作用提取,通过注意力机制突出目标句子中的关键信息。Yi等人[51]基于BiGRU构建了多层注意力机制的关系抽取模型,提供了词级和句级两种注意力权重的计算方式,最终模型在DDI2013数据集上取得了72.20%的F1值。Christopoulou等人[52]基于多任务学习策略构建了集成学习模型,采用基于注意力机制的BiLSTM进行句子内的关系抽取,同时引入Transformer用于提取句子间的实体关系,该模型在临床关系抽取评测任务上取得了较好的性能。上述基于外部知识或注意力机制的关系抽取方法在一定程度上提升了模型性能,但受限于对上下文情境建模不佳等问题,在面对生物医学的复杂长句时未能表现出良好的效果。
近期,生物医学领域使用图神经网络进行图结构表示的关系抽取研究在与日俱增。Song等人[53]在BiLSTM的基础上结合了图循环神经网络GRN,基于图的神经网络架构可以更好地建模层次结构复杂的生物医学长句,有效地提升模型的特征提取能力。Park等人[54]分析了生物医学领域的样本特点并充分地考虑到输入样本的上下文语境信息和空间结构信息,提出了一种注意力图卷积神经网络模型AGCN,基于注意力机制设计了新的剪枝策略用于捕获重要的句法特征,模型在DDI2013数据集上实现了76.86%的SOTA性能。另外,与命名实体识别任务类似的,当前预训练语言模型在诸多领域的先进方法中占据着主导地位,Zhang等人[55]基于ELMo和多头注意力机制构建了BiLSTM模型用于提取化合物-蛋白质相互作用关系(Chemical-Protein Interaction,CPI),通过引入情境化向量表示使模型性能达到65.9%。Sun等人[56]在BERT的基础上引入了包含蛋白质和化合物等概念信息的生物医学领域知识,并通过高斯概率分布对特征表示进行权重重分配,模型在CPI数据集上取得了76.56%的先进性能。上述基于图结构或预训练语言模型的方法通过对上下文情境全局建模,可以大幅地提升模型性能,已成为当前的热门研究点之一,但此类方法对标注训练数据的要求较高且需要依赖高性能的运算环境。
随着远程监督学习在通用领域的长足发展,学者们在生物医学领域亦有尝试。Lamurias等人[57]提出了基于远程监督的miRNA-基因关系抽取模型,可以有效地从未标注的生物医学文献语料中提取关系。为了减少远程知识库中有标记数据的噪声,Li等人[58]提出了多种启发式算法对生物医学样本进行预处理,在一定程度上缓解了远程监督学习标记准确性不佳的问题。Sousa等人[59]将远程监督学习与众包机制相结合,众包可以纠正或丢弃由远程知识库标记产生的噪声数据,将上述方法产生的新的标注数据应用在两个先进的生物医学模型中,二者均实现了更好的性能。
生物医学关系抽取作为信息抽取的核心工作,其重要性对数据挖掘的各项复杂技术影响深远,意义重大,将会继续成为未来的研究热点。对近年来生物医学领域关系抽取任务的研究进展进行概括和分析,归纳出了该任务的基本流程和框架(图5),以及主要深度学习方法的类型、代表工作和优缺点(表3)。最后,总结了该领域的部分代表性数据集的关系类型和下载链接,如表4所示。

表3 生物医学关系抽取方法总结及优缺点概述Table 3 Summary and limitations of biomedical relation extraction

表4 生物医学关系抽取部分代表性数据集Table 4 Mainstream corpora of biomedical relation extraction
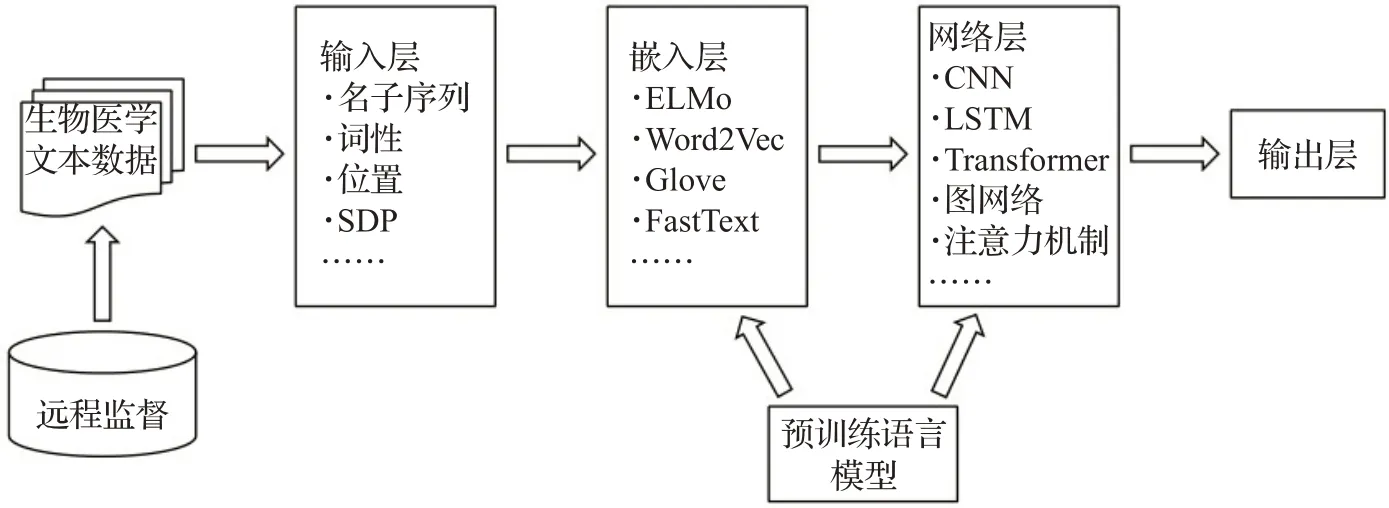
图5 生物医学关系抽取基本框架Fig.5 Framework of biomedical relation extraction
3 生物医学实体关系联合抽取
目前,生物医学实体识别和关系抽取通常被研究者们视为独立存在的任务单独研究,但在实际的生物医学信息抽取和文本挖掘工作中,命名实体识别作为关系抽取的研究基础和关键,二者之间联系密切,实体识别的准确与否对关系抽取结果起着至关重要的作用。当前对于上述两个子任务的处理方式大多为流水线处理,即先对海量生物医学文本数据进行命名实体识别,将识别结果两两组合为实体对,再进行实体对间关系的抽取,最后形成实体对和关系的三元组[60]。
流水线方式不仅会造成误差叠加,导致错误层级传播,而且忽视了两个原本联系密切的子任务间的关系。为了解决上述问题,研究者们考虑对生物医学文本进行实体识别和关系抽取的联合学习,即将二者结合起来,构建一个适合这两个任务的联合模型。
在前期研究中,Kordjamshidi等人[61]提出一种基于SpRL算法[62]的联合学习方法用于生物医学关系提取,根据不同层次构造了四大类共25种生物医学特征用于实体关系联合抽取,在BioNLP-ST 2013任务评测中表现较之前系统有了明显提升。Liu等人[63]基于结构感知机实现药物实体和关系的联合学习,设计打分函数并使用beamsearch方法缩小遍历范围,分别从实体特征和关系特征两方面选取了中心词、模式、领域词典、并列结构、依存结构等适应生物医学领域特性的8种特征用于实体识别和关系抽取,模型在DDI2013的DrugBank部分测评中药品名识别和药物关系抽取F1值达到90.5%和50.3%,均高于基于CRF构建的顺序处理基准模型。基于上述传统机器学习方法构造的生物医学实体和关系联合抽取模型,可以有效地缓解误差叠加问题,但是需要大量的特征工程,对于人力成本要求较高。最近,人们考虑使用较少特征工程的神经网络模型进行生物医学实体识别和关系抽取的联合学习任务。Li等人[64]构建了基于Bi-LSTM-RNN的联合学习模型,用于药物不良事件提取(Adverse Drug Extraction,ADE)和细菌-群落关系提取(Bacteria-Biotope,BB),该模型首先将词向量与字符向量和位置向量结合完成命名实体识别,然后开始抽取实体对之间存在的特定关系,结合依存句法分析通过最短依存路径在同一结构的模型中完成关系抽取工作,通过共享训练参数的方式实现联合学习目的。Bekoulis等人[65]在联合抽取模型中通过使用对抗训练(Adversarial Training,AT)的正则化方法给训练数据增加扰动,提高BiLSTM模型的鲁棒性。以上参数共享的方法虽然使实体识别和关系抽取两个任务之间通过共享训练参数实现共享,但实际还是将两任务先后分开处理,这仍然会产生错误传递和信息冗余问题。Luo等人[66]提出一种基于标注策略的生物医学联合学习模型,将命名实体识别和关系抽取联合抽象为一种序列标注任务,通过合并两个任务的类型标签设计了一种新的标注方案和提取规则,并且以词向量、字符向量和ELMo为输入特征构建了基于注意力机制的BiLSTM-CRF模型,该方法显著提高了联合抽取的性能并在一定程度上缓解了生物医学关系抽取中的实体重叠问题,但在面对复杂关系重叠问题时仍未有良好的解决方案。基于参数共享或标注策略的联合学习方法可以充分利用命名实体识别和关系抽取两个子任务间的密切联系,已逐渐成为信息抽取领域的重点研究方向之一。
4 挑战与展望
近年来,随着深度学习等技术的不断发展,以命名实体识别和关系抽取为代表的生物医学领域信息抽取的各项任务取得了长足的进步,但仍面临诸多问题和挑战,在技术和方法方面仍有广阔的提升空间。基于前述分析,本文认为生物医学领域命名实体识别和关系抽取仍有许多问题值得深入研究。
(1)在命名实体识别方面,由于生物医学文本句式多变、实体边界模糊、无统一的命名规则且存在大量的缩写和简略形式,面对当前指数级增长的数字化文本,通过引入丰富的领域知识表示或通过外部知识库指导等方式对实体进行消歧和链接,以保持命名实体的一致性是后期性能提升需要面临和解决的问题之一。另外,当前生物医学领域的命名实体识别研究仍局限在以文本数据为载体的单模态模式下,然而生物医学研究源远流长,积累了海量的领域资源,为了更充分地结合和利用这些丰富的领域先验知识,引入生物医学图像、音频等文本数据以外的其他多媒体信息的多模态命名实体识别也将是今后的主要研究方向之一。最后,受到语言问题的限制,世界各地的研究者们在生物医学领域的研究所产生的学术文献、电子病历、诊疗记录等数字化数据资源无法实现真正意义上的知识共享。将生物医学命名实体识别研究由单语言模式扩展到跨语言模式下,通过研究不同语言空间的语义相似度,构建跨语言语义表示模型进行多语言环境下的命名实体识别工作将是未来有意义的挑战性工作之一。
(2)在关系抽取方面,远程监督学习虽然可以有效地缓解生物医学这类特定领域带标注的训练样本不足的问题,但仍然存在远程知识库错误标注和错误传播问题,现有的解决方案大都采用多示例学习或启发式学习等方法缓解噪声问题,但此类方法都不可保证初始训练样本的标注准确性,下一步可基于强化学习或主动学习思想,使远程监督模型进行引导性学习,或可成为远程监督在生物医学领域关系抽取任务上新的尝试。另外,当前研究大多集中在单句层面,而在生物医学文本中跨句子间的实体也存在大量复杂多样的语义关系,采用指代消解、实体链接等方法与实体关系联合训练或利用图神经网络的全局建模优势提升跨句子实体间关系抽取效果将是当前需要探索的重要方向之一。最后,当前生物医学领域的关系抽取工作均需要预先定义固定标签的关系类别,如此深度神经网络模型只能抽取特定类别的生物医学关系。面对海量的关系类型复杂多样的生物医学数据,开发和构建基于半监督、弱监督或无监督的开放式关系抽取模型将是当前值得重点探索和研究的方向之一。
(3)当前实体关系抽取方法由先前的流水线模式发展到后来的联合学习模式,有效减少了误差层叠,但在模型内部仍存在无法有效地提取复杂的重叠关系等问题。未来可针对生物医学文本表述特征改进训练样本的标注方法,提出可区分重叠关系的细粒度标注方案,或通过针对每一种关系类型构造单独的特征子空间以避免重叠关系的问题,进一步改进和完善命名实体识别和关系抽取联合学习方法。
(4)除了上述从任务角度的研究展望外,从方法角度来看,学习能力强大的深度神经网络模型往往需要依赖大量标注好的训练样本,由于特定领域下训练语料匮乏且标注语料需要依赖大量的领域专家参与,为了节省标注成本并打破领域知识壁垒,基于小(零)样本学习、自监督学习或是针对现有标注数据引入其他解释性辅助知识的生物医学信息抽取工作将是未来的研究方向之一。另外,当前人工智能研究方兴未艾,深度学习技术迅速发展,深度神经网络模型在诸多领域都取得了优越的表现,但是其“黑盒”机制下参数学习的不可解释性依旧是当前亟待解决的问题之一。在生物医学领域,采用深度学习技术进行数据挖掘和知识发现,除了展示实验结果外,对其背后的生物学机制进行解释显得更为重要。因此,面向生物医学领域开发可解释的深度神经网络架构,例如从神经网络与不确定性知识推理相结合的角度出发展开研究将是当前充满意义和前景的探索方向之一。
(5)中文生物医学信息抽取由于语料库缺乏、中文句式复杂等问题仍落后于英文方面研究,而国内面向中医中药相关领域的数字化文本也在迅速增长,当前药物发现、知识图谱构建、在线医疗等工作正全面展开,信息抽取作为基础工作亟待研究,后期可面向数字化中文生物医学文本构建标注语料或训练大规模语言模型,针对基于中文生物医学文本的命名实体识别和关系抽取等问题展开研究。
5 结束语
作为生物医学文本挖掘的基础工作,信息抽取各项任务的研究价值也正得到越来越多的认可和重视。本文分析了近几年来命名实体识别和关系抽取在生物医学领域的研究现状,总结出国内外研究人员的工作进展,分析了当前面临的主要问题,并探索了未来的研究方向。在了解生物医学信息抽取的发展历程和研究现状的同时,也要总结分析NLP相关领域甚至其他领域的研究成果,为生物医学信息抽取各项任务更加深入地研究带来新的机遇,以创新理念引领信息抽取研究不断取得进步。
