双乾肉羊全基因组SNP/InDel分析及毛色相关候选基因鉴定
2021-10-30刘正喜胡明月白春艳赵中利马惠海闫守庆吉林大学动物科学学院吉林长春3006吉林省农业科学院畜牧兽医研究所吉林公主岭3600
刘正喜,武 斌,胡明月,白春艳,赵中利,曹 阳,马惠海*,闫守庆* (.吉林大学 动物科学学院,吉林 长春 3006;.吉林省农业科学院 畜牧兽医研究所,吉林 公主岭 3600)
单核苷酸多态性(single nucleotide polymorphism,SNP)是指在DNA水平上因单个核苷酸的替换、插入或缺失导致的核酸序列多态性[1]。插入/缺失多态性(insertion/deletion,InDel)是指基因组中某个等位基因座位上因插入或缺失了小片段 DNA所产生的多态性[2]。与其他种类的分子遗传标记相比,SNP和InDel标记具有在基因组中分布广泛、稳定性好和检测易自动化和规模化等优势。随着2代测序技术的快速发展和测序价格的下降,SNP和InDel分子标记已在大量非模式生物中被广泛挖掘,并广泛应用于遗传图谱构建[3]、重要经济性状主效基因定位[4-5]、种属鉴定[6]、个体识别[7]、生物群体遗传结构以及系统发育分析等方面[8-9]。
绵羊属洞角科(Bovidae)绵羊属(Ovis)动物,是人类最早被驯化的家养动物之一,可为人类提供食物和附属品(肉、毛、皮和奶等)[10]。近年来,国内羊肉产品消费需求急剧增长,但是目前在我国肉羊产业发展中,引进品种的繁殖力低和地方品种的生长速度慢是制约肉羊生产的两个主要问题,因此生长速度快且繁殖力高的肉用绵羊新品种或品系的培育势在必行[11]。
双乾肉羊是吉林省农业科学院利用杜泊羊为父本、吉林省本地绵羊为母本,通过杂交、回交以及横交固定等常规育种手段结合分子标记辅助选择方法,选育出的具有采食性好、生长速度快和产仔数高等种质特性的肉用羊新种群。双乾肉羊群体中有黑头、红头和白头3种毛色类型,杂交试验表明不同毛色表型均可稳定遗传。
本研究以红头和黑头双乾肉羊为试验动物,基于基因组重测序技术,检测红头和黑头双乾肉用绵羊全基因组SNP/InDel标记数量和染色体分布,然后基于SNP/InDel做选择信号分析,鉴定影响双乾肉羊毛色的候选基因及因果突变,研究结果可为解析双乾肉羊种质特性以及品种选育提供分子标记和理论依据。
1 材料与方法
1.1 试验材料本试验中52只黑头和60只红头双乾肉羊抗凝血样本采自吉林省乾安肉羊有限责任公司,使用EDTA-K2抗凝,摇匀后送至实验室-20℃ 保存备用。
1.2 主要试剂血液基因组DNA提取试剂盒(EasyPure®Blood Genomic DNA Kit)购自北京全式金生物技术有限公司;2×Taq PCR Mastermix、DL2000 DNA Marker和琼脂糖凝胶DNA回收试剂盒购自天根生化科技(北京)有限公司;其他试剂均为国产或进口分析纯。
1.3 DNA提取、文库构建及测序采用血液DNA提取试剂盒提取基因组DNA,1%琼脂糖凝胶电泳检测DNA的完整性,NanoDrop 2000超微量分光光度计检测DNA的浓度和纯度。将检测合格样品用于构建DNA小片段文库,文库质检合格后,使用Illumina PE150测序平台进行高通量测序,测序深度约12倍。建库及测序工作委托北京安诺优达基因科技有限公司完成。
1.4 原始数据过滤测序原始图像数据文件经 CASAVA碱基识别形成原始数据(Raw Data),对Raw Data进行质控分析、碱基质量分布分析、测序数据过滤后获得高质量数据(Clean Data)。数据过滤主要包括去除接头污染、低质量以及N比例大于5%的reads。
1.5 全基因组SNP和InDel变异检测采用BWA(v0.7.15)软件将Clean Data比对到绵羊参考基因组上(Oar_rambouillet_v1.0)[12],然后通过Picard(v2.21.2)软件进行对比,对结果进行排序和标记重复处理,最后用GATK(v4.1.4.0)软件进行SNP和InDel检测[13]。通过该软件的SelectVariants功能筛选出高质量的变异位点,过滤标准参考其官网推荐阈值,并利用R软件包CMplot绘制SNP密度分布图。
1.6 选择信号分析采用VCFtools[14]软件,以50 kb 作为滑动窗口,25 kb作为滑动步长,计算红头和黑头双乾肉羊之间的群体分化指数FST;将分布在前1%窗口的FST值对应的窗口定义为受选择区域,使用SnpEff(v5.0)软件对受选择区域进行基因注释[15],从而筛选毛色相关基因,并利用重测序数据分析候选基因的多态性。
1.7 MC1R基因多态性与毛色的相关性分析根据NCBI数据库中绵羊MC1R基因序列(NC_040265.1),采用Primer Primier 5.0软件设计引物(MC1R-F:5′-AGGGCGGAAGAGCGACACTGC-3′;MC1R-R:5′-GATGGCACCCAGGAAGCAGA-3′),引物由生工生物工程(上海)股份有限公司合成。采用PCR产物直接测序的方法分别对46只黑头和54只红头双乾肉羊的MC1R基因编码区内2个错义突变位点进行基因分型。PCR 扩增反应体系20 μL:2× PCR Master Mix 10 μL,DNA 模板1 μL,正向和反向引物(10 μmol/L)各0.5 μL, ddH2O 8 μL。PCR扩增条件:95℃预变性2 min;94℃变性30 s,60℃退火30 s,72℃延伸90 s,共30个循环;72℃延伸2 min。PCR产物送生工生物工程(上海)股份有限公司测序,采用Chromas和Haploview软件确定样品2个SNP位点的基因型和单倍型。
2 结果
2.1 测序数据统计及比对通过对6只红头和6只黑头双乾肉羊进行全基因组重测序,红头双乾肉羊命名为RSQ-1~6,黑头双乾肉羊命名为BSQ-1~6,共获得Raw Data 430.15 G,平均测序深度约为12倍,各样本碱基数量在29.85~45.42 G,reads数在198.97~302.78 M;经过过滤获得Clean Data 424.73 G,占原始数据98.74%,所有样品Q30均大于93.19%。将Clean Data同参考基因组进行比对,结果表明,各样本的比对率最低为99.88%,最高为99.91%,平均比对率为99.90%。
2.2 全基因组SNP和InDel变异检测基于GATK软件对12只双乾肉羊全基因组范围内的SNP和长度小于或等于60 bp的InDel进行检测和过滤后,共鉴定了32 338 805个高质量变异位点,平均每kb的变异位点数量为11.51个。变异位点以SNP为主,共计27 877 089个,其在基因组中的平均密度为9.92 SNP/kb(图1),包含错义突变 120 895 个,无义突变1 305个;另外有同义突变206 743 个;SNP主要以转换为主,转换类型以A/G型最多,颠换类型中以G/T型最多。碱基转换的发生频率是碱基颠换的2.45倍。本试验获得InDel位点4 461 716个, InDel长度以1 bp 和2 bp 为主,3~10 bp 的InDel 1 415 519个。不同染色体上的 SNP/InDel 出现频率不同,20号染色体频率最高,平均每kb包含13.95个,X染色体频率最低,平均每 kb仅为7.72个。

注:横坐标代表染色体的物理位置,窗口大小为1 Mbp;纵坐标代表染色体;绿色代表SNP密度小的区域;红色代表SNP密度高的区域图1 各染色体SNP密度分布图
2.3 全基因组选择信号检测按照Holsinger和Weir的统计方法检测红头和黑头双乾肉羊全基因组变异序列的FST值[16],FST值越大表明不同群体间遗传分化程度越大。将1 124个分布在前1%的FST值对应的窗口确定为受选择区域(图2),通过SnpEff软件在受选择区域内共注释到1 977个基因,其中在两种毛色双乾肉羊群体中分化程度最高的毛色相关基因为MC1R。进一步分析测序数据,在MC1R基因编码区发现了5个SNP,即c.218 T>A、c.361 G>A、c.429 C>T、c.600 T>G 和c.735C>T,其中c.218 T>A及c.361 G>A为错义突变,c.218T>A造成第73位氨基酸由蛋氨酸变为赖氨酸;c.361G>A导致其翻译的氨基酸由天冬氨酸变为天冬酰胺,其他3个SNP位点均为同义突变。12个测序样本的5个SNP位点基因型结果见表1。

注:横坐标为染色体号;纵坐标为FST 值图2 红头和黑头双乾肉羊全基因组水平上的FST选择信号分布

表1 重测序个体MC1R基因5个SNP位点的基因型
2.4 群体基因分型与相关性分析以MC1R-F和MC1R-R为引物进行PCR扩增,获得了与预期大小相符的584 bp目的条带,对46只黑头双乾肉羊和54只红头双乾肉羊进行c.218 T>A和c.361 G>A 2个错义突变位点基因分型。PCR产物测序结果表明,2个SNP位点均存在3种基因型(图3)。在c.218 T>A位点,46只黑头羊中,35只为AA基因型,11只为AT基因型,而所有的54只红头羊的基因型均为TT;在c.361 G>A位点,46只黑头羊中,35只为AA基因型,11只为AG基因型,而所有的54只红头羊均为GG基因型;单倍型分析结果表明2个错义突变位点完全连锁,有TG和AA 2种单倍型,单倍型TG频率为59.5%,单倍型AA频率为40.5%。以上结果表明,2个错义突变与双乾肉羊黑头性状完全相关,且黑毛色表型对红毛色表型为显性。
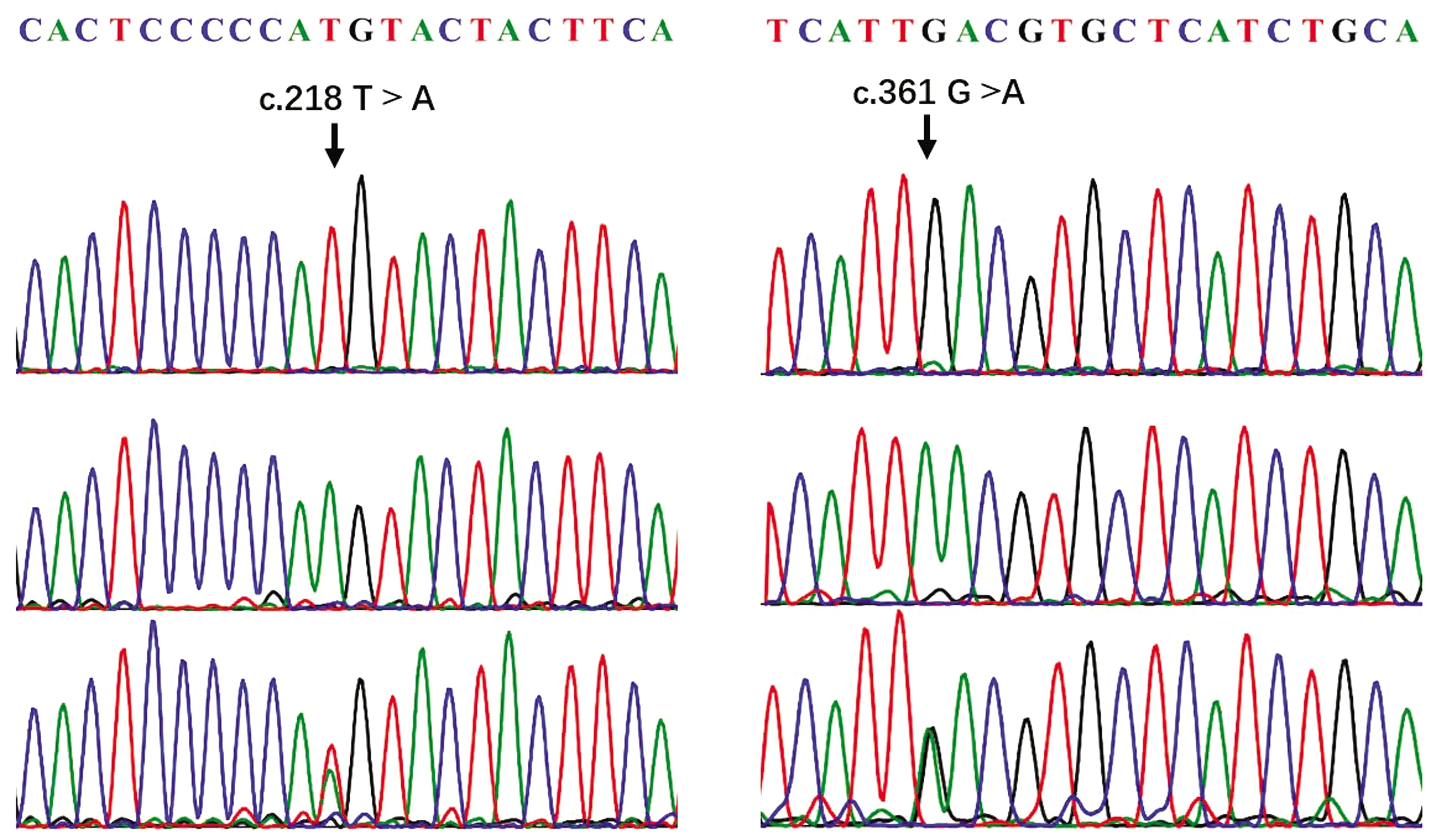
图3 MC1R基因c.218 T>A和c.361 G>A 多态性位点测序峰图
3 讨论
畜禽重要经济性状主效基因的精细定位以及因果突变的解析是当代动物遗传育种的重要任务之一。在2代测序技术出现之前,主效基因定位主要采用RFLP、RAPD和SSR等分子标记,但由于受到标记数量的限制,主效基因定位耗时长、费用高,且难以精细定位[17]。近年来,由于2代基因组测序技术具有高通量、快速和高效等特点,已被广泛应用于动物全基因组范围内SNP和InDel标记的筛选与鉴定[18-19]。JIANG等[5]对8头荷斯坦公牛进行全基因组重测序,共获得插入或缺失长度为1~49 bp 的InDel标记912 302 个,每个个体的标记数量范围为323 490~411 070。SABAHAT等[8]对2个巴基斯坦骆驼品种的103个个体进行简化基因组测序,筛选获得了63 619个SNP,然后基于这些标记对2个品种的群体遗传结构及品种间遗传关系进化了系统分析。双乾肉羊是利用生长速度快且肉品质好的杜泊羊与抗寒能力强且产仔数高的本地绵羊杂交选育而成的肉用绵羊新种群,目前其种群内的遗传结构以及优良种质特性的遗传基础并不清楚。因此,本试验基于重测序技术对12只红头和黑头双乾肉羊全基因组水平上的SNP和InDel标记进行了筛选,分别获得了27 877 089个SNP和4 461 716个InDel多态性标记,为该种群遗传多样性以及选择信号分析提供了丰富的遗传标记。
动物毛色通常是一个品种或物种的重要特征,可作为形态学标记进行物种或品种纯度的鉴别,同时,毛色的差异也会对动物的生长速度、生产性能以及疾病的抵抗力具有一定的影响,因此对动物毛色多样性的形成与调控机制的研究具有重要理论和实践意义[20]。目前基于全基因组SNP/InDel的FST选择信号检测策略被广泛应用于家畜重要经济性状相关候选基因和因果突变位点的筛选与鉴定。ZHOU等[21]基于76只有色羽鸭和30只北京鸭的基因组重测序数据做全基因组关联分析,结果表明MITF基因所在染色体区域具有较强的选择信号,通过比较北京鸭和鹅的基因组发现在MITF的外显子1M和外显子2之间存在约6.6 kb的缺失,该缺失与北京鸭的白毛色完全相关。HENKEl等[22]对1匹斑点马的基因组进行了约19倍深度的重测序,然后对5个斑点候选基因进行结构性变异检测,结果表明MITF基因外显子6~9区间约63 kb的大片段缺失与其斑点毛色以及听力丧失有关。双乾肉羊群体中有3种毛色类型,即红头、黑头和白头,红头和黑头表型个体杂交试验表明黑色对红色为显性,毛色表型与生产性能的关系目前还并不清楚。本试验基于重测序获得的双乾肉羊SNP和InDel标记,计算红头和黑头2个群体之间的FST值,获得了1 124个选择区域,其中哺乳动物毛色调控主效基因MC1R位于14号染色体的其中1个选择区域内。
MC1R基因编码黑素皮质激素受体1(melanocortin receptor 1),该受体是最小的G蛋白偶联受体,具有7个跨膜结构域[23]。黑素皮质激素受体1与黑色素细胞刺激激素α(α-MSH)结合,然后激活膜上的腺苷酸环化酶系统,通过环磷酸腺苷激活酪氨酸激酶催化细胞内的黑色素合成[24]。黑素皮质激素受体1在动物毛色形成与调控中具有重要的作用,研究表明MC1R基因多态性与多种动物毛色多样性相关[25]。赖伟宁等[26]采用候选基因法对野生型和黄毛色豚鼠MC1R基因进行PCR扩增与测序,发现该基因存在2 760 bp缺失多态性,群体基因分型结果表明黄色豚鼠均为缺失纯合子,而野生型豚鼠基因型为野生型纯合子或缺失杂合子,从而推测该基因的缺失与豚鼠的隐性黄毛色有关。本试验基于FST值的选择信号将与双乾肉羊毛色相关的候选基因定位到MC1R基因上,依据重测序数据发现在该基因的编码区内存在5个SNP位点,其中2个为错义突变,3个为同义突变。对2个错义突变位点进行PCR扩增,对产物直接测序并进行群体基因分型,结果表明所有红头毛色个体2个位点均为野生型等位基因纯合子,而黑头个体为突变型等位基因纯合子或杂合子,表明2个错义突变与黑头双乾肉羊毛色相关,且黑色对红色为完全显性。
本研究利用高通量重测序技术在12只双乾肉羊全基因组水平上共获得了27 877 089个SNPs和4 461 716个InDel 多态性标记;基于FST的选择信号以及群体基因分型确定了MC1R为红头和黑头双乾肉羊毛色表型的候选基因以及该基因编码区内的2个错义突变与双乾肉羊黑毛色完全相关。本研究为双乾肉羊种质特性的遗传基础研究以及分子标记辅助选择育种提供了科学依据。
