基于SNP分子标记的泰山/泰科麦系列小麦遗传解析
2021-10-21亓晓蕾李兴锋吕广德王瑞霞君孙宪印孙盈盈陈永军钱兆国
亓晓蕾 李兴锋 吕广德 王瑞霞 王 君孙宪印 孙盈盈 陈永军 钱兆国 吴 科
(1泰安市农业科学院,271000,山东泰安;2国家小麦改良中心泰安分中心,271018,山东泰安;3泰安市农业农村局,山东泰安,271000)
小麦是我国三大粮食作物之一,不断提高小麦育种水平对于促进小麦生产发展、保证国家粮食安全具有重要意义。近年来,由于商业化育种对新品种的迫切需要,在小麦杂交育种中主要利用大面积高产品种作为杂交亲本,导致新育成的品种(系)间遗传相似性不断提高,突破性品种越来越少[1]。对小麦品种(系)进行亲缘关系和遗传差异解析,能够更好地了解不同品种(系)的遗传特点,发掘优异的种质材料,对于指导小麦杂交育种的亲本选配、提高小麦育种水平具有重要意义[2-3]。小麦基因组在DNA水平上的研究成果为分析小麦品种间的亲缘关系提供了新的方法和手段。陆续有RAPD[4]、AFLP[5]和SSR[6-9]等标记应用于小麦种质遗传分析。随着高通量测序技术的出现和发展及后来出现的自动化 SNP芯片分型技术,使通过大规模高通量SNP基因芯片进行小麦遗传组成研究越来越普遍。SNP在定位目标基因、构建高密度遗传图谱和群体结构分类等方面具有越来越大的使用价值和发展空间,如小麦遗传图谱的构建[10-15]、分子标记辅助选择育种[16-18]、全基因组关联分析[19-22]、遗传多样性[23-25]和物种进化[26]研究等方面。曹廷杰等[23]利用Illumina 90k iSelect SNP标记技术分析了河南省自2000年以来近14年审定小麦品种的遗传多样性和遗传基础,结果表明96个小麦品种中,多数品种亲缘关系较近,94.3%的品种间遗传相似系数在0.65~0.81之间;刘易科等[25]利用90K SNP芯片技术对我国主要小麦种植区的240个小麦品种(系)进行全基因组扫描,通过分析参试品种(系)间平均遗传相似系数发现,参试品种(系)间平均遗传相似系数变幅为0.13~0.99,有87.05%的品种(系)遗传相似系数在0.60~0.78之间;聚类分析结果发现我国各省市部分单位因品种(系)交流频繁,育成的品种(系)间出现遗传相似性较高现象。自上世纪90年代至今,山东省泰安市农业科学院在经历了4次育种目标更换,先后育成并审定15个小麦品种,目前还有多个小麦新品系正在参加国家和山东省小麦区域试验,其中以泰山27、泰山28和泰科麦33为代表的小麦品种,在小麦生产中发挥了重要作用。这些小麦品种(系)的遗传基础及遗传多样性差异有待分析明确。利用小麦15 K育种芯片对21个供试小麦品种(系)进行全基因组扫描,分析不同年份育成小麦品种(系)的遗传距离,明确其位点/染色体区段构成差异,揭示全基因组水平上的遗传多样性特点,解析遗传差异的分子基础,以期为小麦新品种组配和选育提供参考依据。
1 材料与方法
1.1 试验材料
供试小麦材料为山东省泰安市农业科学院育成的21个小麦品种(系)。为了探讨不同年份育成小麦品种(系)的遗传构成特点,将21份材料依据育成(审定)年份,分成1990-1999年(90s)期间、2000-2009年(00s)期间、2010-2019年(10s)期间和现在(Present)4个时期进行分析(表1)。

表1 供试小麦材料名称、组合及审(认)定时间Table 1 Wheat materials, combinations and released time
1.2 试验方法
1.2.1 基因组DNA提取 采集供试材料的新鲜叶片组织,利用CTAB法提取基因组DNA。利用琼脂糖凝胶电泳和核酸蛋白测定仪(Nano Drop ND-2000,美国Thermo Fisher Scientific公司)检测DNA质量及浓度,确保达到后续芯片试验要求。
1.2.2 SNP位点分型和质检分析 利用小麦 15 K育种芯片对供试小麦品种(系)进行全基因组扫描,在中玉金标记(北京)生物技术股份有限公司平台进行样本DNA的SNP分型,共获得13 947个SNP位点分型数据。为进一步获取高质量的基因分型结果,对数据进行筛选和过滤,选取在芯片检测中Call rate均大于97%的位点标记,过滤后得到10 101个SNP位点的分型结果,剔除基因型检出率小于80%的SNP位点,剩余8692个SNP位点。此外,由于杂合位点在后代会产生分离而干扰检测结果的精密度,因此进一步筛选去除样品中出现杂合分型的位点,最终在所有检测品种(系)中共获得2082个纯合的SNP位点。结合SNP位点在小麦基因组对应位置分析,最终获得2029个定位于小麦21条染色体上的SNP位点。
1.2.3 数据统计分析 结合小麦15 K育种芯片基因型数据信息,将获得的2029个SNP数据分别转换为0(最小等位基因型)和1(相对等位基因型)格式,建立原始矩阵。统计所有SNP位点多态性信息指数(PIC),同时利用SNP分子数据建立的0~1数据矩阵进行每个 SNP位点的等位变异丰富度(Aij)和PIC的估算,同时估算A、B和D基因组及全基因组的遗传丰富度(Ri)和多样性指数(Ht),具体计算参考郝晨阳等[27]方法。利用 NTSYS-PC ver.2.2软件中的“DICE”计算品种间遗传相似系数,用非加权配对算术平均法(unweighted pair group method arithmetic averages,UPGMA)绘制遗传关系树状图[28]。用“1-DICE”表示遗传距离,计算不同年代育成品种(系)的平均遗传距离。
1.2.4 基因型图谱构建 结合小麦15 K育种芯片物理位置信息,随机选择均匀分布于全基因组的403个SNP标记(每条染色体约20个)绘制21个小麦品种(系)的基因型图谱。根据以上各SNP位点的等位变异的碱基序列在染色体上标记不同颜色,其中AA标记为红色、TT标记为绿色、CC标记为黄色、GG标记为蓝色、NN(缺失)标记为黑色。利用MapChart 2.32绘制21个小麦品种(系)基因型图谱。
2 结果与分析
2.1 SNP位点的遗传多样性
根据基因分型结果,比较2029个多态性SNP位点在A、B、D 3个基因组、7个同源群和21条染色体的分布特点。结果(表2)表明,2029个SNP基因位点在A、B、D基因组中的分布分别为730、820和479个,A、B、D基因组平均等位变异丰富度分别为2.07、2.14和2.13,平均遗传多样性指数分别为0.24、0.24和0.21。综合2个多样性指标可以看出,B基因组的遗传多样性最高,其次是A和D基因组。由图1可知,在7个同源群中,平均等位变异丰富度依次为 H3>H6>H7>H5>H2>H4>H1,平均遗传多样性指数依次为H6>H3>H2>H7>H5>H1>H4。综合2个多样性指数可以看出,二者的分布变化趋势基本一致,第3和第6同源群具有较高的遗传多样性,第1和第4同源群的遗传多样性较低。
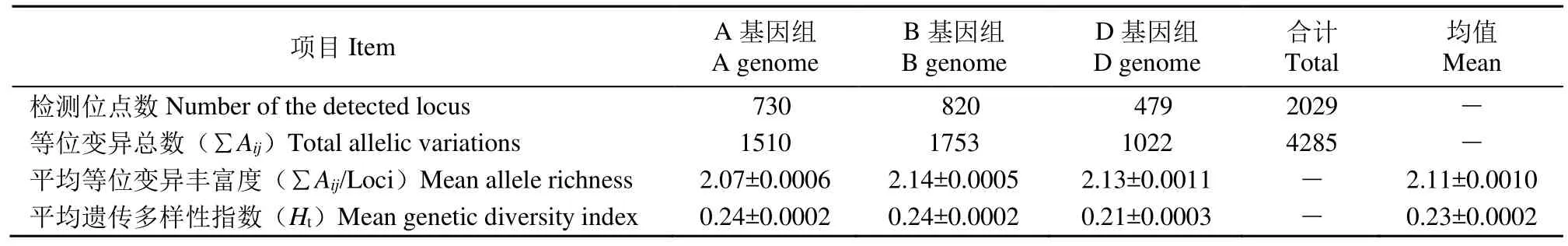
表2 3个基因组的遗传多样性比较Table 2 Genetic diversity comparison of three genomes
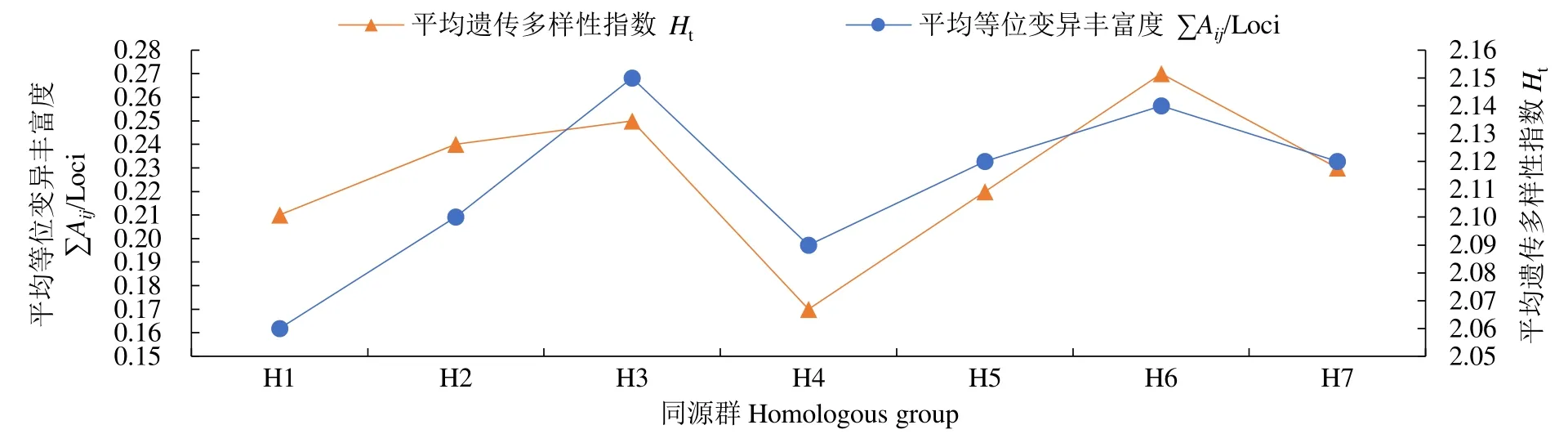
图1 育成品种(系)7个同源群的遗传多样性Fig.1 Genetic diversity of seven homologous groups of the varieties (lines)
由图2可知,在21条染色体中,A基因组的1A染色体的变异丰富度和遗传多样性指数均很低,6A的平均等位变异丰富度偏低;B基因组染色体的平均等位变异丰富度和多样性指数均较高,且2个指标变化趋势基本一致;D基因组的2D和6D染色体的平均等位变异丰富度较高。整体来看,3A、1B、6B染色体的遗传多样性较高,而1A、6A的遗传多样性偏低。

图2 育成品种(系)21条染色体的遗传多样性Fig.2 Genetic diversity of 21 chromosomes of varieties (lines)
2.2 供试材料的遗传距离分析
结果(图3)表明,不同年份小麦品种(系)平均遗传距离的变异范围为0.00~0.37。从平均遗传距离的变化曲线来看,自90s以来,遗传距离总体呈先升后降趋势。由于只有1个90s育成品种,其平均遗传距离为0.00,00s品种(系)间的平均遗传距离为0.10,10s品种(系)间平均遗传距离出现最高值0.37后,现在(Present)品种(系)的平均遗传距离为0.07,降到最低。

图3 不同年代选育品种(系)的平均遗传距离Fig.3 Average genetic distance of wheat varieties (lines) in different ages
2.3 供试材料的聚类分析
为明确21个小麦品种(系)之间的亲缘关系及与系谱关系是否吻合,对其进行聚类分析,结果(图4)表明,21个供试小麦品种(系)间的遗传相似系数在0.69~0.99之间。除早熟高产品种泰山24、优质品种泰山 27、高产品种泰科麦 30、高产节水型品种泰科麦 32和高产优质品种泰科麦 33外,其余品种(系)可以分为4个类群。90s育成的早熟高产品种鲁麦18和00s育成的早熟超高产品种泰山23归为类群Ⅰ;00s育成的早熟高产品种泰山21、大穗高产品种泰山9818和早熟高产优质品种泰山22归为类群Ⅱ;类群Ⅲ为10s和现在育成的小麦品种(系),包括高产品种泰科麦31和特用品种泰科紫麦1号,高产节水品系泰科麦38、高产品系泰科麦308、早熟品系泰科麦6007、优质品系泰科麦36、高产品系TKM0564和TKM7105等;类群Ⅳ主要为10s和现在育成品种(系),包括高产品种泰山28、高产新品系泰科麦34和优质高产抗倒新品系 TKM6215,基本上同一年份育成的品种(系)聚在一起,跟系谱关系(表1)较为吻合。

图4 小麦品种的UPGMA聚类图Fig.4 UPGMA dendrogram of wheat cultivars
2.4 供试材料的共有位点/染色体区段
根据2029个定位的纯合SNP标记的物理位置随机选取均匀分布染色体的403个SNP标记(每条染色体约20个),绘制21个供试小麦品种(系)的基因型图谱。
表3和图5表明,在不同的年份(00s、10s和Present)育成小麦品种(系)间的共有SNP的数量、基因组分布和同源群分布均有差异。00s育成的 5个小麦品种携带的共有SNP为203个,主要分布在A、B基因组和H4、H7同源群;10s育成的7个小麦品种携带的共有SNP为122个,主要分布在D和A基因组和H2、H6同源群;Present育成的8个小麦品种(系)携带的共有SNP为251个,主要分布在A和B基因组和H2、H3同源群。分析同一年份育成的品种(系)共有的染色体区段情况发现:00s育成的5个小麦品种携带的相同染色体区段主要分布在1A、2A、7A、3B、4B、6B和4D染色体上,共包含57个位点;10s育成的7个小麦品种携带的相同染色体区段主要分布在1A、2A、2D、5D、6B、6D和7D染色体上,共包含53个位点;Present育成的8个小麦品种(系)携带的相同染色体区段主要分布在2A、4A、7A、2B、3B、4B、6B和6D染色体上,共包含109个位点(附表1)。

表3 不同育种年份间共有的SNP数量、基因组(A、B、D)分布和同源群(H1~H7)分布Table 3 The number of SNP, genome (A, B and D)distribution and homologous groups (H1-H7)distribution in different breeding year
进一步比较21个供试小麦品种(系)基因型图谱,发现含有25个共同的SNP位点(图5),这些共有SNP位点分布在1A、5A、6A、7A、2B、3B、6B、1D、2D、3D和7D染色体上,且每条染色体上分布SNP位点的数目均不相同,其中1A染色体上的 AX-111786969-AX、109920244-AX、110049336、AX-110094282和AX-109335959位点成簇分布。

图5 21个小麦品种(系)的基因型图谱Fig.5 Genotypic maps of 21 wheat varieties (lines)
3 讨论
高通量测序和DNA分子标记技术已广泛应用于小麦遗传进化研究、种质资源遗传多样性分析和优异基因发掘等方面。很多研究[15, 22-23, 25-26, 30-31]均证明SNP标记是研究小麦遗传多样性、种属间亲缘关系、品种及基因型鉴定和标记辅助育种的有效工具。
本研究结果表明不同年份小麦品种(系)平均遗传距离呈先升高后下降的趋势。在 90s,因仅育成1个小麦品种,平均遗传距离为0.00,无法比较;00s育成品种的平均遗传距离为0.10,可能与较多地利用鲁麦14[32]、鲁麦18及其衍生后代作为杂交亲本有关;10s育成品种平均遗传距离为0.37,达到最高值,在亲本的利用上引入了亲缘关系相对较远的优质材料;现在育成品种(系)间的平均遗传距离出现下降趋势,可能与较多地利用大面积高产品种作为杂交亲本有一定关系。本研究的聚类分析结果可将供试材料大致分为4个类群,且同一年份的品种(系)基本聚在一起,与系谱分析结果吻合。例如,类群Ⅰ的2个品种亲本86026和881414亲缘关系较近;类群Ⅱ中的泰山21和泰山22的亲本之一都是鲁麦18;类群Ⅲ的8个品种(系)中,泰科麦31和TKM7105的共同亲本为泰山26,泰科麦 0311和TKM0564的亲本均含有烟麦系列的血缘,泰科紫麦1号和泰科麦38均含有良星系列的亲本。另外,未被聚类的5个育成小麦品种(系)的亲本中含有郑州8329、藏选1号、漯麦9424、洛旱3号、莱州3279、郑麦366和淮阴9908等的遗传区段或位点,与前面4个类群所用亲本的亲缘关系较远。
目前育成小麦品种(系)间的遗传相似性较高,遗传多样性降低已成为一种普遍现象。郝晨阳等[27]通过分析我国育成品种的遗传多样性,发现从20世纪 50年代起,我国小麦育成品种的遗传多样性指数和品种间平均遗传距离逐步下降;王江春等[6]研究表明,山东省小麦品种遗传距离总体呈下降趋势,50年代品种间的遗传距离最大,之后逐渐降低,80年代达到最低,90年代出现一个高值后又明显降低;程斌等[33]选用53个SSR引物对96个小麦品种(系)进行了遗传多样性分析,证明山东省近年来育成小麦品种(系)遗传基础越来越狭窄、多态性呈下降的趋势。因此,拓宽小麦品种的遗传基础,提高育成品种的遗传多样性,是今后小麦品种遗传改良研究中值得重视和亟待解决的问题。
通过分析不同年份育成小麦品种(系)共有SNP和共有染色体区段基因组分布情况发现,00s、10s和Present育成的小麦品种(系)共有SNP和共有染色体区段分别主要分布在 A、D和 B基因组。查阅相关文献发现A基因组含有丰富的与磷高效[34]、株高[35-36]、千粒重[37]、抽穗期[38]、分蘖数[39]、Glu-A3[40]、产量[41]和籽粒性状[42]等重要性状相关的基因;B基因组含有丰富的与株高[36,43]、穗长[44]、分蘖数[34]、抽穗期[45-47]、成熟期[37,45]、磷高效[34]、籽粒/面粉蛋白质[48]和抗逆[15,41,49]等重要性状相关的基因;D基因组含有丰富的植物抗逆[15,38,49]、高产[41,50]和优质[51-52]等优异基因。以上结果与本文不同年份育种目标吻合,如00s的高产早熟目标、10s高产抗逆目标及现在的高产优质、多抗和节本安全等目标。同时本研究发现21份供试材料均含有25个共同的SNP位点,分布在1A、5A、6A、7A、2B、3B、6B、1D、2D、3D和7D染色体上。通过对比已报道的小麦产量等QTL信息[15, 34, 36-37, 41-43, 45-46, 49-50, 53-58],并选取物理位置10Mb之内的QTL分析发现,25个SNP位点与增加产量的QTL(如产量、千粒重、穗粒数和单株穗数等)、控制株高的QTL、增加分蘖数的QTL、控制开花期和抽穗期的QTL、提高叶锈病和白粉病抗性的 QTL以及提高灌浆速率的QTL等存在密切关系。以上供试材料的遗传分析结果可能与品种选育过程中注重选择携带控制重要性状的关键基因有关,所以在杂种后代中受到较多的选择和保留。
4 结论
21个小麦品种(系)间的遗传差异日趋缩小,遗传多样性逐渐降低;同一年份的品种(系)基本聚在同一类群,与其系谱关系吻合;同一年份育成的小麦品种(系)携带相同的SNP或染色体区段与不同年份的育种目标吻合;在品种(系)组配与选育过程中注重产量、株高、分蘖数、抽穗期、灌浆速率和抗病等性状的选择,可为今后小麦新品种选育提供参考依据。
