侵染青蒿的烟草曲茎病毒的分子鉴定及基因组结构特征分析
2021-10-12钟静赵丽玲李婷婷丁铭
钟静 赵丽玲 李婷婷 丁铭

摘要 菜豆金色花叶病毒属病毒是一类在全球热带及亚热带地区造成严重经济损失的植物病毒,田间杂草是这类病毒重要的中间寄主。本研究从云南省玉溪市采集了表现黄脉的青蒿植株,通过PCR扩增、克隆及测序从样品中获得两条菜豆金色花叶病毒属病毒DNA-A全基因组序列,分别为YN6393-23和YN6393-27,其全長均为2 739 bp,相似性为100%。序列分析发现,YN6393-23和YN6393-27的核苷酸序列与烟草曲茎病毒Tobacco curly shoot virus(TbCSV)的分离物YN4584的核苷酸序列相似性最高,为99.45%。根据国际病毒分类委员会对菜豆金色花叶病毒属病毒种的分类标准,全基因组序列相似性大于91%则为同种病毒,表明此病毒分离物为烟草曲茎病毒的一个分离物。这是菜豆金色花叶病毒属病毒侵染青蒿植株的首次报道。
关键词 青蒿; 烟草曲茎病毒; 杂草
中图分类号: S 432.1
文献标识码: A
DOI: 10.16688/j.zwbh.2020297
Molecular identification and genomic structural characteristics analysis of Tobacco curly shoot virus infecting Artemisia caruifolia
ZHONG Jing#, ZHAO Liling#, LI Tingting, DING Ming*
(Institute of Biotechnology and Germplasm Resources, Yunnan Academy of Agricultural Sciences, Key Laboratory
of Agricultural Biotechnology of Yunnan Province, Key Laboratory of the Southwestern Crop Gene Resources and
Germplasm Innovation, Ministry of Agriculture and Rural Affairs, Kunming 650223, China)
Abstract
Begomoviruses in the family of Geminiviridae have emerged as a major constraint on crop production in tropical and subtropical regions, and weeds can be the intermediate hosts of begomoviruses. Artemisia caruifolia samples with yellow vein symptom were collected in Yuxi, Yunan province. Among which, the DNA-A complete genome sequence of two isolates (YN6393-23 and YN6393-27) belong to Begomovirus were obtained by PCR, cloning and sequencing. The full length of two sequences was 2 739 bp, with the sequence identity of 100%. Sequence analysis showed that these begomoviruses sequences shared the highest identity (99.45%) with Tobacco curly shoot virus YN4584. Based on the ICTV criteria for Begomovirus species demarcation (≥91% sequence identity for the complete genome), the virus was identified as an isolate of TbCSV. This is first report that Artemisia caruifolia plant can be infected by begemovirus.
Key words
Artemisia caruifolia; Tobacco curly shoot virus; weed
双生病毒的病毒粒体为双联体结构,包裹着单分子或双分子环状单链DNA,DNA分子的长度在2.5~3.0 kb之间,双生病毒主要侵染双子叶植物,可危害棉花、木薯、番茄、烟草等多种经济作物[1-4]。近年来,我国广西、云南和海南等地陆续报道了多起因双生病毒危害而造成的经济损失事件,且双生病毒的危害范围及程度呈逐年上升的趋势[5-6]。在双生病毒科Geminiviridae的9个属中,种类最多(包含320多个种)、分布最广(亚洲、非洲及拉丁美洲等地均有分布)、危害最大的属是菜豆金色花叶病毒属Begomovirus。从基因组结构上看,既有双组分病毒又有单组分病毒是该属病毒的一个典型特征,其中双组分病毒的2个基因组大小相近,称为DNA-A和DNA-B,单组分病毒属病毒只含有DNA-A组分,一般编码6~8个开放阅读框(open reading frames,ORFs),包括病毒链编码的V1和V2以及互补链编码的C1、C2、C3和C4,并由一个基因间隔区(intergenic region,IR)分隔开,单组分病毒一般情况下常与beta卫星、alpha卫星以及一些缺陷型小分子DNA卫星形成病害复合体侵染寄主[7-8]。
重组是导致双生病毒快速变异的主要原因。杂草寄主在菜豆金色花叶病毒属病毒的重组与进化过程中起着重要的作用[9]。近年来,菜豆金色花叶病毒属发现的病毒新种较多,其中很大一部分分离自杂草,如黄花稔曲叶病毒Sida leaf curl virus (SiLCV)、紫茉莉曲叶病毒Mirabilis leaf curl virus(MLCV)以及泰國一点红黄脉病毒Emilia yellow vein Thailand virus(EYVTHV)等[10-12],均是从不同杂草中鉴定到的菜豆金色花叶病毒属病毒,这些在杂草中发现的病毒有可能侵染田间经济作物。多年追踪研究发现,很多杂草中发现的菜豆金色花叶病毒属病毒不仅能侵染杂草,也能侵染经济作物并造成经济损失。1995年在新加坡胜红蓟(杂草)中首次分离到胜红蓟黄脉病毒Ageratum yellow vein virus(AYVV),2005年在日本发现其能够侵染番茄,并造成严重经济损失[13-14]。所以,杂草寄主对于菜豆金色花叶病毒属病毒在不同寄主上的扩展非常重要,监测杂草上菜豆金色花叶病毒属病毒的发生流行趋势,对于防控作物上双生病毒病害有重要的科学意义。
青蒿Artemisia caruifolia是菊科蒿属植物,在我国吉林、陕西、山东、浙江、四川及云南等20多个省有分布,是常见的田间杂草,具有药用价值,功效主要为消炎、止血[15]。本研究在云南省玉溪市采集了表现黄脉症状的青蒿植株,从中首次分离到烟草曲茎病毒,并对其全基因组序列特征及其进化和重组进行了系统分析。
1 材料与方法
1.1 供试材料
2017年12月在对云南省玉溪市新平县进行田间双生病毒病害的调查中,发现部分田间杂草青蒿植株上表现出黄脉等疑似双生病毒侵染引起的典型症状,随机采集典型病株叶片(编号为YN6393),保存于-80℃。
1.2 植物总DNA提取
采用CTAB法[16]提取植物总DNA,DNA样品使用超微量分光光度计进行质量检测,将检测合格的样品进行PCR检测或保存于-80℃备用。
1.3 菜豆金色花叶病毒属病毒检测
1.3.1 样品PCR检测
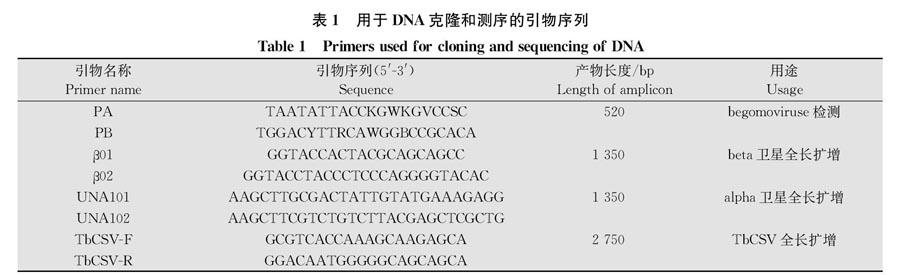
利用菜豆金色花叶病毒属病毒、beta卫星、alpha卫星相对应的通用引物PA/PB[17]、β01/β02[18]以及UNA101/UNA102[19]对样品YN6393进行PCR检测。引物相关信息见表1。
1.3.2 PCR检测体系
PCR检测体系(25 μL):DNA模板0.5 μL、ddH2O 18.5 μL、含Mg2+的10×PCR反应缓冲液2.5 μL、2.5 mmol/L的dNTPs 2 μL、20 μmol/L的上、下游引物各05 μL、Taq Plus DNA聚合酶(5 U/μL, 上海申能博彩生物科技有限公司)0.5 μL。扩增条件:94℃预变性2 min;94℃变性45 s,50℃退火45 s,72℃延伸45 s或90 s(其中采用PA/PB扩增延伸时间为45 s,采用β01/β02和UNA101/UNA102扩增延伸时间为90 s),32个循环;72℃延伸10 min。
1.3.3 PCR产物的回收、克隆及测序
PCR扩增产物经1.0%琼脂糖凝胶电泳检测,将含有目的片段的胶块在紫外灯照射下快速切割,利用Axygen DNA凝胶回收试剂盒回收,将回收产物连接到pGEM-T Easy(Promega, Madison, WI)载体上,连接产物通过热激法转入大肠杆菌DH5α中。将上一步产物均匀涂布于麦康凯培养基(MacConkey Agar,MAC),37℃倒置培养过夜,挑选阳性克隆,送生工生物工程(上海)股份有限公司进行测序。
麦康凯培养基:明胶胰酶水解物17 g/L、胨(肉或酪蛋白)3 g/L、脱氧胆酸钠1.5 g/L、氯化钠5 g/L、乳糖10 g/L、结晶紫1 mg/L、中性红30 mg/L以及琼脂13.5 g/L。
1.4 菜豆金色花叶病毒属病毒全基因组序列克隆
1.4.1 滚环扩增
通过滚环扩增(rolling circle amplification,RCA)能实现对靶核酸的信号放大,本研究用TempliphiTM RCA Kit进行滚环扩增。具体步骤为:将DNA模板(1 μL)与样品缓冲液(5 μL)均匀混合,在95℃下变性3 min;随后立即置冰上冰浴10 min;短暂离心后加入反应缓冲液(5 μL)和DNA聚合酶(0.2 μL),在30℃下反应18 h;在65℃下加热10 min以终止反应。
1.4.2 PCR反应体系
用背靠背引物TbCSV-F/-R(表1)扩增菜豆金色花叶病毒属病毒DNA-A全基因组。PCR反应体系(50 μL):RCA模板0.5 μL、ddH2O 11.5 μL、含Mg2+的10×PCR反应缓冲液25 μL、2 mmol/L的dNTPs 10 μL、20 μmol/L的上、下游引物各1 μL、KOD FX DNA 聚合酶(1 U/μL,日本东洋纺株式会社)1 μL。扩增条件:98℃预变性2 min;98℃变性10 s,50℃退火30s,68℃延伸3 min,32个循环;68℃延伸10 min。
1.4.3 PCR产物的回收、克隆及测序
PCR扩增产物经1.0%琼脂糖凝胶电泳检测。将含有目的片段的胶块在紫外灯照射下快速切割,利用Axygen DNA凝胶回收试剂盒回收,将回收产物连接到pEASY-Blunt zero(北京全式金生物技术有限公司)载体上,连接产物通过热激法转入大肠杆菌DH5α中。将连接产物均匀涂布于麦康凯培养基,37℃倒置培养过夜,挑选阳性克隆,送英潍捷基(上海)贸易有限公司进行测序。
1.5 序列分析、进化树构建及重组分析
菜豆金色花叶病毒属病毒全长基因组核苷酸序列通过SeqMan(DNASTAR. Lasergene.v 7.1)进行拼接,通过DNAMAN 5.22和EditSeq(DNASTAR. Lasergene. v 7.1)进行开放阅读框的寻找;通过BLAST(www.ncbi.nlm.nih.gov)和DNASTAR. Lasergene v 7.1进行核苷酸和氨基酸序列相似性比对;通过MEGA 5软件[20]中的邻接法(neighbor-joining)完成进化树的构建,bootstrap值为1 000;通过RDP v.4.46软件进行重组分析。
2 结果与分析
2.1 菜豆金色花叶病毒属病毒检测结果
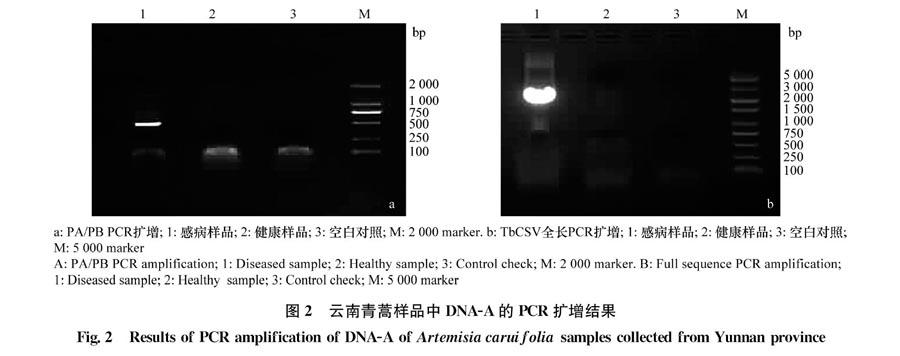
利用菜豆金色花叶病毒属病毒通用引物(PA/PB)对表现黄脉的青蒿植株(YN6393)和健康青蒿植株(CK)总DNA进行PCR扩增,从YN6393总DNA中能够扩增出一条约500 bp的特异性片段,而CK总DNA和双蒸水空白对照均未出现特异性条带(图2)。将目的片段进行回收、克隆和测序,共获得两条序列(YN6393-49和YN6393-50),经BLAST与其他序列比较后发现,YN6393-49的核苷酸序列与烟草曲茎病毒云南分离物YN4501(KU663150.1)的部分核苷酸序列相似性最高,达到98.61%;YN6393-50的核苷酸序列与烟草曲茎病毒的云南分离物YN4524(KU663152.1)的部分核苷酸序列相似性最高,达到98.81%。结果表明,表现黄脉的青蒿样品中检测到菜豆金色花叶病毒属病毒。
利用alpha卫星通用引物UNA101/UNA102及beta卫星通用引物β01/β02对表现黄脉的YN6393和CK总DNA进行PCR扩增,结果表明,YN6393、CK总DNA和双蒸水空白对照均未扩增出特异性条带。在表现黄脉的青蒿样品中未检测到beta卫星和alpha卫星。
2.2 菜豆金色花叶病毒属病毒全长基因组序列扩增结果
菜豆金色花叶病毒属病毒种的确定需要其全基因组序列,所以利用扩增TbCSV全基因组序列的特异引物对表现黄脉的青蒿植株(YN6393)和健康青蒿植株(CK)总DNA进行PCR扩增,结果从YN6393总DNA中能够扩增出一条约2 700 bp的特异性片段,而CK总DNA和双蒸水空白对照均未扩增出特异性条带(图2)。结果表明,从疑似样品中扩增得到了菜豆金色花叶病毒属病毒的全長序列。
2.3 菜豆金色花叶病毒属病毒全长基因组结构分析
将2.2扩增得到的PCR产物进行回收、克隆,挑选阳性克隆进行测序。经过测序从青蒿样品YN6393中得到两条菜豆金色花叶病毒属病毒全长基因组序列:YN6393-23和YN6393-27,DNA-A全长序列均为2 739 bp,两条序列之间的核苷酸相似性为100%,选取其中一条序列(YN6393-23)进行其全基因组结构分析,并在GenBank上进行了登录和注释,序列登录号:MT522159。与大部分菜豆金色花叶病毒属病毒一样,YN6393-23基因组结构具有菜豆金色花叶病毒属中典型的单组分病毒的结构:含有一个保守的基因间隔区和6个开放阅读框(ORFs),在病毒链上编码V1和V2蛋白,在互补链上编码C1、C2、C3以及C4蛋白,其具体位置如下:V1(294-1 064 nt),V2(134-490 nt),C1(1 513-2 598 nt),C2(1 206-1 610 nt),C3(1 060-1 465 nt),C4(2 148-2 570 nt),基因间隔区(IR)包含302 nt核苷酸,位置在2 571-2 739 nt和1-133 nt之间,其中包含有TATA box、保守的9核苷酸序列“TAATATT↓AC”以及重复序列“GGTCCT”。
2.4 菜豆金色花叶病毒属病毒全长基因组序列相似性分析
将分离物YN6393-23与GenBank中的其他同属的全长序列进行比对发现,其2 739 bp的核苷酸序列与TbCSV分离物YN4584(GenBank登录号:MN481144)的核苷酸序列相似性最高,达9945%。根据该属病毒的分类标准,不同种病毒的DNA-A全基因组核苷酸相似性小于91%,同种病毒不同分离物的DNA-A全基因组核苷酸相似性大于91%[21],因此,分离物YN6393-23为烟草曲茎病毒的一个分离物,将其简写为TbCSV-YN6393-23。
将分离物TbCSV-YN6393-23与GenBank中相似性较高以及国内发生的菜豆金色花叶病毒属病毒进一步比对发现,TbCSV-YN6393-23的IR区与TbCSV-YN2247分离物(GenBank登录号:KX290925)核苷酸相似性最高(99.0%);AV1与TbCSV-YN4530分离物(GenBank登录号:KU934097)核苷酸相似性最高(99.6%);AV2与TbCSV-YN4501分离物(GenBank登录号:KU934094)和TYLCSbV-Y4536分离物(GenBank登录号:NC_029995)核苷酸相似性最高(均为100%);AC1与TbCSV-YN4584分离物(GenBank登录号:MN481144)核苷酸相似性最高(994%);AC2与TbCSV-YN2247、TbCSV-YN4584以及TbCSV-YN4519 3个分离物(GenBank登录号:KX290925、MN481144、KU934095)核苷酸相似性最高(均为99.8%);AC3与TbCSV-YN4584分离物(GenBank登录号:MN481144)核苷酸相似性最高(998%);AC4与TbCSV-YN2247、TbCSV-YN4584以及TbCSV-YN4519 3个分离物(GenBank登录号:KX290925、MN481144、KU934095)核苷酸相似性最高(均为99.3%)。结果表明,TbCSV-YN6393-23分离物与云南获得的其他离物在不同编码区相似性均较高(表2)。
2.5 菜豆金色花叶病毒属病毒全长基因组序列系统进化与重组分析
将TbCSV-YN6393-23与GenBank中其他菜豆金色花叶病毒属病毒构建系统进化树,结果发现,TbCSV-YN6393-23与TbCSV的不同分离物聚在一起形成一个大的分支,其中与TbCSV YN4584聚于一小支,说明TbCSV-YN6393-23与TbCSV YN4584亲缘关系最近,该分离物是TbCSV的一个分离物(图3)。这些菜豆金色花叶病毒属病毒都是从云南分离获得的,它们具有非常明显的地理分布特征,即相同地域的TbCSV进化关系非常紧密。经过RDP重组分析发现,TbCSV-YN6393-23并无明显的重组事件发生。
3 讨论
菜豆金色花叶病毒属病毒分布于热带及亚热地区,为害多种经济作物及杂草。近年来国际上对各种经济作物上的菜豆金色花叶病毒属病毒开展了广泛的研究,取得了重要的进展[22-24],而田间农作物周边的杂草中也分离到不少菜豆金色花叶病毒属病毒,并且杂草中此类病毒种类非常丰富,已报道有30多种杂草可被菜豆金色花叶病毒属病毒侵染[25-29]。本研究从表现黄脉症状的田间杂草青蒿中通过分子生物学手段分离到两条菜豆金色花叶病毒属病毒的全基因组序列(YN6393-23和YN6393-27),分析发现,该分离物与TbCSV-YN4584相似性最高,且此分离物为菜豆金色花叶病毒属典型的单组分病毒,不伴随任何卫星分子,这是首次在青蒿上检测到菜豆金色花叶病毒属病毒。
进一步分析发现TbCSV-YN6393-23与其他分离自云南的TbCSV分物的相似性较高,亲缘关系也较近;其中与TbCSV-YN2247的相似性较高(993%),而TbCSV-YN2247也是同样分离自玉溪市新平县,但是寄主为番茄。前期研究发现,田间杂草常常是菜豆金色花叶病毒属病毒病害发生流行过程中的中间寄主,在菜豆金色花叶病毒属病毒的侵染循环过程中起着重要的作用[28-30];在同一地区的经济作物(番茄)和杂草(青蒿)上都鉴定到TbCSV,而且两个分离物之间的相似性较高,表明青蒿很有可能作为TbCSV的中间寄主。在云南地区,青蒿是常见的田间杂草,在番茄种植区的田间地头均有发生,所以青蒿可作为双生病毒的中间寄主,通过烟粉虱传播侵染其周边经济作物(番茄、辣椒、茄子等)。
TbCSV首次发现是在云南省保山市的烟草上,其全基因组序列于2000年在GenBank上进行登录和注释(登錄号:AF240675),并于2002年进行了报道[31],此后该病毒便在云南省多个地区的不同寄主上发现,并造成一定的经济损失。到目前为止,只在中国[32]、印度[33]、孟加拉国(登录号:KM383757)以及缅甸(登录号:MK920411)这4个亚洲国家报道过TbCSV。主要在茄科的番茄[34]、烟草[31]和辣椒[35]、豆科的菜豆[23]以及葫芦科的西瓜[36]等作物上造成严重危害;而在杂草上,TbCSV能够侵染锦葵科的赛葵Malvastrum coromandelianum[32]和黄花棯Sida acuta(登录号:LC316184)、菊科的野向日葵Helianthus spp.[37]和胜红蓟Ageratum conyzoides[38]、紫茉莉科的紫茉莉Mirabilis jalapa[39]以及苋科的喜旱莲子草Alternanthera philoxeroides(登录号:GU199583),可见其寄主范围非常广泛。同属菊科的青蒿、野向日葵和胜红蓟能够被TbCSV侵染,而胜红蓟不但能被TbCSV侵染,也能被AYVCNV[11]、AYVV(登录号:AF307861.1)、PaLCuCNV(登录号:JX294075.1)以及胜红蓟耳突病毒Ageratum enation virus(AEV)[29]等菜豆金色花叶病毒属病毒侵染,是一种易感菜豆金色花叶病毒属病毒的植物。目前已报道有3种菊科植物能被TbCSV侵染,同属菊科的金盏花Calendula officinalis[40]、紫茎泽兰Ageratina adenophora[41]、豨莶Sigesbeckia orientalis[42]、鳢肠Eclipta prostrata[30]和一点红Emilia sonchifolia[12]也都被证实能被菜豆金色花叶病毒属病毒侵染;那么菊科植物中的莴笋、茼蒿、油麦菜、向日葵、非洲菊和蒲公英等具有经济价值的植物是否也能被菜豆金色花叶病毒属病毒侵染?所以对菊科植物上菜豆金色花叶病毒属病毒的检测及监测非常有必要。
杂草是双生病毒重要的中间寄主,对这类病毒发生流行起到至关重要的作用。青蒿是我国各地田间常见杂草,其分布范围相当广泛,因此有必要对其感染的病毒种类进行调查与监测,其结果对防控该类病毒病害起到科学的指导作用。
参考文献
[1] BOTTCHER B, UNSELD S, CEULEMANS H, et al. Geminate structures of African cassava mosaic virus [J].Journal of Virology, 2004, 78(13): 6758-6765.
[2] BRIDDON R W. Cotton leaf curl disease, a multicomponent begomovirus complex [J]. Molecular Plant Pathology, 2003, 4(6): 427-434.
[3] ZHOU Xueping, XIE Yan, ZHANG Zhongkai. Molecular characterization of a distinct begomovirus infecting tobacco in Yunnan, China [J]. Archives of Virology, 2001, 146(8): 1599-1606.
[4] CASTILLO-URQUIZA G P, BESERRA J E A, BRUCKNER F P, et al. Six novel begomoviruses infecting tomato and associated weeds in Southeastern Brazil [J]. Archives of Virology, 2008, 153(10): 1985-1989.
[5] 赵丽玲, 钟静, 尹跃艳, 等. 两种菜豆金色花叶病毒属病毒复合侵染番茄及重组特征[J]. 园艺学报, 2016, 43(7): 1305-1314.
[6] 刘勇, 李凡, 李月月, 等. 侵染我国主要蔬菜作物的病毒种类、分布与发生趋势[J].中国农业科学, 2019, 52(2): 239-261.
[7] HARRISON B D, ROBINSON D J. Natural genomic and antigenic variation in whitefly-transmitted geminiviruses (begomoviruses) [J]. Annual Review of Phytopathology, 1999, 37(1): 369-398.
[8] NAWAZ-UL-REHMAN M S, FAUQUET C M. Evolution of geminiviruses and their satellites [J]. FEBS Letters, 2009, 583(12):1825-1832.
[9] MONCI F, SANCHEZ-CAMPOS S, NAVAS-CASTILLO J, et al. A natural recombinant between the geminiviruses Tomato yellow leaf curl sardinia virus and Tomato yellow leaf curl virus exhibits a novel pathogenic phenotype and is becoming prevalent in Spanish populations [J]. Virology, 2002, 303(2): 317-326.
[10]GUO Xiaojian, ZHOU Xueping. Molecular characterization of a new begomovirus infecting Sida cordifolia and its associated satellite DNA molecules [J]. Virus Genes, 2006, 33(3): 279-285.
[11]KULSHRESHTHA A, ROSHA P, SHARMA D, et al. Molecular characterization of a new begomovirus infecting Mirabilis jalapain northern India [J]. Archives of Virology, 2017, 162(7):2163-2167.
[12]ZHAO Liling, ZHONG Jing, ZHANG Xiaoyun, et al. Two distinct begomoviruses associated with an alphasatellite coinfecting Emilia sonchifolia in Thailand [J]. Archives of Virology, 2018, 163(6):1695-1699.
[13]TAN P H N, WONG S M, WU Mian, et al. Genome organization of ageratum yellow vein virus, a monopartite whitefly-transmitted geminivirus isolated from a common weed [J]. Journal of General Virology, 1995, 76(12): 2915-2922.
[14]ANDOU T, YAMAGUCH A, KAWANO S, et al. Ageratum yellow vein virus isolated from tomato plants with leaf curl on Ishigaki island, Okinawa, Japan [J]. Journal of General Plant Pathology, 2010, 76(4): 287-291.
[15]中國医药信息查询平台 [DB/OL]. [2020-06-08]. http:∥www.dayi.org.cn/cmedical/detail/1008545.
[16]DOYLE J J, DOYLE J L. A rapid DNA isolation procedure for small quantities of fresh leaf tissue [J]. Phytochemistry Bulletin, 1987, 19: 11-15.
[17]DENG D, MCGRATH P F, ROBINSON D J, et al. Detection and differentiation of whitefly-transmitted geminiviruses in plants and vector insects by the polymerase chain-reaction with degenerate primers [J]. Annals of Applied Biology, 1994, 125(2): 327-336.
[18]BRIDDON R W, BULL S E, MANSOOR S, et al. Universal primers for the PCR-mediated amplification of DNA beta-a molecule associated with some monopartite begomoviruses [J]. Molecular Biotechnology, 2002, 20(3): 315-318.
[19]BULL S E, BRIDDON R W, MARKHAM P G. Universal primers for the PCR-mediated amplification of DNA 1: a satellite-like molecule associated with begomovirus-DNA β complexes [J]. Molecular Biotechnology, 2003, 23(1): 83-86.
[20]TAMURA K, PETERSON D, PETERSON N, et al. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods [J]. Molecular Biology and Evolution, 2011, 28(10): 2731-2739.
[21]BROWN J K, ZERBINI F M, NAVAS-CASTILLO J, et al. Revision of Begomovirus taxonomy based on pairwise sequence comparisons [J]. Archives of Virology, 2015, 160(6): 1593-1619.
[22]KHAN A J, AKHTAR S, ALZAIDI A M, et al. Genetic diversity and distribution of a distinct strain of Chili leaf curl virus and associated betasatellite infecting tomato and pepper in Oman [J]. Virus Research, 2013, 177(1): 87-97.
[23]LI Pengbai, JING Chenchen, WANG Zhiyuan, et al. First report of Tobacco curly shoot virus infecting Phaseolus vulgaris in China [J]. Plant Disease, 2019, 103(1): 165.
[24]董家红, 李光西, 罗延青, 等. 香料烟粉虱传双生病毒的分子检测[J]. 西南农业学报, 2007, 20(3): 417-420.
[25]赵丽玲, 钟静, 张晓云, 等. 侵染苣荬菜的中国胜红蓟黄脉病毒及伴随的卫星基因组结构特征[J]. 植物保护, 2017, 43(5): 67-73.
[26]王向阳. 广西胜红蓟黄脉病样中发现多种菜豆金色花叶病毒属的病毒[J]. 植物病理学报, 2007, 37(6): 679-682.
[27]XIONG Q, FAN S, GUO X, et al. Stachytarpheta leaf curl virus is a novel monopartite Begomovirus species [J]. Archives of Virology, 2005, 150(11): 2257-2270.
[28]MA X Y, CAI J H, LI G X, et al. Molecular characterization of a distinct begomovirus infecting Euphorbia pulcherrima in China [J]. Journal of Phytopathology, 2004, 152(4): 215-218.
[29]KHAN M S, TIWARI A K, JI S H, et al. Ageratum conyzoides and its role in begomoviral epidemics, Ageratum enation virus: an emerging threat in India [J]. International Journal of Plant Research, 2012, 24(2): 20-28.
[30]何自福, 虞皓,毛明杰,等. 鱧肠黄脉病样中烟粉虱传双生病毒的检测[J]. 西北农林科技大学报(自然科学版), 2005, 33(S1): 137-139.
[31]XIE Yie, ZHOU Xueping, ZHANG Zhongkai, et al. Tobacco curly shoot virus isolated in Yunnan is a distinct species of Begomovirus [J]. Chinese Science Bulletin, 2002, 47(3): 199-201.
[32]LI Pengbai, JING Chenchen, LI Ke, et al. First report of Tobacco curly shoot virus infecting Malvastrum coromandelianum in China [J]. Journal of Plant Pathology, 2018, 100(2): 339.
[33]VENKATARAVANAPPA V, SWARANALATHA P, LAKSHMINARAYANA R C N, et al. Molecular evidence for association of Tobacco curly shoot virus and a betasatellite with curly shoot disease of common bean (Phaseolus vulgaris L.) from India [J]. Journal of Plant Pathology & Microbiology, 2012, 3(7):148-157.
[34]SWARNALATHA P, KANAKALA S, MANASA M, et al. Molecular characterization of Tobacco curly shoot virus infecting tomato (Solanum lycopersicum L.) in India [J]. Pest Management in Horticultural Ecosystems, 2013, 19(1): 73-84.
[35]QING L, XIONG Y, SUN X C, et al. First report of Tobacco curly shoot virus infecting pepper in China [J]. Plant Disease, 2010, 94(5): 637.
[36]ZHAO L L, DING M, ZHANG X Y, et al. First report of Tobacco curly shoot virus (TbCSV) and its associated satellites from watermelon in China [J]. Journal of Plant Pathology, 2017, 99(3): 761-764.
[37]KUMAR S, JAIDI M, RAJ S K. Molecular characterization of a begomovirus and betasatellite infecting wild sunflower (Helianthus spp.) in India [J]. Virology Research Journal, 2016, 1(1): 1-9.
[38]JIAO Xiaoyang, GONG Huanran, LIU Xuejian, et al. Etiology of ageratum yellow vein diseases in South China [J]. Plant Disease, 2013, 97(11): 1497-1503.
[39]XIONG Y, QING L, REN F, et al. First report of Tobacco curly shoot virus on Mirabilis jalapa Linn. in China [J]. Journal of Plant Pathology, 2010, 92(2): 543-546.
[40]JAIDI M, KUMAR S, SRIVASTAVA A, et al. First report of Ageratum enation virus and Ageratum leaf curl betasatellite infecting Calendula officinalis in India [J]. New Disease Reports, 2015, 32(1): 6.
[41]ONUKII M, HANADA K. Genomic structure of a geminivirus in the genus Begomovirus from yellow vein-affected Eupatorium makinor [J]. Journal of General Plant Pathology, 2000, 66(2): 176-181.
[42]YANG C X, LUO J S, ZHENG L M, et al. Mixed infection of Papaya leaf curl China virus and Siegesbeckia yellow vein virus in Siegesbeckia orientalis in China [J]. Journal of Plant Pathology, 2011, 93(4): S81.
(責任编辑:杨明丽)
