基于环境DNA-宏条形码技术的底栖动物监测及水质评价研究进展
2021-10-11金小伟林晓龙杜丽娜崔永德吴小平孙红英谢志才王新华王备新
王 萌,金小伟,林晓龙,杜丽娜,崔永德,吴小平,孙红英,谢志才,王新华,王备新,*
1 南京农业大学植物保护学院,南京 210095 2 中国环境监测总站,北京 100012 3 南开大学生命科学学院,天津 300071 4 广西师范大学珍稀濒危动植物生态与环境保护教育部重点实验室,桂林 541006 5 中国科学院水生生物研究所淡水生态与生物技术国家重点实验室,武汉 430072 6 南昌大学生命科学学院,南昌 330031 7 南京师范大学生命科学学院,南京 210023
淡水大型底栖无脊椎动物简称底栖动物,是指生活史的全部或部分生活在水体底部,个体大小不能通过500 μm孔径网筛的无脊椎动物[1]。常见的有节肢动物门昆虫纲的水生昆虫、软甲纲的淡水蟹、钩虾,软体动物门的蚌类、螺类,环节动物门的寡毛纲、蛭纲,以及扁形动物门的涡虫等[1]。底栖动物是维护水生态系统结构与功能完整性的重要生物类群。它具有分布广泛,生活周期长,对水质变化敏感等特点,因此是水质监测与评价中应用最广泛的生物类群[2]。近些年,日益增强的人类活动干扰、水污染和气候变化导致底栖动物多样性锐减,严重威胁水生态系统健康[3]。如何科学、快速和准确地为淡水生物多样性和水质评价的研究与实践提供大量底栖动物数据,已是当前水生态环境管理关注的重要问题。
传统以形态学鉴定为基础的底栖动物监测方法存在成本高、耗时长、代表性低、专业性强和精确性不够高等缺陷。首先,以生物个体为目标的采样方法需要消耗较大的人力财力,并在采样空间方面存在局限性[4]。其次,跨越数个不同动物门类的底栖动物形态学鉴定高度依赖分类专业人员,然而动物分类研究者数量正在不断减少。再者,采集到的水生昆虫绝大多数是幼体,科级水平以下的鉴定特征模糊甚至缺乏,往往难以准确鉴定到属和种,因此无法准确识别物种与胁迫因子间的专一响应关系,有可能降低评价等级的准确性和精确性[5-6]。综上,传统监测方法的缺陷使其难以适应当前水质监测要求的大规模、高频率、高要求的管理需求,因此急需新的调查方法满足现阶段日益增长的对水生态环境和生物多样性的评估需求。
近年来,环境DNA-宏条形码技术通过对环境样品中混合DNA进行高通量测序和比对,能够快速大规模地得到生物多样性鉴定信息和群落组成,极大得革新了生物多样性调查方法,现已被广泛应用于土壤和水生态环境的生物多样性监测中[7-10]。相比于传统方法,环境DNA-宏条形码技术不需大量的人力物力,成本低、操作简便,因此更适合于大尺度的流域生物多样性监测[11]。同时,该方法灵敏度高,即使在物种密度很低的情况下也能准确检测到目标物种的存在[4]。此外,环境DNA-宏条形码技术可以不依赖分类人员同时对跨门类物种进行监测,并且不受发育时期的限制,完成物种的准确鉴定[4]。因此,环境DNA-宏条形码技术被认为是现阶段实现水生态环境快速、大尺度和大样本物种监测最有发展前景的方法[11]。
目前,环境DNA-宏条形码技术对底栖动物的检测和鉴定仍存在部分问题和不足,制约其进一步在水质监测中的应用。主要问题有低丰度物种漏检、物种鉴定错误、采样流程与方法不统一以及生物量精确估测困难等[11]。这些问题在技术层面体现在样品采集与处理流程、分子标记选择、引物设计、PCR偏好性,以及参考数据库的完整性等影响因素[12-14]。因此在推动环境DNA-宏条形码技术应用底栖动物监测时应重点关注这些关键影响因素。近十年来,针对以上关键影响因素国内外开展了比较多的优化研究[14-19]。然而其中大部分研究较为分散,部分结论不一致,且缺少系统性的分析和比较,制约了底栖动物环境DNA-宏条形码监测技术方案的规范和优化,因此亟需对相关问题进行系统性的梳理与整合。鉴于此,本文对环境DNA-宏条形码的概念和在底栖动物监测的应用进行了综述,重点针对影响环境DNA-宏条形码技术用于底栖动物监测和水质评价的关键因素及其相应优化进行了分析总结,并基于此提出提高环境DNA-宏条形码技术监测底栖动物效率和准确率的有效途径,期望为基于环境DNA-宏条形码技术的底栖动物监测方案的标准化和规范化提供建议和参考,最后对该技术在底栖动物监测和水质评价中的最新发展方向进行了展望。
1 环境DNA-宏条形码技术简介及在底栖动物监测中的应用
环境DNA概念最早于1987年提出,用于从沉积物中提取微生物的DNA,2000年左右开始引入生态学领域[20]。环境DNA(environmental DNA,eDNA)是指从土壤、水和大气等环境样品中直接提取的不做任何分离得到的生物释放的游离DNA片段的总和[20]。具体包括微生物、动物和植物不同物种本身以及向环境中释放的分泌物、唾液、精子、粪便和脱落的皮肤等一系列降解程度不同的各种DNA混合物[20]。环境DNA-宏条形码技术(eDNA metabarcoding)指从土壤、水等环境采样提取总DNA片段,使用通用引物进行PCR扩增并结合高通量测序,得到上百万条序列,再与已有数据库中的序列信息进行比对,从而一次性对环境样品中的多个物种或类群进行快速、大规模鉴定[10]。该方法能够有效检测出传统方法未检测到的种类,同时能够对大部分物种进行属或种级别的准确鉴定。目前环境DNA-宏条形码技术在水生生态系统中的应用主要集中在物种多样性调查[21]、入侵和稀有物种检测[22]以及目标生物相对丰度、种群大小、分布和发生动态检测等方面[23]。2008年,环境DNA-宏条形码技术首次应用于入侵水生生物美国牛蛙(Ranacatesbeiana)的监测[22]。此后其他研究人员先后利用该技术对亚洲鲤科鱼类中的鲢(Hypophthalmichthysmolitrix)和鳙(H.nobilis)[24]、蓝腮太阳鱼(Lepomismacrochirus)[25]和新西兰泥蜗牛(Potamopyrgusantipodarum)[26]等物种成功进行了监测。
2011年,Hajibabaei等[27]首次证明环境DNA-宏条形码技术适用于底栖动物多样性监测。随后的研究进一步证明该技术对环境敏感类群蜉蝣目、毛翅目和襀翅目(蜉蝣目Ephemeroptera、襀翅目Plecoptera和毛翅目Trichoptera昆虫,简称EPT昆虫)物种有良好的检出效果[28-29]。Elbrecht和Leese[30]的模拟实验进一步证明环境DNA-宏条形码技术对大部分底栖动物的检出率较高,总体检出率达98%,其中EPT的检出率均为100%。Beermann等[31]对湿地摇蚊的研究表明,环境DNA-宏条形码技术对摇蚊的检出率比传统调查方法高70%以上。当前,环境DNA-宏条形码技术已经开始从独立样点或者特定物种监测向大尺度、大样本监测和评估转变(表1)[32,39]。基于环境DNA-宏条形码技术的生物监测被称为“Biomonitoring 2.0”[40],该技术的广泛应用标志着生物多样性监测进入全新的分子时代。环境DNA-宏条形码技术进行底栖动物多样性监测和水质评价具有广阔的发展前景,然而相比国外目前国内相关研究较少[41-42],因此需要更多的方法学和应用研究来推动我国基于环境DNA-宏条形码的水质监测的发展。
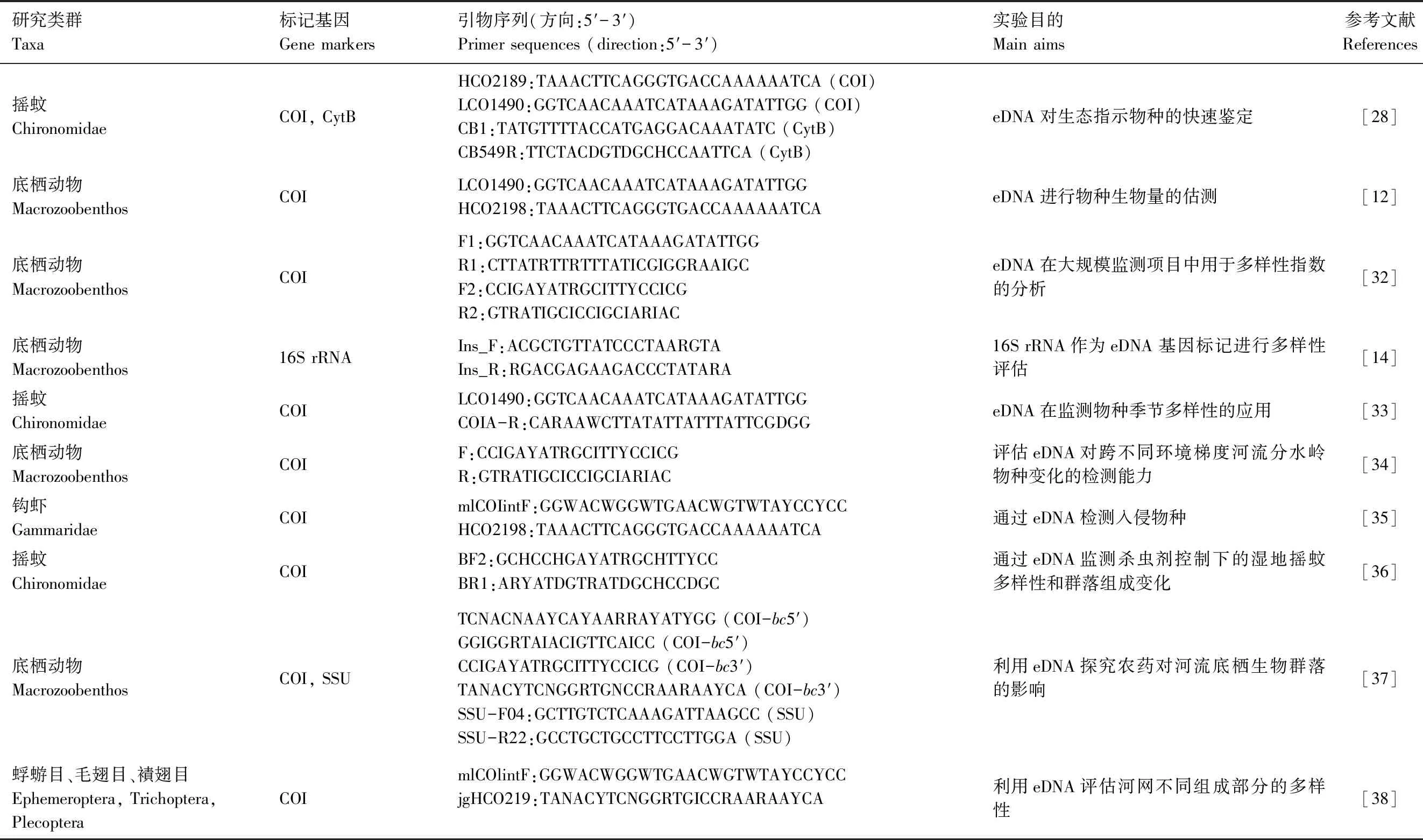
表1 环境DNA-宏条形码技术监测淡水底栖动物典型研究示例Table 1 Examples of environmental DNA (eDNA) metabarcoding technique for biomonitoring of freshwater macrozoobenthos
2 环境DNA-宏条形码技术在底栖动物监测中的关键技术问题及优化策略
环境DNA-宏条形码技术对水生态环境监测的一般操作流程主要包括:样品采集、eDNA提取、PCR扩增、eDNA高通量测序和序列生物信息学分析(图1),技术层面上影响环境DNA-宏条形码技术监测效率和准确性的关键因素主要在于不同的采样方法与流程、分子标记选择、引物设计、PCR偏好性以及DNA条形码参考数据库的完整性等[11-12](图2)。
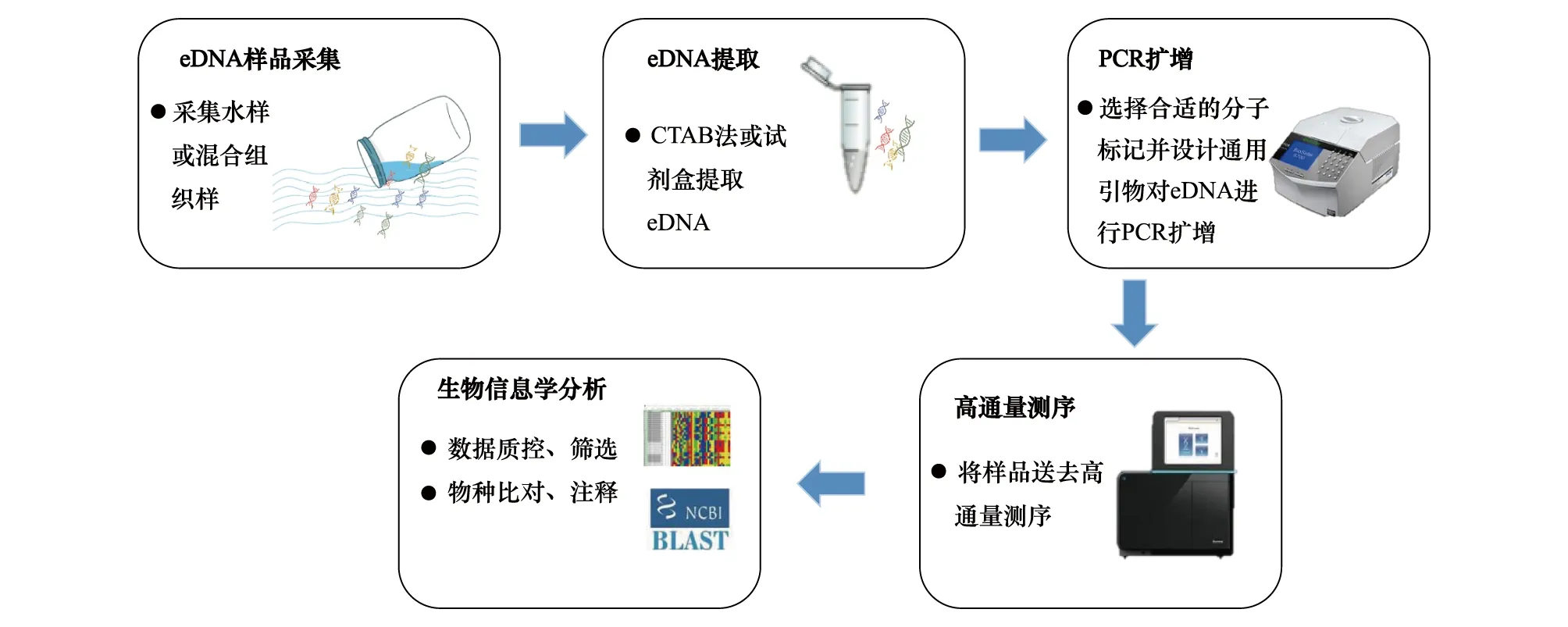
图1 环境DNA-宏条形码技术的一般操作流程Fig.1 The basic workflow of environmental DNA (eDNA) metabarcoding
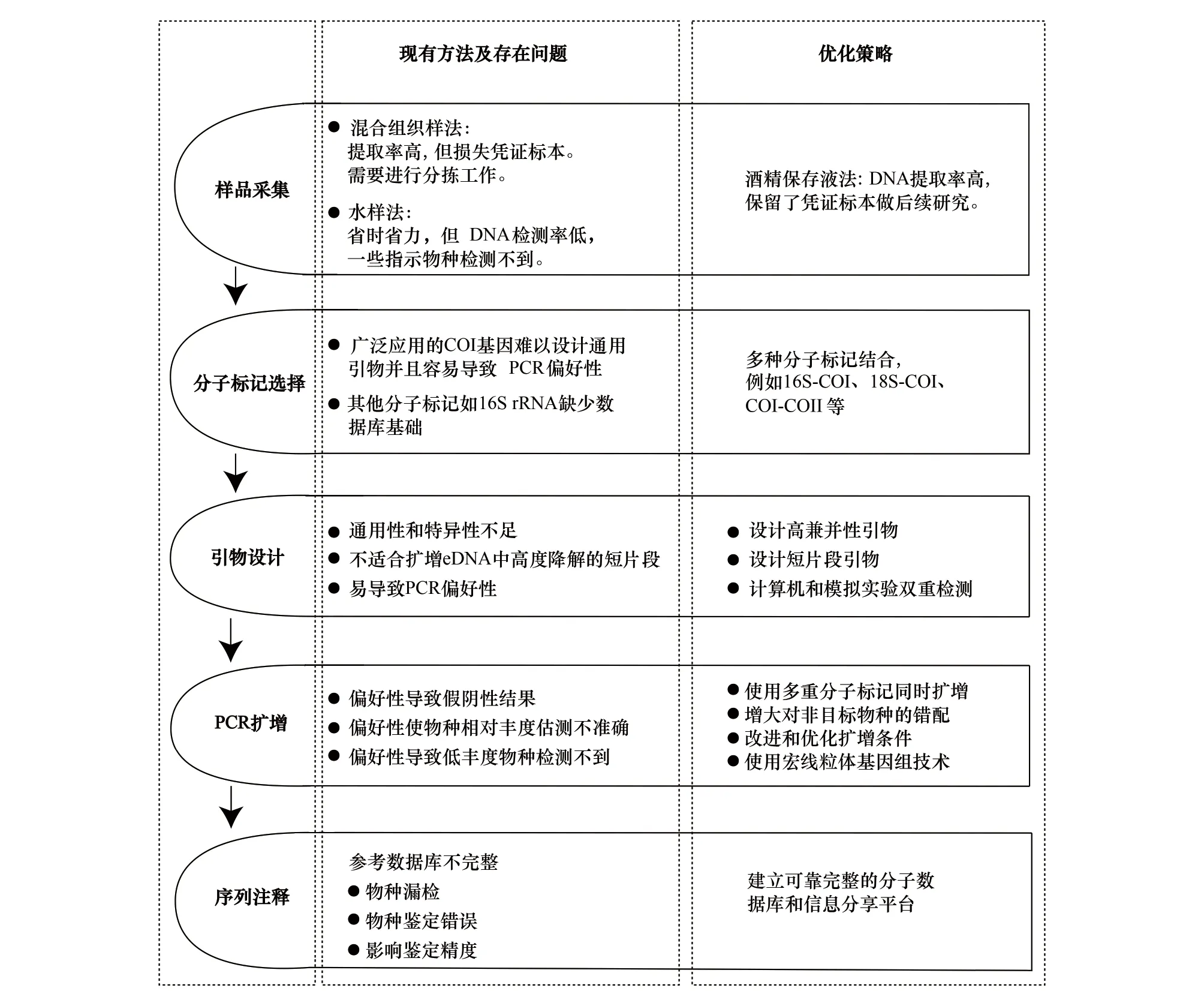
图2 环境DNA-宏条形码技术在底栖动物监测应用中的关键问题与优化策略Fig.2 Critical factors influencing the application of environmental DNA metabarcoding in macrozoobenthos monitoring and their optimized strategies
2.1 采样方法与处理流程
与eDNA样品采集方法相关的优化研究是环境DNA-宏条形码研究的热点方向之一[20,40]。底栖动物的采样方法对最后的检测结果有着显著影响,目前水样法和混合组织样法是获取底栖动物eDNA的两种主要方法。
2.1.1水样法
水样法指通过水环境样本进行eDNA的富集,现阶段水样eDNA的采集方法主要有两种:沉淀法和过滤法[43]。沉淀法[21]特点是不过滤水样,直接加入氯化钠和乙醇并结合离心操作获取eDNA,更适用于原核生物细菌等的获取。过滤法则通过过滤水样将eDNA截留在滤膜上,效果取决于过滤孔径大小[43]。无论在静水和流水中,对于底栖动物而言,采用过滤法eDNA捕获率最高[43]。采集水样量也是影响物种检测准确度和检出率的重要因素。根据研究对象不同,每个重复水样的采集量从15 mL到10 L不等[16,44]。一般来说采集量大时样品代表性较好,但同时采样难度和后续工作量也显著增加,目前底栖动物eDNA的水样采集量一般是1 L或2 L[45]。当目标生物密度较低或调查江河、湖泊等大型水体时,应适当增加采样重复数以及单次水样取样量[46],过少的水样量以及无重复样本都可能会导致物种检出率降低甚至数据无效。
水样法是淡水生态多样性监测的常用方法。然而由于水的流动性,流水中的eDNA会沿河网向下游扩散,从而影响下游真实的物种丰富度和多样性[47]。因此水样法更适合流域尺度的物种多样性调查,但无法重建微观尺度下的生物多样性空间分布模式[48]。此外,水环境样本的异质性和采样的随机性也常导致不稳定的检出结果[49,50]。近期一些研究表明水样中的底栖动物检测率仅有传统形态学采样方法的1/3,且对一些关键指示生物的检测较为困难[51]。
2.1.2混合组织样法
混合组织样法是将全部个体或部分混合后进行研磨,然后基于均质化后的混合组织匀浆(bulk sample)进行总DNA提取的方法。大部分底栖动物调查时先获得踢网样品,然后进行分拣,去除其中的杂质、底泥以及植物和藻类等非目标有机物后再对目标样品研磨均一化后提取eDNA[32,52]。少数研究不进行分拣,底栖动物样品和杂质混合在一起研磨后提取eDNA[15,27,37]。一般认为不进行分拣时样品成分往往较为复杂,包含多种PCR抑制剂,影响DNA的提取和PCR扩增效率[18,53]。但近期一些研究发现样品中的杂质不影响重要指示生物的检测[54],甚至未分拣的样品检测率高于分拣样品与水环境样本[55]。
混合组织样法的优点在于DNA的提取率高,但缺点是损失了相应的凭证标本,导致无法进行后续研究。鉴于此,Hajibabaei等[52]提出了一种无损提取法(non-deconstructive method),即通过样品酒精保存液中提取eDNA来替代从组织混合样中提取eDNA。该方法能够成功检测到大部分底栖物种,其底栖动物检测率仅略低于混合组织样法(87%vs89%)[17,56-58]。研究表明70%—96%浓度的酒精保存液都适用于提取混合DNA[17,57,59],且酒精浸泡后取样时间的长短对于物种检测率影响并不显著[17]。早期酒精保存液法提取eDNA要先进行酒精的蒸发再进行DNA的提取[52,57],步骤繁琐不利于大规模酒精样品的处理。近期一些研究者对此进行了优化,一方面省略样品分拣,对酒精保存液进行二次过滤来提取eDNA,显著缩减处理的时间和步骤[59];另一方面通过一系列方法(超声波、震荡、冷冻)增加DNA的释放率,进一步提高该方法的检测率[59]。酒精保存液法的优点在于保存了凭证标本的完整性,但其在物种检测率上仍低于混合组织样法[17,52]。此外,该方法容易造成少数物种漏检,尤其是外壳骨化强烈的物种如鞘翅目、带壳的软体动物以及带巢生活的一些毛翅目种类[17,59]。
综上,当进行一些重点指示生物如EPT昆虫多样性监测时,酒精保存液法对凭证标本无损伤、节省时间,是可替代组织样法一种方法。而当对整个底栖动物类群多样性做评估时建议采用常规的混合组织样方法进行监测[17]。
2.2 分子标记选择
选取合适的分子标记与扩增引物是环境DNA-宏条形码扩增成功的关键。理想的适用于环境DNA-宏条形码监测的分子标记应同时具备高度保守区域和高度可变区域,前者便于通用引物设计,后者有助于物种识别[60-61]。动物线粒体细胞色素氧化酶COI基因保守度适中,在不同物种之间有良好的解析度,并且现有数据库最为全面,是目前最常用的标记基因[30]。然而COI作为分子标记也有着显而易见的缺陷,体现在COI基因作为一个蛋白编码基因,第三位密码子的突变率较高,在不同物种中差异较大,导致难以设计物种覆盖度较为全面的通用引物[62]。此外,基于COI基因的引物容易引起一些优势物种的过度扩增,从而导致一些低丰度和难扩增物种检测不到[13]和物种生物量估测不准确[12,63]。
鉴于以上COI基因的缺陷,一部分研究者致力于寻找能够替代COI基因的其他分子标记。此类研究主要集中在16S rRNA、18S rRNA等基因标记[14,61]。Elbrecht等[14]通过模拟实验验证16S rRNA相较COI基因在底栖动物中具有更高的检测率,尤其昆虫纲的检测率接近100%。此外,16S rRNA引物偏好性较小,对生物量的估测也更为准确,但其对除昆虫纲以外的其他底栖动物检测率不佳[14,64]。Ficetola等[64]比对16S rRNA、18S rRNA和COI基因标记在环境DNA监测中的效果发现,18S rRNA通用性最强但分辨率较差,更适合对生物多样性做整体评估,而COI基因种级界定效果最佳。16S rRNA、18S rRNA相较COI基因具有更广泛的物种覆盖度,然而缺少像COI基因一样强大而广泛的参考数据库,物种比对难以达到满意的精度[14]。
综上可知,没有单独一个基因标记能够完美实现宏条形码技术监测对于物种覆盖度和解析度的要求。因此我们建议在进行eDNA监测时将16S rRNA或其他分子标记扩增的结果作为COI基因的补充[14]。或者在PCR扩增时中采用多分子标记(multiple primers)的策略[64-65](例如16S-COI、18S-COI、COI-COII),兼顾物种的覆盖度和精确性[66-67],并利用COI基因强有力的参考数据库进行物种的注释[14]。
2.3 引物设计
引物的设计在eDNA监测中至关重要,合适的引物可以显著提高eDNA的扩增质量并减少引物的偏好性[19]。现阶段引物设计的主要思路是设计COI基因的高简并性引物,增加通用性的同时尽可能地减少其引物偏好性。目前效果较好的有Geller等[68]设计的COI基因通用引物jgHCO2198和jgLCO1490,该引物适用于所有后生动物;Hajibabaei等[52]针对底栖动物设计的三组高简并性COI引物。目前由Elbrecht和Leese[30]设计的高兼并性COI引物BF1/BF2和BR1/BR2不仅对底栖动物检测率显著高于传统的COI引物和16S rRNA,同时减少了其扩增偏好性。
引物设计的另一思路是采用短片段引物(mini-barcode)。由于环境DNA-宏条形码研究中广泛存在高度降解的短片段DNA和高通量测序短的特点,短片段引物在底栖动物环境DNA-宏条形码检测中更有优势[35]。当前应用的有Leray等[69]设计出的短片段引物(313 bp)mlCOlintF/mlCOlintR和Vamos等[70]设计出的短片段引物fwh1 set、fwh2 set(扩增长度分别为178 bp和205 bp)。相比传统通用引物(如LCO1490/HCO2198)与较为成熟的高兼并性引物(如BF1/BF2和BR1/BR)[30],短片段引物所含信息相对较少,引物偏好性较高,但对于短片段DNA扩增效率更高。因此在样品保存较好、eDNA降解程度小时,建议使用高兼并性COI引物如BF1/BR1和BF2/BR2。如果样品DNA高度降解,使用短片段引物会使扩增更为有效。
引物设计好后更重要的是如何评价其扩增的效率和特异性。新设计的引物应通过模拟实验和电脑检测(e.g.In silicon PCR软件)的双重验证后方可大规模地应用[30,60-61,71]。MacDonald等[72]总结出在环境DNA-宏条形码研究中引物设计和评价的九个步骤,按步骤进行引物的设计和评价能够有效地降低阴性错误和阳性错误发生的概率。这9个步骤包括:(1)定义eDNA实验涉及到的生物范围,并据此确定合适的基因标记,如COI;(2)建立物种类群的基因比对数据库;(3)通过系统发育关系检验目标基因区别物种的能力;(4)针对目标物种进行特异性引物的设计,注意引物序列中包含非目标生物的碱基错配;(5)通过遗传距离分析找到容易混淆的近似种;(6)通过模拟实验检验引物的特异性,评估假阳性和假阴性错误的风险;(7)通过连续稀释的DNA样本检验引物的灵敏性;(8)基于野外实验样品评估引物性能及PCR条件是否合适、有效;(9)实施大范围的实验来制定具有使用限制和可能错误的标准调查原则。
2.4 PCR偏好性
PCR偏好性是由于引物和不同样品模板之间亲和力不同,某些亲和力强的样品模板在PCR的每轮循环中更容易被扩增,导致其在最终的PCR产物中过量表达的现象。引物和模板之间存在不同碱基数量的错配是造成PCR偏好性的重要原因,即引物和模板错配数量少比错配数目多的更容易扩增[46]。此外,基因片段长度与碱基组成也影响引物结合效率(比如短片段更容易被扩增,GC含量过高的模板不易扩增)[19]。
PCR偏好性是造成环境DNA-宏条形码技术对低丰度物种检测率低[28]和物种生物量估测错误[12,30]的最主要因素。在生态研究中,生物多样性和生态系统功能的评估都离不开对物种相对丰度的准确的分析。然而,现阶段环境DNA-宏条形码基于分子数据对物种的定量分析无法准确反映物种真实的生物量和相对丰度[11]。尽管很多研究都证明环境DNA-宏条形码扩增的序列数和生物量之间具有正相关的线性关系[12,23,73-74],但这种线性关系只有在PCR偏好性没有显著影响时才成立[75]。因此,控制和减轻PCR的偏好性是环境DNA-宏条形码技术应用于水生态环境监测的主要挑战之一。建议可以采用以下方法来避免或减弱PCR偏好性。
(1)使用多重分子标记同时扩增,即使用不同分子标记基因的保守区设计多对通用引物。不同引物配套使用,能够在一定程度上减轻PCR偏好性[65,67]。但此方法获得的种类往往较为复杂,给序列比对和多套数据整合等带来困难,同时也增加了假阳性率和监测成本。
(2)引物设计上,增大对非目标物种的错配[46]。同时,针对优势种设计封闭引物,减少不必要的DNA扩增,对低丰度物种设计更为特异性的引物。
(3)在PCR扩增阶段,改进和优化扩增条件,包括降低引物量、适当增加模板量、减少循环次数以及适当提高退火温度等[19]。或者采用降落PCR(Touch-down PCR)和巢式PCR提高扩增的特异性[19]。
(4)使用宏线粒体基因组技术,直接从混合样品中获取总DNA并测序,避开PCR扩增环节[19]
2.5 参考数据库
参考数据库的完整性和质量直接决定了运用环境DNA-宏条形码技术进行物种鉴定的可靠性和准确度[76]。虽然近年来GenBank、BOLD(The Barcode of life Data System)等公共数据库收录了大量数据,但其中水生生物类群收录不足。除此之外,文库中收录的数据多为COI、16S rRNA等序列片段,而基于其他分子标记基因的数据收录较少[12,77]。数据库的不完善使得大量的环境DNA-宏条形码数据不能得到注释或正确注释,造成物种漏检或鉴定错误,这是很多环境DNA-宏条形码检测结果低于形态学检测结果的首要原因[12]。此外,注释数据中超过50%的数据只能鉴定到门或目,对物种的辨识度很低[77],无法满足大多数水生生物多样性监测和水质监测需要鉴定到属或种的要求。
针对此问题,全球很多研究机构开始着手建立可靠完整的分子数据库和信息分享平台。澳大利亚专门建立了针对底栖动物的数据库Aquatic Invertebrates of Australia[78]。加拿大建立了EPT数据库,收录了112个EPT物种,2277条COI序列[79],近期又建立了包含150万条条形码数据和凭证标本的参考数据库[80]。美国建立了收录209种毛翅目昆虫和超过1000多条条形码数据的参考库[81]。同时,德国也建立了EPT数据库,收录了363个物种和2000多条序列[82]。
目前我国条形码数据库发展相对较慢,尚未建立针对底栖动物的条形码数据库。虽然有GenBank和BOLD等公共数据库,但其数据在地理空间和物种覆盖度方面极不平衡,导致我国特有底栖动物种类鲜有收录,且已有的序列还存在着大量鉴定和标记错误[83]。因此急需一个由专业分类学家通过准确鉴定和测序而建立的更为完备的,特别是包含我国种群数据的底栖动物条形码数据库。鉴于我国受限于缺乏条形码数据库的现状,2019年11月由南京农业大学水生昆虫团队首倡召开的中国底栖动物条形码数据库研讨会在南京举行。会议上确定由南京农业大学、南开大学、中国科学院水生生物研究所、中山大学、南京师范大学、广西师范大学等15个高校与科研院所的分类专家及团队共同携手参与构建中国首个具有自主知识产权的底栖动物条形码数据库。目前,数据库已完成昆虫纲(毛翅目、蜉蝣目、襀翅目、蜻蜓目等)、寡毛纲、软体动物门(腹足纲和瓣鳃纲)与软甲纲(溪蟹和钩虾)等合计400余种的条形码测序工作,收录共计1000余条形码序列。该条形码数据库的建立有利于摸清我国底栖动物多样性家底,并为我国底栖动物多样性保护研究和水生态的健康评价提供坚实的数据基础和技术保障。
3 基于环境DNA-宏条形码技术的水质监测应用展望
3.1 环境DNA监测标准化及评价体系建立
当前底栖动物水质监测的取样及分析流程多样,然而不同方法获得的结果可比性较差。采集过程中不同水样的采集位置、过滤水量、重复次数以及后期实验室不同的PCR重复次数、测序深度等都会导致测定结果之间有较大差异[67]。此外,采样过程中的防交叉污染,对照设立以及样品存放条件等都对结果有重要影响,然而这些标准至今仍未统一[54]。采样方法与流程的标准化是底栖动物环境DNA监测结果具有可重复性与可比性的重要前提。因此探索有效的采样方法和流程并统一标准对今后开展基于环境DNA的水生态系统的大规模日常监测具有重要意义,后续应加强对底栖动物环境DNA方法标准化的研究。
环境DNA-宏条形码技术监测在调查物种多样性中具有诸多优势,但在水质监测和评估方面尚未建立独有的评价体系。传统的水质监测和评价建立在形态学鉴定的基础上,检测的单位是生物个体,通过计算一系列以指示生物有无和丰度等为基础的生物指数来进行[6]。而基于环境DNA-宏条形码技术的分子监测得到的是大量DNA序列,以分子可操作单元(Molecular Operational Taxonomic Units,MOTUs)为单位。然而,MOTUs的划分取决于主观设定的阈值,不同的阈值划分可导致MOTUs的数量和归类显著不同[67]。其次,由于缺少较为完整的数据库,MOTUs中只有小部分能精确注释到属或种进而与传统方法对接而被利用,使得大部分生物多样性监测和生态学研究结果停留在MOTUs阶段,难以将获得的多样性分布数据与现有的生物学和生态学指标结合起来,从而分析多样性现状、空间分布格局及胁迫因素的相关关系[83]。此外,环境DNA-宏条形码技术较形态学方法更易检测出稀有物种。这类物种可参考资料较少,未纳入传统的生物指数评价范围,无法用于生物指数的计算。因此,环境DNA-宏条形码技术在水质监测的广泛应用尚需于建立起其独特的评价体系[77]。
针对以上问题,可以考虑建立基于宏条形码数据的生物指数及评价体系。直接利用序列的相对丰度和MOTUs数计算的生物指数,已在细菌microgAMBI[84]、硅藻[85-86]和海洋底栖动物gAMBI等[87-88]中得到应用,并已显示出与传统手段相似的生态评价效果[89]。同时,该方法可进一步与人工智能相结合,利用监督式机器学习(Supervised Machine Learning,SML),通过输入大量序列和MOTUs数样本训练模型进行分子生物指数计算[85],进而用来评估水生态健康状况和预测水体质量[90]。该方法能快速地从海量的宏条形码数据中重建生态网络,提升水质监测效率。未来,大数据和智能化技术为基础的分子生物指数将成为水环境生态管理的重要指标。
3.2 宏线粒体基因组技术
宏线粒体基因组技术采用免PCR扩增法(如短核苷酸链捕获探针法、全基因组鸟枪法等)直接对多物种混合样品进行高通量测序,并通过生物信息学分析获得样品中物种的线粒体全基因组,进而对物种类群进行注释[91]。线粒体基因组比传统分子标记基因包含更长的序列片段,可大幅提高混合物种的鉴定灵敏度和分辨率。该方法的有效性已在无脊椎动物中已得到验证[92]。如Tang等[93]开发出一套针对昆虫的基于高通量Illumina测序平台的宏线粒体基因组对混合物种相对丰度的分析流程,并利用该方法替代传统的COI条形码进行物种鉴定,研究显示物种鉴定的位点信息扩大了约20倍。目前该方法由于测序成本高,对下游生物信息分析技术要求较高等问题,仍处于探索阶段。但该方法解决了由于PCR扩增造成的物种偏倚性,同时获得更多信息位点,可更灵敏和准确地评估物种组成,进而实现各物种生物量乃至相对丰度的分析[92],因此,宏线粒体基因组技术无疑是未来宏条形码技术发展的重要方向之一。
3.3 环境RNA的应用
环境DNA-宏条形码相对于传统生物调查方法的劣势在于无法区分物种的生活个体和死亡个体,也无法判定检测到的物种所处发育阶段以及群体的性别比例等信息。近年来出现的环境RNA(Environmental RNA,eRNA)技术以环境中的RNA为研究对象,能够弥补这一劣势,不仅能够判断物种存在与否,还能更好地反映物种在环境中的活跃程度[94]。
少数结合eDNA和eRNA技术同时进行监测的研究发现,eDNA和eRNA两者得到的MOTUs数以及物种数有较大差异[95]。研究表明eDNA对真菌有更高的检测率,而eRNA对后生动物有更高的检测率,其中19.5%的OTUs为eDNA特有,17.7%为eRNA特有[95]。由于只在eRNA数据中出现的OTUs有可能是某些生物代谢过量表达或者反转录和建库过程中出现的假序列,因此一些研究者倡导联合使用eDNA和eRNA的数据,以期更好地对生态环境进行评估[94,96]。由于RNA在环境中相对于DNA更容易降解,其特殊的样品采集、保存、处理以及反转录等操作都会增加调查的难度和成本。但该方法对活体的检测能力能够进一步促进对生物环境和群落结构的认知,是一项非常有潜力的环境分析方法。
