蒺藜苜蓿MtPEAMT基因克隆及其序列比对分析
2021-09-28季晓敏沈益新迟英俊
季晓敏 沈益新 迟英俊

摘要:从蒺藜苜蓿中克隆得到磷酸乙醇胺甲基转移酶(PEAMT)的编码序列和启动子序列,通过序列对比,对蒺藜苜蓿、拟南芥、番茄等14种植物PEAMT的核苷酸序列及氨基酸序列进行分析。结果显示,蒺藜苜蓿与其他13种PEAMT的基因结构十分相似,除大豆PEAMT外,均含有12个外显子和11个内含子。14种PEAMT蛋白质长度为 475~501个氨基酸,分子量为53.86~57.09 ku,等电点均小于7,为亲水性蛋白。14种PEAMT均无跨膜结构,无信号肽,均定位于細胞质。蒺藜苜蓿PAEMT在其N端和C端各有一个保守的甲基转移酶结构域,并包含4个SAM依赖性基序(I、p-I、II、III)。14种植物PAEMT严格按照生物种属进行聚类,其中蒺藜苜蓿PEAMT和木豆PEAMT的亲缘关系最近。蒺藜苜蓿PEAMT易被蛋白激酶C、EGFR激酶、蛋白激酶A、cdc2等激酶磷酸化。蒺藜苜蓿与其他13种PEAMT的启动子区域均含有一些逆境响应元件和植物激素响应元件。综上所述,植物PEAMT在进化过程中十分保守,都具有响应植物非生物胁迫的特性。
关键词:磷酸乙醇胺甲基转移酶;蒺藜苜蓿;磷脂酰胆碱;甜菜碱甘氨酸;胁迫响应
中图分类号: S541+.901 文献标志码: A
文章编号:1002-1302(2021)18-0054-11
收稿日期:2021-01-24
基金项目:国家自然科学基金(编号:31601324);作物遗传与种质创新国家重点实验室开放课题(编号:ZW201904)。
作者简介:季晓敏(1994—),男,江苏南通人,硕士,主要从事草类植物分子生物学研究。E-mail:xiaominji941219@163.com。
通信作者:迟英俊,博士,讲师,主要从事分子遗传学研究。E-mail:yingjunchi@njau.edu.cn。
磷酸乙醇胺甲基转移酶(PEAMT)是一种S-腺苷甲硫氨酸依赖性的甲基转移酶。在植物体内,它主要催化3步连续的甲基化反应,从磷酸乙醇胺经由磷酸单甲基乙醇胺和磷酸二甲基乙醇胺2种中间产物最终转化成磷酸胆碱[1]。磷酸胆碱是磷脂酰胆碱和甘氨酸甜菜碱的合成前体[2-3]。磷脂酰胆碱作为真核生物体内一种重要的结构膜脂质,有利于增加膜的流动性,提高机体的抗氧化活性,防止膜损伤[4]。此外,磷脂酰胆碱还是磷脂酸等信号分子的前体[5],在哺乳动物体内,磷脂酰胆碱通过与膜蛋白结合,直接参与膜介导的信号传导[6]。甘氨酸甜菜碱是一种渗透调节物质,它能提高细胞质中的渗透压,在盐、干旱、低温等胁迫环境下保护植物的细胞膜,维持其脂膜的完整性[7-8]。由此可见,PEAMT在膜生物发生、发育和胁迫适应中扮演着重要角色。
鉴于PEAMT在植物磷脂合成和胁迫响应中的关键作用,已陆续在拟南芥[2]、小麦[9]、玉米[10]等植物中被深入研究。Cruz-Ramírez等发现PEAMT1在拟南芥根系发育[11]和维持外胚层细胞完整性的磷脂代谢中十分重要。AtPEAMT1还可以通过影响活性氧-生长素信号传导模块调节细胞分化,维持根尖分生组织的正常生长[12]。而在对小麦PEAMT的研究中发现,低温处理和盐处理都会诱导PEAMT的表达,导致其蛋白质表达量上调以及酶活性的提升[9],这也为通过转基因手段提高植物抗逆性提供了一条可行之策。
然而,目前的研究多集中于对单个植物PEAMT的分离鉴定及功能表征,缺乏对多种植物PAEMT的共性认识。此外,有生化证据表明不同植物,尤其是豆科植物与其他植物间磷脂酰胆碱合成通路差异很大[13-14],而不同植物PEAMT之间的功能差异尚不明确。
蒺藜苜蓿(Medicago truncatula)是豆科生物学和基因组学研究的模式植物[15],因此本研究通过对蒺藜苜蓿PEAMT的克隆,以及与其他植物PAEMT的对比分析,阐明不同植物PEAMT间的进化关系,初步分析豆科植物与其他植物间功能分化的内在原因,以期为挖掘优质基因和分子育种提供理论依据。
1 材料与方法
1.1 试验材料
植物材料为蒺藜苜蓿(Medicago truncatula cv. R108)。菌株为大肠杆菌DH5α感受态(南京鼎思生物技术有限公司)。载体为克隆载体pMD-19T[宝生物工程(大连)有限公司]。
本研究分析所用植物磷酸乙醇胺甲基转移酶的核苷酸序列和氨基酸序列来源于美国国家生物信息中心(NCBI)数据库。
1.2 试验方法
1.2.1 蒺藜苜蓿的培养
挑选籽粒饱满的蒺藜苜蓿种子,用6%NaClO溶液对种子消毒5 min,用去离子水漂洗5遍,随后将其均匀分布在铺有2层滤纸的培养皿上,添加去离子水使培养皿内部保持湿润,放入恒温培养箱中,24 ℃、黑暗条件下萌发。3 d 后,挑选长势一致的苜蓿幼苗,移入土中,采用盆栽土培,营养土、蛭石体积比为1 ∶1,光照—黑暗时间为12 h—12 h,温度为光照24 ℃、黑暗20 ℃。
1.2.2 植物总RNA的提取及反转录
待蒺藜苜蓿生长至1个月左右,取2~3张新鲜幼嫩叶片,采用RNA simple 总RNA提取试剂盒[DP419,天根生化科技(北京)有限公司]提取RNA,用琼脂糖凝胶电泳检测提取的总RNA的完整性。随后采用HiScript Ⅱ 1st Strand cDNA Synthesis Kit(南京诺唯赞生物科技股份有限公司)合成cDNA第1链。
1.2.3 植物基因组DNA的提取
待蒺藜苜蓿生长至1个月左右,取2~3张新鲜幼嫩叶片,采用新型植物基因组提取试剂盒[DP320,天根生化科技(北京)有限公司]提取基因组DNA,用1%琼脂糖凝胶电泳及微型分光光度计确定提取质量。
1.2.4 蒺藜苜蓿PEAMT基因及其启动子的克隆
以拟南芥PEAMT1(At3g18000)序列为探针,在NCBI数据库(https://www.ncbi.nlm.nih.gov/)中搜索与之同源性最高的蒺藜苜蓿基因序列,将其命名为MtPEAMT。
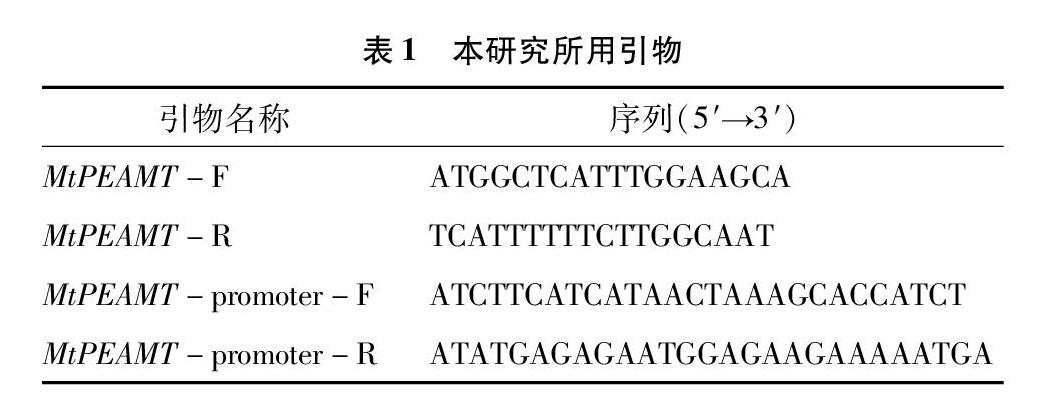
根据蒺藜苜蓿PEAMT的预测序列,利用软件Premier 5.0设计引物,引物序列见表1。以蒺藜苜蓿叶片cDNA为模板,扩增基因的CDS区域。反应程序为95 ℃ 3 min;95 ℃ 15 s,56 ℃ 15 s,72 ℃ 2 min,32个循环;72 ℃ 5 min。
截取蒺藜苜蓿PEAMT起始密码子ATG上游 1 500~2 000 bp为目标片段,利用Premier 5.0软件设计引物,引物序列见表1。以蒺藜苜蓿基因组DNA为模板,扩增基因的启动子区域。反应程序:94 ℃ 1 min;94 ℃ 30 s,56 ℃ 30 s,72 ℃ 32 min,30个循环;72 ℃ 5 min。
将MtPEAMT基因及其启动子的PCR产物进行纯化回收,与pMD-19T载体进行连接,并转化至大肠杆菌,对通过菌液PCR验证后的阳性单克隆菌种进行测序。
1.2.5 植物PEAMT基因的对比分析
本研究对蒺藜苜蓿等14种植物PEAMT基因进行对比分析。对植物PEAMT基因结构分析通过GSDS网站(http://gsds.cbi.pku.edu.cn/)进行;蛋白分子量及等电点等理化性质预测使用BioXM 2.7软件进行。总平均亲水性,脂肪族指数及氨基酸分布预测通过ExPASy在线程序(https://web.expasy.org/protparam/)进行;亚细胞定位预测通过ProtComp 9.0在线程序(http://linux1.softberry.com/)进行;信号肽预测通过SignalP-5.0在线程序(http://www.cbs.dtu.dk/services/SignalP/)进行;跨膜结构预测通过TMHMM工具(http://www.cbs.dtu.dk/services/TMHMM/)进行;蛋白序列比对通过MEGA 10软件和GENEDOC软件进行;蛋白质结构域预测通过Pfam网站(http://pfam.xfam.org/)进行,并使用TBtools对数据进行可视化分析;磷酸化位点预测使用NetPhos3.1网站(http://www.cbs.dtu.dk/services/NetPhos/)进行;系统进化树使用MEGA10软件的邻接法(Neighbor-Joining)构建,自展值(bootstrap)设定为1000。启动子顺式作用元件预测使用PlantCARE程序(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)进行,经过筛选与整理后,使用TBtools对数据进行可视化分析;使用Excel 2019、Powerpoint 2019、Adobe Illustrator 2020作图。
2 结果与分析
2.1 蒺藜苜蓿PEAMT及其启动子的克隆
以蒺藜苜蓿叶片cDNA为模板,通过PCR反应,特异性扩增出推定的PEAMT(图1)。蒺藜苜蓿PEAMT(Medtr6g069600)的CDS长度为1 491 bp,推测编码497个氨基酸,蛋白分子量为56.99 ku,等电点为6.05,总平均亲水性小于0,为亲水性蛋白。
以蒺藜苜蓿基因组DNA为模板,通过PCR反应,特异性扩增出推定的PEAMT启动子区域(图1)。蒺藜苜蓿PEAMT的启动子长度为1 558 bp。
2.2 不同植物PEAMT基因的初步鉴定
以拟南芥PEAMT的氨基酸序列为探针,利用BLAST功能,在NCBI蛋白数据库进行目的基因搜索,共得到28个注释为磷酸乙醇胺甲基转移酶的植物PEAMT基因。再次通过其蛋白序列,搜索NCBI基因组数据库,发现有部分PEAMT的基因组数据缺失。为保证数据分析的完整性与一致性,本研究选取基因组序列和蛋白序列均完整存在的13种植物PEAMT(表2)及蒺藜苜蓿PEAMT进行序列比较分析。
2.3 蒺藜苜蓿與其他植物PEAMT基因结构的比较分析
由图2可知,蒺藜苜蓿PEAMT的CDS长度为1 491 bp,基因组长度为7 864 bp。通过GSDS网站在线分析基因结构,蒺藜苜蓿PEAMT具有12个外显子和11个内含子。另外13种植物PEAMT的CDS长度为1 425~1 503 bp,基因组长度为4 179~9 518 bp。不同植物PEAMT基因结构十分相似,除大豆PEAMT含有11个外显子和10个内含子以外,其他13种植物PEAMT基因均含有12个外显子和11个内含子。不同植物之间内含子的长度差异较大,导致了基因全长的差异。
2.4 蒺藜苜蓿与其他植物PEAMT基本性质比较分析
由表3可知,蒺藜苜蓿与其他13种植物PAEMT在蛋白质长度、分子量、等电点等蛋白质的基本性质方面表现出高度的一致性。植物PEAMT的蛋白长度范围为475~501个氨基酸,分子量范围为53.86~57.09 ku。14种植物PEAMT的等电点均小于7,总平均亲水性均小于0,表现为亲水性蛋白,脂肪族指数范围为76.62~83.24。预测的亚细胞定位结果表明,14种植物PEAMT均定位于细胞质,无信号肽,而跨膜预测结果也表明14种植物PEAMT均无跨膜结构域的存在。氨基酸序列对比结果(表4)表明,PEAMT序列之间的相似性很高,为76.26%~95.95%,其中蒺藜苜蓿PEAMT与木豆PEAMT的相似度最高,为90.34%。
2.5 蒺藜苜蓿与其他植物PEAMT的结构域比对分析
通过Pfam数据库预测结果(图3)得知,蒺藜苜蓿PEAMT在蛋白的N端和C端各包含1个典型的甲基转移酶结构域,14种植物PEAMT其N端的甲基转移酶结构域的大小和位置均高度一致,而C端的甲基转移酶结构域的大小差异明显。其中蒺藜苜蓿的C端结构域最大,与木豆的C端结构域较为相似。
进一步选取PEAMT结构域区域序列进行比对,结果(图4)表明,2个甲基转移酶结构域序列在14种植物之间具有一定的保守性,2个结构域的N端保守性均高于C端。每个结构域均含有Ⅰ、p-Ⅰ、Ⅱ、Ⅲ 4个基序。且14种植物PEAMT基序I中的排列符合(LIV)-(VL)-(ED)-xG-(APC)-GxG[16]的排列方式。通过序列比对发现,同样符合要求。同时,在对线虫、疟原虫PEAMT结构研究中发现的大多数催化残基和磷酸化位点[17],它们在植物PAEMT中依旧存在并且序列没有发生变化。
2.6 蒺藜苜蓿与其他植物PEAMT进化分析
为探究不同植物PEAMT的系统进化关系,本研究利用MEGA 10軟件的Neighbor-joining(邻接)法初步构建了植物PEAMT的系统进化树,结果见图5,14种植物PEAMT严格按照生物种属进行聚类。单子叶植物分支由禾本科的水稻、高粱和玉米PEAMT组成。双子叶植物分支由豆科植物PEAMT、葫芦科植物PEAMT、藜科植物PEAMT、茄科植物PEAMT、胡麻科植物PEAMT、十字花科植物PEAMT和锦葵科植物PEAMT组成。其中蒺藜苜蓿PEAMT与木豆PEAMT的亲缘关系最为接近。
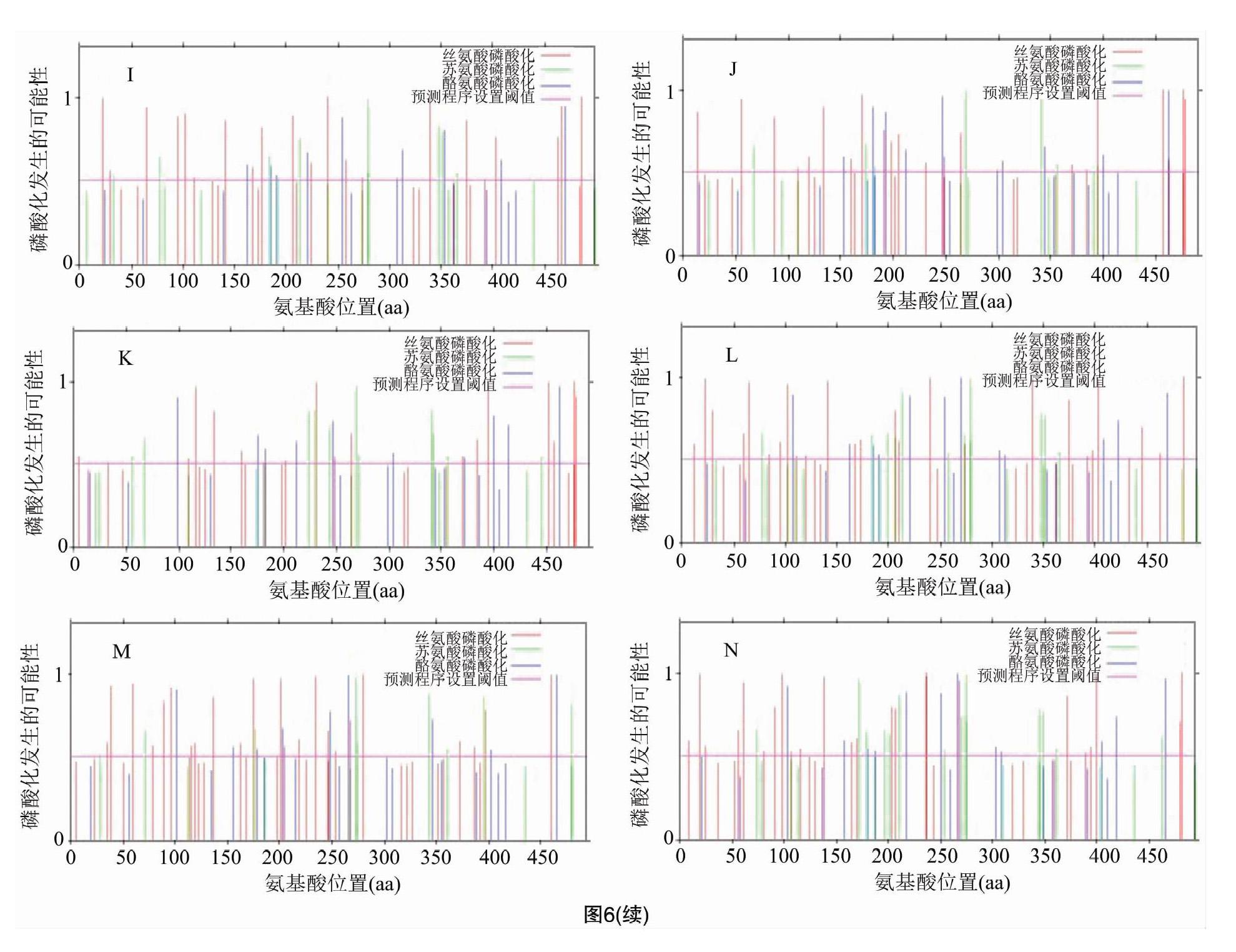
2.7 蒺藜苜蓿与其他植物PEAMT的磷酸化位点预测分析
本研究通过NetPhos对PEAMT的磷酸化位点进行预测分析,结果见图6、表5。预测的磷酸化位点均匀分布于蛋白全长,14种PEAMT均含有大量的蛋白激酶C和EGFR激酶的磷酸化位点,蒺藜苜蓿PEAMT最易受蛋白激酶C磷酸化。此外,大部分PEAMT还易受蛋白激酶A、蛋白激酶G、CKⅠ、CKⅡ、cdc2等激酶磷酸化。
2.8 不同植物PEAMT的启动子分析
通过PlantCare对PEAMT基因启动子区域可能存在的顺式作用元件进行预测,数据经分类整理后使用TBtools进行数据可视化处理。由图7可知,蒺藜苜蓿PEAMT的启动子区域存在光响应元件、干旱诱导元件、 脱落酸响应元件和分生组织特异表达元件。其他植物PEAMT启动子均含有大量的光响应元件,以及一些逆境响应元件,如干旱诱导元件、防御和应激响应元件、厌氧诱导原件和低温响应元件。此外还有很多例如脱落酸响应元件、茉莉酸响应元件和水杨酸响应元件的植物激素响应元件和一些组织特异性表达元件。
3 讨论与结论
测序技术的飞速发展,产生了大量生物序列信息,而大数据的到来给数据挖掘和管理提出了新的挑战,为有效地使用这些数据,需要有效的方法来存储和分析此类数据,这也为推动生物信息学的发展创造了契机[18-20]。磷酸乙醇胺甲基转移酶在植物的许多生物学功能中发挥重要作用,是磷脂酰胆碱合成途径中的关键酶。前人研究表明,不同植物的磷脂酰胆碱合成通路存在很大差异,暗示不同物种的PEAMT可能存在特异性功能[13]。因此,本研究选择豆科模式植物蒺藜苜蓿作为研究对象,成功从蒺藜苜蓿中克隆了PEAMT的CDS序列和启动子区域,并通过序列对比,系统地分析了蒺藜苜蓿PEAMT和其他13种植物PEAMT的核苷酸序列和氨基酸序列,为确定其功能差异的形成原因提供了理论基础。
蒺藜苜蓿PEAMT和其他13种植物的PEAMT在生化性质如蛋白质长度、等电点、亲水性等方面十分相似。亚细胞定位预测结果表明,14种PEAMT均定位于细胞质,而亲水性、信号肽及跨膜预测结果也与之保持一致。目前已表征功能的PEAMT均定位于细胞质[2],这也从侧面说明了预测结果的准确性。系统进化分析结果显示,植物PEAMT严格按照其种属分类关系进行聚类,显示了其在进化过程中的保守性。本研究还发现,不同植物之间内含子的长度差异较大,其中蒺藜苜蓿、木豆、藜麦和菠菜PEAMT的第2个内含子明显长于其他植物PEAMT的第2个内含子,这可能是造成不同物种PEAMT功能差异的原因之一。
迄今为止,已经在26种生物中鉴定出30多种PEAMT。根据其结构域的分布,可将已发现的磷酸乙醇胺甲基转移酶分为三大类。第1类是植物特有的甲基转移酶,包含2个SAM依赖性的甲基转移酶催化结构域MT1和MT2,其N端的MT1结构域催化由磷酸乙醇胺转化为磷酸单甲基乙醇胺的初始甲基化反应;其C端的MT2结构域则催化由磷酸单甲基乙醇胺经磷酸二甲基乙醇胺转化为磷酸胆碱的2步后续甲基化反应[21]。第2类磷酸乙醇胺甲基转移酶是在恶性疟原虫(Plasmodium falciparum)中发现的,其只具备MT2结构域的特征,并且能催化连续3步甲基化反应[22]。第3类磷酸乙醇胺甲基转移酶是在线虫(Caenorhabditis elegans)中发现的,它也有2个不同的结构域,但是只有其中1个甲基转移酶结构域起作用,而另1个并不具有催化活性[23]。本研究的蛋白结构域分析结果表明,植物PEAMT在其N端和C端各有1个甲基转移酶结构域,符合典型的植物PEAMT蛋白结构域排布方式。此外,鉴于PEAMT甲基转移酶结构域的串联形式以及其相当于典型小分子甲基转移酶2倍大小的分子量,结合系统进化分析结果,本研究推断PEAMT可能是2个甲基转移酶基因的融合产物,这种融合可能发生于被子植物分化之前。多序列比对结果表明,一些在线虫或者疟原虫中发现的催化残基和磷酸化位点,它们在植物PEAMT中同样存在,且序列未发生变化[17],表明不同物种的磷酸乙醇胺甲基转移酶在进化过程中也具有高度的保守性。本研究还发现,不同PEAMT的C端序列保守性较差,推测在物种进化过程中可能发生了某些突变,这也有可能是造成不同物种PEAMT功能差异的原因之一。
磷酸化作為一种常见的蛋白翻译后修饰方式,其位点活性与状态对植物生长发育和信号传导十分重要[24]。通过NetPhos预测磷酸化位点可知,植物PEAMT易被蛋白激酶C、蛋白激酶A和酪氨酸激酶Ⅱ等磷酸化。前人研究表明,蛋白激酶C,蛋白激酶A,酪蛋白激酶Ⅱ等激酶在生物磷脂代谢过程中扮演着十分重要的角色。例如Opi1是酵母脂质合成相关的转录调控中的一个关键阻遏物,酪蛋白激酶Ⅱ和蛋白激酶A的磷酸化会激活其阻遏功能,而蛋白激酶C的磷酸化则会抑制阻遏功能。而在磷脂酸介导的脂质代谢过程中十分重要的磷脂酸水解酶也会被酪氨酸激酶Ⅱ和蛋白激酶C磷酸化[25]。磷脂酸作为脂质合成的重要前体物质[26],其水解酶与PEAMT在受到同种激酶磷酸化时是否存在竞争关系,以及是否会对生物的脂质代谢造成影响值得进一步探究。此外,某些蛋白会通过磷酸化和去磷酸化来调节其位置和活性状态[25],因此蛋白激酶C和蛋白激酶A等对PEAMT磷酸化作用的差异性也值得深思。
启动子是重要的基因表达调控区域,不同环境下,植物会通过自身的信号传导机制,诱导特定基因的表达,而这种诱导表达与基因上游启动子区域的顺式作用元件以及相应的转录因子之间存在密不可分的关系。本研究在对植物PEAMT基因启动子区域顺式作用元件的预测中发现,它们的启动子区域除了大量的光响应元件以外,还分布着一些逆境响应元件和脱落酸响应元件等一系列植物激素响应元件。脱落酸介导植物对很多非生物胁迫的响应,如盐胁迫和干旱胁迫,而脱落酸响应元件是其信号传导通路中的关键一环[27-28]。马赛箭在对盐角草PEAMT启动子的分析过程中同样发现其启动子区域包含诸如低温响应元件,热胁迫响应元件一类的逆境响应元件和诸如脱落酸响应元件、生长素响应元件的植物激素响应元件[29]。目前已有部分报道证实了PEAMT是植物抗性相关基因。谢瑾卉在对辽宁碱蓬的研究中发现,PEAMT启动子驱动下GUS的表达会受盐、冷、干旱和ABA等胁迫的诱导[30],而在对玉米PEAMT启动子的研究中也得出相似的结果[31]。Nuccio等发现盐处理会诱导菠菜PEAMT的大量表达,继而提高Cho的产量以促进甜菜碱的积累[32]。这些发现也与本研究中的对比分析结果一致,暗示植物PEAMT除了调控磷脂酰胆碱的生物合成外,可能还具有响应非生物胁迫的能力。
综上所述,本研究通过对蒺藜苜蓿等14种植物PEAMT的对比分析,初步确定了蒺藜苜蓿PEAMT具有响应非生物胁迫的特性,推测了造成不同植物PEAMT功能差异的可能原因,但是对于解释不同植物PEAMT之间功能差异的形成原因仍然需要进一步试验证据的支撑。植物体内的脂质合成通路十分复杂,PEAMT是脂质合成通路种的关键限速酶,但除此以外,还需要多种酶的共同参与,如磷脂甲基转移酶和磷酸胞苷酰转移酶[33],而它们与PEAMT的上下游关系及对底物的竞争势必会对自身活性及脂质代谢造成影响。此外,基因差异功能的形成与其表达模式也存在密切关系。因此,探究PEAMT与其他基因之间的互作关系以及确定其时空表达特性将有助于更好了解PEAMT的功能。
蒺藜苜蓿PEAMT的cDNA全长1 491 bp,编码蛋白分子量56.99 ku,理论等电点为6.05,为亲水性蛋白。植物PEAMT在进化过程中十分保守,都具有响应植物逆境胁迫的特性。
参考文献:
[1]Lykidis A. Comparative genomics and evolution of eukaryotic phospholipid biosynthesis[J]. Progress in Lipid Research,2007,46(3/4):171-199.
[2]Bolognese C P,Mcgraw P. The isolation and characterization in yeast of a gene for Arabidopsis S-adenosylmethionine:phospho-ethanolamine N-methyltransferase[J]. Plant Physiology,2000,124(4):1800-1813.
[3]Mcneil S D,Nuccio M L,Ziemak M J,et al. Enhanced synthesis of choline and glycine betaine in transgenic tobacco plants that overexpress phosphoethanolamine N-methyltransferase[J]. Proceedings of the National Academy of Sciences of the United States of America,2001,98(17):10001-10005.
[4]Asano T,Hayashi N,Kobayashi M,et al. A rice calcium-dependent protein kinase OsCPK12 oppositely modulates salt-stress tolerance and blast disease resistance[J]. Plant Journal,2012,69(1):26-36.
[5]Hong Y Y,Zhang W H,Wang X M. Phospholipase D and phosphatidic acid signalling in plant response to drought and salinity[J]. Plant,Cell & Environment,2010,33(4):627-635.
[6]Yeagle P L. The membranes of cells Ⅱ the lipids of biological membranes[M]. Pittsburgh:Academic Press,2016.
[7]Sakamoto A,Valverde R,Chen T H,et al. Transformation of arabidopsis with the codA gene for choline oxidase enhances freezing tolerance of plants[J]. Plant Journal,2000,22(5):449-453.
[8]Sakamoto A,Murata N. Genetic engineering of glycinebetaine synthesis in plants:current status and implications for enhancement of stress tolerance[J]. Journal of Experimental Botany,2000,51(342):81-88.
[9]Charron J F,Breton G,Danyluk J,et al. Molecular and biochemical characterization of a cold-regulated phosphoethanolamineN-methyltransferase from wheat[J]. Plant Physiology,2002,129(1):363-373.
[10]Wu S W,Yu Z W,Wang F G,et al. Cloning,characterization,and transformation of the phosphoethanolamine N-methyltransferase gene (ZmPEAMT1) in maize (Zea mays L.)[J]. Molecular Biotechnology,2007,36(2):102-112.
[11]Cruz-Ramírez A,López-Bucio J,Ramírez-Pimentel G,et al. The xipotl mutant of Arabidopsis reveals a critical role for phospholipid metabolism in root system development and epidermal cell integrity[J]. The Plant Cell,2004,16(8):2020-2034.
[12]Zou Y,Zhang X J,Tan Y Y,et al. Phosphoethanolamine N-methyltransferase 1 contributes to maintenance of root apical meristem by affecting ROS and auxin-regulated cell differentiation in Arabidopsis[J]. The New Phytologist,2019,224(1):258-273.
[13]Datko A H,Mudd S H. Phosphatidylcholine synthesis:differing patterns in soybean and carrot[J]. Plant Physiology,1988,88(3):854-861.
[14]Williams M,Harwood J L. Alternative pathways for phosphatidylcholine synthesis in olive (Olea europaea L.) callus cultures[J]. The Biochemical Journal,1994,304(2):463-468.
[15]Cook D R. Medicago truncatula-a model in the making![J]. Current Opinion in Plant Biology,1999,2(4):301-304.
[16]Clawson G A,Willis D B,Weissbach A,et al. Proceedings of a Hoffman-La Roche-UCLA colloquium on nucleic acid methylation[C]. Frisco,1990.
[17]Lee S G,Jez J M. Nematode phospholipid metabolism:an example of closing the genome-structure-function circle[J]. Trends in Parasitology,2014,30(5):241-250.
[18]Min S,Lee B,Yoon S. Deep learning in bioinformatics[J]. Briefings in Bioinformatics,2017,18(5):851-869.
[19]Guo R X,Zhao Y,Zou Q,et al. Bioinformatics applications on Apache Spark[J]. GigaScience,2018,7(8):giy098.
[20]Gauthier J,Vincent A T,Charette S J,et al. A brief history of bioinformatics[J]. Briefings in Bioinformatics,2019,20(6):1981-1996.
[21]Begora M D,Macleod M J,Mccarry B E,et al. Identification of phosphomethylethanolamine N-methyltransferase from Arabidopsis and its role in choline and phospholipid metabolism[J]. The Journal of Biological Chemistry,2010,285(38):29147-29155.
[22]Reynolds J M,Takebe S,Jy C,et al. Biochemical and genetic analysis of the phosphoethanolamine methyltransferase of the human malaria parasite Plasmodium falciparum[J]. The Journal of Biological Chemistry,2008,283(12):7894-7900.
[23]Lee S G,Jez J M. Evolution of structure and mechanistic divergence in di-domain methyltransferases from nematode phosphocholine biosynthesis[J]. Structure,2013,21(10):1778-1787.
[24]van Bentem S F,Hirt H. Using phosphoproteomics to reveal signalling dynamics in plants[J]. Trends in Plant Science,2007,12(9):404-411.
[25]Kwiatek J M,Han G S,Carman G M. Phosphatidate-mediated regulation of lipid synthesis at the nuclear/endoplasmic reticulum membrane[J]. Biochimica et Biophysica Acta-Molecular and Cell Biology of Lipids,2020,1865(1):158434.
[26]Craddock C P,Adams N,Bryant F M,et al. PHOSPHATIDIC ACID PHOSPHOHYDROLASE regulates phosphatidylcholine biosynthesis in Arabidopsis by phosphatidic acid-mediated activation of CTP:PHOSPHOCHOLINE CYTIDYLYLTRANSFERASE activity[J]. The Plant Cell,2015,27(4):1251-1264.
[27]Watanabe K A,Homayouni A,Gu L,et al. Transcriptomic analysis of rice aleurone cells identified a novel abscisic acid response element[J]. Plant,Cell & Environment,2017,40(9):2004-2016.
[28]張一冉,王雅楠,杨 杨,等. 脱落酸与水杨酸处理调节李果实抗冷性及氧化酶活性[J]. 江苏农业学报,2020,36(2):471-476.
[29]马赛箭. 盐角草磷酸乙醇胺N-甲基转移酶基因(PEAMT)及其启动子的克隆和表达分析[D]. 大连:大连理工大学,2008.
[30]谢瑾卉. 辽宁碱蓬SIPEAMT基因表达特性及SIPEAMT启动子功能分析[D]. 大连:辽宁师范大学,2013.
[31]勾 薇. 玉米磷酸乙醇胺氮甲基转移酶(PEAMT)基因启动子的克隆和功能分析[D]. 杨凌:西北农林科技大学,2017.
[32]Nuccio M L,Ziemak M J,Henry S A,et al. cDNA cloning of phosphoethanolamine N-methyltransferase from spinach by complementation in Schizosaccharomyces pombe and characterization of the recombinant enzyme[J]. The Journal of Biological Chemistry,2000,275(19):14095-14101.
[33]Keogh M R,Courtney P D,Kinney A J,et al. Functional characterization of phospholipid N-methyltransferases from Arabidopsis and soybean[J]. Journal of Biological Chemistry,2009,284(23):15439-15447.
