小球藻属DNA条形码的鉴定研究
2021-09-13张稚兰邢炳鹏
张稚兰 邢炳鹏
摘要:为探讨小球藻鉴定的新方法以及评估小球藻属D NA条形码候选序列的鉴定作用,文章对2 0株小球藻的线粒体基因细胞色素C氧化酶亚基Ⅰ( C O I)、核基因组r D NA的内转录间隔区( I T S)以及核酮糖1, 5 -二磷酸羧化酶( r b c L) 3个片段进行P C R扩增测序,并对各序列进行生物信息学分析。研究结果表明: C O I、I T S和r b c L序列在2 0株小球藻中的扩增和测序效率均呈陽性,扩增成功率分别为7 6%、8 3%和7 0%;经评估, C O I、I T S和r b c L序列作为D NA条形码在小球藻分类鉴定中均不适合较低的分类单位,但可参考动植物的分类方法,将多个片段组合应用,从而筛选不同进化级别的D NA条形码。
关键词:小球藻; D NA条形码;分类鉴定;物种;基因
中图分类号: Q 9 4 9.2 1 文献标志码: A 文章编号: 1 0 0 5-9 8 5 7( 2 0 2 1) 0 2-0 0 6 9-0 8
基金项目:自然资源部第三海洋研究所基本科研业务费专项资金资助项目(海三科2 0 1 5 0 1 2、海三科2 0 1 9 0 1 5).
A s s e s s m e n t o fC a n d i d a t eD N AS e q u e n c e s f o rB a r c o d i n go f t h eG e n u sC h l o r e l l a
Z HAN GZ h i l a n1, X I N GB i n g p e n g1, 2
( 1 .T h i r dI n s t i t u t eo fO c e a n o g r a p h y,MN R, X i a m e n3 6 1 0 0 5, C h i n a; 2 .O b s e r v a t i o na n dR e s e a r c hS t a t i o no fC o a s t a lW e t l a n dE c o s y s t e mi nB e i b uG u l f,MN R, B e i h a i 5 3 6 0 1 5, C h i n a)
A b s t r a c t:T o e v a l u a t et h e v a l i d a t i o n o f D NA b a r c o d i n g c a n d i d a t e s e q u e n c ei ni d e n t i f y i n g C h l o r e l l a, t od i s c u s s t h en e ww a y so f i d e n t i f y i n g, t h i sp a p e r t o o k2 0 i n d i v i d u a l s s a m p l e d f o r a n a l y -s i s, 3s e q u e n c e sc y t o c h r o m ec o x i d a s eⅠ( C O I) , i n t e r n a l t r a n s c r i b e ds p a c e r( I T S)a n dr i b u l o s e - 1, 5 - b i s p h o s p h a t eo f c a r b o x y l a s e( r b c L)w e r ea m p l i f i e da n ds e q u e n c e d . T h e s es e q u e n c e sw e r ea n a -l y z e db yb i o i n f o r m a t i c sm e t h o d s . T h er e s u l ts h o w e db o t ha m p l i f y i n ga n ds e q u e n c i n ge f f e c t so f C O I, I T Sa n dr b c Lt o w a r d st h es a m p l eo fc h l o r e l l aw e r ep o s i t i v e, t h ea m p l i f i c a t i o ns u c c e s sr a t e w a s7 6%, 8 3%a n d7 0%, t h e s e 3s e q u e n c e sw e r en o t s u i t a b l e f o r l o w e r t a x a l e v e l .I n t h e f u t u r e t h e c o m b i n a t i o no fm a n ys e g m e n t s c o u l db e c o n s i d e r e da n d t e s t e d, t op i c ko u t t h ed i f f e r e n t e v o l u t i o n -a r y l e v e l so fD NAb a r c o d i n g .
K e y w o r d s: C h l o r e l l a, D NAb a r c o d i n g, I d e n t i f i c a t i o n, S p e c i e s, G e n e
0 引言
小球藻( C h l o r e l l a)是球形单细胞真核微藻,隶属绿藻门( C h l o r o p h y t a) ,是地球上最早出现的生命之一。目前已发现的小球藻有十数种,变种达数百种,生态分布广泛。小球藻生长速度快且光合效率高,既可以光能自养,又可以使用有机碳源异养生长和繁殖,有很高的营养价值,其油脂还可转化为生物柴油,在医药、化工、食品和生物能源等领域有广阔的应用前景[ 1-3]。
关于小球藻的分类,已有的方法侧重于形态结构特征、生理生化特征、超微结构以及细胞壁成分等指标[ 4-5],这些指标均是小球藻鉴定的重要依据,但易受环境影响。小球藻体积小、变种多且形态特征简单,分属于共球藻纲( T r e b o u x i o p h y c e a e)和绿藻纲( C h l o r o p h y c e a e)[ 4],类群之间的分类特征交叉重叠且界限模糊不清,大大增加小球藻鉴定的难度,甚至可能导致出现错误的鉴定结果。根据对小球藻属基因组成的研究,其属内成员G C含量值的分布范围很广,几乎涵盖整个真核生物的G C图谱,表明目前对小球藻的分类并不能代表自然界的真实情况[ 4],因此准确对小球藻进行分类鉴定目前仍是难题。
D NA条形码技术直接利用生物的D NA信息作为分类鉴定的方法,具有独特的优势,为小球藻的鉴定拓宽道路。D NA条形码的概念于2 0 0 3年被首次提出[ 6],此后相关研究快速发展,并成为生物分类的研究热点。D NA条形码是利用1段标准的短序列进行物种鉴定的技术[ 6],不受个体形态和生长阶段的影响,具有操作简单、鉴定周期短、可重复性强以及可高效和准确地鉴定单个物种、辨别相近物种和发现隐形种等优点。目前在藻类分子分类研究中,学者已陆续将1 0余种基因序列作为藻类的候选D NA条形码并在不同类群中应用,但尚未取得统一的藻类条形码标准片段[ 7-1 2],研究的基因主要包括C O I、I T S、U P A( 2 3 S)、r b c L、L S U( 2 8 S r D NA)、S S U( 1 8 Sr D NA)、t u f A( e l o n g a t i o nf a c t o r T ug e n e)。其中, C O I和I T S由于具有較高的进化速率,常被用于近缘群体或物种的鉴定[ 1 3]; R u b i s c o酶在光合作用和光呼吸过程中发挥重要作用[ 1 4],其大亚基基因r b c L在物种之间的保守性强,因此成为分子系统学研究中被广泛使用的标准片段之一[ 1 5]。
本研究尝试将C O I、I T S和r b c L基因作为小球藻D NA条形码的候选序列,评估3条D NA条形码候选序列在小球藻属中的鉴定能力,以期为深入研究小球藻条形码和选择标准条形码提供理论和数据支持。
1 材料与方法
1 . 1 材料
本研究选取的所有藻种均购置于中国科学院淡水藻种库,共有2 0株小球藻样品。培养基为B G - 1 1,培养温度为2 5℃,光照照度为45 0 0l x,光暗比为1 2h∶1 2h。小球藻株系来源如表1所示。

1 . 2 样品D N A提取以及P C R扩增和测序
离心采集对数期藻液,使用天根生化科技(北京)有限公司的植物基因组D NA提取试剂盒,提取基因组总D NA。P C R反应的总体积为2 5mm3,其中包括2×T a qP l u sP C R M i x1 2 . 5 mm3、引物( 0 . 0 1mm o l /d m3)各1mm3、D NA模板2mm3以及超纯水8 . 5mm3。P C R扩增的引物序列如表2所示。
C O I基因P C R循环参数设定为9 5℃, 3m i n; 3 0个循环包括: 9 5℃、0 . 5m i n, 5 0℃、1m i n, 7 4℃、1 . 5m i n, 7 4℃、7m i n。I T S基因P C R循环参数设定为9 4℃, 5 m i n; 3 0个循环包括: 9 4℃、1 m i n, 5 0℃、1m i n, 7 2℃、1 . 5 m i n, 7 2℃、8 m i n。r b c L基因P C R循环参数设定为9 4℃, 5m i n; 3 0个循环包括: 9 4℃、1 m i n, 5 3℃、1 m i n, 7 2℃、1 m i n, 7 2℃、1 0m i n。P C R产物经1%琼脂糖电泳检测后送至生工生物工程(上海)股份有限公司测序。
利用S e q u e n c h e r软件拼接样品的双向测序结果,经人工校对确定无误后提交至G e n B a n k数据库。利用C l u s t a lX2 . 0软件进行多序列比对并查错。利用ME GA7 . 0软件对D NA序列特征进行分析,包括序列长度、G C含量、多态位点和简约信息位点等参数。采用最大似然法进行序列饱和性检验,使用D AMB E软件建立碱基替换饱和曲线[ 1 6]。利用K i m u r a - 2 - p a r a m e t e r( K 2 P)[ 1 7]模型计算种内和种间的遗传距离。将实验所得数据信息与G e n -B a n k中的数据进行比对,采用邻接法构建不同序列的小球藻系统发育树。采用自举检验法检验邻接法分支树的可信度,各分支经10 0 0次重复抽样检验得到支持率。以计算机P e r l语言统计各序列种内和种间遗传距离的分布频度,评估3条D NA条形码候选序列的间隔( b a r c o d i n gg a p)。
2 结果分析
2 . 1 C O I、I T S和r b c L的序列结构特征
3条D NA条形码候选序列在2 0株小球藻中的扩增均呈阳性,扩增成功率分别为7 6%、8 3%和7 0%。各条候选D NA条形码的序列特征如表3所示。
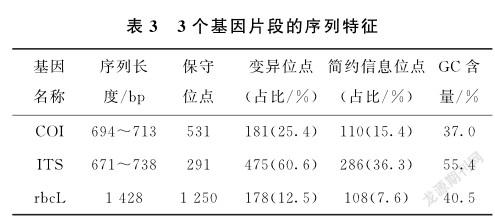
各序列长度的范围为6 7 1~14 2 8b p,其中I T S的变化范围最大( 6 7 1~7 3 8b p)。实验所得的I T S序列包含完整的I T S 1、5 . 8 Sr D NA和I T S 2, 2 0条I T S的序列长度并不一致,其中5 . 8 Sr D NA最为保守( 1 5 9b p) ,而I T S 1和I T S 2出现一定的变化。3条序列的G C含量也不同,其中I T S最高( 5 5 . 4%) , C O I最低( 3 7 . 0%)。
3条D NA条形码候选序列的遗传差异是通过p距离和简约信息位点占比( P I %)计算的,其中C O I、I T S和r b c L的平均p距离分别是0 . 0 5 7、0 . 2 2 0和0 . 0 3 3,而其P I %值分别为1 5 . 4%、3 6 . 3%和7 . 6%。其中I T S的平均p距离和P I %值最高,表明I T S在3条D NA条形码候选序列中的遗传变异度最高。
2.2 碱基替换饱和性
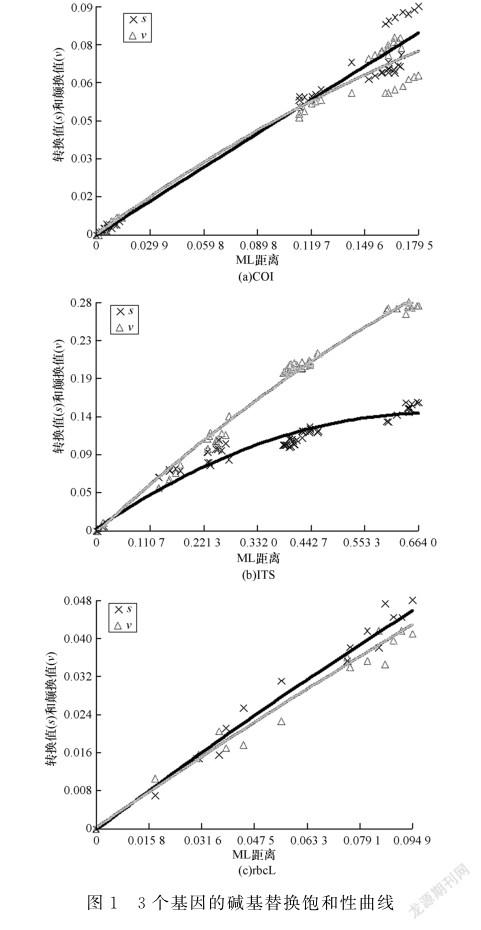
C O I、I T S和r b c L的碱基替换饱和性曲线如图1所示。
由图1可以看出: C O I和r b c L的转换值略高于颠换值,而I T S的颠换值高于转换值; I T S转换曲线已进入平稳期,表明小球藻的I T S序列的转换已达到饱和;与之相比, C O I和r b c L的转换曲线和颠换曲线均为线性关系,表明这2个序列之间的替换尚未达到饱和,更适合用于系统树的构建。
2 . 3 C O I、I T S和r b c L的b a r c o d i n gg a p
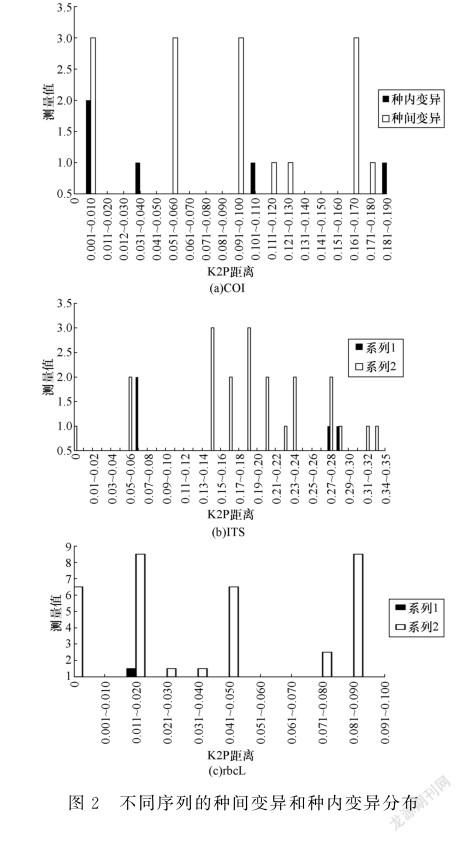
標准D NA条形码的种间遗传变异应显著大于种内遗传变异,并形成1个间隔区即b a r c o d i n g g a p[ 1 8-1 9],因此种内变异应集中分布于b a r c o d i n g g a p图中数值较小的一侧,而数值较大的一侧应集中显示种间变异[ 2 0]。小球藻的3条D NA条码候选序列的b a r c o d i n gg a p如图2所示。
由图2可以看出, C O I、I T S和r b c L的种内变异和种间变异存在不同程度的重叠。其中: C O I的种内变异超过种间变异,因此不利于小球藻的分类鉴定;而I T S和r b c L的种内变异和种间变异虽有一定的重叠,但仍存在间隔区,且相比之下r b c L的间隔区更大,因此有利于小球藻的分类鉴定。
2 . 4 C O I、I T S和r b c L的系统发育
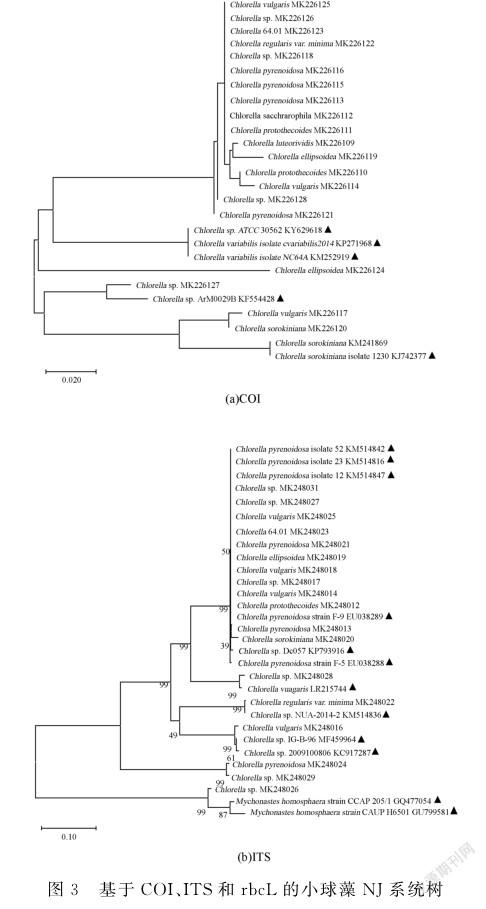
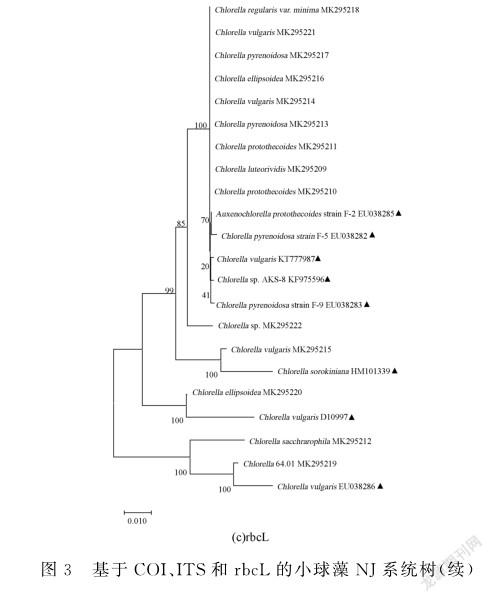
在目前对3个候选序列分别进行的系统发育分析中,各分支内的聚类情况都是无序的,与各自的形态学分类地位不符。在形态学分类上,同种的小球藻没有被完全聚为同一类,而不同种的小球藻又没有被分开,与传统分类学存在较大差异。由于3个候选片段都不能完全区分所有的小球藻种类,可能其并不适合较低的分类单位。
基于C O I、I T S和r b c L的小球藻N J系统树如图3所示,其中▲标记的为G e n B a n k中的数据。
3 讨论
理想的D NA条形码序列应具有易于高效扩增、种间差异显著和种内变异较小等特点[ 2 1]。根据对小球藻不同序列的分析结果, 3条候选D NA条形码序列均不适合单独完成对小球藻到种水平的鉴定,但其各具特点。H e b e r t等[ 2 2]对动物界中除刺胞动物门外的所有动物门进行C O I基因研究,发现C O I基因的长度较短、易于扩增且变异率适度,适合作为物种D NA条形码。C O I基因是目前公认的动物D NA条形码基因,其有效性在对软体动物、鸟类[ 2 3-2 6]和鳞翅目昆虫[ 2 7]等的研究中得到验证。多名学者和国际机构分别对陆生植物D NA条形码进行研究,发现C O I基因不仅进化速率低,而且基因含量和结构改变很大,因此其作为植物D NA条形码并不合适[ 2 1, 2 8-3 0]。在藻类D NA条形码研究中, C O I基因在硅藻、甲藻、褐藻和红藻研究中都得到成功应用[ 8, 3 1-3 3];但C O I基因在绿藻中不能设计出有效的引物,因此不适合作为绿藻的条形码[ 3 4]。本研究的分析结果也表明C O I序列不适合作为小球藻D NA条形码,其原因可能有2点:①缺乏数据,即G e n B a n k数据库中缺乏本研究实验所采用藻种的C O I基因序列;②在形态分类时,由于样本缺少明确的形态特征或形态指标不适合作为种的分类标准,导致分类出现偏差[ 3 5]。
I T S序列是核糖体r R NA基因的转录间隔区,包含I T S 1、5 . 8 Sr D NA和I T S 2,其中I T S 1和I T S 2的进化速率较快,而5 . 8 Sr D NA最为保守。较高的变异度使I T S在区分物种方面的能力较强,其在一些原生生物[ 1 0]、真菌[ 1 1-1 2]、藻类[ 1 3, 3 6-3 7]、植物[ 3 8-3 9]和动物[ 4 0]的分类鉴定中得到成功应用,而本研究基于I T S基因构建的系统树无法将所有种类的小球藻完全分开。M u l l e r等[ 4 1]对2 9株普通小球藻的I T S序列长度和基因扩增片段长度的多态性( A F L P)进行分析,提出有些已由传统分类方法确定的普通小球藻并不属于此分类; H u s s等[ 4]基于小球藻的生化和分子信息,认为蛋白核小球藻和普通小球藻同种异名,因此在系统树中表现为I T S长度相同的普通小球藻和蛋白核小球藻被聚在同一个分枝中。而在依赖小球藻类1 8 Sr R NA基因构建的系统发育树中, C h l o r e l as a c c h a r o p h i l a、C h l o r e l l a e l i p s o i d e a、C h l o r e l a l u t e o v i r i d i s和C h l o r e l am i r a b -i l i s各自以很高的自展值聚在一起[ 4],与本研究实验根据I T S基因序列构建的系统发育树的结果类似,表明这些小球藻具有很近的亲缘关系。在本研究实验中, I T S的遗传距离(p距离)和P I %值均比C O I和r b c L高很多,表明其具有较高的变异度,而碱基替换饱和性分析也证明I T S具有很高的变异度,这可能导致I T S序列不适合单独用于小球藻的系统发育分析。
r b c L基因全长约1 . 4k b,能提供较多的分子信息,在光合作用和光呼吸中占据重要地位。受到功能的限制, r b c L的进化速率较慢,因此更适合较高分类阶元的研究[ 4 2-4 4]。本研究实验中r b c L基因的遗传距离(p距离)和P I %值在3个条形码候选片段中均最低,表明其较为保守。但r b c L的引物通用性强且序列易比对,可参考将其与多个片段组合,以提升物种鉴定的正确率[ 4 3-4 5]。F r i e d l[ 4 6]基于1 8 Sr D NA序列的分析以及H u s s等[ 4]对生物化学、生理学、超微结构和1 8 Sr D NA的研究,均将小球藻类群划分为2个不同的纲,即绿藻纲( C h l o r o p h y c e a e)和共球藻纲( T r e b o u x i o p h y c e a e) ,其中H u s s等[ 4]又将共球藻纲中的小球藻分为2个类群。本研究实验根据r b c L基因所构建的系统发育树与H u s s等[ 4]的分类相似,即类群Ⅰ中的C h l o r e l av u l g a r i s和C h l o r e l l ap r o t o t h e c o i d e s聚在一起,而类群Ⅱ中的C h l o r e l l ae l i p s o i d e a和C h l o r e l l as a c c h r a r o p h i l a分布在外围;但又与H u s s等[ 4]的分类略有差别,如属于类群Ⅱ的1株C h l o r e l ae l l i p s o i d e a和C h l o -r e l l al u t e o r i v i d i s与类群Ⅰ聚在一起,而这与孙雪等[ 2]的研究结果类似。出现这种情况的原因可能为实验所采用的藻株不同、数据库中的信息错误、样品D NA污染和分类命名错误等,导致分析结果的可信度降低或找不到可参考的数据[ 7]。
随着时代的发展,小球藻属的成员不断变化,而目前对于藻类D NA条形码的研究还不成熟,小球藻D NA条形码尚没有统一的标准基因片段。但可参考动植物的分类方法,将多个片段组合应用,以筛选不同进化级别的D NA条形码[ 7]。随着小球藻类不同D NA片段信息的不断丰富、规范化和规模化以及D NA条形码技术的不断成熟, D NA条形码将在小球藻分类鉴定中得到更广泛的应用。
参考文献
[ 1] 黄燕娟,王小芬,陈向凡.小球藻的营养及药用价值[ J].现代生物医学进展, 2 0 1 3, 1 3( 3 2) : 6 3 9 6-6 3 9 8.
[ 2] 孙雪,吴晓微,李兴文,等.小球藻核r D NAI T S与叶绿体r b c L基因序列分析及应用[ J].水产学报, 2 0 0 9, 3 3( 4) : 5 6 5-5 7 1.
[ 3] B L A N CG, D UN C A NG, A G A R K O V AI, e t a l . T h eC h l o r e l a v a r i -a b i l i sN C 6 4 Ag e n o m e r e v e a l s a d a p t a t i o nt op h o t o s y m b i o s i s, c o e v o -l u t i o nw i t hv i r u s e s, a n dc r y p t i cs e x[ J]. P l a n tC e l l, 2 0 1 0, 2 2( 9) : 2 9 4 3-2 9 5 5.
[ 4] HU S SVAR, F R A N KC,HA R TMA N NEC, e ta l . B i o c h e m i c a l t a x o n o m ya n d m o l e c u l a rp h y l o g e n y o ft h e g e n u sc h l o r e l a s e n s u l a t o( c h l o r o p h y t a) [ J]. J o u r n a lo fP h y c o l o g y, 1 9 9 9, 3 5( 3) : 5 8 7-5 9 8.
[ 5] K R I E N I T ZL, HE G EWA L DEH, HE P P E R L ED, e t a l . P h y l o -g e n e t i cr e l a t i o n s h i p o fc h l o r e l l aa n d p a r a c h l o r e l ag e n . n o v .( c h l o r o p h y t a, t r e b o u x i o p h y c e a e) [ J]. P h y c o l o g i a, 2 0 0 4, 4 3( 5) : 5 2 9-5 4 2.
[ 6] HE B E R TPD, R A T NA S I N GHAM S, D EWAA R DJR. B a r -c o d i n ga n i m a l l i f e: c y t o c h r o m eco x i d a s es u b u n i t1d i v e r g e n c e s a m o n gc l o s e l yr e l a t e ds p e c i e s[ J]. P r o cB i o l S c i, 2 0 0 3, 2 7 0( S 1) : 9 6-9 9.
[ 7] 崔翠菊,張立楠,王娜,等.藻类D NA条形码研究进展[ J].中国生物工程杂志, 2 0 1 2, 3 2( 9) : 1 1 3-1 1 7.
[ 8] S A UN D E R SGW. A p p l y i n gD NAb a r c o d i n g t o r e dm a c r o a l g a e: ap r e l i m i n a r ya p p r a i s a lh o l d sp r o m i s ef o rf u t u r ea p p l i c a t i o n s[ J]. P h i l o sT r a n sRS o cL o n dBB i o lS c i, 2 0 0 5, 3 6 0( 1 4 6 2) : 1 8 7 9-1 8 8 8.
[ 9] KU C E R AH, S AUN D E R SG W. A s s i g n i n gm o r p h o l o g i c a l v a r i -a n t so ff u c u s( f u c a l e s,p h a e o p h y c e a e)i nC a n a d i a nw a t e r st o r e c o g n i z e ds p e c i e su s i n gD NAb a r c o d i n g[ J]. B o t a n y, 2 0 0 8, 8 6( 9) : 1 0 6 5-1 0 7 9.
[ 1 0] S T E R N R F,AN D E R S E N R A,J AME S ON I,e t a l . E v a l u a t i n gt h e r i b o s o m a l i n t e r n a l t r a n s c r i b e ds p a c e r( I T S)a s ac a n d i d a t ed i n o f l a g e l a t eb a r c o d e m a r k e r[ J]. P L o S O n e, 2 0 1 2, 7( 8) : e 4 2 7 8 0.
[ 1 1] S E I F E R TKA. P r o g r e s s t o w a r d sD NAb a r c o d i n go f f u n g i[ J]. M o lE c o lR e s o u r, 2 0 0 9, 9( S 1) : 8 3-8 9.
[ 1 2] S C HO C HCL, S E I F E R TKA, HUHN D O R FS, e t a l . N u c l e a r r i b o s o m a l i n t e r n a l t r a n s c r i b e ds p a c e r( I T S)r e g i o na sau n i -v e r s a lD NAb a r c o d em a r k e r f o rF u n g i[ J]. P r o c e e d i n g so f t h e N a t i o n a lA c a d e m yo fS c i e n c e so f t h eU n i t e dS t a t e so fAm e r i -c a, 2 0 1 2, 1 0 9( 1 6) : 6 2 4 1-6 2 4 6.
[ 1 3] 郭立亮.海洋微藻D NA條形码基因的评估及环境样本多样性分析和定量基因的开发[ D].青岛:中国海洋大学, 2 0 1 5.
[ 1 4] 孙雪,马斌,周成旭.蛋白核小球藻核酮糖- 1, 5 -二磷酸羧化/加氧酶小亚基部分基因序列的克隆与分析[ J].海洋科学, 2 0 0 8( 3) : 4 0-4 3.
[ 1 5] HA S E B E M, I T O M, KO F U J IR, e ta l . P h y l o g e n e t i cr e l a t i o n -s h i p s i ng n e t o p h y t ad e d u c e df r o mr b c Lg e n es e q u e n c e s[ J]. T h eB o t a n i c a lM a g a z i n e, T o k y o, 1 9 9 2, 1 0 5( 3) : 3 8 5-3 9 1.
[ 1 6] X I AX. D a m b e7: n e wa n d i m p r o v e dt o o l s f o rd a t aa n a l y s i s i n m o l e c u l a rb i o l o g ya n de v o l u t i o n[ J]. M o lB i o lE v o l, 2 0 1 8, 3 5( 6) : 1 5 5 0-1 5 5 2.
[ 1 7] K I MUR A M. As i m p l em e t h o df o re s t i m a t i n ge v o l u t i o n a r y r a t e so f b a s e s u b s t i t u t i o n s t h r o u g hc o m p a r a t i v e s t u d i e s o f n u -c l e o t i d es e q u e n c e s[ J].JM o lE v o l, 1 9 8 0, 1 6( 2) : 1 1 1-1 2 0.
[ 1 8] L AHAY ER, VAND E RB ANK M, B O GA R I ND, e t a l . D NA b a r c o d i n gt h ef l o r a so fb i o d i v e r s i t yh o t s p o t s[ J]. P r o c e e d i n g s o f t h eN a t i o n a lA c a d e m yo fS c i e n c e so f t h eU n i t e dS t a t e so f Am e r i c a, 2 0 0 8, 1 0 5( 8) : 2 9 2 3-2 9 2 8.
[ 1 9] ME Y E RCP, P AU L AYG. D NAb a r c o d i n g: e r r o rr a t e sb a s e d o nc o m p r e h e n s i v es a m p l i n g[ J]. P L o SB i o l, 2 0 0 5, 3( 1 2) : e 4 2 2.
[ 2 0] 姜黎.基于D NA條形码技术的重楼正伪品鉴定方法研究[ D].泸州:泸州医学院, 2 0 1 3.
[ 2 1] C B O LP l a n tW o r k i n gG r o u p1, P E T E R M H, L AUR AL, e t a l . AD NAb a r c o d e f o r l a n dp l a n t s[ J]. P r o c e e d i n g so f t h eN a -t i o n a lA c a d e m yo fS c i e n c e so f t h eU n i t e dS t a t e so fAm e r i c a, 2 0 0 9, 1 0 6( 3 1) : 1 2 7 9 4-1 2 7 9 7.
[ 2 2] HE B E R TPD, C YW I N S KAA, B A L LSL, e ta l . B i o l o g i c a l i -d e n t i f i c a t i o n s t h r o u g hD NAb a r c o d e s[ J]. P r o cB i o lS c i, 2 0 0 3, 2 7 0( 1 5 1 2) : 3 1 3-3 2 1.
[ 2 3] J OHN S E NA, R I N D A LE, E R I C S ONPGP, e ta l . D NAb a r -c o d i n go fs c a n d i n a v i a n b i r d sr e v e a l sd i v e r g e n tl i n e a g e si n t r a n s - a t l a n t i cs p e c i e s[ J]. J o u r n a lo fO r n i t h o l o g y, 2 0 1 0, 1 5 1( 3) : 5 6 5-5 7 8.
[ 2 4] P A R K H, YOO HS, J UN GG, e ta l . N e wD NAb a r c o d e s f o r i d e n t i f i c a t i o no fK o r e a nb i r d s[ J]. G e n e s&G e n o m i c s, 2 0 1 1, 3 3( 2) : 9 1-9 5.
[ 2 5] L I J TMA E RDA, K E R RKC, S T O E C K L E M Y, e ta l . D NA b a r c o d i n gb i r d s: f r o m f i e l dc o l l e c t i o nt od a t aa n a l y s i s[ J]. M e t h o d sM o lB i o l, 2 0 1 2, 8 5 8: 1 2 7-1 5 2.
[ 2 6] A L I A B A D I AN M, B E E N T J E SK K, R O S E L AA RCS, e ta l . D NAb a r c o d i n go fD u t c h b i r d s[ J]. Z o o k e y s,2 0 1 3( 3 6 5) : 2 5-4 8.
[ 2 7] HE B E R TPD, P E N T ONEH, B UR N SJM, e t a l . T e ns p e c i e s i n o n e:D NA b a r c o d i n g r e v e a l s c r y p t i c s p e c i e s i n t h e n e o t r o p i c a ls k i p p e rb u t t e r f l y A s t r a p t e sf u l g e r a t o r[ J]. P r o -c e e d i n g so f t h eN a t i o n a lA c a d e m yo fS c i e n c e so ft h eU n i t e d S t a t e so fAm e r i c a, 2 0 0 4, 1 0 1( 4 1) : 1 4 8 1 2-1 4 8 1 7.
[ 2 8] V I J A Y A NK, T S O UCH. D N Ab a r c o d i n g i np l a n t s: t a x o n o m y i n an e wp e r s p e c t i v e[ J]. C u r r e n tS c i e n e, 2 0 1 0, 9 9( 1 1) : 1 5 3 0.
[ 2 9] 闫化学,于杰. D NA条形码技术在植物中的研究现状[ J].植物学报, 2 0 1 0, 4 5( 1) : 1 0 2-1 0 8.
[ 3 0] 唐建阳,周先治.植物D NA条形码研究现状及应用前景[ J].中国农学通报, 2 0 0 9, 2 5( 2 4) : 3 5-4 3.
[ 3 1] E VAN SK M,MANNDG. Ap r o p o s e dp r o t o c o l f o rn o m e n -c l a t u r a l l ye f f e c t i v eD NAb a r c o d i n go fm i c r o a l g a e[ J]. P h y c o l o -g i a, 2 0 0 9, 4 8( 1) : 7 0-7 4.
[ 3 2] E VAN SK M,WO R T L E YA H,MANNDG. A na s s e s s m e n t o fp o t e n t i a l d i a t o m“b a r c o d e”g e n e s( c o x 1, r b c L, 1 8 Sa n d I T S r D NA) a n dt h e i re f f e c t i v e n e s s i nd e t e r m i n i n gr e l a t i o n s h i p s i n s e l l a p h o r a(b a c i l l a r i o p h y t a)[ J]. P r o t i s t,2 0 0 7,1 5 8(3) : 3 4 9-3 6 4.
[ 3 3] R O B B AL, RU S S E L LSJ, B A R K E RGL, e t a l . A s s e s s i n g t h e u s eo f t h em i t o c h o n d r i a l c o x 1m a r k e r f o ru s e i nD NAb a r c o d -i n go fr e da l g a e( r h o d o p h y t a) [ J]. AmJB o t, 2 0 0 6, 9 3( 8) : 1 1 0 1-1 1 0 8.
[ 3 4] 郭盛華. D NA条形码技术的概况及其在大型海藻中的应用[ J].科技资讯, 2 0 1 2( 1 8) : 2 4 7.
[ 3 5] F UNK D J,OML AN D K E. S p e c i e s - l e v e lp a r a p h y l y a n d p o l y p h y l y: f r e q u e n c y, c a u s e s, a n dc o n s e q u e n c e s,w i t hi n s i g h t s f r o m a n i m a l m i t o c h o n d r i a l D NA[ J]. A n n u a l R e v i e w o f E c o l o g yE v o l u t i o na n dS y s t e m a t i c s, 2 0 0 3, 3 4: 3 9 7-4 2 3.
[ 3 6] MON I Z M B,KA C ZMA R S KA I . B a r c o d i n g o f d i a t o m s: n u c l e a re n c o d e dI T Sr e v i s i t e d[ J]. P r o t i s t,2 0 1 0,1 6 1( 1) : 7-3 4.
[ 3 7] MON I ZMB, KA C ZMA R S KAI . B a r c o d i n gd i a t o m s: i s t h e r e a g o o dm a r k e r? [ J]. M o lE c o lR e s o u r, 2 0 0 9, 9( S 1) : 6 5-7 4.
[ 3 8] YAN GJB, GAOLM, L I UJQ, e t a l . C o m p a r a t i v e a n a l y s i s o f al a r g e d a t a s e ti n d i c a t e st h a ti n t e r n a lt r a n s c r i b e d s p a c e r( I T S)s h o u l db e i n c o r p o r a t e di n t ot h ec o r eb a r c o d ef o rs e e d p l a n t s[ J]. P r o c e e d i n g so f t h eN a t i o n a lA c a d e m yo fS c i e n c e so f t h eU n i t e dS t a t e so fAm e r i c a, 2 0 1 1, 1 0 8( 4 9) : 1 9 6 4 1-1 9 6 4 6.
[ 3 9] D UZ,Q I M I K E A,YAN G C, e ta l . T e s t i n gf o u rb a r c o d i n g m a r k e r sf o rs p e c i e si d e n t i f i c a t i o no fP o t a m o g e t o n a c e a e[ J]. J o u r n a l o fS y s t e m a t i c sa n dE v o l u t i o n, 2 0 1 1, 4 9( 3) : 2 4 6-2 5 1.
[ 4 0] MO R I T ZG, P AU L S E N M,D E L K E R C, e ta l . I d e n t i f i c a t i o n o f t h r i p s u s i n g I T S - R F L Pa n a l y s i s[ J]. T h r i p s a n dT o s p o v i r u s -e s: P r o c e e d i n g so f t h e7 t hI n t e r n a t i o n a lS y m p o s i u mo nT h y s -a n o p t e r a, 2 0 0 2: 3 6 5-3 6 7.
[ 4 1] MU L L E RJ, F R I E D LT, HE P P E R L ED, e ta l . D i s t i n c t i o nb e -t w e e n m u l t i p l ei s o l a t e so fc h l o r e l av u l g a r i s( c h l o r o p h y t a, t r e b o u x i o p h y c e a e)a n d t e s t i n g f o r c o n s p e c i f i c i t y u s i n g a m p l i f i e df r a g m e n t l e n g t hp o l y m o r p h i s ma n dI T Sr D NAs e -q u e n c e s[ J].J o u r n a l o fP h y c o l o g y, 2 0 0 5( 4 1) : 1 2 3 6-1 2 4 7.
[ 4 2] D AUG B J E R GN, AN D E R S E NRA. P h y l o g e n e t i ca n a l y s e so f t h er b c Ls e q u e n c e sf r o m h a p t o p h y t e sa n dh e t e r o k o n ta l g a e s u g g e s t t h e i rc h l o r o p l a s t sa r eu n r e l a t e d[ J]. M o lB i o lE v o l, 1 9 9 7, 1 4( 1 2) : 1 2 4 2-1 2 5 1.
[ 4 3] N EWMA S T E RSG, F A Z E KA SAJ, S T E E V E SR A, e ta l . T e s t i n gc a n d i d a t ep l a n tb a r c o d er e g i o n si nt h em y r i s t i c a c e a e[ J]. M o lE c o lR e s o u r, 2 0 0 8, 8( 3) : 4 8 0-4 9 0.
[ 4 4] S A S SC, L I T T L EDP, S T E V E N S ON D W, e ta l . D NAb a r -c o d i n g i nt h ec y c a d a l e s: t e s t i n gt h ep o t e n t i a l o fp r o p o s e db a r -c o d i n gm a r k e r sf o rs p e c i e si d e n t i f i c a t i o no fc y c a d s[ J]. P L o S O n e, 2 0 0 7, 2( 1 1) : e 1 1 5 4.
[ 4 5] F A Z E KA SAJ, B UR G E S SKS, K E S ANAKUR T IPR, e t a l . M u l t i p l em u l t i l o c u sD NAb a r c o d e sf r o mt h ep l a s t i dg e n o m e d i s c r i m i n a t ep l a n t s p e c i e se q u a l l yw e l l[ J]. P L o SO n e, 2 0 0 8, 3( 7) : e 2 8 0 2.
[ 4 6] F R I E D L T. I n f e r r i n gt a x o n o m i cp o s i t i o n sa n dt e s t i n gg e n u s l e v e l a s s i g n m e n t s i nc o c c o i dg r e e n l i c h e na l g a e: ap h y l o g e n e t i c a n a l y s i so f1 8 Sr i b o s o m a lRNAs e q u e n c e sf r o m d i c t y o c h l o -r o p s i sr e t i c u l a t aa n df r o m m e m b e r so ft h eg e n u sm y r m e c i a( c h l o r o p h y t a, t r e b o u x i o p h y c e a ec l . n o v . ) [ J]. J o u r n a lo fP h y -c o l o g y, 1 9 9 5, 3 1( 4) : 6 3 2-6 3 9.
