牡丹的基因组DNA提取与ISSR引物筛选
2021-09-12李会云程孟雪李勇慧刘忠艳
李会云 程孟雪 李勇慧 刘忠艳

摘要:为了研究牡丹种质资源的遗传多样性和DNA鉴定,采用ISSR标记的方法来进行试验,即采用改良的CTAB法从成熟干燥的牡丹叶片中提取基因组DNA,其质量检测采用琼脂糖凝胶电泳法和超微量分光光度计法,用优化的ISSR-PCR反应系统,对加拿大哥伦比亚大学(University of British Columbia,UBC)公布的220条ISSR引物进行筛选。结果表明,从6个牡丹品种的叶片中提取的DNA浓度范围在30×10-3 ~100×10-3 μg/μL之间,DNA样品纯度 D260 nm/D280 nm 比值在1.7~1.9之间;从220条ISSR引物中筛选出了12条适合牡丹的扩增结果好的引物,选出的引物扩增条带清晰、多态性高、重复性好。说明提取的DNA质量较好,该改良的CTAB法适用于从蛋白质和酚类物质含量较高的植物材料中提取基因组DNA;筛选出的12条ISSR引物为进一步研究牡丹资源的遗传多样性奠定了基础。
关键词:牡丹;DNA提取;ISSR引物筛选;PCR扩增;改良的CTAB法
中图分类号:S685.110.1 文献标志码: A
文章编号:1002-1302(2021)15-0058-06
收稿日期:2020-12-21
基金项目:河南省科技攻关项目(编号:192102110035)。
作者简介:李会云(1982—),女,河南安阳人,博士,讲师,从事植物生理及分子生物学的教学与科研工作。E-mail:30226972@qq.com。
牡丹(Paeonia suffruticosa),是芍药科芍药属多年生落叶灌木[1],是一种传统中国花卉,也是世界著名花卉,姿态万千、色彩丰富,不但在园林和花卉文化中占有重要地位,深受中国人民喜爱,而且在对外传播和交流时受到世界人民喜爱。同时,牡丹也是一种重要的花卉植物和资源植物,其药用、文化和观赏历史已约有2 000余年。中国牡丹主要分布在洛阳、兰州、彭州、菏泽、延安、铜陵等地。据调查,不同产地的牡丹共计约有1 500个品种[2-4]。
近年来,RAPD、SSR、ISSR、AFLP和SRAP 等分子标记已广泛用于牡丹的遗传多样性分析、核心种质构建、种质资源分类鉴定等方面[5]。ISSR(inter-simple sequence repeat)标记是由Zietkiewicz等开发的分子标记[6]。SSR( simple sequence repeat) 广泛分布在真核植物基因组中,由1~6个碱基对组成,其两端序列通常是相对保守的单拷贝序列,因此,可以设计特异的寡核苷酸引物进行SSR-PCR,根据串联重复序列的数量,通过扩增串联重复序列揭示了SSR的长度多态性[7]。ISSR是基于SSR发展起来的一种新型分子标记技术,它根据基因组内广泛存在的微卫星序列设计单一引物,扩增出DNA序列在SSR之间的反向排列,然后对扩增产物进行电泳、染色,根据条带的存在及相对位置来分析不同样品间ISSR标记的多态性[8-9]。ISSR标记方法由于其高效性、重复性好、操作简单等特点,被广泛应用于种质资源鉴定、遗传多样性分析、杂种后代快速鉴定等领域[10]。目前,ISSR标记已经用于湖南、云南香格里拉、福建、四川[11-14]牡丹的遗传多样性分析,然而,ISSR标记尚未用于分析洛阳牡丹的遗传多样性。本试验采用优化的ISSR-PCR反应体系篩选ISSR引物,选出12条引物,为研究洛阳牡丹的遗传多样性奠定基础。
1 材料与方法
1.1 试验材料
本试验植物材料采自洛阳市牡丹研究院,共有6个牡丹品种:鹤顶红、文培紫、海棠争润、银粉金鳞、似荷莲、锦袍江,对这6个牡丹品种从1~6依次进行编号,干燥处理后保存于自封袋备用。本试验于2019年在洛阳师范学院生命科学学院生科楼406和509实验室实施。
1.2 主要仪器设备
YXQ-SG46-280S型手提式压力蒸汽灭菌器(施都凯仪器设备有限公司);101-1AB型电热鼓风干燥箱(天津市泰斯特仪器有限公司);SCIENTZ-48型高通量组织研磨机(宁波新芝生物科技股份有限公司);JA2003A型电子天平(上海精天电子仪器有限公司);LX-200迷你型离心机(海门市其林贝尔仪器);TGL-16B台式离心机(上海安亭科学仪器厂制造);DYY-6C型电泳仪(北京市六一仪器厂);ChampChemi 610 Plus型全自动多色荧光及化学发光凝胶成像系统(北京赛智创业科技有限公司);THZ-82A数显恒温振荡器(常州朗越仪器制造有限公司);SEDIG型梯度PCR仪;Eppendorf AG型PCR仪;K5500 Plus型超微量分光光度计。
1.3 主要试剂
CTAB、EDTA,均购自生工生物工程(上海)股份有限公司;Tris-HCl(pH值=8.0)、三氯甲烷、异戊醇、乙酸钠、冰乙醇、β-巯基乙醇、GoldView I型核酸染色剂、10×DNA loading buffer,均购自北京索莱宝科技有限公司;2×Taq MasterMix混合液(康为,CW0690M)。
1.4 试验方法
1.4.1 牡丹DNA的提取
根据改良的CTAB磁珠法[15]提取牡丹基因组DNA。取少量干燥叶片于 2 mL 的研磨管中,并放入直径3 mm的钢珠,再将研磨管放进组织研磨机中进行研磨,60 s后取出。加入1 000 μL 2% CTAB溶液和8 μL β-羟基乙醇,放入水浴锅中65 ℃水浴1 h,每10 min倒置1次以混均匀。水浴锅中取出后,加入500 μL CI(三氯甲烷 ∶异戊醇=24 ∶:1),摇床10 min(强度约160 r/min左右)。然后在室温下1 200 r/min离心10 min,取上清液650 μL(视具体情况而定)于 1.5 mL 离心管中(弃去有机相)。再加入500 μL CI于1.5 mL的离心管中,摇床摇10 min。然后在室温下1 200 r/min离心10 min,取上清液500 μL(视情况而定)于1.5 mL离心管中(吸取时小心仔细)。加入1/10体积3 mol/L乙酸钠和2倍体积冰乙醇(体积与吸取的上清液相比)低温静置24 h(-20 ℃)。保留白色球状物,倒去溶液(可先离心)。加入 500 μL 75%乙醇清洗2次,倒去溶液(根据情况离心,尽量第1次不离心,第2次离心。1 200 r/min离心2~3 min)。将离心管倒扣在吸水纸或卫生纸上,约5~10 min,常温下干燥DNA,之后再开盖放在烘干机中,50 ℃烘干 20 min 左右。加入适量无菌水(50~100 μL)溶解DNA。
1.4.2 DNA浓度测定
通过超微量分光光度计测定DNA的浓度,通过测定DNA的D260 nm/D280 nm比确定DNA的纯度和完整程度。最后将适用牡丹DNA保存于4 ℃备用。
1.4.3 PCR扩增反应
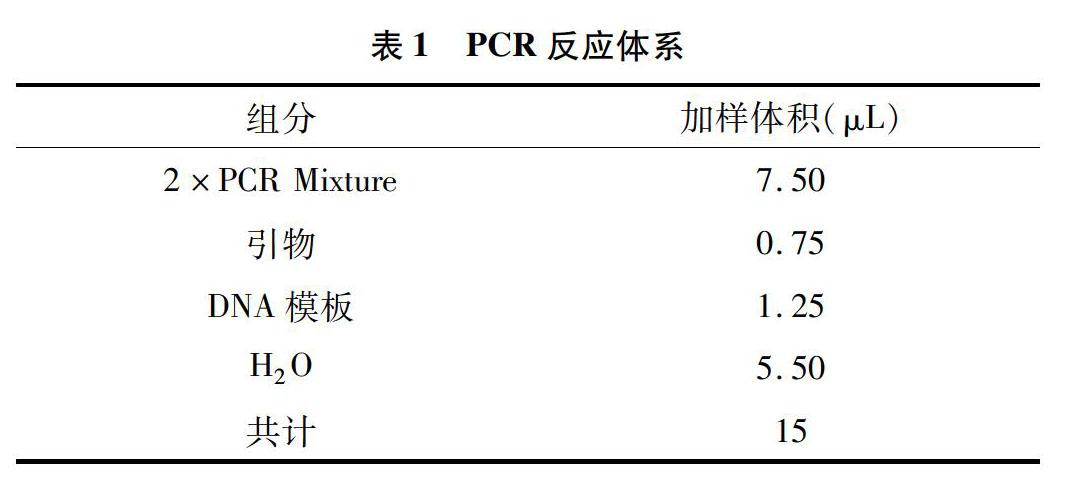
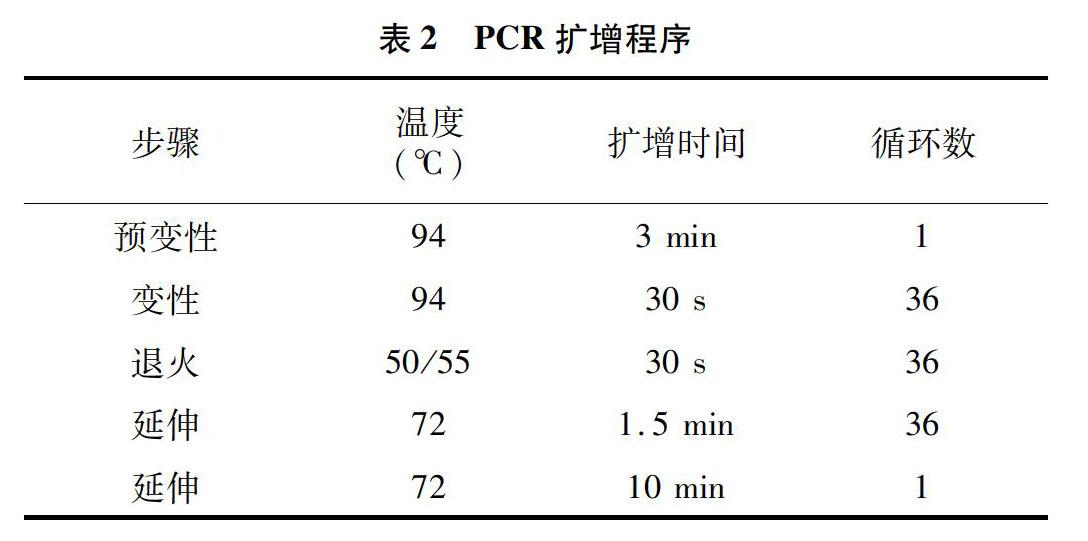
牡丹PCR反应体系见表1(DNA模板是从35种牡丹中随机选取的6个牡丹基因组DNA)。PCR扩增程序见表2。
1.4.4 琼脂糖凝胶电泳
在锥形瓶中称取0.3 g的琼脂糖,加入30 mL 1×TAE缓冲液,配制成溶液,加热混匀,向溶液中加入2 μL GoldView I型核酸染色剂,混合,倒入模具中(注意不能有气泡,否则会影响跑胶结果),冷却10 min。等琼脂糖胶凝固后,将梳子垂直拔出。在电泳槽内加入1×TAE缓冲液,将配好的胶放入电泳槽中,准备点样。从冰箱拿出扩增好的产物,第1个孔内加入 1 μL 10×Loading buffer和4 μL Marker,之后每个孔内加入5 μL扩增好的产物。按照顺序依次迅速精准点样,以防止样品扩散,导致跑胶条带不清晰。加样完毕后,设置电压为120~130 V,电流为110 mA,电泳时间30~45 min。电泳结束后,轻轻取出凝胶并放入凝胶成像系统中观察成像结果。
1.4.5 引物筛选及退火温度的确定
本试验根据已优化的ISSR-PCR反应体系,用鹤顶红、文培紫、海棠争润、银粉金鳞、似荷莲、锦袍江6个牡丹DNA样品,对UBC公布的220条ISSR引物(表3)进行初筛和复筛,使用温度梯度模式选择合适的退火温度。温度设定为45、50、55、59 ℃4个温度梯度。
1.4.6 数据处理
各个DNA样品的浓度和D260 nm/D280 nm比值采用K5500 Plus型超微量分光光度计进行测定,各组引物的ISSR-PCR扩增图通过Microsoft PowerPoint 2013和 Adobe Photoshop CS5 软件进行制作。
2 结果与分析
2.1 基因组DNA提取质量检测结果
通过改良的CTAB法提取牡丹DNA,用超微量分光光度计测DNA浓度。测得DNA浓度范围在30~100 ng/μL,DNA样品纯度D260 nm/D280 nm比值范围在1.7~1.9之间,提取的DNA质量较好,无蛋白质、酚类的污染。
2.2 ISSR引物筛选结果及退火温度的确定
如果退火温度太低,可能会发生非特异性扩增,并降低重复率;但是,如果温度过高又会使条带减少,导致一些可能反应多态性的条带消失[16]。因此确定合适的退火温度十分必要。本研究将温度设定为45、50、55、59 ℃ 4个温度梯度,选择最佳退火温度。最后选出2个退火温度:50、55 ℃。用优化的PCR反应体系对220条引物进行扩增、筛选。根据PCR产物的凝胶成像,最终从220条引物中共筛选出12条扩增条带清晰、多态性好的ISSR引物,引物筛选结果和退火温度的确定(表4、图1、图2和图3)。
3 讨论与结论
牡丹不仅具有较好的观赏价值,同时还有很高的药用价值。为了筛选出适合牡丹遗传多样性分析的ISSR引物,本试验利用改良的CTAB法从牡丹成熟干燥的叶片中提取DNA,與Rawat等的ISSR-PCR反应体系优化研究中同样是选用成熟干叶的试验材料[17]相一致。
利用ISSR分子标记技术研究牡丹遗传多样性和品种鉴定,可以获得 DNA水平上的遗传信息,不受外界环境的影响[18]。ISSR分子标记受多种内在因素的影响,测定的可靠性、稳定性与PCR反应条件密切相关[19-21]。退火温度决定着 PCR 的特异性,它是引物和模板组合时的一个温度参数,过高或过低都会影响 ISSR-PCR 扩增的清晰度及条带的多少[22]。因此,在本试验中采用温度梯度PCR模式,将温度设定为45、50、55、59 ℃4个温度梯度,寻找合适的退火温度。
为了能够获得所需的扩增产物,在该试验中优化了ISSR-PCR反应体系。PCR反应体系中5种成分(Taq DNA聚合酶、Mg2+、dNTP、引物与模板DNA) 的浓度与反应结果有很大的关系。 TaqDNA
聚合酶是Mg2+依赖性酶,其活性会受到Mg2+浓度变化的影响,对Mg2+浓度比较敏感。Taq DNA 聚合酶用量过多时,不仅会造成成本的浪费,而且会出现非特异性扩增的条带,产生弥散现象,用量过低时会降低扩增效率[23]。本试验刚开始采用10 μL的体系,包括引物0.4 μL,10×Buffer 1 μL,dNTP 08 μL,Taq DNA聚合酶0.2 μL,双蒸水5.6 μL,模板DNA 2 μL。凝胶成像显示其扩增结果并不理想。之后用2×Taq MasterMix混合液来代替10×Buffer、dNTP和Taq DNA聚合酶,换为15 μL反应体系:2×Taq MasterMix 7.50 μL,引物0.75 μL,模板DNA 1.25 μL,双蒸水5.50 μL。凝胶成像显示其扩增条带清晰、多态性高、重复性好。利用此体系对所有引物进行引物筛选, 最终获得ISSR-11、ISSR-44、
ISSR-45、ISSR-48、ISSR-59、ISSR-60、ISSR-81、ISSR-91、ISSR-93、ISSR-94、ISSR-97、ISSR-125共12条ISSR引物。本试验用2×Taq MasterMix混合液来代替10×Buffer、dNTP和Taq DNA聚合酶,不仅可以有效解决它们之间的浓度配比问题,还可以节省操作时间、避免污染,与李振山在枣研究中使用的ISSR-PCR反应体系和试验结果[24]相似。
本研究结果表明,从鹤顶红、文培紫、海棠争润、银粉金鳞、似荷莲和锦袍江6个牡丹品种的叶片中提取的基因组DNA浓度范围在30~100 ng/μL之间,DNA样品纯度D260 nm/D280 nm比值在1.7~1.9之间,表明所提取的6个样品的DNA质量较好,没有蛋白质和酚类物质的污染,证实了该改良的CTAB法适用于从蛋白质和酚类物质含量较高的植物材料中提取基因组DNA。从220条ISSR引物中筛选出了12条引物,以6个牡丹品种的基因组DNA为模板,通过优化的ISSR-PCR反应体系进行扩增,凝胶成像结果显示,扩增条带清晰、多态性高、重复性好,共筛选出了12条引物,为研究牡丹遗传多样性分析奠定了基础。
参考文献:
[1]张 琳,王佩佩,韩雅祯,等. 油用牡丹主要相关性状与SSR分子标记的关联分析[J]. 东北林业大学学报,2019,47(3):31-37.
[2]史倩倩. 中原牡丹传统品种遗传多样性研究[D]. 北京:中国林业科学研究院,2012.
[3]李保印. 中原牡丹品种遗传多样性与核心种质构建研究[D]. 北京:北京林业大学,2007.
[4]陈富慧,索志立,赵孝庆,等. 中国牡丹品种的花期[J]. 东北林业大学学报,2005,33(6):55-61.
[5]韩丽晓,王 莹. 牡丹分子标记研究进展[J]. 安徽农业科学,2012,40(16):8850-8851,8996.
[6]Zietkiewicz E,Rafalski A,Labuda D. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification[J]. Genomics,1994,20(2):176-183.
[7]方治偉,李论.SSR分型技术研究进展[J]. 生物化工,2018,4(1):118-121.
[8]Godwin I D,Aitken E A,Smith L W. Application of inter simple sequence repeat (ISSR) markers to plant genetics[J]. Electrophoresis,1997,18(9):1524-1528.
[9]Condit R,Hubbell S P. Abundance and DNA sequence of two-base repeat regions in tropical tree genomes[J]. Genome,1991,34(1):66-71.
[10]谢佳燕,张知彬. ISSR标记技术及其在遗传多样性研究中的应用[J]. 兽类学报,2004,24(1):71-77.
[11]钟丽凡,吕长平,许文婷,等. 湖南本土牡丹遗传多样性与亲缘关系的ISSR分析[J]. 江苏农业科学,2018,46(10):34-37.
[12]刘 通,冯 丹,陈少瑜,等. 4个滇牡丹天然居群遗传多样性的ISSR分析[J]. 西部林业科学,2014,43(3):31-36.
[13]郑 涛,陈振东,林秀香,等. 福建省野牡丹属种质资源的ISSR分析[J]. 热带亚热带植物学报,2013,21(5):406-413.
[14]童 芬,谢登峰,曾心美,等. 四川牡丹和圆裂四川牡丹遗传多样性的ISSR分析[J]. 西北植物学报,2016,36(10):1968-1976.
[15]白凤虎,谢晓美,李德芳,等. 改良CTAB法用于提取红麻成熟叶片高质量DNA的研究[J]. 中国麻业科学,2007,29(3):158-161,165.
[16]罗登花,刘军民,韩正洲,等. 三桠苦ISSR反应体系的优化与引物筛选[J]. 时珍国医国药,2017,28(8):1988-1991.
[17]Rawat S,Joshi G,Annapurna D,et al. Standardization of DNA extraction method from mature dried leaves and ISSR-PCR conditions for Melia dubia Cav.—A fast growing multipurpose tree species [J]. American Journal of Plant Sciences,2016,7(3):437-445.
[18]李勇慧,于相丽,马会萍,等. 不同品种牡丹ISSR遗传多样性分析[J]. 生物技术通报,2020,36(4):78-83.
[19]徐 君,李静会,李 欣,等. 荷花ISSR引物筛选及反应体系优化[J]. 江苏农业科学,2014,42(10):42-44.
[20]胡凤荣,王 斐,王志强,等. 风信子ISSR-PCR体系的优化及引物筛选[J]. 分子植物育种,2013,11(1):139-144.
[21]陆秀娟,潘 虹,李祥栋,等. 薏苡种质资源ISSR分子标记筛选及亲缘关系分析[J]. 江苏农业科学,2019,47(5):32-36.
[22]吴 敏,贺 晓,贺一鸣,等. 蒙古高原特有植物蒙古莸ISSR-PCR反应体系优化及引物筛选[J]. 分子植物育种,2019,17(5):1583-1588.
[23]黄丽华,陈秋婷,肖雨沙,等. 半枫荷ISSR-PCR体系优化及引物筛选[J]. 分子植物育种,2020:1-18
[24]李振山. 枣ISSR反应体系的建立及其在新郑枣区优良单株早期鉴定上的应用[D]. 郑州:河南农业大学,2008:1-54.
