盐碱胁迫下青稞全转录组分析
2021-08-23王玉林原红军杨春葆徐齐君
扎 桑,王玉林,原红军,杨春葆,徐齐君*
(1. 省部共建青稞和牦牛种质资源与遗传改良国家重点实验室,西藏 拉萨 850002; 2. 西藏自治区农牧科学院农业研究所,西藏 拉萨 850002)
【研究意义】大麦不仅是全球最早的驯化作物之一,也是世界第四大谷类作物[1]。青稞(HordeumvulgareL.,qingke)是我国青藏高原地区的大麦属主栽作物,也称裸大麦,占西藏粮食总产的 80%以上,能适应高海拔地区干旱、寒冷、缺氧等恶劣环境,在长期的自然进化过程中积累了较多的特异强抗逆遗传资源[2]。盐碱化正在成为影响全球环境和土地资源的主要问题[3]。土壤盐碱化会大大降低农作物的产量[4-6]。研究表明,青稞对盐胁迫的耐受能力高于普通大麦[7-8],同时,青稞营养成分丰富,兼备粮食、饲料、工业原料等多种用途[9],因而可以作为盐碱地利用和改良的优选作物之一。植物具有应对不同非生物胁迫的多种防御系统,包括渗透调节、抗氧化防御、激素调节和细胞膜的脂质饱和[10]。培育耐盐碱品种对有效利用盐碱化土地具有重要意义。【前人研究进展】植物对盐碱的耐受性可以通过调节不同代谢途径的多个基因进行改良。这些基因主要分为四类:第一类是渗透调节相关基因,如由P5CS酶合成的脯氨酸,是一种渗透保护性氨基酸[11]。第二类是离子转运蛋白编码基因,如拟南芥中Na(+)/ H(+)反转运蛋白基因AtNHX1,可增强植物的耐盐性[12]。第三类是抗氧化相关基因,如能激活拟南芥中抗坏血酸(植物中重要的抗氧化剂)合成的AtERF98[13]。最后一类是信号转导相关基因,如RrANR能通过调节植物响应非生物胁迫的内源关键信号因子ABA,增强烟草的耐受性[14]。近年来,长度大于200 bp的非编码RNA(lncRNA)已渐渐成为动植物的重要调节因子[15]。例如,长链非编码RNA MuLnc1可以通过产生siRNAs沉默类钙调蛋白基因(CML27)的表达,参与桑树的耐盐分和干旱胁迫[16]。在拟南芥中,定位于细胞核的干旱诱导lncRNA(DRIR)是植物响应干旱和盐胁迫的正向调节因子[17]。拟南芥中的Npc536通过增加初生根和次生根响应盐胁迫[18]。【本研究切入点】然而,青稞中盐碱抗性相关的lncRNA尚未见报道。全转录组适合同时研究mRNA和lncRNA及其相关性。为进一步阐明青稞基因在盐碱胁迫中的作用,本文对2个不同盐碱耐受青稞品种盐碱胁迫2,8,24,48和72 h的响应基因和lncRNAs进行了研究。高抗盐碱品种中特异表达的差异基因的功能富集分析表明,它们与“防御反应”、“压力反应”、“离子转运”和“膜”相关。此外,通过lncRNA顺式和反式靶基因预测构建了lncRNA与mRNA之间的调控网络。【拟解决的关键问题】该研究有助于进一步了解青稞盐碱耐受性的机制,提高青稞对盐碱胁迫的耐受性。
1 材料与方法
1.1 试验材料
选取青稞耐盐碱的0119和对盐碱敏感的0226作为实验材料[19]。材料均由西藏自治区农牧科学院农业研究所提供。
1.2 试验方法
1.2.1 材料盐碱处理 用次氯酸钠溶液将种子灭菌15 min,用无菌水冲洗,最后在25 ℃黑暗、相对湿度60%的培养箱中放置2 d,使其发芽。幼苗在22/20 ℃(白天/夜晚)、14/10 h(亮/暗)光周期的温室中生长3周后,转移至带有蛭石栓塞的器皿中。用含150 mmol/L NaHCO3的Hoagland营养液浇灌盐碱胁迫处理组的幼苗。用Hoagland营养液浇灌对照组幼苗。
1.2.2 总RNA提取和lncRNA文库的构建 取盐碱胁迫处理2、8、24、48和72 h和相应对照组植株的新鲜叶片,使用Trizol试剂(Invitrogen,卡尔斯巴德,加利福尼亚州,美国)从中提取总RNA。并将总RNA中的rRNA去除来富集mRNA和非编码RNA。加入片段化缓冲液,将mRNA和非编码RNA片段化,通过琼脂糖凝胶电泳纯化,并通过PCR扩增进行富集。使用HiSeq 2000(Illumina)平台对所有文库(2个品种×3个生物学重复×2种处理×5个时间点)分别进行测序,产生长度为150 bp的双末端测序数据。每个时间点的不同处理取3个生物学重复。
1.2.3 转录组数据的比对和组装 使用fastqc(0.11.4)和fastp[20](0.12.5)对过滤后数据进行质量控制。首先,使用HISAT2[21](2.1.0)将过滤后的RNA-seq数据分别与青稞的参考基因组进行比对[2],参数设置为“置--no-mixed --no-discordant --no-”。然后使用stringtie[21](1.3.3b)进行转录本组装,设置参数进行转录本组装,以对低覆盖度的数据进行过滤。
1.2.4 LncRNA鉴定 使用cuffcompare[22](2.2.1)将所有样品组装得到的转录本与参考注释文件中已知的mRNA转录本进行比较[2]。保留可能是非编码的class_code为i,j,o,u和x的转录本,过滤掉含单个外显子、低reads覆盖度以及短于200 bp的转录本。然后使用gffread(参数为参数为e)[23]将剩余的转录本转换为fasta格式,并使用编码潜能计算工具(CPC 0.9)[23]对这些转录本进行编码潜能评估,其中使用UniRef100蛋白质数构建BLAST数据库。最终,CPC得分<0的所有转录本均被视为鉴定出的lncRNA。
1.2.5 差异表达和功能富集分析 将参考mRNA注释文件[2]与预测的lncRNA合并成一个gtf文件,使用HTSeq (参数为“参数为“能富集分析定出的、低di)[24]分别统计bam文件中比对到每个转录本的reads数目。使用DESeq2[25](1.10.1)R包进行差异表达分析和原始reads数目的归一化;若满足padj <0.05且|log2FoldChange |> 1,则被认为是差异表达的转录本。使用TopGO[26](2.22.0)R包进行GO富集分析。
1.2.6 通过qRT-PCR验证RNA-seq数据 采用9700(美国应用生物系统公司)PCR系统对RNA进行逆转录(RT),合成cDNA。采用LightCycler®480使荧光实时定量PCR仪(Roche,Swiss)进行PCR反应,将装有反应液的384孔光学平板(Roche,Swiss)放入仪器中,反应条件为:95 ℃孵育5 min,然后进行95 ℃持续10 s和60 ℃持续30 s的40个循环。每个样品设置3个生物学重复。使用Primer Premier 5.0(http://www.premierbiosoft.com)设计引物,并由Invitrogen(美国卡尔斯巴德)公司合成。将基因的表达水平与GAPDH(参照)比较,用2-照)Ct方法计算其表达量[27]。
1.2.7 lncRNA靶基因预测 为鉴定反向靶基因,用R中的cor函数计算所有mRNA和lncRNA组合之间的表达相关系数,相关系数大于0.9的lncRNA-mRNA组合则被定义为是共表达的。用cytoscape将mRNA-lncRNA构成的共表达网络进行可视化[28](3.6.0版)。为了鉴定顺式靶基因,将位于lncRNA上游和下游5 kb内的蛋白质编码基因作为lncRNA的靶基因。
2 结果与分析
2.1 2个青稞品种对盐碱胁迫的应答表型
胁迫48 h以前,0119和0226品种的叶片颜色差异不大(图1),胁迫72 h后,0226品种的黄色叶片数量明显多于0119品种。0119品种较0226品种具有更好的盐碱耐受性,符合实验预期。
2.2 青稞中lncRNA的全基因组鉴定
所有样品过滤后的reads共比对到27 641个基因,占总基因数的76.5%,总体平均比对率为62.02%。将转录组数据组装结果与已知注释数据进行比较,共鉴定出76 749种可能的新转录本。
用固定的流程鉴定青稞中的lncRNA,共鉴定出6704个可靠的lncRNA,其CPC得分<0。j,i,o,u和x类型的lncRNA分别代表新型、内含子、正义、基因间和反义的lncRNA,数量分别为1104、655、826、2741和1378个。其中,基因间长链非编码转录本(lincRNA)和反义长链非编码转录本是lncRNA的两大主要类型。
将lncRNA的特征与已知的mRNA进行比较。青稞LncRNA的外显子数量少于mRNA(图2-A,平均分别为2.54和4.41个),但是lncRNA的外显子长度(图2-B,平均404个核苷酸)长于mRNA(平均245个核苷酸)。青稞全长lncRNA转录本(平均长度为1026个核苷酸)略短于mRNA转录本(平均长度为1081个核苷酸,图2-C)。
2.3 盐碱胁迫应答基因的鉴定
为了鉴定应答盐碱胁迫的基因,通过比较所有时间点处理样品和对照样品的差异获得差异表达基因(DEG)。
在0119品种中,有463个(320个上调和143个下调,表1)、7277个(2589个上调和4688个下调)、2579个(1611个上调和968个下调)、7133个(2873个上调和4260个下调)和8622个(3877个上调和4745个下调)mRNA分别在2 、8、24、48和72 h差异表达。在0226品种中,有3587个(1844个上调和1743个下调)、4795个(1943个上调和2852个下调)、1699个(1003个上调和696个下调)、8456个(3905个上调和4551个下调)和6403个(3091个上调和3312个下调)mRNA分别在2 、8 、24 、48 和72 h差异表达(图3-A)。此外,在0119和0226品种中分别有76和233个mRNA在5个时间点都上调,14和56 个mRNAs在5个时间点都下调。

表1 盐碱胁迫下差异表达的mRNAs
在0119品种中,有78个(66个上调和12个下调,表2)、805个(299个上调和506个下调)、 336个(208个上调和128个下调)、883个(333个上调和550个下调)和1214个(645个上调和569个下调)lncRNA分别在2、8、24、48和72 h差异表达。在0226品种中,有403个(244个上调和159个下调)、504个(261个上调和243个下调)、176个(114个上调和62个下调)、947个(509个上调和438个下调)和734个(354个上调和380个下调)lncRNA分别在2,8,24,48和72 h差异表达(图3-B)。此外,在0119和0226品种中分别有8和26个lncRNA 在5个时间点都上调,1和5个lncRNA在5个时间点都下调。
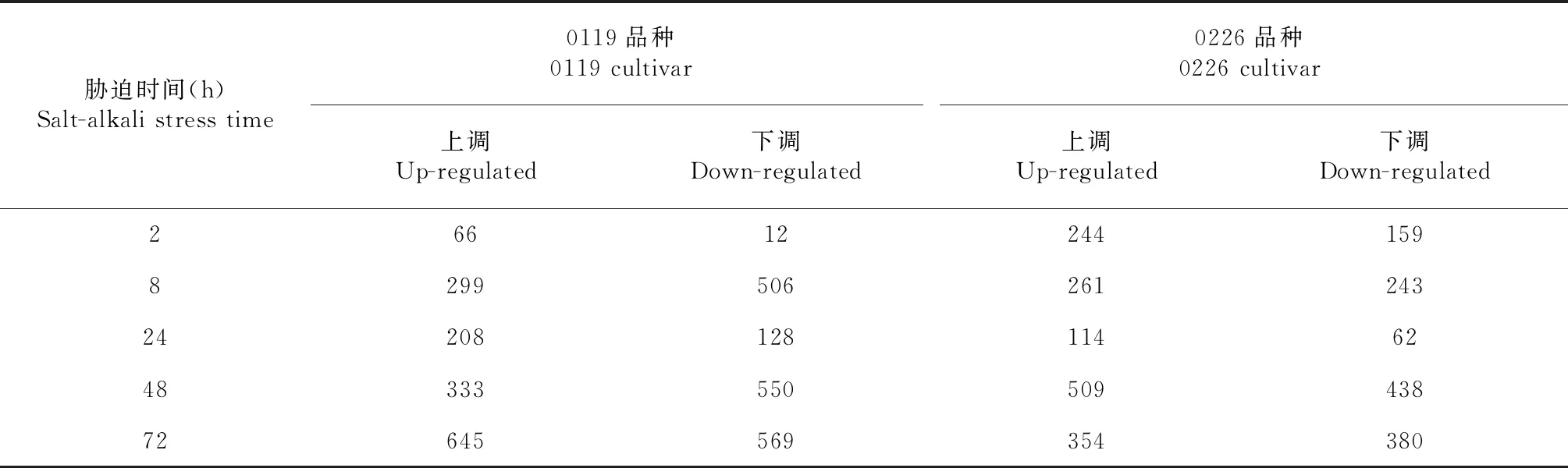
表2 盐碱胁迫下差异表达的lncRNAs
随着胁迫时间的增加,不同基因参与了胁迫应答(图3-C和3-D)。在2 h时,0226品种中差异表达基因的数量(3587个mRNA,403个lncRNA)明显高于0119品种中差异表达基因的数量(463个mRNA,78个lncRNA)。因此,0226品种能比0119品种更快、更强的应答盐碱胁迫。2个品种的DEGs数量(包括mRNA和lncRNAs)均在24 h减少。DEGs数量随时间变化的趋势说明盐碱胁迫应答包含两个阶段。第一阶段为渗透期,本质上是由于细胞外部的盐分而引起的水响应;第二阶段是离子毒性阶段,由于盐分的积累而产生[29]。24 h可能是两个阶段的分界点。
2.4 2个青稞品种中盐碱应答基因的比较分析
为了研究盐碱耐受性的机制,对0119和0226品种胁迫应答的差异基因进行了比较(图4-A和4-B),上调或下调表示在盐碱胁迫下在一个或多个时间点上调或下调表达的基因。其中,盐碱胁迫下73.75%在0119中上调的mRNA在0226中也上调,71.30%在0119中下调的mRNA在0226中也下调。在0119品种(耐盐碱)中,有1303个mRNA特异上调表达, 1815个mRNA特异下调表达。盐碱胁迫下58.22%在0119中上调的lncRNAs在0226中也上调,50.82%在0119下调的lncRNAs在0226也下调。在0119品种(耐盐碱)中,有346个lncRNA特异上调表达,346个lncRNA特异下调表达。在2个青稞品种中,超过一半的盐碱应答基因是共有的。
进而对0119品种中3118个特异的DEG进行GO富集分析和KEGG分析。基因ID和GO条目的映射关系来自已发表的数据[22]。特异富集的生物学过程,包括“防卫反应”、“压力反应”、“刺激反应”、“离子运输”、“金属离子运输”和“阴离子运输”;富集的分子功能,包括“催化活性”和“蛋白激酶活性”;富集的细胞组分主要与“膜”相关(图5-A)。对其进行KEGG分析发现5个主要通路:“植物-病原体相互作用”、“植物激素信号转导”、“泛素系统”、“复制和修复”和“ABA响应元件转录因子”。其中,脱落酸(ABA)是高盐和干旱胁迫的关键调节因子[30]。
从图4-C和4-D可知,0119品种中特异上调或下调的mRNA和lncRNA的log2 Fold Change值。胁迫2 h时,0119品种中差异表达基因较少,这些基因在胁迫8,24,48和72 h时的差异表达具有一定的时间特异性。功能富集分析表明它们可能与盐碱耐受性有关。差异表达模式在8 和48 h以及在24 和72 h相似。
2.5 0119品种中特异DEG的功能注释
0119品种耐盐碱胁迫,而0226品种对盐碱胁迫敏感。在0119品种中,有3118个特异差异表达的mRNA和798个特异差异表达的lncRNA,它们可能与盐碱耐受性有关。根据SwissProt数据库,对3118个mRNA的功能进行注释(表4),对798个lncRNA的靶基因的功能进行注释(表5)。其中,HVUL7H37356.2(8 和48 h上调)被注释为NHX6,属于Na+/ H+反转运蛋白(NHXs),在Na+和K+稳定和pH调节中起重要作用[32]。 HVUL5H41848.2(8 h上调)被注释为HKT9,HKT转运蛋白[高K(+)亲和力转运蛋白]介导Na(+)-K(+)共转运或Na(+)特异性转运[32]。HVUL3H14791.2(72 h时上调)和HVUL2H11056.2(42 和72 h时下调)被注释为HAK1,HVUL2H07884.2(TCONS_00057912的顺式靶基因)、HVUL2H11056.2(TCONS_00073 142的顺式靶基因))和HVUL4H58544.2(TCONS_00134336和TCONS_00150546的顺式靶基因)被注释为HAK家族,而HAK(高K+亲和力)转运蛋白主要通过促进K+转运提高植物的耐盐性[33]。其次, HVUL7H33659.2(8 h时上调)、HVUL2H09286.2(8 h时下调)和HVUL6H08720.2(48 h时下调)被注释为APX家族,抗坏血酸过氧化物酶(APXs)可以保护植物免受氧化损伤[34]。HVUL6H20868.2(TC ONS_00225173的顺式靶基因)被注释为CAT2,过氧化氢酶(CAT)是一种高活性酶,可在光呼吸过程中去除过氧化氢[35]。HVUL5H36534.2(8 h下调)、HVUL3H06057.2(8 h下调)、HVUL6H19324.2(8 h下调)和HVUL7H42896.2(48 h下调)被注释为盐耐受性蛋白。还有几个应对各种胁迫的重要转录因子(TF)家族,如AP2 / ERF(APETALA2 /乙烯响应因子)、WRKY(属于锌指蛋白)、NAC、MYB、bHLH(基本螺旋-环-螺旋)和bZIP(碱性域亮氨酸拉链)蛋白[36-37]。总之,0119品种中特异DEG(mRNAs)在转录因子家族中的分布如下:AP2 / ERF(3个上调基因,1个下调基因)、WRKY(5个上调基因,9个下调基因)、NAC(7个上调基因,10个下调基因)、MYB(8个上调基因,8个下调基因)、bHLH(2个上调基因,15个下调基因)、bZIP( 1个基因)和其他锌指蛋白(14个上调基因, 17个下调基因)。这些TFs也可能与0119品种的耐盐碱性有关。
2.6 盐碱应答lncRNA的靶基因预测
lncRNA可以顺式作用于其转录位点附近,或反式作用于独立的基因座[38]。例如,X染色体失活的特异性转录本(XIST)起顺式作用,通过诱导抑制性异染色质的形成,这是启动X染色体失活所必需的[39];HOX反义转录RNA(HOTAIR)反式抑制位于不同染色体上的HOXD簇[40]。
0119和0226品种在5个时间点差异表达的lncRNA有2255个。通过2种策略预测这2255个lncRNA的靶基因。顺式靶基因预测获得2639个mRNA-lncRNA对,包括1685个lncRNA和2191个mRNA。反式靶基因预测获得25 383个mRNA-lncRNA对,包括1193个lncRNA和5798个mRNA。这些mRNA中的291个与GO:0055114(氧化还原过程)相关(图6)。此外,有 711个mRNA-lncRNA对在两种策略中重叠。
对0119品种中特异的差异表达lncRNA的靶基因进行了GO富集分析和KEGG路径富集分析。GO富集的生物过程包括“防御反应”、“光合作用”和“压力反应”,0119品种优异的光合作用系统可能是其抗盐碱性的关键因素;富集的分子功能包括“激酶活性”和“转移酶活性”;细胞组分主要与“膜”相关(图5-B)。此外,富集的KEGG通路包括“植物-病原体相互作用”、“翻译”、“植物激素信号转导”和“苯丙烷生物合成”。
2.7 基因qRT-PCR验证
为了验证RNA-seq数据计算基因表达量的可靠性,利用qRT-PCR随机挑选6个mRNA和4个lncRNA在盐碱胁迫8、24、48 h的0119品种中进行验证。6个mRNA的qRT-PCR结果和RNA-seq结果相对一致(图7),并且具有相似的表达模式。
4个lncRNA在选定3个时间点的qRT-PCR结果和RNA-seq结果也一致(图8)。说明RNA-seq数据获得的mRNA和lncRNA的表达模式是可靠的。
3 讨 论
植物为适应盐度、寒冷、高温和辐射等各种非生物胁迫,进化出了复杂的防御机制。近年来,许多研究表明lncRNA、蛋白质编码基因和miRNA参与植物应答非生物胁迫。胁迫诱导基因的研究不仅有助于了解胁迫耐受的分子机制,而且有助于利用基因工程培育胁迫耐受性品种[41]。近年来,随着测序技术的快速发展,新一代代测序(NGS)技术已广泛应用于各个领域[42]。本研究中,使用RNA-seq技术全面分析了青稞盐碱胁迫相关基因。
植物的盐胁迫包含两个不同的阶段。在渗透阶段,气孔通过缩小孔径的大小来平衡水分胁迫,最终导致光合作用速率降低,进而导致活性氧(ROS)积累[43]。因此,植物必须产生足够的氧化还原酶,如过氧化物酶(POD)、超氧化物歧化酶(SOD)、过氧化氢酶(CAT)和抗坏血酸过氧化物酶(APX)来应对氧毒害[44]。
从60个0119和0226品种样品中鉴定出6704个lncRNA。此外,耐盐碱的0119品种中分别有3118和798个盐碱胁迫差异表达mRNA和lncRNA,其功能富集包括“离子转运”、“植物激素信号转导”、和“ABA响应元件结合因子”,表明它们可能与青稞的盐碱抗性有关。NHX(HVUL7H37356.2)、HKT(HVUL5H41848.2)和HAK(HVUL3H14791.2)基因家族虽然可以将Na+从质膜中排出或泵入液泡中,但重要的维持离子稳定的盐过度敏感(SOS)信号传导通路基因[46]没在本文的DEG中。另外,2个品种中差异基因的数量在胁迫24 h减少,并且2个品种中一半以上的差异基因是共有的。耐盐性通常由数十种生理机制和数十万个基因调节。 HAK,APX家族的一些基因和可能的耐盐性蛋白或特异TF在0119品种中表现出相反的差异表达趋势。盐碱抗性相关的调控机制需要通过敲除或过表达实验进行深入研究。
某些lncRNA可通过顺式作用调控邻近基因,而某些lncRNA可通过反式作用远程调控基因表达。预测了lncRNA的顺式和反式靶基因,并根据相关的编码基因间接预测这些lncRNA的功能,但并未通过直接捕获与RNA相关的染色质的CHART[46]或ChARP[47]技术预测靶基因。另外,lncRNA如何找到靶基因以及哪些蛋白质参与该过程均不清楚。远程三维染色体结构技术可以帮助寻找lncRNA的反式靶基因[48]。 lncRNA在远程调控基因表达中的作用可以通过染色体构象捕获(3C)[49]和其染色质相互作用的衍生方法进行进一步研究。
lncRNA的序列包含潜在的转录因子(TF)结合位点,并且TF与lncRNA之间的相互作用可能调节基因的表达。在拟南芥中,光敏色素相互作用因子(PIF)胁迫相关的TF可能在强光下正向和负向调节lncRNA的表达[50]。 lncRNA也可能与TF相互作用,促进靶基因的表达[51]。 RNA蛋白质相互作用的RNA免疫沉淀(RIP)[52]、交联和免疫沉淀(CLIP)[53]方法可用于研究与lncRNA结合的TF。从另一个角度看,RNA-RNA相互作用也有助于lncRNA功能的研究,如补骨脂素对RNA相互作用和结构的分析(PARIS)[54],连接相互作用的RNA的高通量测序(LIGR-seq)[55]和补骨脂素交联、连接和选择杂交(SPLASH)[56]的技术。
lincRNA和miRNA在调节防御相关基因表达的过程中存在复杂的交互作用。在毛果杨(Populustrichocarpa)中,一些lincRNA被认为是miRNA的靶基因而发挥功能[57]。同样,lncRNA被鉴定为应答番茄病毒感染的miRNA的靶基因[58]。但是,本文中并未对lncRNA作为miRNA的靶基因进行研究。因此,需要整合更多的相关数据,进一步分析lncRNA功能。
