SeqType® P52人类祖先识别SNP检测试剂盒性能验证
2021-08-20王颖希马原李甫焦章平
王颖希,马原,李甫,焦章平
1.北京市公安司法鉴定中心,北京 100192;2.北京市朝阳区公安司法鉴定中心,北京 100123;3.北京市东城区公安司法鉴定中心,北京100061
祖先信息标记(ancestry informative marker,AIM)是指不同人群之间基因频率差异非常大的多态性基因位点[1]。而单核苷酸多态性(single nucleotide poly⁃morphisms,SNP)在人类基因组中广泛分布,是分析人群遗传信息、筛选AIM 位点的重要遗传标记之一[2]。国内外的法庭科学工作者已在此领域做了大量工作,并应用于部分案件[3]。ENOCH 等[3]认为35~45 个AIM位点可以区分2~3 个洲际群体,绝大部分已报道的AIM 组合可以区分3~8 个不同地区人群,且多数以洲际间人群区分为主,如PHILLIPS 等[1]报道的34 个SNP位点和李彩霞等[4]报道的30 个SNP 位点均主要用于区分亚洲、欧洲和非洲人群。
为同时实现大区域划分和来源细化,并尽量精简AIM 个数,可先采用洲际SNP 大概判定人员来源的大方向,然后再结合使用分辨率更高、更有针对性的遗传标记进行区域细化。如PHILLIPS 等[5]采用由23 个SNP 组成的具有欧洲、南亚区分能力的Eurasiaplex 试剂盒,结合FONDEVILA 等[6]已报道的34-plex 对来自欧洲、中东、南亚、东亚的人群区分效率进行了验证。LI等[7]报道了可以更加有效区分东亚人群的74个SNP,NIEVERGELT 等[8]报道了区分非洲、中东、欧洲、中/南亚、东亚、美洲、大洋洲人群的41 个AIM。
本研究使用的祖先信息SNP 位点组合,涵盖可以区分亚、欧、非人群的SNP 位点15 个,区分亚洲各部(东亚、北亚、南亚、东南亚、中南亚、中东)的SNP 位点19 个,区分汉族、藏族人群的SNP 位点16 个,区分汉族、蒙古族人群的SNP 位点2 个。通过对未知样本的检测,以获得亚欧非-亚洲-汉蒙藏的溯源信息。
1 材料与方法
1.1 材料
中国汉族、藏族、蒙古族、维吾尔族、彝族人员血样均为日常案件收集,每个民族各70 人。血样采集均在知情同意的情况下进行,签署知情同意书,符合伦理学相关规范,同时符合《法庭科学DNA 数据库人员样本采集规范》(GA/T 1380—2018)。血样采集后使用血样采集专用卡(北京达博创新科技开发有限公司)制备成350 份干血片保存。阳性对照为2800M标准DNA(质量浓度10 ng/μL,美国Promega 公司)。
1.2 方法
1.2.1 DNA提取
血卡使用WD-超微量磁珠法DNA 提取试剂盒(长春市博坤生物科技有限公司)在全自动96 道微量DNA 提取工作站(长春市博坤生物科技有限公司)上进行DNA 提取,经Qubit®3.0 荧光定量仪(美国Thermo Fisher Scientific 公司)定量质量浓度为0.1~0.4 ng/μL。
1.2.2 SNP文库制备
采用SeqType®P52 人类祖先识别SNP 检测试剂盒(北京爱普益生物科技有限公司)进行文库制备。上述位点的入选依据,主要参考不同人群的等位基因频率差(值),洲际间种群值≥0.7,亚洲各部值≥0.5,汉族、藏族人群值≥0.4。
文库制备基于以直接扩增法文库构建技术为核心的SeqType®系统[9],即采用带有接头、样本标签、引物的融合引物进行扩增,聚合酶链反应(polymerase chain reaction,PCR)扩增直接完成SNP 文库制备。
PCR包括DNA聚合酶0.3 μL,2.5×PCR缓冲液4 μL(含dNTPs),融合引物混合液1.0 μL,0.1~0.4 ng/L DNA 模板2 μL,双蒸水补足至10 μL。PCR 扩增程序:95 ℃5 min;95 ℃30 s,60 ℃30 s,72 ℃1 min,22 个循环;72 ℃10 min。
将350 份样本的PCR 产物分成10 组混合液进行纯化,每组混合液含35 份样本的扩增产物。每份混合液分别取50 μL 进行磁珠纯化,纯化产物经Qubit®3.0 荧光定量仪定量后,每次纯化产物分别取0.4 ng 测序,总共使用10 个芯片测序。
1.2.3 高通量测序
采 用Ion Chef 系 统、Ion TorrentTMIon S5TMXL系统和Ion 520TM芯片(美国Thermo Fisher Scientific公司)进行高通量测序,操作参照Ion TorrentTMIon 510TM&Ion 520TM&Ion 530TMKit-Chef 试剂盒(美国Thermo Fisher Scientific 公司)说明书进行。
1.2.4 数据分析
采用SeqVision V1.5 软件[9-10](北京爱普益生物科技有限公司)对测序结果进行分析,分析流程包括长度质量控制、样本归类、基因座归类、基因分型,通过等位基因频率差(值)评价位点区分效果。
群体聚类采用Structure2.3.4(http://web.stanford.edu/group/pritchardlab/structure.html)进行。以数字编码替代样本已知族群分类和基因分型完成数据准备,项目建立后填写样本数量、基因座的数量等基本信息,按照实际情况设置K值、预设族群数后运行distructversion 1.1(https://web.stanford.edu/group/rosen berglab/distruct.html)完成族群聚类簇。
主成分分析(pricipal component analysis,PCA)采用python 软件包(https://pypi.tuna.tsinghua.edu.cn/simple/sklearn/)进行。主成分个数设置为3 个,通过数据训练模型得到主成分变量,同时获取每个主成分的贡献度,并在二维坐标系中绘制相应的散点图。
2 结果
2.1 SNP位点选取
如表1 所示,SeqType®P52 人类祖先识别SNP 检测试剂盒涵盖可以区分亚、欧、非人群的SNP 位点15 个(P1~P15),区分亚洲各部(东亚、北亚、南亚、东南亚、中南亚、中东)的SNP 位点19 个(P16~P35),区分汉族、藏族人群的SNP 位点16 个(P36~P50),区分汉族、蒙古族人群的SNP 位点2 个(P51、P52)。

表1 SeqType® P52人类祖先识别SNP检测试剂盒位点信息Tab.1 Locus information of SeqType® P52 Human Ancestry Identification SNP Detection Kit
2.2 测序数据过滤
测序总条数为1.8×106~6.1×106条,分析后获得1.7×106~4.9×106条有效比中序列,数据比中率71%,单个样本单个位点有效测序深度≥720×(表2),平均检出率96%。
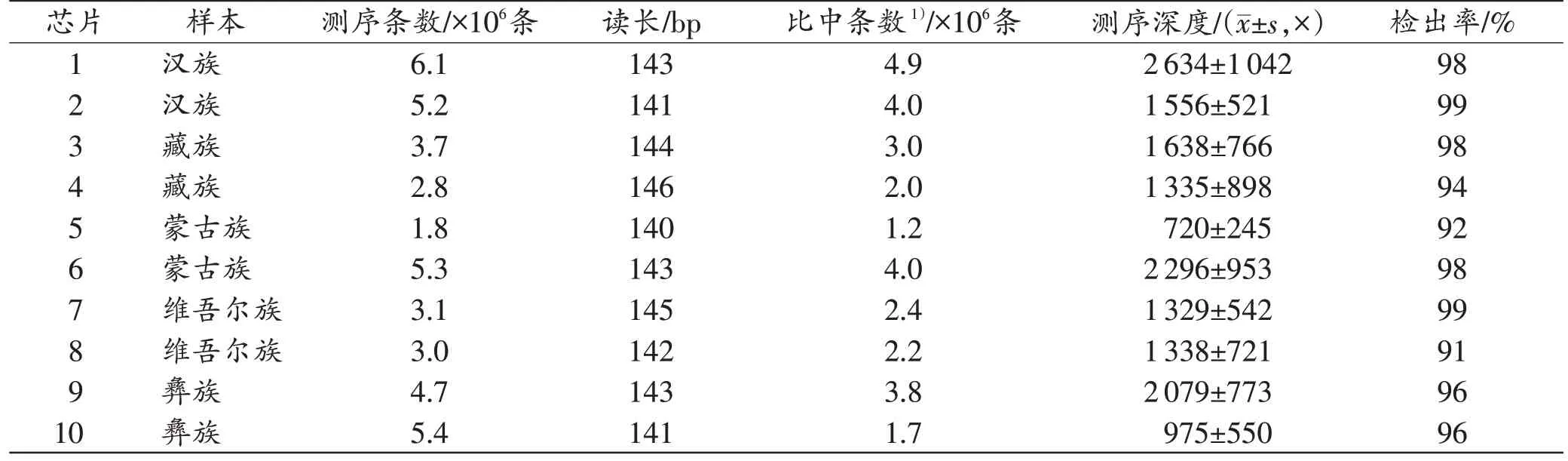
表2 测序及数据分析整体情况Tab.2 Overview of sequencing and data analysis (n=35)
2.3 体系构建与样本检验结果
以汉族、藏族、蒙古族、维吾尔族、彝族各70 份样本,对SeqType®P52 人类祖先识别SNP 检测试剂盒进行验证。结果显示,SeqType®P52 人类祖先识别SNP 检测试剂盒体系可以有效区分汉族、藏族、维吾尔族人群,其中藏族、维吾尔族特异性SNP 均为17 个。以等位基因1 的分布频率作为等位基因频率计算依据,统计5 个民族的等位基因频率(表3)。以汉族为对照,分别计算藏族、蒙古族、维吾尔族、彝族4 个少数民族和汉族的群体间等位基因频率差。结果显示,藏族、蒙古族、维吾尔族、彝族4 个民族和汉族群体的等位基因频率差均值分别为0.20、0.05、0.24和0.11,蒙古族、彝族和汉族群体差异较小,藏族、维吾尔族和汉族差异最大(附表2)。

表3 各民族等位基因频率统计Tab.3 Summary of allele frequencies in each population
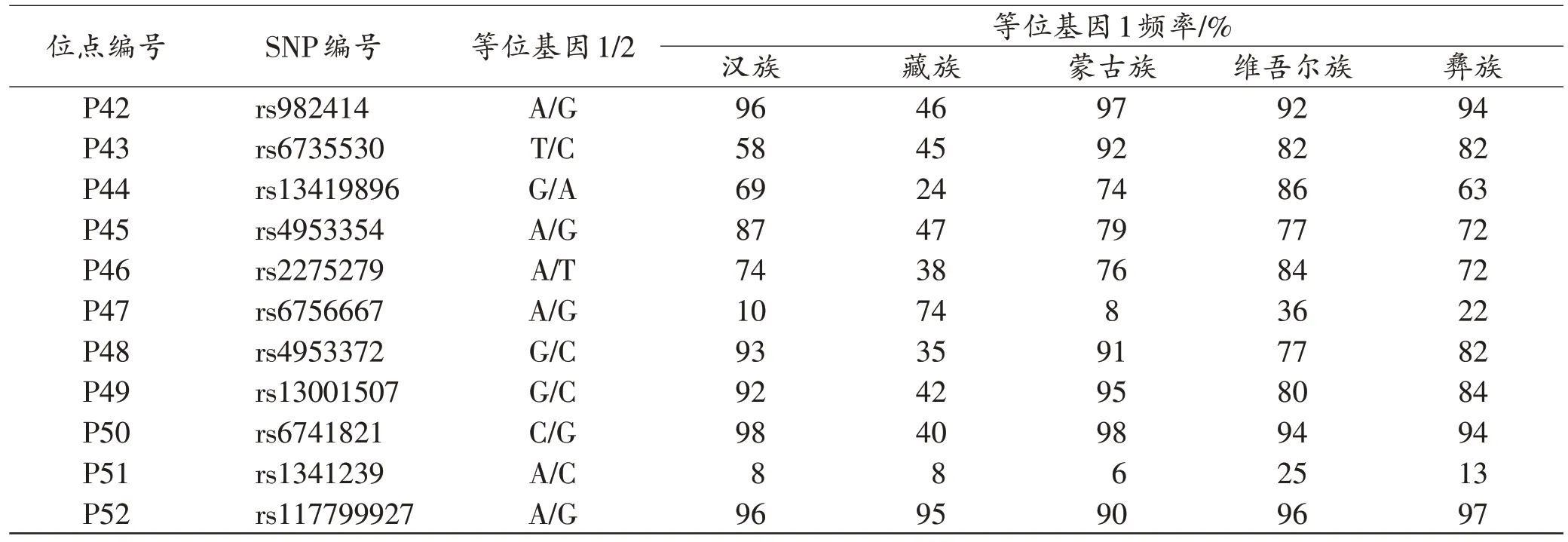
续表3Continued Tab.3
从差异位点的分布来看,17个SNP位点在藏族、汉族人群的值≥0.3(附表2),其中rs1800414、rs1229984为北亚、东亚之间差异位点,rs3811801 为东南亚、东亚之间差异位点[1],其余14 个位点均为前人[11,14-16]报道的藏族群体特异的耐高海拔相关SNP。维吾尔族、汉族人群值≥0.3 的SNP 位点17 个(附表2),其中7 个为WEI 等[11]已报道的欧洲和东亚之间差异SNP,10 个为区分亚洲各部的SNP,包括4 个东亚、北亚之间差异位点,1 个东亚、东南亚之间差异位点和3 个东亚、中-南亚之间差异位点,2 个东亚、南亚之间差异位点。相比之下,分别只有1个和3个SNP位点在蒙古族和汉族,彝族与汉族人群的值≥0.3。其中rs6735530为耐高海拔相关SNP[11],以等位基因1 频率为准,rs6735530在蒙古族、维吾尔族、彝族人群的频率,均高于汉族。
利用Structure2.3.4 在参数K=5 模式下对5 个人群全部样本进行聚类分析。结果显示,藏族和维吾尔族均有比较独立的祖先成分。对于彝族而言,其2/3拥有相对独立的与藏族人群接近的祖先成分,1/3 成分和维吾尔族相似。蒙古族拥有与汉族相似的祖先来源成分。
由此推断,SeqType®P52 人类祖先识别SNP 检测试剂盒可以较为准确地区分汉族、藏族、维吾尔族群体。对这3 个群体进行PCA 分析(图2)显示,3 个人群可在二维图上清晰区分,汉族、维吾尔族边界几乎没有交叉,汉族、藏族和维吾尔族、藏族群体边缘有少量交汇。

图1 汉族、藏族、蒙古族、维吾尔族、彝族人群聚类Fig.1 Clusters of Han,Tibetan,Mongolian,Uygur and Yi population
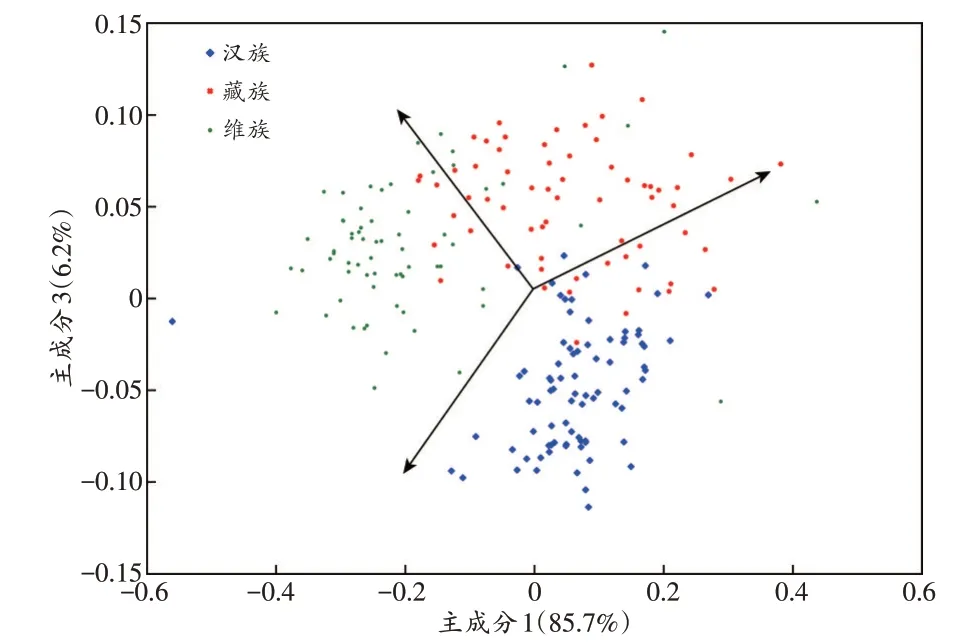
图2 汉族、藏族、维吾尔族人群的主成分分析二维点状图Fig.2 PCA 2D point graph of Han,Tibetan and Uygur population
3 讨论
中华民族是多元文化、多民族统一体。藏族是由历史上的吐蕃直接发展而来的(www.China.org),吐蕃是青藏高原上土生土长的一个民族,青藏高原地势高、气候寒冷,不适于发展农耕业,在长期的发展中很少有其他民族迁入,因此藏族是相对隔离的群体。本研究所采用的SeqType®P52 人类祖先识别SNP 检测试剂盒可以有效区分中国汉族、藏族人群遗传结构成分,藏族特异性SNP 也主要来自藏族群体特异的耐高海拔相关SNP[12-15]。
表型差异的相关功能基因可以作为祖先信息SNP 位点筛选的基础,李彩霞等[4]报道的30 个用于区分东亚、欧洲和非洲人群的AIM,来自30 个与肤色、瞳孔颜色、黑色素代谢等表型相关基因。SeqType®P52人类祖先识别SNP 检测试剂盒中的17 个维吾尔族和汉族特异SNP 位点中有7 个来自WEI 等[11]报道的27 个SNP,其余10 个主要来自北亚,中-南亚和南亚特异的亚洲地域相关SNP[6-8]。这与新疆地区古代居民主要表现出欧罗巴人种和蒙古人种以及上述两大人种混合类型的体质特征和基因类型相吻合[17]。
本研究样本聚类结果显示,彝族拥有相对独立的祖先成分,同时又和藏族人群比较接近。这与黎彦才等[18]提出的藏彝走廊类型(甘肃南部到云南西部、南部生活的藏缅语系少数民族)结论一致。
全基因组范围的比较筛查是目前为止AIM 获得的最有效方法,但从筛选频率来看,即使相对隔离的藏族群体和汉族群体的祖先SNP 筛选频率仍低至0.000 1%,90 万个SNP 仅有不足100 个SNP 符合Fst≥30%的筛选标准[15]。成吉思汗统一蒙古各部后的迁徙和融合,使得区分汉族、蒙古民族的AIM 的筛选尤为困难,SeqType®P52 人类祖先识别SNP 检测试剂盒中的遗传标记也无法对汉族、蒙古族人群进行有效区分。
