微生物修复被化学除草剂乙草胺污染的环境研究进展
2021-08-10徐安明董维亮
王 彤,许 斌,徐安明,周 杰,董维亮
(南京工业大学 生物与制药工程学院,江苏 南京 211800)
乙草胺(2-乙基-6-甲基-N-(乙氧基甲基)-2-氯代乙酰替苯胺)是由美国孟山都公司于1971年开发的一类氯乙酰苯胺类除草剂,其分子结构主要由苯环和氯乙酰氨基团组成。乙草胺是一种长链脂肪酸抑制剂,通过抑制焦磷酸环化酶的活性来抑制杂草幼苗的生长,可用于杂草芽前防治,例如一年生禾本科杂草(马唐、稗草等)和一年生阔叶杂草(野苋等),对多年生杂草无效[1]。由于除草活性高、应用范围广且持效期适中,乙草胺在我国应用面积不断扩大,自1977年以来的年使用量超过1万t,是我国生产量和使用量最多的除草剂之一[2]。
乙草胺的化学结构稳定,在农业应用过程中只有少量发挥除草活性,而绝大部分残留于植物表面或土壤中,随着雨水迁移到地下水、地表水和河流等生态环境中。数据调查显示,松花江流域沉积物中乙草胺含量可达0.47~11.76 μg/kg,其河岸土壤中含量高达0.03~709.37 μg/kg[3];淮河流域沉积物中乙草胺含量高达3 900~6 600 μg/kg[4]。进一步研究表明,暴露在环境中的乙草胺可诱导多种细胞生理毒性进而影响生物体健康,包括对生殖、内分泌和心血管系统的毒性效应[4],并且其与炎症因子IL-1β直接结合后可形成免疫毒性[5]。除此之外,最近研究表明乙草胺是一种强致癌物和神经毒素,通过评估乙草胺对斑马鱼胚胎细胞的神经毒性以及对幼虫运动行为和基因表达的影响,表明其在神经发育和神经传导过程中也具有一定毒性[4, 6]。目前,研究人员在鲤鱼、白鲢和虹鳟等水产品中已经发现乙草胺残留[7],这表明乙草胺可以通过生物链的富集作用进入生物体内,最终有可能威胁人类健康。因此,对环境中残留的乙草胺进行有效清除对于保护生态平衡、保障人类健康都至关重要。
近年来,利用环境微生物来清除化学农药残留因具有效率高、成本低、生态效益高等优点,逐渐成为环境污染物治理最具有发展潜力的修复方法,也是当前环境科学领域的研究热点。本文综述了国内外报道的乙草胺高效降解微生物资源和微生物分解代谢机制的最新研究进展,并对乙草胺等化学农药污染环境微生物修复技术的未来研究方向与应用屏障进行讨论和展望。
1 乙草胺高效降解微生物资源
丰富的微生物资源是开展乙草胺降解机制研究并实现污染环境生物修复的基础。在早期研究中,一些能利用乙草胺作为唯一碳源生长的土著原生菌群是主要的降解性微生物资源。例如,Xu等[8]从乙草胺污染土壤中富集了4种高效降解乙草胺的微生物菌群,其中菌群D可以在9 d内降解80 mg/L的乙草胺,降解效率达到了99%。近年来,国内外研究人员从污染土壤和活性污泥中筛选及鉴定出大量具备乙草胺降解能力的纯培养微生物资源,包括Pseudomonas、Rhodococcus、Bacillus和Sphingomonas等多个菌属,并在实验室的模拟环境中验证了它们对乙草胺的降解性能(表1)[9-26]。

表1 乙草胺生物降解的微生物资源
然而,真实污染环境中的乙草胺生物降解面临更复杂、更苛刻的降解条件,这对降解菌株的环境耐受性提出了更高要求。王苑力等[24]从乙草胺污染的农田土壤中分离出一株耐盐的乙草胺降解菌Bacillussp.ACD-9,在3 d内对初始质量浓度为50 mg/L的乙草胺降解率可达56.8%,并能够在温度4~50 ℃、pH 4.5~10.0、NaCl质量浓度为0~150 g/L的条件下生长。此外,构建分工协作的多细胞体系从而减轻单细胞体系的代谢压力也是另一种有效的方法。Duc等[25]筛选的菌株PseudomonasfluorescensKT3能将乙草胺转化为2-甲基-6-乙基苯胺(MEA),并且MEA进一步被微生物Bacillussubtilis2M6E降解及完全矿化;Hou等[27]以乙草胺为唯一碳源构建了含有Rhodococcussp. T3-1、Delftiasp. T3-6和Sphingobiumsp.MEA3-1这3种微生物的多细胞降解体系,在6 d内能够降解100 mg/L的乙草胺,降解效率达到了96%。
2 乙草胺生物降解途径解析
有机污染物的微生物降解途径是评价污染物环境迁移行为、降解菌株生物修复应用潜力的重要参考指标。目前,已报道的乙草胺微生物降解途径主要有两种:1)好氧脱烷基途径;2)厌氧脱氯途径。
2.1 好氧脱烷基途径
乙草胺的好氧脱烷基生物降解途径已经相对明确。首先,乙草胺经过N-脱烷基反应被转化为2-氯-N-(2-乙基-6-甲基苯)乙酰胺(CMEPA),CMEPA进一步发生酰胺水解反应转化为MEA。在此基础上,MEA能够通过自发水解和羟基化反应形成2-甲基-3-羟基-6-乙基对苯二酚(3-OH-MEHQ),并随着苯环的开环反应进入三羧酸循环(TCA),最终被降解为CO2和H2O。Cheng等[28]以双细胞体系探索了乙草胺的好氧降解途径,基于农药厂污泥中分离出的菌株Sphingomonassp.DC-6,能够将乙草胺转化为CMEPA和水解产物苯胺衍生物MEA,进一步,MEA在菌株SphingobiumbaderiDE-13的作用下最终实现乙草胺完全矿化。Hou等[27]构建了三细胞体系来解析乙草胺的好氧代谢途径,由于菌株Rhodococcussp. T3-1和Delftiasp. T3-6将乙草胺降解为MEA后不能深入降解,在此基础上,他们进一步筛选了菌株Sphingobiumsp.MEA3-1,它能够将降解中间体MEA转化为3-OH-MEHQ,最终通过邻苯二酚开环途径完全降解为CO2和H2O(图1)。
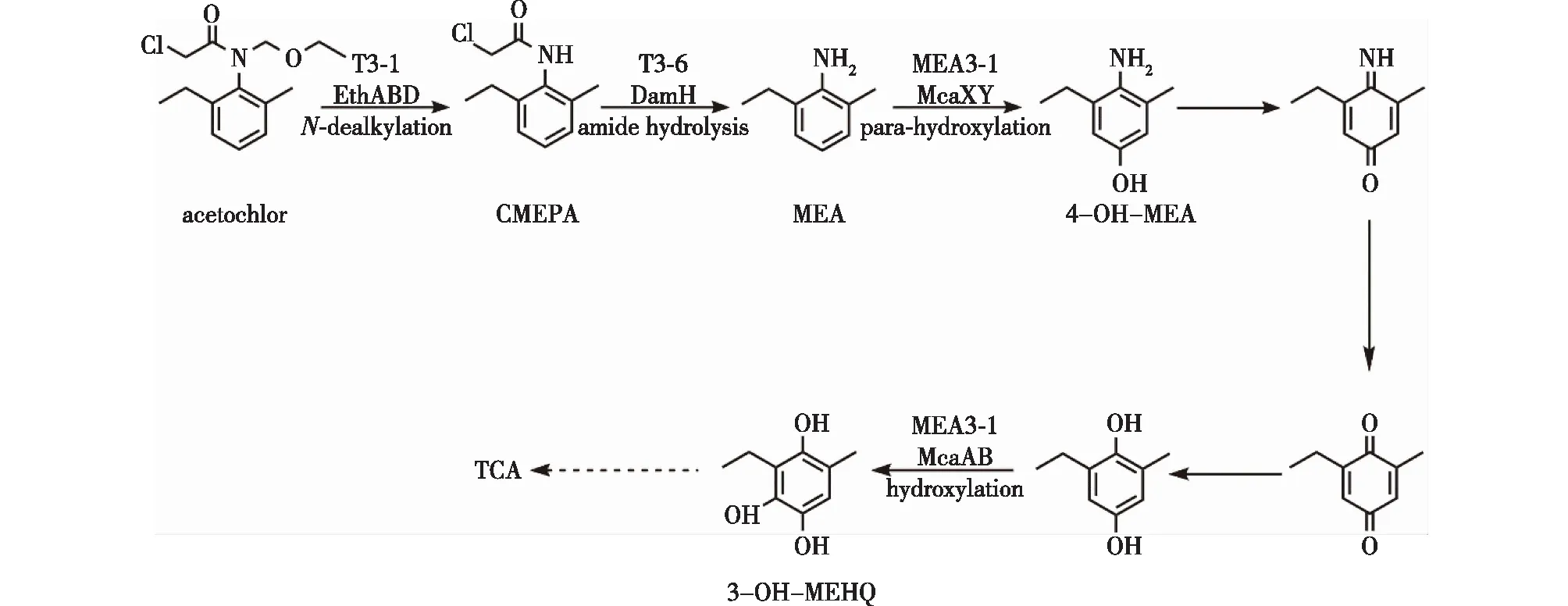
图1 乙草胺好氧脱烷基微生物降解途径[27]Fig.1 Aerobic dealkylation pathway of acetochlor[27]
2.2 厌氧脱氯途径
目前,针对乙草胺厌氧脱氯降解途径的研究相对较少。与乙草胺好氧脱烷基途径起始反应不同,厌氧途径的初始反应为脱氯。首先,乙草胺通过脱除氯乙酰基中的氯原子能够形成2-乙基-6-甲基-N-(乙氧基甲基)-乙酰苯胺(EMEMA),并进一步通过乙氧基甲基的去除形成N-(2-甲基-6-乙基苯基)乙酰胺(MEPA)。所得中间产物MEPA则可以通过芳香环甲基的去除及乙酰基的甲基化而被转化为N-2-乙基苯基甲酰胺(EPF),并进一步通过甲酰基的羟基化生成2-乙基-N-羧基苯胺(ECA)(图2)。Liu等[29]通过在活性污泥中外源添加乙草胺进行压力驯化,获得了具有乙草胺降解效果的厌氧污泥;其中,Sporomusa、Sporobacterium、Dechloromonas、Azotobacter和Methanobacterium属的细菌丰度显著增加,表明这些菌属可能参与乙草胺的厌氧生物降解过程。然而,乙草胺厌氧脱氯途径中的关键催化酶目前还未被挖掘和鉴定,这也有待于国内外研究人员在后续的研究中进行完善。

图2 乙草胺厌氧脱氯微生物降解途径Fig.2 Anaerobic dechlorination pathway of acetochlor
3 乙草胺降解的生化机制
脱烷基反应是乙草胺好氧微生物降解的起始反应,N-脱烷基酶和C-脱烷基酶能够催化乙草胺的脱烷基反应。Chen等[30]鉴定了一种来源于SphingomonadsDC-6和DC-2中催化乙草胺N-脱烷基化反应的三组分Rieske非血红素铁加氧酶(RHO)体系,由同(源)寡聚体加氧酶、[2Fe-2S]铁氧化还原蛋白、谷胱甘肽型还原酶3种成分组成。Gao等[31]从菌株Bacillussp. Hys-1中克隆了一个编码脱羧酶的Debutoxylase基因(Dbo),该基因编码的C-脱烷基酶Dbo可催化乙草胺发生C-脱烷基反应。除此之外,N-乙氧基甲基化酶也能够催化乙草胺的N-乙氧基甲基化反应生成CMEPA。Wang等[32]采用硫酸铵沉淀法和疏水相互作用色谱法,从菌株Rhodococcussp. T3-1中纯化了属于细胞色素P450体系的N-乙氧基甲基化酶,并证明其为一种可催化乙草胺转化为CMEPA的三组分酶,该团队进一步在大肠杆菌中成功异源表达了ethBAD基因,验证其编码的酶具有N-乙氧基甲基化活性。
酰胺水解反应可使CMEPA水解形成MEA。Li等[17]通过将菌株SphingobiumquisquiliarumDC-2的基因文库转化到大肠杆菌中,获得了CMEPA酰胺水解酶基因cmeH,并克隆表达了cmeH基因,由cmeH编码的酰胺水解酶CmeH能催化CMEPA水解为MEA。Wang等[33]采用硫酸铵沉淀法、二乙氨乙基阴离子交换法、疏水作用色谱法和Sephadex G-200凝胶过滤等蛋白质纯化方法,从Delftiasp. T3-6中纯化出一种高活性的酰胺水解酶DamH,并克隆其编码基因于大肠杆菌中进行表达,结果表明:DamH是一种具有酰胺键和酯键活性的双功能水解酶,其酰胺水解酶功能的比酶活达到5 036 U/mg,可高效将底物CMEPA水解为MEA,是乙草胺微生物降解过程中第二个关键酶。
MEA将经过一系列苯环羟化过程转化为3-OH-MEAQ,最终通过邻苯二酚降解途径完全矿化。Dong等[34]通过比较基因组学的方法挖掘及鉴定了参与MEA降解过程的关键基因meaA和meaB,对应编码的加氧酶MeaA和还原酶MeaB能够构成新型黄素单加氧酶体系,催化MEHQ羟基化反应,将其转化为3-OH-MEHQ;并且,他们进一步推测,MEA首先通过P450单加氧酶作用转化为4-羟基-2-甲基-6-乙基苯胺(4-OH-MEA),然后进一步自发水解脱氨为2-甲基-6-乙基苯胺(MEHQ)。Cheng等[28]通过分析比较MEA降解菌株SphingobiumbaderiDE-13与其MEA降解缺陷突变体的基因组时发现,关键基因meaXY作为一种双组分单氧酶系统,能够利用NADH和黄素单核苷酸作为辅助因子催化MEA的羟基化,将MEA转化为4-OH-MEA。
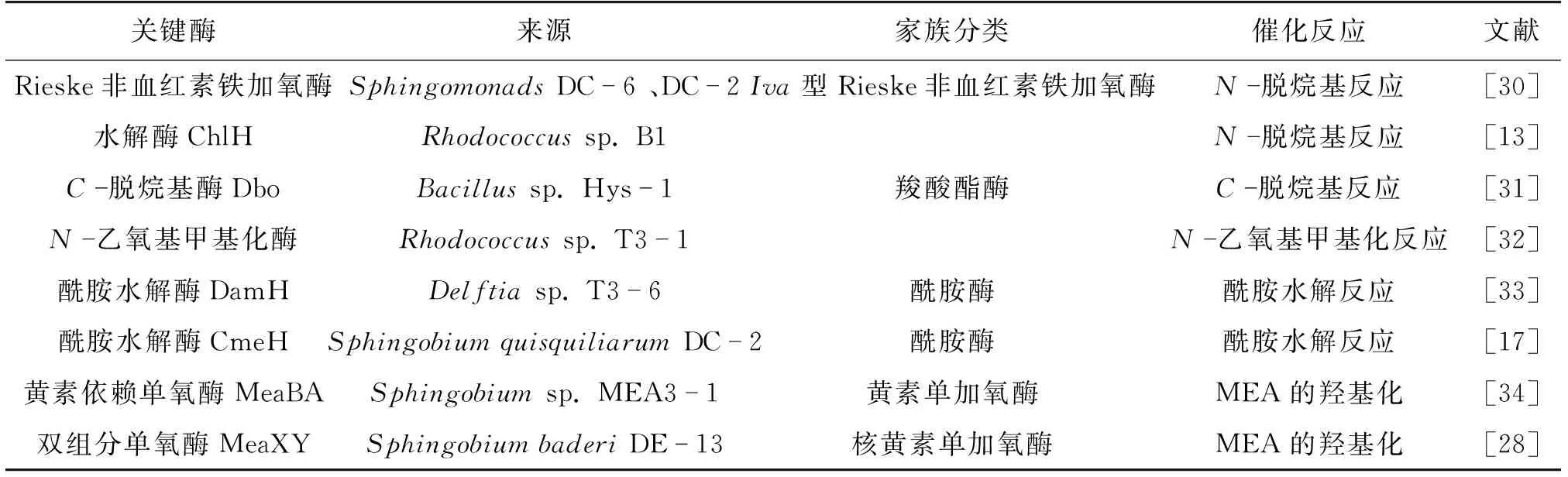
表2 乙草胺微生物降解过程关键酶
4 乙草胺污染环境的生物修复
鉴于乙草胺严重的环境毒性和生态威胁,开发合理、高效的乙草胺污染环境修复技术已经刻不容缓。目前,最具应用潜力的乙草胺污染环境生物修复方式有微生物修复和植物修复2种。
基于乙草胺高效降解微生物资源以及相对清晰的乙草胺微生物降解机制,发展乙草胺等化学农药污染环境的微生物修复技术是研究热点(图3)。李强[35]利用盆钵实验证明菌株Sphingomonassp.或DC-6可有效解除1.0 mg/kg及更高浓度的残留乙草胺对玉米的药害,并使乙草胺污染土壤的微生物群落结构维持平衡。张宁[36]从长期施用乙草胺的花生连作障碍田花生根瘤中,筛选出1株乙草胺高效降解菌Rhizobiumsp. ZL-27,在2.37 mg/kg乙草胺的盆栽土壤中培养40 d后,成功清除了27%的乙草胺。尽管这些已研究的乙草胺污染环境的微生物修复技术效率有待提高,但在模拟环境中的成功修复为乙草胺微生物修复的未来实际应用提供了宝贵的经验和指导。
图3 微生物修复乙草胺污染环境Fig.3 Microbial remediation of environmental pollution by acetochlor
利用常见植物对乙草胺等化学农药进行植物修复是另一种有效的生物修复手段。乙草胺的植物修复本质是将可实现乙草胺催化降解的关键基因元件导入植物中,构建具有乙草胺降解能力的转基因植物,从而实现对于乙草胺污染土壤的植物修复(图4)。在植物细胞中引入谷胱甘肽合成酶,提高谷胱甘肽的产量,能够让所构建的转基因植物具备乙草胺等化学农药的同化能力。Gullner等[37]通过在植物中表达γ-谷胱甘肽合成酶,发现这些转基因植物在乙草胺存在的情况下生产情况显著强于野生型植物。Chu等[38]将SphingomonaswittichiiDC-6中编码乙草胺N-脱烷基酶系统的cndA基因导入拟南芥中,并分别在细胞质和叶绿体中实现了活性表达,结果表明:当cndA定位于叶绿体时,拟南芥对乙草胺具有较强的耐受性,并且在48 h内可将水中20 μmol/L乙草胺中的94.3%转化为无毒物质;在30 d内可清除含5 mg/kg乙草胺土壤中的80.2%。这一研究结果对于乙草胺真实污染环境中植物修复的实际应用具有重要参考价值。

图4 植物修复乙草胺污染土壤Fig.4 Phytoremediation of acetochlor contamination
5 总结与展望
综上所述,随着我国乙草胺的生产量和使用量不断增加,具有稳定结构的乙草胺大量残留于土壤、水体等生态环境中,严重影响生态平衡甚至人类健康。近年来,针对乙草胺不合理使用导致的环境问题及健康问题,国内外研究人员围绕乙草胺微生物降解技术开展了大量研究工作。从早期的复杂混菌体系到纯菌体系,再到分工合作的人工多细胞体系,研究人员筛选出大量具备乙草胺高效降解及矿化能力的微生物资源,解析了乙草胺的微生物降解路径并挖掘出与降解相关的关键酶元件,为真实污染环境中乙草胺微生物修复技术的应用打下了坚实的基础。
目前,许多具有优异乙草胺降解能力的菌株已经被广泛报道,然而其降解功能评价大多停留在实验室模拟体系研究阶段。由于真实的污染环境中存在许多易模拟因素(例如,水分、pH、氧化还原电势、可溶氧和土著微生物等),对微生物活性具有重要影响,多数降解菌株的生物修复效率往往并不理想,甚至无效。因此,对于真实污染环境的生物修复过程,降解微生物的生存动态、与土著微生物的相互作用以及真实复杂环境的影响因素都需要被深入评价。
从自然环境中筛选出的野生型乙草胺降解菌株普遍存在降解效率低、环境适应能力差、降解能力不稳定等缺点,因此,如何提升真实污染环境下微生物对于乙草胺的降解及矿化效率是突破乙草胺生物修复应用技术瓶颈的关键。近年来,随着合成生物学的不断发展,基于代谢路径改造、定向进化等分子生物学手段对菌株进行改造为乙草胺生物修复效率的提升提供了新的思路。针对高效降解菌株的缺乏,利用液滴微流控、水凝胶制备、荧光探针设计等技术构建乙草胺降解微生物的高通量筛选平台,能够大大提升目标微生物的筛选效率;对于已筛选出的乙草胺降解菌株,利用细胞融合及代谢路径改造等技术,通过基因克隆、基因突变、基因组编辑等策略,能够调控乙草胺降解微生物的代谢路径,扩大菌株对污染物种类的降解范围,获得遗传稳定、环境适应能力强的乙草胺高效降解模式菌株。
由于真实修复环境与微生物体内环境间存在巨大差异,所以在实际生物修复应用中的乙草胺降解酶具有催化效率低、稳定差等不足。针对关键降解路径中酶催化效率低这一关键瓶颈,利用定向进化、理性设计、化学修饰等技术对酶进行改造,能够实现酶催化效率和稳定性的显著增强。酶的定向进化主要包括易错PCR、DNA改组、随机引发重组等技术。酶的理性设计主要基于酶与底物的相互作用,理性选择氨基酸残基为靶点对酶的关键氨基酸进行突变,改变底物结合能力,实现酶对污染物降解能力的强化。基于化学反应,以功能分子对酶进行化学修饰也是提升酶催化活性及稳定性的重要方式。这些酶改造技术的不断进步,也为真实污染环境下乙草胺等环境污染物生物修复的实际应用提供了更多的选择。
与此同时,对乙草胺降解途径解析后发现,乙草胺完全降解及矿化的实现往往是多种微生物协同合作、劳动分工的结果。当微生物菌株以菌群的形式发挥降解作用时,微生物菌群中不同微生物各有分工,才能够完成复杂的工作;而且,微生物菌群中微生物往往处于动态平衡状态,对复杂环境具有更强的适应能力;除此之外,劳动分工有利于减轻纯培养微生物的代谢负荷,保证微生物菌群的正常运转。然而,通过简单的混合微生物菌株构建混菌体系并不能实现菌株之间分工合作效率的最大化,因此,如何构建高效、可控的人工多细胞体系实现多细胞之间的分工合作优化是以后研究的重点。
近年来,随着新型材料制备技术的发展,通过静电纺丝、3D生物打印等技术能够实现微生物细胞的固定化,也为环境污染物降解人工多细胞体系的构建提供了技术支持。基于新型功能材料构建的细胞固定化能够实现目标微生物的接种比例、空间分布、外部环境调控,从而满足环境污染物降解人工多细胞体系的构建需求。此外,微生物固定化材料能够为降解微生物提供温和的微环境,将降解微生物与复杂的外部环境隔绝开来,对于提升降解微生物的环境耐受性及降解效率、拓展真实污染环境中的乙草胺生物修复具有重要意义。
