北极地区萨氏真蛇尾(Ophiura sarsii)群体遗传多样性研究
2021-08-09史文阁李一璇NAZAROVASophia范士亮徐勤增
史文阁,李一璇,NAZAROVA Sophia A.,赵 磊,郭 健,范士亮,徐勤增*
(1.自然资源部 第一海洋研究所,
自然资源部海洋生态环境科学与技术重点实验室,山东 青岛266061;2.青岛海洋科学与技术试点国家实验室 海洋生态与环境科学功能实验室,山东 青岛266237;3.俄罗斯科学院动物研究所 海洋研究室,圣彼得堡199034;4.贵州医科大学 生物与工程学院,贵州 贵阳550025)
萨氏真蛇尾(Ophiura sarsii)属于蛇尾纲(Ophiuroidea)真蛇尾目(Ophiurina)真蛇尾科(Ophiuridae)真蛇尾属(Ophiura),主要分布于北太平洋、北极各边缘海和北大西洋海域[1],其最南端分布海域为日本海(36°N)。有关萨氏真蛇尾的研究通常随大型底栖生物调查而展开。Toshihiko等[2]对日本海萨氏真蛇尾体长频率、空间分布进行了调查,发现大个体数量随着水深的增加而增加。Anisimova[3]对巴伦支海南部的萨氏真蛇尾群体结构进行了分析。Steffens等[4]发现拉普捷夫海萨氏真蛇尾主要分布在水深30 m以内的海域。Ravelo[5]对楚科奇海东北部萨氏真蛇尾的年龄组成、空间分布和次级生产力等种群特征进行了详细描述。Li等[6]基于线粒体COI和核基因标记ITS2片段分析了萨氏真蛇尾及其亚种浅水萨氏真蛇尾(Ophiurasarsii vadicola)的遗传特征。目前,对北极萨氏真蛇尾群体的遗传特征认识较少,有待深入开展研究。
线粒体控制区(Control Region,CR)具有相对其他分子标记突变较快的特点,是脊椎动物群体遗传研究中常用的分子标记,但在其他动物中开展的研究较少。Omr等[7]发现棘皮动物控制区序列有突变速率快且受环境选择小的特点,是研究棘皮动物物种内部遗传特征的理想分子标记。申欣等[8]研究发现,相对于其他基因,以蛇尾为代表的棘皮动物线粒体NADH脱氢酶亚基(nad)基因具有较多的变异位点,因而nad系列基因适合作为棘皮动物系统发育分析的分子标记。
北极太平洋扇区白令海-楚科奇海域地质构造复杂,处于亚洲、北美洲和太平洋与北冰洋的交汇处,历史上白令海峡出现了多次开启与关闭的地质变化事件[9]。此外,该区域的洋流分布复杂,主要有阿拉斯加海岸流、西伯利亚海岸流和白令海峡洋流等,使得该海域拥有比较特殊的物种迁移与基因交流现象[10]。本研究基于不同方式获得了白令海、楚科奇海和巴伦支海三个海域萨氏真蛇尾样品,选取线粒体控制区,nad2和nad6三种分子标记,研究3个区域大型底栖生物优势种——萨氏真蛇尾的群体遗传与连通性,并检测在北极太平洋及大西洋扇区是否存在基因交流以及相关程度,讨论不同分子标记在该种类群体进化研究中的适用性,同时揭示北极海域3个地理群体间萨氏真蛇尾的群体遗传特征,为其迁移提供科学依据。
1 材料与方法
1.1 样本采集
萨氏真蛇尾来自第十次北极考察获得的40个白令海(BL)、白令海峡至楚科奇海(R)站位海域样本(图1),以及俄罗斯科学院动物研究所提供的19个巴伦支海(BAS)样本。通过阿氏网将白令海、楚科奇海蛇尾样品在所处海域分两站进行采集(BL09、BL12、R01和R04),采集到的一部分样本使用乙醇固定,于-20℃保存;另一部分样品用低温海水清洗,使用液氮超低温暂存,返回到实验室后转移至超低温冰箱(-80℃),长期保存。

图1 第10次北极考察萨氏真蛇尾采样白令海、楚科奇海与巴伦支海采样站位Fig.1 Sampling sites of Ophiura sarsii from Bering Sea Chukchi Sea and Barents Sea
1.2 DNA提取、扩增与测序
取长约1 cm的萨氏真蛇尾腕部组织,使用两种试剂盒:QIAamp Fast DNA Tissue试剂盒(QIAGEN公司,德国)和OMEGA Tissue DNA试剂盒(OMEGA公司,美国)用于基因组DNA提取。萨氏真蛇尾腕部组织经液氮研磨后进行消化,其余提取步骤参照说明书。将提取后的总基因组DNA进行琼脂糖凝胶电泳,检测DNA完整性,检测合格的DNA置于-20℃保存。将59个个体进行线粒体控制区、nad2和nad6序列扩增。
根据GenBank中萨氏真蛇尾线粒体基因组全序列(GenBank编号:MH780492),采用Oligo 7.0软件设计用于扩增线粒体控制区、nad2和nad6区域的引物(表1)。引物由上海生工生物工程技术有限公司合成。PCR反应体系共50μL,包含双蒸水30μL、10×PCR buffer(without Mg2+,Takara)5μL、MgCl2(25 mm,Takara)4μL、d NTP Mix(2.5 mm,Takara)4μL、每种引物(浓度10μM)2μL、牛血清蛋白溶液(BSA)0.75 μL、rTaq酶(Takara)0.25μL和模板DNA 2μL。CR的扩增程序为95℃预变性3 min;95℃变性30 s,48℃退火30 s,72℃延伸1 min,35个循环;72℃延伸10 min,7℃保存。nad2的扩增程序为95℃预变性3 min;95℃变性30 s,48℃退火30 s,72℃延伸1 min,35个循环;72℃延伸10 min,7℃保存。nad6的扩增程序为95℃预变性3 min;95℃变性30 s,50℃退火30 s,72℃延伸1 min,35个循环;72℃延伸10 min,7℃保存。

表1 扩增线粒体控制区,nad2和nad6分子标记的PCR引物序列Table 1 The PCR primers for amplication of CR,nad 2 and nad 6 sequences
将PCR扩增产物置于1%琼脂糖凝胶中进行电泳检测,结果显示线粒体控制区、nad2和nad6检测合格的条带数分别为59、21和31条,检测合格条带清晰的PCR产物送至青岛生工生物工程技术有限公司切胶纯化,并利用自动测序仪进行双向测序,测序引物即PCR扩增引物。
1.3 数据处理
使用Dnastar Lasergene v7.1软件[11]对测序结果进行拼接。利用DnaSP 6.12.03软件[12]分别计算线粒体控制区、nad2和nad6基因的分离位点数目(Number of Segregating,S)、单倍型数量(Number of Haplotype,H)、核酸差异数(Nucleotide Differences,K)、核苷酸多样性(Nucleotide Diversity,PI)、单倍型多样性(Haplotype Diversity,HD)以及群体间的基因交流值。将上述分析获得的3个分子标记的单倍型序列分别构建系统发生树,使用近缘种Ophiura lutkeni(GenBank编号:AY184223.1)的线粒体控制区、nad2和nad6序列作为外群,通过PhyloSuite v1.2.1软件[13]中的MAFFT模块[14]进行序列比对,G-blocks软件[15]获得对齐的线粒体控制区、nad2和nad6数据集。使用IQTREE软件[16]进行最大似然法(Maximum-Likelihood,ML)系统发育分析,设置20 000次快速自展分析(ultrafast booststrap)。Figtree软件(http:∥tree.bio.ed.ac.uk/software/figtree/)进行进化树美化。通过中介邻接法(Median-Joining)构建单倍型网络图。Popart 4.8.4软件[17]进行单倍型的谱系结构分析,由于BL海域样品较多,将BL09与BL12在单倍型网络图上做了区分。
使用Arlequin ver 3.5.2.2软件[18]计算群体间的遗传分化指数(Fst)和分子方差分析(Analysis of Molecular Variances,AMOVA),检验单倍型在群体间分布频率差异,AMOVA分群将BL09与BL12定为一组,R与BAS海域各定为一组。利用Tajima'sD和Fu'sFs中性检验推测萨氏真蛇尾的群体历史动态。以上分析均通过20 000次重复抽样检验其显著性。
2 结 果
2.1 序列特征及遗传多样性
线粒体控制区序列长度为322 bp,A、T、G和C含量分别为35.4%、30.4%、14.3%和19.9%,A+T含量(65.84%)高于G+C含量(34.16%),其中A含量最高,碱基组成没有明显的偏向性。在59条序列中共检测到15个单倍型,分离位点数差异为0~8。3个地理群体间线粒体控制区单倍型多样性相差较大,其中巴伦支海海域单倍型多样性最高(0.731±0.066),白令海峡向北至楚科奇海域单倍型多样性最低(0.000±0.000);巴伦支海核苷酸多样性最高(0.005 58±0.000 063),白令海峡向北至楚科奇海域海域核苷酸多样性最低。
nad2序列长度为598 bp,A、T、G和C含量分别为30.9%、34.4%、17.1%和17.6%,G+C含量为34.62%,其中T含量最高,碱基偏向性不明显。在3个地理群体32条序列中,共检测到27个单倍型,且各群体单倍型均具有较高多样性,巴伦支海萨氏真蛇尾单倍型最高(1.000±0.250),白令海萨氏真蛇尾核苷酸多样性(0.009 33±0.006 80)与平均核酸差异数(5.457)最高。
nad6序列长度为449 bp,A、T、G和C含量分别为45.7%、18.5%、12.0%和23.8%,G+C含量为35.86%,其中A含量最高。在3个地理群体31个序列中,共检测到24个单倍型。3个地理群体的单倍型多样性均较高,接近1;核苷酸多样性相近,其中白令海域最低(0.009 02±0.005 59),巴伦支海最高(0.026 85±0.00028)(表2)。
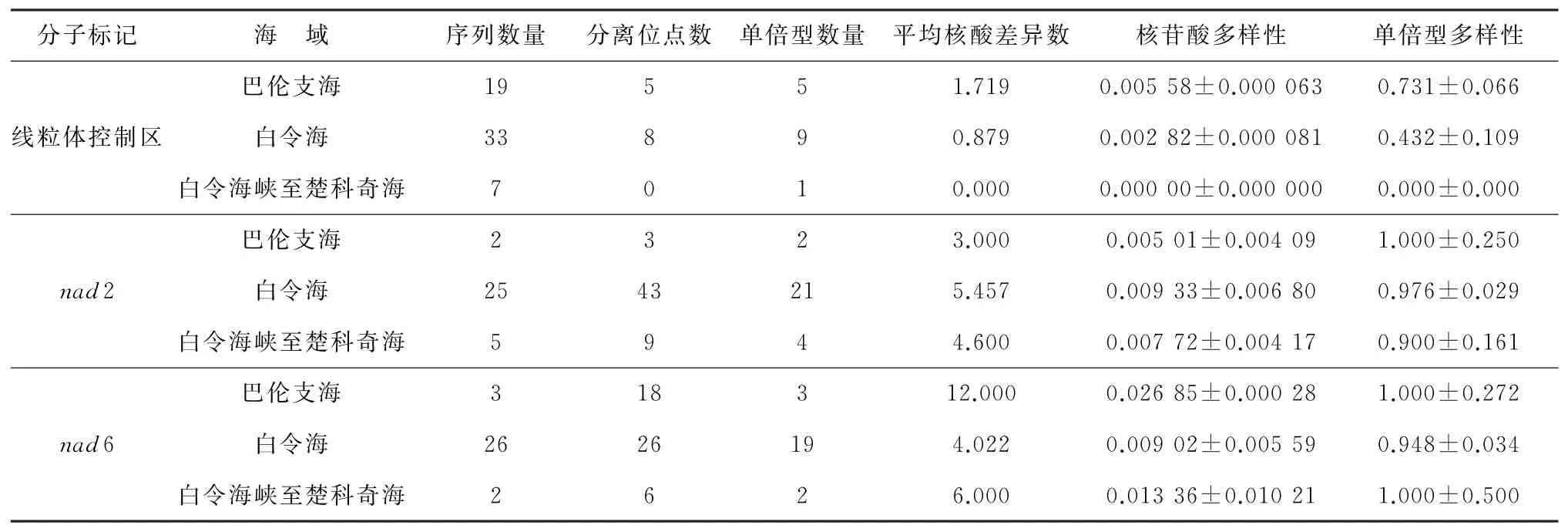
表2 萨氏真蛇尾线粒体控制区、nad 2、nad 6标记的遗传多样性参数Table 2 Genetic diversity parameters of Ophiura sarsii based on CR,nad 2 and nad 6 markers
在所用的3个分子标记中,线粒体控制区的扩增效果最好,但nad2与nad6分离位点数、单倍型数量与核苷酸多样性均高于控制区,且揭示的单倍型多样性也明显高于控制区片段。
2.2 系统发生
基于最大似然法构建的线粒体控制区单倍型系统发生树(图2a),形成显著分化的2个支系:巴仑支海群体样本明显聚在一起,另外3个群体交叉分布于系统发生树中。线粒体控制区结果显示,北极巴伦支海海域与白令海、楚科奇海之间的萨氏真蛇尾存在显著的遗传分化,而白令海与楚科奇海之间遗传分化不明显。线粒体控制区单倍型网络图(图3a)显示,3个海域萨氏真蛇尾群体间的分群结果与系统发生树结果基本一致,巴伦支海海域聚为L2谱系,另外2个海域相互交叉形成L1谱系。
基于nad2和nad6构建的系统发生树(图2b和图2c)与控制区树结构一致,巴伦支海海域聚为一支,剩余群体交叉分布。nad2和nad6单倍型图(图3b和图3c)展示的群体结构与相应系统发生树结构相近,也分为L1和L2两个谱系。

图2 基于控制区、nad 2和nad 6序列构建的ML树Fig.2 ML phylogenetic tree based on CR,nad 2 and nad 6 sequences

图3 基于控制区、nad 2和nad 6序列构建的萨氏真蛇尾群体单倍型网络图Fig.3 Haplotype network of Ophiura sarsii populations based on CR,nad 2 and nad 6 sequences
2.3 群体遗传结构
使用分化固定指数(Fst)评估3个群体间分化程度(表3),巴伦支海海域与另外2个海域间Fst指数较大(0.744 87~0.944 38),而白令海与楚科奇海海域间Fst指数均较低,且呈现负值(-0.070 53~-0.009 09),P值均不显著(P为0.324 32~0.747 75,P>0.05)。由此说明,巴伦支海萨氏真蛇尾与白令海和楚科奇海萨氏真蛇尾存在较为显著的分化。白令海和楚科奇海之间Fst指数为负值,表明这2个海域间萨氏真蛇尾可能有较强的遗传同质性。AMOVA结果显示3个海域控制区变异主要来源于群体间(表3),且P值呈极显著的结果(P<0.01)。

表3 基于线粒体控制区、nad 2和nad 6序列的萨氏真蛇尾群体间遗传分化系数F st及相应P值Table 3 F st and P-value from haplotype frequencies of Ophiura sarsii based on CR,nad 2 and nad 6 sequences
2.4 群体历史动态
3个标记的中性检验结果(表4)表明白令海海域的Tajima'sD与Fu'sFs值呈现负值,但统计检验不显著,巴伦支海与楚科奇海海域中性检验呈现正值或等于0,结合错配分布呈现多峰分布的结果,说明3个海域之间可能不存在群体扩张事件。

表4 基于线粒体控制区、nad 2和nad 6序列的萨氏真蛇尾群体AMOVA分析结果Table 4 AMOVA analysis of Ophiura sarsii based on CR,nad 2 and nad 6 sequences

表5 基于线粒体控制区、nad 2和nad 6序列的萨氏真蛇尾群体中性检验值Table 5 Neutral test value of Opsiura sarsii based on CR,nad 2 and nad 6 sequences
3 讨 论
遗传多样性的高低决定物种对环境变化的响应能力,因此它是物种生存进化的前提,是研究区域物种遗传多样性的基础。单倍型多样性和核苷酸多样性是遗传多样性的重要指标[19],本研究使用线粒体控制区、nad2和nad6三个标记分析了巴伦支海、楚科奇海与白令海三个海域萨氏真蛇尾群体遗传结构及遗传谱系分化特征。萨氏真蛇尾是北极大型底栖生物优势种[1],生物量高,本研究中3个海域的单倍型多样性与核苷酸多样性都较高,表明该种类在北极海域有较高的遗传多样性。
海洋环流和环境适应性等因素会影响海洋生物分布及群体遗传结构[20]。底栖生物尤其是其幼虫在冷水环境中能够比最适环境条件下迁移更长距离[21],北极的冷水环境更适合萨氏真蛇尾幼体迁移,并且北极底栖生物分布有高度的可变性[22]。北极地区特别是白令海峡附近有复杂的洋流环境[23],可促进萨氏真蛇尾的基因交流,这也与Ershova等[24]关于白令海与楚科奇海萨氏真蛇尾生物群体呈分散状分布的研究结果一致。此外,食物也是影响生物分布和迁移的重要因素。萨氏真蛇尾在楚科奇海呈现沿岸分布、部分分散的特点可能与该地区浮游植物分布格局有关[24],这可能是白令海与楚科奇海萨氏真蛇尾分化程度不明显,形成同一个谱系的原因之一。目前,化石证据表明萨氏真蛇尾在北太平洋出现的时间要早于大西洋海域。北太平洋萨氏真蛇尾的化石记录从晚中新世持续至晚更新世[25],而大西洋萨氏真蛇尾最早只记录至更新世[26]。因此,我们推测巴伦支海的萨氏真蛇尾是由太平洋迁移而来。
Fst能够衡量种群间遗传分化的尺度[27],本研究表明3个海域中巴伦支海的萨氏真蛇尾与白令海和楚科奇海海域种群相比获得的Fst值较高,表明该海域与其他海域的萨氏真蛇尾存在较明显分化。AMOVA分析表明白令海楚、科奇海与巴伦支海海域萨氏真蛇尾变异主要来源于群体间,变异的主要来源可能是巴伦支海与另外两个海域地理距离导致的遗传差异。本研究也对3个海域的萨氏真蛇尾进行了Exact检验和基因流分析。Exact检验结果显示三海域中单倍型在群体间分布频率差异不显著(P>0.05),结合遗传分化系数与AMOVA分析结果,说明萨氏真蛇尾在各群体内部随机交配。
4 结 论
本研究选取萨氏真蛇尾线粒体控制区、nad2和nad6三个标记描述北极白令海、楚科奇海、巴伦支海三个海域萨氏真蛇尾群体遗传特征,结果表明:
①3个海域萨氏真蛇尾nad2与nad6区域分离位点多于线粒体控制区,同时这2个区域揭示的单倍型多样性也明显高于控制区片段。
②北极地区萨氏真蛇尾的遗传多样性较高,群体数量较大,但本研究中的3个海域萨氏真蛇尾在各群体内部随机交配。
③3个海域萨氏真蛇尾分为两个谱系,巴伦支海成为一个单独谱系,白令海与楚科奇海成为另一个谱系。巴伦支海与其他海域的萨氏真蛇尾存在明显的分化,而白令海与楚科奇海海域萨氏真蛇尾遗传分化不明显。白令海与楚科奇海萨氏真蛇尾分化程度不明显的原因可能与两海域浮游植物的分布有关。
本文分析了北极3个萨氏真蛇尾群体的遗传差异,揭示了北极地区萨氏真蛇尾的群体遗传结构,为萨氏真蛇尾在太平洋与大西洋之间的迁移提供了科学依据。
