菠萝NAC转录因子基因AcoNAC1生物信息学及表达分析
2021-08-06谭秦亮蔡元保杨祥燕李穆李季东黄思婕程琴庞新华朱鹏锦周全光
谭秦亮 蔡元保 杨祥燕 李穆 李季东 黄思婕 程琴 庞新华 朱鹏锦 周全光

摘 要:NAC转录因子作为植物中数量最大的转录因子家族之一,在植物生长发育及各种逆境胁迫中发挥非常重要的调控作用。本研究以菠萝为试验材料,采用RT-PCR技术克隆得到基因AcoNAC1(GenBank登录号:XM_020225830),对其进行生物信息学及表达分析。生物信息学分析表明,该基因cDNA序列全长1723 bp,ORF为1062 bp,编码353个氨基酸,相对分子量为38.34 kDa,理论等电点为8.88;其编码蛋白的二级结构由20.40%的α-螺旋(H)和15.30%的β-折叠(E)以及59.21%的无规则卷曲(C)组成,具有NAC典型结构保守域。基因表达分析表明,AcoNAC1基因的表达受低温和干旱胁迫诱导,低温胁迫24 h和干旱胁迫12 h时的表达量最高;在不同成熟期品种中表现出持续的诱导表达,但诱导强度逐渐下降,其中早熟品种谢花后20~50 d及晚熟品种谢花后20~60 d基因表达量均处于较高水平。因此,推测AcoNAC1基因可能参与菠萝逆境胁迫响应以及果实发育与成熟的调控。
关键词:菠萝;NAC转录因子;生物信息学分析;逆境胁迫
Abstract: NAC is one of the largest families of transcription factors in plants, which plays an important role in plant growth, development and all kinds of adversity stress response. Mature pineapple pulp was used as the research material. AcoNAC1 (GenBank accession number: XM_020225830) was cloned by RT-PCR from Ananas comosus, then bioinformatics and expression were analyzed. Bioinformatics analysis showed that the full length cDNA of AcoNAC1 was 1723 bp and contained an ORF of 1062 bp, and was predicted to encode a protein of 353 amino acids, with a theoretical molecular weight of 38.34 kDa and an isoelectric point of 8.88. Secondary structure prediction revealed AcoNAC1 had 20.40% alpha helix (H), 15.30% beta folded (E) and 59.21% random curl (C), with typical NAC conservative domain. Gene expression analysis showed that the expression of AcoNAC1 was induced by low temperature and drought stress, and the highest expression was observed in low temperature stress 24 h and drought stress for 12 h. Different maturity varieties showed a continuous induction expression, and the expression was at a higher level after the precocious variety blossom 20-50 d and late-maturing variety blossom 20-60 d. Therefore, AcoNAC1 maight participate in response to adversity stress reaction and fruit development or maturity of regulation.
Keywords: Ananas comosus; NAC transcription factor; bioinformatics analysis; adversity stress
NAC(NAM、ATAF1/2、CUC2)转录因子是一类植物特有的蛋白家族,最早在矮牵牛(Petunia hybrida)和拟南芥(Arabidopsis thaliana)中被发现[1],在其他真核生物中尚未发现该家族成员的存在[2]。NAC转录因子结构复杂,大多数含有1个保守的N端DNA功能结合域和C端多变结构,N端保守的NAC功能结构域根据不同的基序可进一步分为A、B、C、D、E 5个保守的亚结构域,具有结合DNA、蛋白质及功能二聚体的特性,并决定着NAC转录因子的主要生化功能[3-4]。与N端的高度保守性不同,NAC转录因子的C端序列存在多变性,且富含酸性氨基酸、苏氨酸、脯氨酸、丝氨酸等,无论是在編码氨基酸还是序列长度上均表现出变化多样性,因此C端具有转录激活、抑制或结合蛋白质活性的特点[5]。
大量研究表明,首先NAC转录因子在植物生长发育中起重要的作用[6],通过此类基因的表达可以调控植物侧根的发育[7]、花的形态建成[8]、果实成熟[9]、叶片衰老[10]以及次生壁的形成[11]等。例如在拟南芥中,AtNAC2主要在花和根中高度表达,超表达AtNAC2能够促进侧根的发育[12]。其次是可以响应植物生长过程中遇到的逆境胁迫,激活多种生物和非生物胁迫过程中相关基因的表达,提高植物对逆境胁迫的耐受性[13]。例如在拟南芥中AtANAC019、AtANAC055和AtANAC072 的表达均受到干旱、高盐或脱落酸(ABA)的诱导,超表达可提高转基因植株对干旱的耐受性[14]。在小麦中TaNAC5基因参与逆境胁迫的应答,在渗透和低温胁迫下TaNAC5基因上调表达,而高盐胁迫则抑制TaNAC5基因表达[15]。在水稻中ONAC045过表达可以提高转基因水稻植株对冷和盐胁迫的抗性,还增强植株对ABA的灵敏度[16];过表达SNAC1基因既可增加水稻对ABA的敏感性,又可提高转基因水稻对干旱和盐碱化的耐受性,还可以通过调控保卫细胞的闭合来增强水稻的抗旱性[17-18]。
菠萝(Ananas comosus)是凤梨科凤梨属多年生草本植物,广泛分布于世界上的热带、亚热带地区,目前全球80多个国家与地区种植菠萝,但98%产自亚洲、美洲和非洲[19-20]。菠萝是我国热带、亚热带地区最具特色和优势的热带水果之一[21],与荔枝、香蕉及木瓜合称为我国岭南四大热带水果。目前,NAC转录因子在菠萝中的生物学功能尚不清楚,其相关研究未见报道。基于前期的研究基础[22-24],本研究从菠萝中克隆到1个NAC转录因子基因AcoNAC1,利用生物信息学方法对该基因的序列及其编码蛋白进行分析,预测其蛋白的理化性质、结构及功能等,并利用RT-qPCR分析该基因在不同胁迫和不同成熟期的表达模式,为今后深入研究该基因的功能提供理论依据。
1 材料与方法
1.1 材料
试验材料为广西亚热带作物研究所菠萝种质资源圃提供的菠萝品种‘金菠萝(MD-2)以及早熟品种‘巴厘(Comte de Paris)和晚熟品种‘pérola。选取处于幼苗期,苗龄为60 d的‘金菠萝组培苗作为胁迫处理对象,低温胁迫选择4 ℃环境,干旱模拟胁迫选择20%浓度的PEG 6000浇灌土壤至饱和。在‘金菠萝幼苗期分别采集经4 ℃低温胁迫和20% PEG 6000干旱胁迫0、1、3、6、12、24 h后的叶片,未进行处理的植株作为对照。早熟品种‘巴厘谢花(自然开花,以第一朵小花谢花开始算起,下同)后20、35、50、60、65、70 d分别采集果肉,晚熟品种‘pérola谢花后20、40、60、80、100、120 d分别采集果肉。以上所有材料取样后液氮速冻,保存于?80 ℃冰箱中,用于后期试验。
1.2 方法
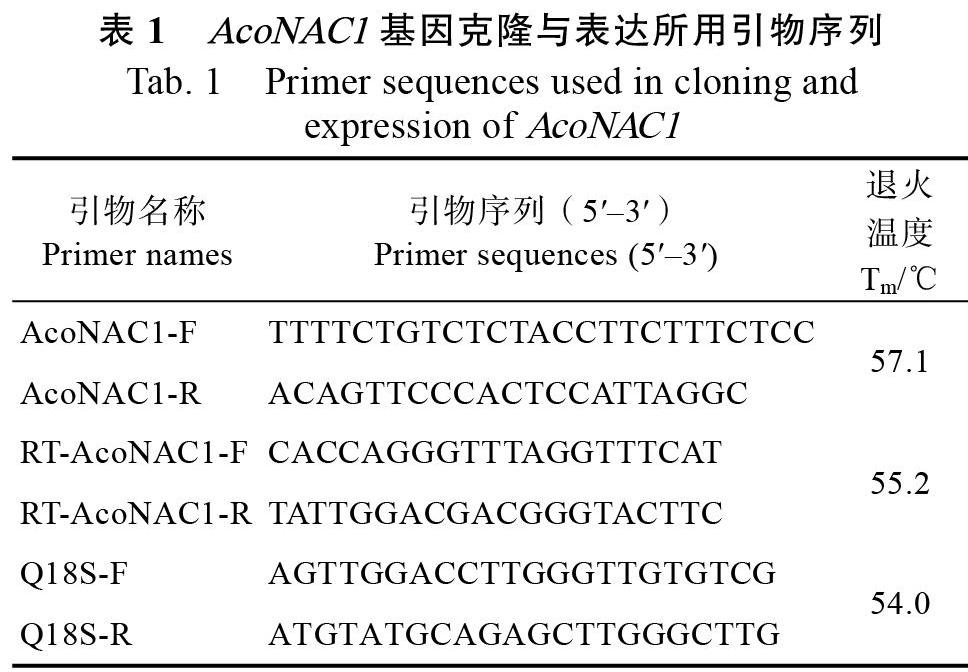
1.2.1 菠萝AcoNAC1基因的克隆 采用CTAB改良法提取菠萝叶片和果肉的总RNA,具体操作参照蔡元保等[24]的方法。利用课题组前期对菠萝转录组测序获得的编码NAC转录因子的EST序列,设计全长扩增引物AcoNAC1-F和AcoNAC1- R,扩增AcoNAC1基因的全长cDNA序列,所用引物(表1)全部由生工生物工程(上海)股份有限公司合成。
1.2.2 AcoNAC1蛋白性质分析 参考蔡元保等[24]和马海洋等[25]采用的在线工具和软件分析AcoNAC1蛋白的NAC保守结构域、基本理化特性、信号肽、亚细胞定位、蛋白质二级结构及蛋白质跨膜区段等结构和功能。
1.2.3 AcoNAC1蛋白同源性及系統进化树分析 利用DNAMAN6.0软件将菠萝AcoNAC1与NCBI数据库中搜索的菠萝AcNAC(OAY83360.1)、水稻OsNAC(XP-006660793.1)、海枣PdNACX2(XP- 017699777.1)、油棕EgNAC(XP-010929381.1)、玉米ZmNAC(PWZ38257.1)、高粱SbNAC(XP- 002462604.1)、大豆GsNACX3(XP_028199935.1)序列进行同源蛋白多序列比对。并利用DNAMAN 6.0软件,对菠萝和22个不同植物蛋白进行系统进化树的构建。
1.2.4 菠萝AcoNAC1基因表达分析 采用qRT- PCR方法分析AcoNAC1基因的实时表达情况,AcoNAC1基因的特异引物为RT-AcoNAC1-F和RT-AcoNAC1-R,内参基因选择菠萝18S rRNA基因,引物为Q18S-F和Q18S-R(表1)。RT-qPCR反应体系及程序参考蔡元保等[24]的方法。每个试验设3次重复,采用2?ΔΔCT方法分析处理数据,确定AcoNAC1基因的相对表达量。
2 结果与分析
2.1 菠萝AcoNAC1基因的克隆及序列分析
根据前期研究的菠萝转录组测序获得编码NAC转录因子的EST序列,利用RT-PCR技术扩增获得该EST的全长cDNA序列。经过序列分析表明,该基因的全长cDNA为1723 bp,最大开放阅读框(ORF)为1062 bp,可编码353个氨基酸。SMART在线工具预测表明,该编码蛋白含有一个典型的NAC转录因子的结构域(图1),位于该蛋白的N端,初步推测该蛋白属于NAC转录因子,因此将该基因命名为AcoNAC1(GenBank登录号:XM_020225830)。
2.2 编码蛋白AcoNAC1的理化性质分析
利用ProtParam在线工具预测编码蛋白AcoNAC1的理化性质,AcoNAC1蛋白的分子式为C1702H2604N484O503S14,相对分子量为38.34 kDa,理论等电点(pI)为8.88。该蛋白由353个氨基酸残基组成,富含强碱性氨基酸(Arg、Lys,共36个)和强酸性氨基酸(Asp、Glu,共31个),占比较高的氨基酸有甘氨酸(Gly)、丙氨酸(Ala)、异亮氨酸(Leu)、脯氨酸(Pro),分别占总氨基酸数的10.8%、10.2%、7.9%、7.9%。SignaIP4.0软件分析结果显示,AcoNAC1蛋白中不存在信号肽,亚细胞定位在细胞质中,平均亲水系数(GRAVY)为?0.543,不稳定指数为43.29,因此预测该蛋白为不稳定亲水蛋白。
2.3 编码蛋白AcoNAC1的二级结构及跨膜性预测
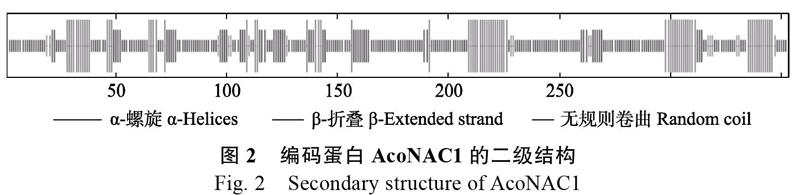
SOPMA软件分析结果显示(图2),AcoNAC1蛋白结构中主要含有α-螺旋(H)和β-折叠(E)以及无规则卷曲(C)这3种二级结构,其百分比分别20.40%、15.30%和59.21%。TMpred软件跨膜区域预测表明(图3),AcoNAC1蛋白有1个比较明显的跨膜结构,为246~273位点。
2.4 菠萝AcoNAC1蛋白的同源性分析
通过Blastp搜索和DNAMAN软件分析显示,菠萝AcoNAC1蛋白与其同源的菠萝AcNAC(OAY83360.1)序列一致性最高,同源性达到95.18%;其次为水稻OsNAC(XP-006660793.1)、海枣PdNACX2(XP-017699777.1)、油棕EgNAC(XP-010929381.1),同源性分别达到65.23%、63.01%、61.43%。而且,AcoNAC1与其他NAC转录因子一样,N-端的氨基酸序列高度保守,可以进一步划分为A、B、C、D、E 5个保守的亚结构域(图4)。
2.5 菠萝AcoNAC1蛋白的系统进化分析
利用DNAMAN软件分析AcoNAC1蛋白及其同源蛋白的系统进化表明,菠萝AcoNAC1蛋白与其同源的菠萝AcNAC(OAY83360.1)亲缘关系最近,与海枣PdNACX2(XP-017699777.1)和油棕EgNAC(XP-010929381.1)的亲缘关系次之;与拟南芥AtNAC(NP_188169.1)、亚麻荞CsNAC(XP_0104873776.1)和烟草NtNAC(XP_ 009625026.1)的亲缘关系最远(图5)。
2.6 菠萝AcoNAC1基因表达分析
2.6.1 AcoNAC1基因在逆境胁迫下的表达与分析 為研究AcoNAC1基因的表达是否受逆境胁迫的诱导,本研究采用低温、干旱2种不同逆境胁迫对菠萝幼苗进行处理。结果表明(图6),在低温处理1 h后,AcoNAC1基因表达量有明显上调,随后表达逐渐减少,到6 h后表达量再次明显上调,并在处理12 h后再次回落,之后随着处理时间的推迟,该基因的表达在处理24 h后达到最高值,表达量为对照(处理时间0 h)的11.25倍;在干旱处理3 h后,AcoNAC1基因表达量明显上调,且在12 h后达到最高值,为对照(处理时间0 h)的10.8倍。
2.6.2 AcoNAC1基因在菠萝不同成熟期的表达与分析 为研究AcoNAC1基因的表达是否能调控菠萝成熟期,本研究选用早熟品种(‘巴厘,Comte de Paris)、晚熟品种(pérola)的不同成熟期果肉进行检测。结果表明(图7),在不同成熟期2个品种中AcoNAC1基因表现出持续的诱导表达,且整体表达量呈逐渐减少趋势。早熟品种‘巴厘(Comte de Paris)谢花20 d 后AcoNAC1基因表达量达到最高峰值,随后到60 d后表达量下降到最低峰值,之后该基因的表达量一直未出现较大的增加;晚熟品种‘pérola谢花20 d后AcoNAC1基因表达量较高,且在谢花60 d后再次达到最高值,随后逐渐下降,到谢花100 d后下降到最低峰值,120 d时又有所回升。
3 讨论
NAC转录因子是一类基因调控蛋白,植物一系列生理生化反应均离不开NAC的调控,NAC不仅可以调节植物基本的新陈代谢和生长发育,而且在响应逆境胁迫方面也有着重要的作用。目前有关NAC转录因子家族的研究越来越热门,已在拟南芥[26]、水稻[27]、大豆[28]、番茄[29]、苹果[30]、葡萄[31]等多种植物中鉴别出NAC转录因子家族的大量基因,其中拟南芥中存在117个、水稻151个、大豆152个、番茄104个、苹果180个、葡萄74个。本研究利用转录组学等方法从菠萝中成功获得AcoNAC1基因(GenBank登录号:XM_ 020225830)的全长序列,该序列长度为1723 bp,开放阅读框(ORF)为1062 bp,可编码353个氨基酸,是一种不稳定亲水蛋白,有跨膜结构但不存在信号肽,亚细胞定位在细胞质中。序列分析表明,AcoNAC1蛋白和其他已知的NAC转录因子一样,N-端含有一个典型的NAC类转录因子的结构域,可分为5个亚结构域,与大部分前人研究结果完全一致。已有研究表明[32-33],NAC转录因子的N端具有典型的功能结构域特征,由150~160个氨基酸残基共同构成包含A~E共5个亚结构域,其中A亚结构域与蛋白互作二聚体的形成有关,B和E亚结构域与NAC转录因子的功能分化有关,C和D亚结构域与DNA的结合有关,其中A、C和D亚结构域高度保守,B和E亚结构域保守性不强。二级结构预测可知,该蛋白主要以无规则卷曲为主,这与梁芳等[34]对太行菊NAC转录因子的研究结果基本一致。
植物非生物胁迫作用主要包括干旱、低温、高盐等环境胁迫作用,植物受到一定的环境胁迫时就会通过多种信号转导途径响应胁迫信号,进而启动下游相关胁迫应答因子发挥作用,从而提高植物抵御逆境胁迫的能力。对香蕉研究发现,过表达MusaNAC042的香蕉转基因植株经干旱胁迫处理后,干旱指标脯氨酸含量升高,丙二醛含量降低,相对含水量高于对照植株,此外还引起多个下游相关基因的表达上调,说明MusaNAC042基因能够较好的提高香蕉的抗旱性[35]。水稻中OsNAC5和OsNAC095基因受干旱、高盐等胁迫诱导后上调表达,但受低温胁迫后下调表达,过表达这些基因能够增强转基因株系的抗干旱和抗高盐水平[36-37]。过表达OsNAC022的转基因水稻植株在干旱胁迫下较野生型(WT)植株耐旱性强,成活率高,生长性能好,其中蒸腾速率会降低,脯氨酸和可溶性糖含量会增加,且可通过ABA依赖通路调控转基因植物中OsNAC022基因的表达[38]。综上所述,NAC基因成员确实参与了植物的各种逆境应答。本研究发现,干旱和低温都对AcoNAC1基因的表达具有诱导作用,在低温处理1、6、24 h与干旱处理3、12、24 h后AcoNAC1基因的表达均有明显的增强趋势,这些变化说明AcoNAC1基因参与了菠萝非生物胁迫的调控响应过程,但其在不同胁迫条件下参与调控的途径和方式存在差异。
开花和果实成熟是植物生长发育中特有的一个发育阶段,是受到多种因素共同调控的复杂过程,在不同植物中相关基因调控的作用存在差异。近年来研究表明,NAC(NAM、ATAF1/2、CUC2)是一种具有特异性且可调控多种生物功能的植物转录因子,在转录水平上NAC转录因子可根据自身的特异性结合靶基因启动子上的顺式作用元件,激活或抑制下游靶基因的表达,进而调控果实的成熟期、外观、风味及品质的相关变化。在脐橙晚熟突变体中发现1个NAC基因CitNAC[39],其在成熟柑橘的果皮和果肉中表达较高,而在果实发育的其他阶段表达量相对较低,推测其可能与果实的成熟和组织的衰老密切相关。本研究中菠萝AcoNAC1基因在早熟和晚熟品种的果实成熟期中,均受到诱导表达,但表达量整体呈下降的趋势,推测其在菠萝果实形成过程中有促进作用,但是在菠萝果实成熟后期具有一定的负调控作用。
参考文献
Aida M, Ishida T, Fukaki H, et al. Genes involved in organ separation in Arabidopsis: An analysis of the cup-shaped cotyledon mutant[J]. Plant Cell, 1997, 9(6): 841-857.
刘美英, 冶晓芳, 唐益苗, 等. TaNAC提高转基因烟草的抗旱功能[J]. 中国烟草学报, 2010, 16(6): 82-88.
张慧珍, 白雪芹, 曾幼玲. 植物NAC转录因子的生物学功能[J]. 植物生理学报, 2019, 55(7): 915-924.
Jensen M K, Kjaersgaard T, Nielsen M M, et al. The Arabidopsis thaliana NAC transcription factor family: Structure-function relationships and determinants of ANAC019 stress signalling[J]. Biochemical Journal, 2010, 426(2): 183-196.
Puranik S, Sahu P P, Srivastava P S. NAC proteins: regulation and role in stress tolerance[J]. Trends in Plant Science, 2012, 17(6): 369-381.
Olsen A N, Ernst H A, Leggio L L, et al. NAC transcription factors: Structurally distinct, functionally diverse[J]. Trends Plant Science, 2005, 10(2): 79-87.
Hao Y J, Wei W, Song Q X, et al. Soybean NAC transcription factors promote abiotic stress tolerance and lateral root formation in transgenic plants[J]. The Plant Journal, 2011, 68(2): 302-313.
Hendelman A, Stav R, Zemach H, et al. The tomato NAC transcription factor SINAM2 is involved in flower-boundary morphogenesis[J]. Journal of Experimental Botany, 2013, 64(18): 5497-5507.
Ma N N, Feng H L, Meng X, et al. Overexpression of tomato SINAC1 transcription factor alters fruit pigmentation and softening[J]. BMC Plant biology, 2014, 14: 351.
Guo Y F, Gan S S. AtNAP, a NAC family transcription factor, has an important role in leaf senescence[J]. Plant Journal, 2006, 46(4): 601-612.
Chai M F, Bellizzi M, Wan C X, et al. The NAC transcription factor OsSWN1 regulates secondary cell wall development in Oryza sativa[J]. Journal of Plant Biology, 2015, 58(1): 44-51.
He X J, Mu R L, Cao W H, et al. AtNAC2, a transcription factor downstream of ethylene and auxin signaling pathways, is involved in salt stress response and lateral root development[J]. The Plant Journal, 2005, 44(6): 903-916.
張 丹, 马玉花. NAC转录因子在植物响应非生物胁迫中的作用[J]. 生物技术通报, 2019, 35(12): 144-151.
Tran L S P, Nakashima K, Sakuma Y, et al. Isolation and functional analysis of Arabidopsis stress inducible NAC transcription factors that bind to a drought-responsive Cis-element in the early responsive to dehydration stress 1 promoter[J]. Plant Cell, 2004, 16(9): 2481-2498.
胡 伟, 颜 彦, 韦运谢, 等. 小麦TaNAC5基因克隆及表达分析[J]. 西北植物学报, 2014, 34(12): 2394-2398.
Zheng X, Chen B, Lu G, et al. Overexpression of a NAC transcription factor enhances rice drought and salt tolerance[J]. Biochemical and Biophysical Research Communications, 2009, 379(4): 985-989.
Saad A S, Li X, Li H P, et al. A rice stress-responsive NAC gene enhances tolerance of transgenic wheat to drought and salt stresses[J]. Plant Science, 2013, 203-204: 33-40.
Hu H, Dai M, Yao J, et al. Overexpressing a NAM, ATAF, and CUC (NAC) transcription factor enhances drought resistance and salt tolerance in rice[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(35): 12987-12992.
Botella J R, Smith M. Genomics of pineapple, crowning the king of tropical fruits, genomics of tropical crop plants[J]. Genomics of Tropical Crop Plants, 2008: 441-451.
刘海清, 刘志颐. 中国菠萝鲜果产业的国际竞争力分析[J]. 江苏农业科学, 2017, 45(17): 334-337.
杨文秀, 赵维峰, 邓大华, 等. 云南省菠萝产业发展现状分析[J]. 农学学报, 2014, 4(1): 60-63.
杨祥燕, 蔡元保, 张治礼, 等. 菠萝MADS-box基因AcMADS2的克隆與表达分析[J]. 植物生理学报, 2016, 52(9): 1406-1412.
蔡元保, 杨祥燕, 孙光明, 等. 菠萝叶片色泽、色素及抗氧化活性的关系[J]. 植物科学学报, 2017, 35(2): 283-290.
蔡元保, 杨祥燕, 孙光明, 等. 菠萝花发育相关基因AcMADS1的克隆与组织表达特性分析[J]. 植物学报, 2014, 49(6): 692-703.
马海洋, 赵秋芳, 陈 曙, 等. 菠萝PEPC基因家族生物信息学分析[J]. 热带作物学报, 2020, 41(1): 97-103.
Ooka H, Satoh K, Doi K, et al. Comprehensive analysis of NAC family genes in Oryza sativa and Arabidopsis thaliana[J]. DNA Research, 2003, 10(6): 239-247.
王春雨, 张 茜. 植物NAC转录因子功能研究进展[J]. 生物技术通报, 2018, 34(11): 14-20.
王 洋, 柏 锡. 大豆NAC基因家族生物信息学分析[J]. 大豆科学, 2014, 33(3): 325-333.
姜秀明, 牛义岭, 许向阳. 番茄NAC基因家族的系统进化及表达分析[J]. 分子植物育种, 2016, 14(8): 1948-1964.
李德龙, 周 军, 王大玮, 等. 苹果NAC转录因子的克隆与全基因组生物信息学分析[J]. 基因组学与应用生物学, 2017, 36(6): 2507-2517.
Su H, Zhang S, Yuan X, et al. Genome-wide analysis and identification of stress-responsive genes of the NAM-ATAF1, 2-CUC2 transcription factor family in apple[J]. Plant Physiology and Biochemistry, 2013, 71: 11-21.
Ernst H A, Olsen A N, Larsen S, et al. Structure of the conserved domain of ANAC, a member of the NAC family of transcription factors[J]. EMBO Reports, 2004, 5(3): 297-303.
Hao Y J, Song Q X, Chen H W, et al. Plant NAC-type transcription factor proteins contain a NARD domain for repression of transcriptional activation[J]. Planta, 2010, 232(5): 1033-1043.
梁 芳, 张 燕, 郝平安, 等. 太行菊NAC转录因子基因OpNAC1的克隆及生物信息学分析[J]. 分子植物育种, 2018, 16(11): 3477-3486.
Tak H, Negi S, Ganapathi T R. Banana NAC transcription factor MusaNAC042 is positively associated with drought and salinity tolerance[J]. Protoplasma, 2017, 254(2): 803-816.
Jeong J S, Kim Y S, Redillas M C, et al. OsNAC5 overexpression enlarges root diameter in rice plants leading to enhanced drought tolerance and increased grain yield in the field[J]. Plant Biotechnology Journal, 2013, 11(1): 101-114.
Huang L, Hong Y, Zhang H, et al. Rice NAC transcription factor ONAC095 plays opposite roles in drought and cold stress tolerance[J]. BMC Plant Biology, 2016, 16(1): 203.
Hong Y B, Zhang H J, Huang L, et al. Overexpression of a stress-responsive NAC transcription factor gene ONAC022 improves drought and salt tolerance in rice[J]. Frontiers Plant Science, 2016, 7: 4.
Liu Y Z, Baig M N R, Fan R, et al. Identification and expression pattern of a novel NAM, ATAF, and CUC-like gene from Citrus sinensis Osbeck[J]. Plant Molecular Biology Reporter, 2009, 27(3): 292-297.
責任编辑:谢龙莲
