基于TCGA构建胃癌免疫基因预后风险评估模型
2021-07-28王永强彭际奎姜洪伟王举
王永强 彭际奎 姜洪伟 王举
内蒙古自治区人民医院胃肠外科,呼和浩特 010017
胃癌是严重威胁人类健康的消化道恶性肿瘤之一,我国胃癌发病率高,在男性、女性分别居恶性肿瘤第2、第3位,病死率均居恶性肿瘤第2位[1]。早期胃癌,手术治疗能有效控制疾病的恶化。然而,胃癌发病隐匿,早期症状不明显,就诊时大多属晚期,已出现腹膜、肝、肺等远处转移,失去手术机会。另外,虽然化疗联合靶向治疗是晚期胃癌或胃癌术后的主要手段,但受耐药的影响,其疗效有限[2-3]。数据显示,胃癌5年生存率仅为31.5%[4]。因此,探索胃癌发生发展新机制以及制定治疗新策略迫在眉睫。近年来,胃癌肿瘤微环境免疫抑制状态以及胃癌对免疫治疗响应的研究备受关注。
目前认为,免疫微环境影响肿瘤发生、发展、转移及耐药[5-6],肿瘤浸润淋巴细胞可用来评估肿瘤患者复发及死亡风险[7-8]。研究表明微卫星不稳定的胃癌患者接受抗PD-1/PD-L1单抗的免疫治疗可获得明显的生存受益[9-10]。另外,对于肿瘤微环境中效应T细胞浸润增多、肿瘤突变负荷高的患者,临床预后也更佳[11-12]。然而,受限于免疫细胞标志物,传统检测免疫浸润的方法,例如流式细胞计数、免疫组化法等,并不能全面反应免疫细胞的浸润情况。利用转录组数据,通过单样本基因集富集分析,可充分展示肿瘤患者28种免疫细胞浸润特征,从而指导临床实践。对于胃癌,目前还没有基于免疫基因数据库分析其免疫微环境以及评估其预后的分子模型。因此,本研究基于癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库获取大样本的胃癌转录数据,以期构建胃癌免疫相关生存模型,并探索免疫微环境生存相关性以及其影响胃癌发生发展的重要调控通路。
1 材料与方法
1.1 原始数据下载及数据预处理 从TCGA官网(https://portal.gdc.cancer.gov/repository)下载胃癌测序数据,依次选择STAD→RNA-Seq→FPKM,并整理成行名为基因名、列名为样本名的矩阵文件。免疫相关基因列表从ImmPort网站(https.//www.immport.org/home)下载并整理,总共1 739个免疫基因。从总矩阵中提取免疫相关基因表达量,用于后续分析。此外,从TCGA网站下载胃癌临床数据,整理成矩阵文件,并排除:(1)临床信息不完整的病例;(2)术后随访时间小于90 d的病例。
1.2 构建风险预测模型
1.2.1 分组 将免疫相关基因、临床预后矩阵合并成行名为样本名,列名为生存时间、生存状态和基因的新矩阵,采用R“caret”包,按6∶4比例分为训练集、验证集,并采用卡方检验分析两组样本集临床病理特征的差异性。
1.2.2 构建风险评估模型 首先采用单因素Cox分析在训练集中筛选预后相关免疫基因,在此基础上,利用多因素Cox分析,得到胃癌风险评估模型。进一步利用模型对样本进行评分,以中位风险评分将样本分为高、低风险组,运用R“survminer”包绘制高、低风险Kaplan-Meier生存曲线从而分析该模型预测效果;运用R“survival ROC”包绘制受试者工作特征曲线(ROC)从而验证分析模型可靠性;再运用以R“pheatmap”包绘制风险状态图。
1.2.3 验证风险评估模型 利用上述风险模型对验证集样本进行评分,并以测试集中位风险评分将验证集样本分为高、低风险组,再分别运用R“survminer”“survival ROC”及“pheatmap”包绘图,进一步验证模型预测的有效性及可靠性。
1.3 Cox回归分析筛选影响胃癌预后的独立危险因素 将临床病理特征及生存矩阵、风险评分(risk score)矩阵整理合并成包含生存时间、生存状态、性别、年龄、分化程度、TNM分期以及风险评分的新矩阵。先采用单因素Cox分析,筛选预后相关变量,再预后相关的变量纳入多因素Cox回归分析,最终筛选影响胃癌预后的独立危险因素。
1.4 免疫细胞浸润分析 利用单样本富集分析(single sample gene set enrichment analysis,ssGSEA),将基因表达矩阵转化成28种免疫细胞相对浸润比例矩阵,行名是免疫细胞类型,列名是样本名。预设特征基因集,即各免疫细胞的特征基因集,从最近发表的2篇文献整理[13-14]。卡方检验分析高、低风险组与各免疫细胞浸润比例、各临床病理特征的关系,采用R“pheatmap”包绘制高、低风险热图。
1.5 高、低风险组差异分析 运用R“edger”包分析高、低风险组差异表达基因后,再利用R“clusterProfiler”对差异基因进行基因本体论(GO)、京都基因与基因组大百科全书数据库(KEGG)富集分析,以探索影响胃癌预后的重要调控通路以及相关分子机制。
1.6 统计学分析 采用R语言对本研究进行统计学分析,采用survival包进行单因素、多因素Cox回归分析。计数资料采用χ2检验,以R“chisq.test”函数分析。计量资料采用t检验或Wilcoxon秩和检验,以R“t.test”或“wilcox.test”函数分析。
2 结 果
2.1 分组 按照排除标准,将胃癌样本分为训练集(221例)、验证集(147例),χ2检验结果显示,训练集、验证集样本在年龄、性别、病理分级、临床分期、淋巴结转移及远处转移方面差异均无统计学意义(均P>0.05),说明两组样本具有可比性,见表1。
2.2 Cox比例风险回归模型分析结果 采用单因素Cox分析在训练集中筛选预后相关免疫基因(P<0.05),再纳入多因素Cox分析,得到由9个免疫基因构成的胃癌预后风险评估模型(图1),风险值(risk score)=PROC×0.076+IGKV1D-43×0.032+CLCF1×0.049+IL21RA×-0.074+TAFA4×0.061+NOX4×0.446+INHA×0.133+ITGAV×0.025+FABP3×0.019,按训练集中位风险值分别将训练集、验证集样本分为高、低风险组。
2.3 Kaplan-Meier生存及ROC分析 采用R“survminer”包对高、低风险组进行Kaplan-Meier分析,检测风险评估模型预测胃癌患者预后的有效性,结果显示,训练集中高、低风险组5年总生存率(OS)分别为20.0%(22/110)、50.5%(55/111);验证集中高、低风险组5年OS分别为24.7%(18/73)、43.2%(32/74)(图2A、B);两组样本集高风险组OS均显著低于低风险组(均P<0.05)。采用R“time ROC”包进行ROC绘制,检测风险评估模型预测胃癌患者预后的可靠性,结果显示,训练集1、3、5年ROC的AUC值为0.69、0.71、0.78,验证集1、3、5年ROC曲线的AUC为0.56、0.71、0.78(图2C、D)。
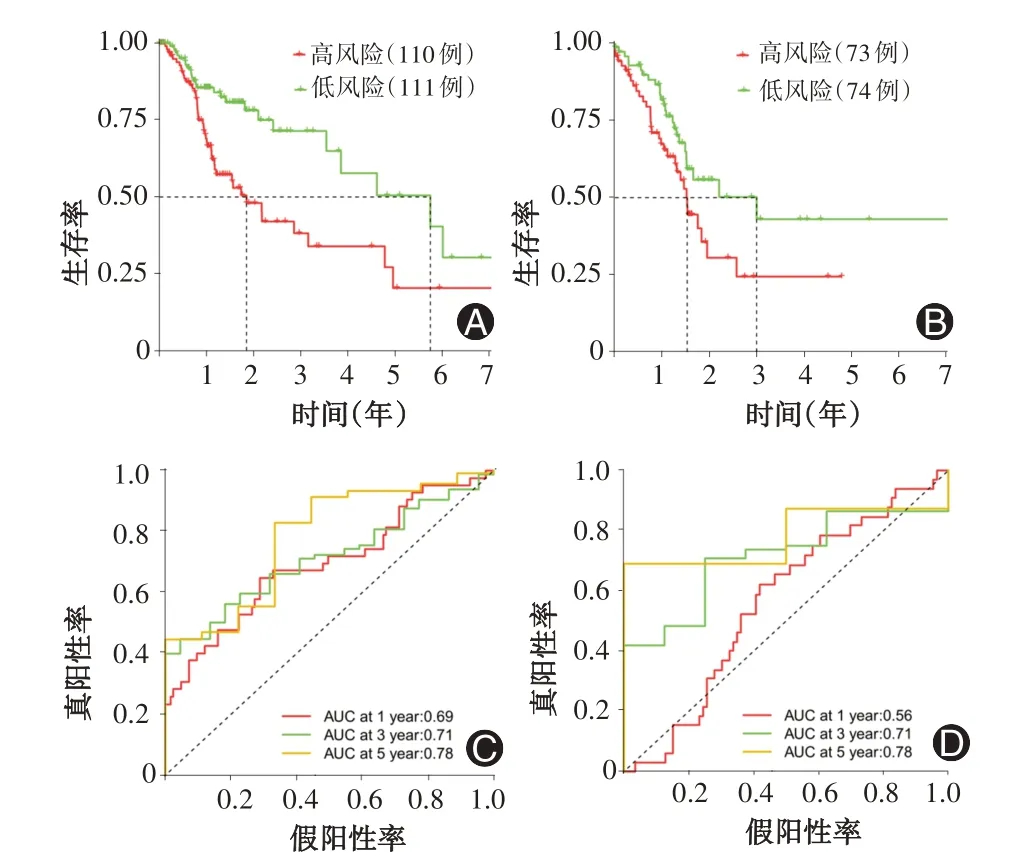
图2 训练集、测试集胃癌患者中高、低风险组Kaplan-Meier生存曲线及ROC(A为训练集的生存曲线,B为验证集的生存曲线,C为训练集的ROC,D为验证集的ROC)
2.4 免疫风险评估模型Cox回归分析 以构建的免疫风险评估模型联合胃癌临床病理特征,包括性别、年龄、分化程度、TNM分期,先做Cox单因素分析,再纳入预后相关的因素做Cox多因素分析,筛选影响胃癌预后的独立因素,结果显示,在训练集、验证集中免疫风险评估模型、TNM分期都是影响胃癌预后的独立因素(表2)。
2.5 免疫风险评估模型与免疫细胞浸润、临床病理特征的关系 利用Cibersort反卷积算法,将每个样本基因表达矩阵转化成28种免疫细胞浸润比例的矩阵,并采用χ2检验分析训练集、验证集中高、低风险组与临床病理特征的关系,采用Wilcoxon秩和检验(属于非参数检验)分析训练集、验证集中高、低风险组与免疫细胞浸润的关系。结果如图3所示,训练集、验证集中免疫风险评分均与活化的CD4+T细胞的浸润有关,高风险组活化CD4+T细胞的浸润比例显著降低(P<0.05)。
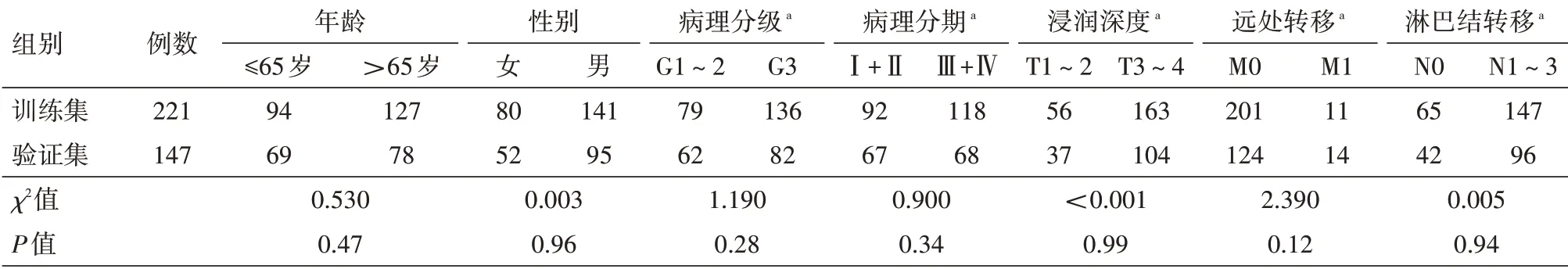
表1 训练集、测试集胃癌患者的临床病理特征分布(例)
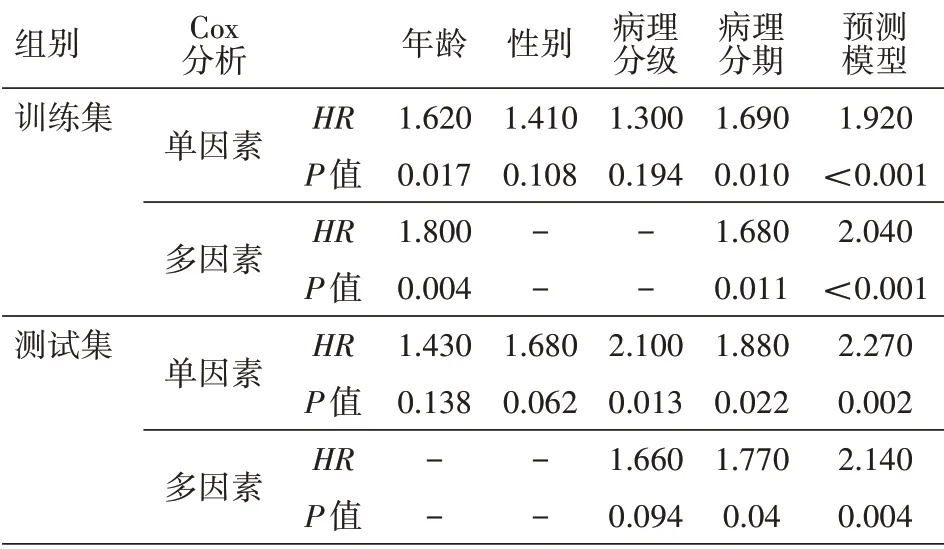
表2 胃癌临床病理特征及风险预测模型在训练集、测试集的单因素及多因素Cox回归分析
2.6 风险差异基因GO、KEGG富集分析 利用非参数检验,分别在训练集、验证集中,筛选高、低风险组差异基因,再以R“clusterProfiler”包对差异基因进行KEGG富集分析,以初步探索模型中基因促进胃癌进展的分子机制。如图4所示,训练集、验证集的差异基因均富集于PI3K-Akt、cGMP-PKG、ECM-受体结合、黏着斑激酶、肿瘤蛋白多糖等肿瘤相关信号通路。
3 讨 论
作为构成肿瘤微环境的重要组成部分,免疫细胞在肿瘤发生、转移、耐药、预后评估、治疗评估等方面起重要作用。免疫评分已作为胃肠道肿瘤预后判断的重要依据。本研究基于TCGA数据库,利用Cox比例回归模型,构建了由9个免疫基因组成的胃癌预测模型,利用该模型可准确、有效地评价胃癌预后。
利用公共数据库如TCGA、GEO测序或芯片、临床数据构建胃癌预后模型的研究较多,包括利用编码蛋白基因、lncRNA、miRNA构建模型。此类预测模型存在以下问题:(1)为减少纳入构建模型的基因数,先设定严格阈值,筛选差异表达基因。然而,预后相关基因不一定是差异基因。因此,一些关键基因可能被剔除。随着生物信息学发展,功能基因被进一步注释、分类,如免疫、代谢以及RNA结合蛋白相关基因。(2)利用全部样本构建模型,缺少外部和/或内部数据的交叉验证,模型的可靠性有待商榷。鉴于以上问题,本研究纳入全部免疫相关基因(1 739例)建模,而不筛选差异基因;TCGA胃癌样本分为训练集、验证集,在训练集中建模,在验证集中检测,设置循环,满足条件后输出结果。本研究构建的模型在训练集、验证集中均能有效评估胃癌患者预后,随访时间越长,准确性越高,且该模型是胃癌预后的独立危险因素。高风险组患者活化CD4+T细胞浸润比例减少,CD4+T细胞浸润是影响结肠癌预后的独立危险因素,部分解释了该组患者预后差的原因[15]。此外,本研究分别在训练集、验证集中筛选高、低风险组差异基因,纳入GO、KEGG富集分析以初步探索模型基因导致患者预后差的分子机制,结果显示模型基因可能通过激活PI3K-Akt通路促进胃癌转移、耐药。有文献报道,PI3K-Akt参与胃癌转移及化疗耐药[16-17]。

图3 训练集、验证集胃癌患者高低风险组临床及免疫细胞浸润热图(A为训练集,221例,B为验证集,147例)

图4 训练集、验证集胃癌患者风险差异基因KEGG富集图(A为训练集,B为验证集)
该胃癌预后评估模型包含8个风险基因PROC、IGKV1D-43、CLCF1、TAFA4、NOX4、INHA、ITGAV、FABP3和1个保护基因IL27RA,其中4个风险基因被报道与胃癌或其他恶性肿瘤发生、发展及预后相关。心肌营养因子样细胞因子1(CLCF1)属于Gp130细胞因子家族成员,与细胞因子受体因子1(CRLF1)形成异源二聚体,与神经营养因子(CNTF)竞争性结合其受体CNTFR,从而激活JAK-STAT增殖相关信号通路,与肝细胞癌索拉菲尼耐药、有氧糖酵解有关[18]。另外,肿瘤相关巨噬细胞通过CLCF1/CXCL6/TGF-β轴协调肝癌细胞与中性粒细胞的“对话”[19]。目前尚无CLCF1与胃癌发生、转移、预后及耐药的报道。本研究提示CLCF1是影响胃癌预后的危险因素,CLCF1高表达的患者预后差,然而,CLCF1促进胃癌进展的作用及机制需要进一步研究。NAPDH氧化酶4(NOX4)作为催化亚基,可促进活性氧(ROS)的产生,而ROS伴随肿瘤代谢重编程产生,可作为第二信使,参与多条信号通路的激活及氧化还原信号调控与肿瘤代谢。沉默NOX4或使用抑制剂可逆转由肿瘤相关成纤维细胞引起的CD8+T细胞耗竭而出现的免疫抑制状态,从而改善免疫治疗耐药情况[20]。本研究显示,NOX4在模型中所占权重最高,高表达个体罹患胃癌的风险增高1.56倍,说明NOX在胃癌预后评估中的作用最大。整合素α5(integrinαV,ITGAV)属于整合素家族成员之一,与整合素β亚基形成异源二聚体,调节新生血管生成及肿瘤发展。细胞外基质通过ITGAV激活JAK2/STAT5通路,而该通路参与细胞干性维持及肿瘤发生,IL-32γ通过抑制ITGAV介导的STAT5通路从而抑制肺癌干细胞的增殖[21]。作为转录调控因子YAP、WWTR1靶基因的ITGAV不仅直接激活Hippo通路,促进肝癌转移,而且正反馈调节YAP、WWTR1的活性。而且,有文献报道,ITGAV促进胃癌细胞增殖、迁移及侵袭的能力[22]。本研究发现,ITGAV高表达个体预后差,提示ITGAV可能促进胃癌的转移从而影响预后。脂肪酸结合蛋白3(fatty acid binding protein 3,FABP3),在脂肪酸转运、细胞增殖以及基因调控方面起重要作用,其高表达被认为是非小细胞肺癌的不良预后因素[23],与本研究显示的FABP3高表达是胃癌不良预后因素相似。
综上所述,本研究利用TCGA胃癌转录组数据,构建了由9个免疫基因组成的预后评估模型,训练集、验证集验证结果进一步证实了该模型具有良好的预测性能,其能准确区分高、低风险的病例,具有潜在临床应用价值。
