水稻粒重粒形QTL的定位及qTGW1.2/qGL1.2的验证
2021-07-19杜成兴张华丽戴冬青吴明月梁敏敏陈俊宇马良勇
杜成兴 张华丽 戴冬青 吴明月 梁敏敏 陈俊宇 马良勇
水稻粒重粒形QTL的定位及的验证
杜成兴#张华丽#戴冬青 吴明月 梁敏敏 陈俊宇*马良勇*
(中国水稻研究所 水稻生物学国家重点实验室/国家水稻改良中心,杭州 310006;#共同第一作者;*通信联系人,E-mail: chenjunyu@caas.cn,maliangyong@caas.cn)
【目的】粒重粒形对水稻的产量和品质均有重要的影响。本研究通过开展水稻粒重粒形QTL的初步定位,并对新鉴定的第1染色体长臂区间进行验证,旨在进一步揭示水稻粒重粒形的遗传调控机制。【方法】以大粒的FM9为父本,小粒的EFT为母本,配组衍生遗传群体,先后获得包含277个株系的F2:3群体和211个株系的重组自交系群体(Recombinant Inbred Lines, RILs),测定千粒重、粒长和粒宽,采用完备区间作图法进行QTL初定位;针对新鉴定的区间,筛选2个剩余杂合体单株,自交衍生分离群体,开展QTL效应验证。【结果】初定位分析共检测到35个调控千粒重、粒长和粒宽的QTL,其中,11个能同时在两个群体中被检测到,18个仅在F2:3群体中被检测到,6个仅在RIL群体中被检测到;应用两个剩余杂合体衍生的两套分离群体验证了新鉴定的区间对千粒重和粒长的效应,并观察到颖壳细胞长度的显著变化。通过qPCR分析,观察到与细胞周期、生长素代谢和粒形相关基因表达发生了显著变化。【结论】初步定位的35个QTL以及验证的有利于进一步揭示水稻粒重粒形的遗传控制基础,也为后续的基因克隆及分子标记辅助选择奠定了基础。
水稻;粒重;粒形;QTL;剩余杂合体
水稻是世界三大粮食作物之一,是亚洲最重要的主食作物[1]。水稻产量相关性状的遗传研究是当前作物科学研究的热门领域。水稻粒重作为稻谷产量三要素之一,主要由粒长和粒宽等粒形性状决定,同时粒形也是影响稻米外观和加工品质的关键因素之一,是商品稻米的重要指标[2-3]。
粒重与粒形均属于典型的多基因控制的数量性状,相互之间高度相关[4-5]。过去20多年间,数量性状基因座(Quantitative Trait Locus, QTL)的研究一直受到重视,目前为止,已鉴定出500多个粒重和粒形的相关QTL(http://www.gramene.org/),广泛分布于水稻所有12条染色体上,并已有20多个QTL被克隆[6]。这些研究提升了我们对粒重与粒形遗传组成的理解,尤其通过基因克隆进一步揭示了粒重粒形的分子调控机制,这些克隆的基因主要涉及植物激素、泛素-蛋白酶体通路、MAPK信号通路、G-蛋白信号通路以及一些转录调控因子等途径[7-8]。因此,挖掘新的水稻粒重粒形QTL,将进一步完善粒重粒形的遗传调控网络,促进水稻产量与品质的遗传改良。
水稻粒重粒形主要由少数主效与多数微效基因共同调控,在QTL初定位分析中,主效位点由于对群体的表型变异贡献大,其分离往往会对其他微效位点的检测功效及遗传效应估计产生较大的影响[9-11]。构建次级定位群体,排除主效位点的干扰,是新位点验证、精细定位乃至克隆的有效途径[12-13]。次级群体的构建,除了通过连续回交(6~7代)使遗传背景与轮回亲本基本一致外,也可通过筛选剩余杂合体自交衍生;后者相对前者,仅需1次分子标记检测,筛选出目标单株即可,无需重新构建群体,省时省力,已有较普遍的应用,比如在排除主效粒重粒型QTL和影响的基础上,完成了多个微效粒重粒形QTL的鉴定[14-16]。

图1 群体构建过程
Fig.1.Development of the rice materials used in the study.
本研究筛选粒重和粒形具有显著差异的两个水稻材料进行杂交配组,构建了F2:3群体和重组自交系群体,用于粒重和粒形的QTL初定位分析。并且,针对在两套初定位群体中均呈显著作用的区间,从重组自交系群体中筛选出2个不同遗传背景的剩余杂合体单株,自交衍生了2套分离群体,开展该区间对粒重粒形的效应验证,并初步分析了该QTL区间对颖壳细胞分裂生长的影响,以及部分细胞周期相关基因和3个主效粒形基因的表达变化。
1 材料与方法
1.1 群体材料
以中优早81为轮回亲本的谷梅2号导入系FM9为父本(大粒),春江06和TN1杂交衍生的后代株系EFT为母本(小粒),通过杂交衍生构建了具有277个株系的F2:3群体和211个株系的F7代 RIL群体,用于粒重粒形QTL的初定位分析。针对在两套初定位群体中对千粒重和粒长都呈显著作用的第1染色体长臂上的区间(RM315−RM12138),从RIL群体中挑选到2个剩余杂合体单株RHL17和RHL124,衍生分离群体,用于QTL效应的验证。这两个单株遗传背景不同,在与RM315−RM12138区间交叠的FIR3596−FIR3892区间(2.96 Mb)表现为杂合,在其他背景区间上高度纯合。通过自交,获得2套F8分离群体,命名为D1和D2,分别包含48和45个单株;进一步从每个群体中挑选区间呈父本纯合型(即FM9纯合型)和母本纯合型(即EFT纯合型)的单株,自交后获得2套相应的F8:9家系群体,命名为E1和E2;E1包含EFT纯合型株系12个和FM9纯合型株系10个,E2包含EFT纯合型株系12个和FM9纯合型株系11个。群体构建的具体流程如图1所示。两个剩余杂合体单株RHL17和RHL124的基因型组成如图2所示。
1.2 田间试验及性状考查
F2:3群体、RIL群体和两套F8:9近等基因系群体分别于2015年、2019年和2020年种植在浙江杭州(5月−9月);两套F8分离群体于2019年种植在海南陵水(2019年12月−2020年4月)。F8:9近等基因系群体每株系种2行,每行8个单株,株行距16.7 cm×26.7 cm,正常田间管理。成熟后混收中间4株。将每株系的种子在自然条件下风干后用18%的生理盐水浸种,取实粒于39℃烘箱烘干20 h[17]。使用SC-G型万深考种自动分析仪测量千粒重,粒长和粒宽,每次150粒,2次重复取平均值,并进行数据分析。

图2 两个剩余杂合体单株RHL17和RHL124与目标qTGW1.2/qGL1.2区间的基因型组成
Fig.2.Schematic genotypes of two residual heterozygotes RHL17 and RHL124 and the targeted region of.
1.3 遗传图谱构建与QTL分析
将在双亲FM9和EFT之间具有多态性的148对SSR(simple sequence repeats)标记和通过亲本间重测序结果获得的58对InDel标记,用于F2:3群体和RIL群体的基因型检测,使用QTL IciMapping 4.2构建遗传连锁图谱。F2:3群体的遗传图谱包含其中的155对标记,总遗传距离为2362.3 cM,平均距离为15.0 cM;RIL群体的遗传图谱包含其中的170对标记,总遗传距离为 1742.7 cM,平均距离为10.3 cM。两套群体共用129对标记,F2:3群体单独使用的标记26对,RIL群体单独使用的标记41对。
采用QTL IciMapping 4.2软件[18]中的完备区间作图法(Inclusive Composite Interval Mapping, ICIM),模拟计算1000次,获得F2:3和RIL群体中QTL存在的阈值,分别为3.80和2.88;在全基因组范围内检测千粒重、粒长和粒宽的QTL;QTL命名遵照McCouch等[19]建议的规则命名;将2个群体在同一染色体不同区间检测到的QTL按物理位置依次命名。
1.4 颖壳扫描电镜观察
选取E1群体中的两种亲本纯合型(FM9和EFT)的成熟干燥种子颖壳,经导电处理后,置于扫描电子显微镜下观察、采图。为了保证两个近等基因系之间的遗传背景尽量保持一致,我们所挑选的两个近等基因系衍生于同一个剩余杂合体单株RHL17;针对RHL17遗传背景中的5个剩余杂合区间,我们进一步分析了该对近等基因系的基因型。结果显示,除了第2和第3染色体长臂的2个杂合区间已通过自交纯合,呈FM9纯合型,剩余的3个杂合区间仍为杂合。
1.5 RT-PCR表达分析
利用颖壳扫描电镜观察所用到的近等基因系进行相关基因的表达分析。取5 cm左右的幼穗提取总RNA[7, 20],cDNA合成反应使用ReverTra Ace®反转录试剂盒(TOYOBO)。qRT-PCR使用SYBR®实时定量PCR试剂盒(TOYOBO),在Applied Biosystems™ 7900HT快速实时荧光定量PCR仪上进行。使用水稻()作为内参对照,每个基因反应均设置3个技术重复,3个生物学重复,所用到的各基因引物参见附表1。

**表示在 0.01水平上显著。
Fig.3.Comparison of 1000-grain weight, grain length and grain width of the parents EFT and FM9.
1.6 数据处理
使用Excel 2010进行表型数据的录入、整理,作图,并进行数据相关性和方差分析。
2 结果与分析
2.1 亲本、F2:3和RIL群体的粒重和粒形
亲本EFT和FM9在千粒重、粒长和粒宽性状上均存在极显著的差异(图3);大粒亲本FM9的千粒重高达41.01 g,粒长10.96 mm,粒宽3.38 mm,相比小粒亲本EFT分别增加103%、57%和24%。EFT/FM9衍生的F2:3和RIL群体的千粒重、粒长和粒宽的频率分布较为接近,均呈连续分布;粒宽表现出明显的双向超亲分离现象,但千粒重、粒长的超亲现象并不明显(图4)。此外,同一群体的三个性状之间均呈极显著正相关(<0.01)(表1)。
2.2 粒重和粒形QTL的初定位结果
应用同样衍生于EFT/FM9的F2:3和RIL群体开展千粒重、粒长、粒宽的QTL初定位。两套群体分别检测到29个和17个QTL。其中,11个QTL在两个群体中均能检测到,18个QTL仅在F2:3群体中检测到,6个QTL仅在RIL群体中检测到,因此,共定位到35个调控粒重粒形的QTL,分布在除第4和第8染色体外的其余10条染色体上。增效等位基因来自大粒亲本FM9的QTL占到29个(83%)。并且,有多个调控不同粒重粒形性状的QTL位于同一区间或相近位置。各性状的具体QTL定位结果如下(表2和图5)。
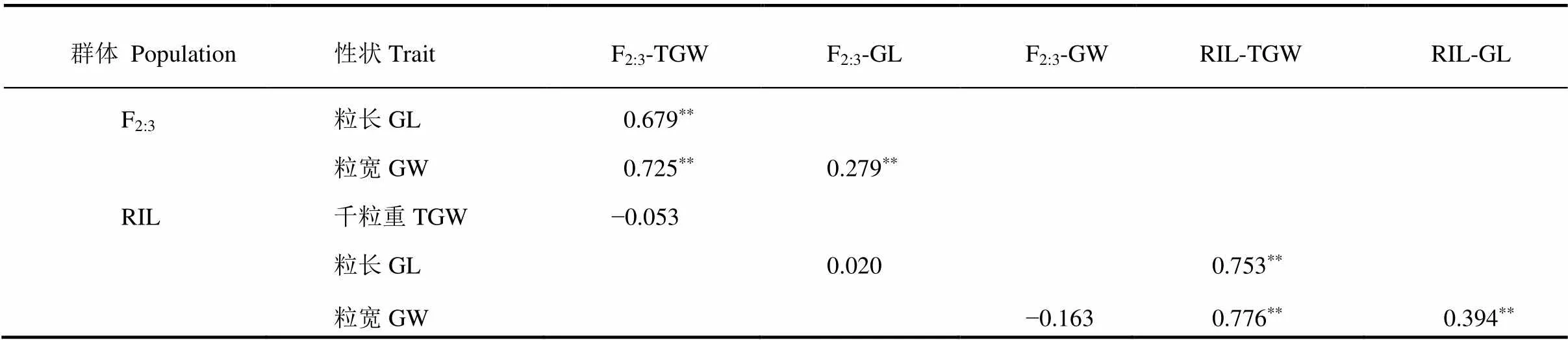
表1 EFT/FM9 F2:3和RIL群体粒型性状相关性分析
**表示在0.01水平上显著相关。
** Significantly correlated at 0.01 level.GL, Grain length; GW, Grain width; TGW, 1000-grain weight.
千粒重。分别在F2:3和RIL群体中检测到12和9个QTL,其中,两套群体同时检测到的QTL有5个,F2:3群体单独检测到的有7个,RIL群体单独检测到的有4个,因此,共定位到16个调控千粒重变异的QTL,分布在除第4、8和11染色体外的9条染色体上。其中,除了和之外,14个QTL的增效等位基因均来自大粒亲本FM9。的作用最强,在F2:3和RIL群体中均呈主效作用,来自FM9的等位基因增加粒重,加性效应分别为3.7和3.0 g,贡献率分别高达38.33%和34.24%;其余QTL的加性效应和贡献率均大幅减小,在F2:3群体中的加性效应为0.76~1.49 g,贡献率为1.76%~6.74%,在RIL群体中的加性效应为0.91~1.41 g,贡献率为3.16%~9.87%;此外,我们注意到QTL效应排名在2~5位的、、和也能同时在两套群体中起显著作用。
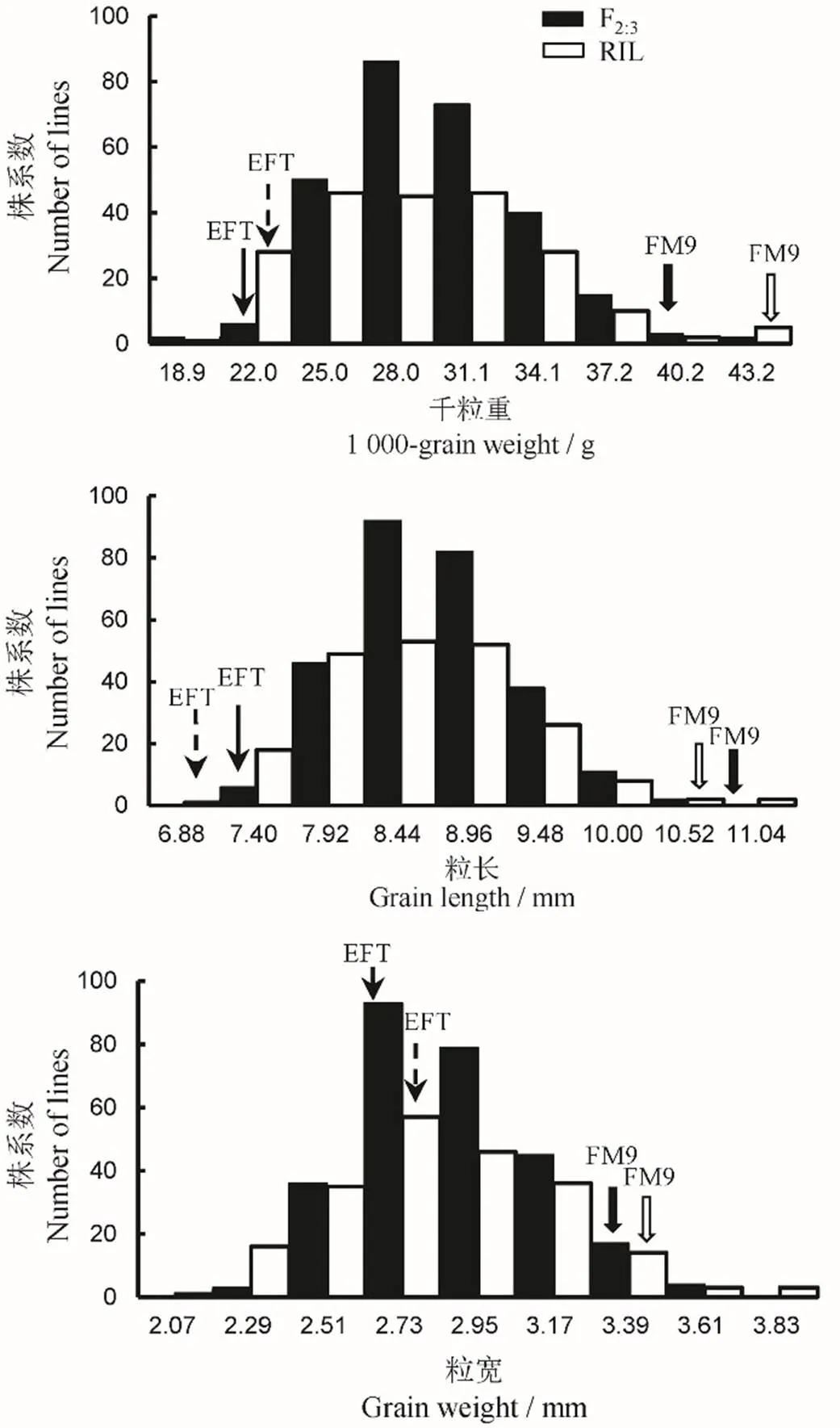
图4 EFT/FM9衍生的F2:3和RIL群体的粒重粒形性状的频率分布
Fig.4.Frequency distribution of grain weight and shape of EFT/FM9 derived F2:3and RIL populations.
粒长。分别在F2:3和RIL群体中检测到11和6个QTL,其中,两套群体同时检测到的QTL有4个,F2:3群体单独检测到的有7个,RIL群体单独检测到的有2个,因此,共定位到13个调控粒长变异的QTL,分布在除第4、8、10和12染色体外的8条染色体上。其中,除了和之外,11个QTL的增效等位基因均来自大粒亲本FM9。在与效应最大的千粒重QTL相同的区间上也检测到调控粒长的QTL,该位点同时在F2:3和RIL群体中起作用,加性效应分别为0.21和0.32 mm,贡献率分别为6.35%和18.24%。此外,我们还检测到另一个效应更大的粒长QTL,该位点在两群体中的加性效应分别为0.44和0.38 mm,贡献率分别高达32.65%和25.95 %。和两个位点的累加对两套群体的粒长变异的贡献分别可达39.00%和44.19%。其余位点的作用较弱,加性效应变幅为0.12~0.22 mm,贡献率介于2.27%~8.25%。

图5 在F2:3和RIL群体中检测到的粒重和粒形QTL
Fig.5.QTLs for grain weight and shape detected in F2:3and RIL populations.

表2 F2:3和RIL群体中检测到的粒重粒形QTL
Add−加性效应,指一个EFT等位基因取代一个FM9等位基因所产生的遗传效应。PVE-贡献率,指相应QTL所解释的群体表型方差的比例。a初定位QTL区间包含或相邻的已克隆粒重粒形QTL,其中,穗粒数基因对粒重粒形具有多效性。
Add, Additive effect measured as the genetic effect when the FM9 allele is replaced by the EFT allele.PVE, Proportion of phenotypic variance explained by the QTL effect.aCloned QTL for grain weight and shape located in the given region, among whichare grainnumber genes showing pleiotropic effects on grain weight and shape.
粒宽。分别在F2:3和RIL群体中检测到6个和2个QTL,其中,两套群体同时检测到的QTL有2个,F2:3群体单独检测到的有4个,因此,共定位到6个调控粒宽变异的QTL,分布在第2、3、5、11和12染色体上。其中,和的增效等位基因来源于小粒亲本EFT,与相邻及相同区间的和的等位效应方向相同,其余4个粒宽QTL的增效等位基因来自大粒亲本FM9。在被检测到的6个QTL中,存在2个主效位点的分离,即和,这两个位点在两套群体中均起显著作用,但效应方向相反。在F2:3和RIL群体中的加性效应分别为0.20和0.17 mm,贡献率分别为30.95%和24.96%,大粒亲本FM9的等位基因起增加粒宽的作用;的加性效应分别为0.15 mm和0.13 mm,贡献率分别为19.25%和15.49%,粒宽的增效等位基因却来源于小粒亲本EFT。这两个效应方向相反的主效位点的分离,可能是造成F2:3和RIL群体粒宽变异双向超亲分离现象相比千粒重和粒长变异更加明显的原因。其余4个QTL的遗传效应均大幅减小,加性效应变幅为0.05~0.07 mm,贡献率为2.93%~4.42%。
2.3 对qTGW1.2/ qGL1.1的验证
鉴于第1染色体长臂RM315−RM12138区间能同时在F2:3和RIL群体中检测到对千粒重()和粒长()的显著作用,并且暂未见该区间的进一步验证与基因克隆研究,我们随即检测重组自交系群体,从中挑选到2个不同遗传背景的剩余杂合体单株RHL17和RHL124。它们在与RM315−RM12138交叠的FIR3596−FIR3892区间上(2.96 Mb)呈杂合,在其他背景区间上高度纯合(图2),通过自交,获得了2套F8分离群体,并进一步筛选目标区间的纯合单株,自交衍生相应的2套F8:9家系群体,用于区间的粒重和粒长效应验证。
在初定位的F2:3和RIL群体中,区间对千粒重的加性效应分别为1.08和0.94 g,平均1.01 g;对粒长的加性效应分别为0.15和0.21 mm,平均0.18 mm。在两套F8分离群体D1和D2中,区间的大粒亲本FM9纯合型、小粒亲本EFT纯合型以及杂合型材料之间的千粒重和粒长均有极显著的表型差异,其中杂合型株系的表型值居中;D1和D2群体均未在该区间检测到对粒宽的显著作用,与初定位的结果吻合(图6)。该QTL区间在D1和D2群体中的增效等位基因都来自大粒亲本FM9,对千粒重的加性效应分别为1.16和1.22 g,对粒长的加性效应分别为0.22和0.23 mm。在两套F8:9家系群体E1和E2中,两个亲本纯合型材料之间的千粒重和粒长亦均呈极显著的差异,同样对粒宽无显著效应,加性效应方向不变,对千粒重的加性效应分别为0.89 g和1.18 g,对粒长的加性效应分别为0.21 mm和0.22 mm(表3)。以上结果验证了区间对千粒重和粒长的遗传效应。
2.4 颖壳表皮细胞的扫描电镜观察及细胞周期和粒形相关基因的表达分析
水稻谷粒大小通常由颖壳细胞的大小和数目决定[7, 39]。为了探究是如何通过调控颖壳细胞的分裂和生长影响粒长,我们利用扫描电镜比较了E1群体中两种亲本纯合型FM9和EFT之间成熟谷粒外颖细胞的数目和大小(图7-A、B)。结果表明,相较于短粒形EFT,长粒形FM9外颖细胞长度极显著增加,细胞宽度却显著减小,我们推测长粒形FM9的粒长增加可能主要由其颖壳细胞的长度增加所造成,或也有可能通过调控细胞周期,影响细胞增殖而造成粒长的变化。因此,我们进一步检测了18个细胞周期相关基因、6个生长素相关基因和3个主效粒形基因(,,)在FM9和EFT的幼穗中的表达情况。结果显示,16个相关基因表达水平表现出显著变化,包括9个细胞周期相关基因、4个生长素相关基因和3个主效粒形基因(图7-C)。其中,针对3个主效粒形基因,我们发现相比小粒EFT,在大粒FM9中,调控粒宽的和的表达水平分别显著下降与上升,而调控粒长的显著上升,该结果与主要调控粒长的变化相符。以上结果表明,可能参与、、介导的粒形调控途径,通过调控细胞周期和细胞大小的变化造成颖壳细胞长度以及谷粒长度的改变。

**表示在0.01水平上差异显著。
Fig.6.Comparison of plant architecture, grain shape and grain weight among different genotypes ofin the D1 population (A-C) and the D2 population (D-F) of advanced generations.
A−FM9和EFT的颖壳细胞(标尺:200 μm);B−外颖细胞长度和宽度的比较(*, **分别表示在 0.05 和 0.01 水平上显著相关,=30);C−FM9和EFT幼穗中细胞周期与粒形相关基因的表达水平比较。−A型细胞周期蛋白依赖性激酶1;−B型周期蛋白依赖性激酶2;− A型周期蛋白依赖性激酶1-1;−A型周期蛋白依赖性激酶2-1;−B型周期蛋白依赖性激酶1-1;−B型周期蛋白依赖性激酶2-2;−D型周期蛋白依赖性激酶4-1;−D型周期蛋白依赖性激酶4-2;−突触融合蛋白相关蛋白KNOLLE;− T型细胞周期蛋白依赖性激酶1;−U型细胞周期蛋白依赖性激酶4-3;−U型细胞周期蛋白依赖性激酶4-1;MAD2−细胞扩增相关基因MAD2;− E2F转录因子2;−丝裂原活化蛋白激酶;−微型染色体维持蛋白2;−微型染色体维持蛋白3;−微型染色体维持蛋白4;−粒宽基因;−粒长基因;−粒宽基因;−PIN蛋白1a;− PIN蛋白1;−大粒基因;−Aux/IAA蛋白11;−生长素应答因子19;−色氨酸转氨酶。
A, Local outer surfaces ofFM9andEFTspikelet hulls.(Bars = 200 µm); B, Comparison of the cell length and width of outer glume cells (*, **Significant at 0.05 and 0.01 levels, respectively,=30); C, Two parental homozygous genotypesFM9andEFTare involved in the expression levels of cell cycle- and grain shape-related genes in young panicles.,Cyclin-dependent kinase A-1;,B-type cyclin-dependent kinase 2;1;, A-type cyclin 1;1;,A-type cyclin 3;1;,B-type cyclin 1;,B-type cyclin 2;2;,D-type cyclin 4;1;,D-type cyclin 4;2;,Syntaxin-related protein KNOLLE;, T-type cyclin 1;,U-type cyclin 4;3;,Cadmium tolerant 2;,Cell expansion-related gene MAD2;, E2F transcription factor 2;,Mitogen-activated protein kinase;,Mini-chromosome maintenance protein 2;,Mini-chromosome maintenance protein 3;,Mini-chromosome maintenance protein 4;, Grain weight 2;, Grain size 3;, Grain width 5;,Pin protein 1a;, Pin-formed 1b;,Big grain 1;,Aux/IAA protein 11;,Auxin response factor 19;,Tillering and small grain 1.
图7 携带的FM9纯合型(FM9)和EFT纯合型(EFT)材料的颖壳表皮细胞扫描电镜观察和相关基因的表达分析
Fig.7.Scanning electron observation and quantitative expression analysis of related genes inFM9andEFT.
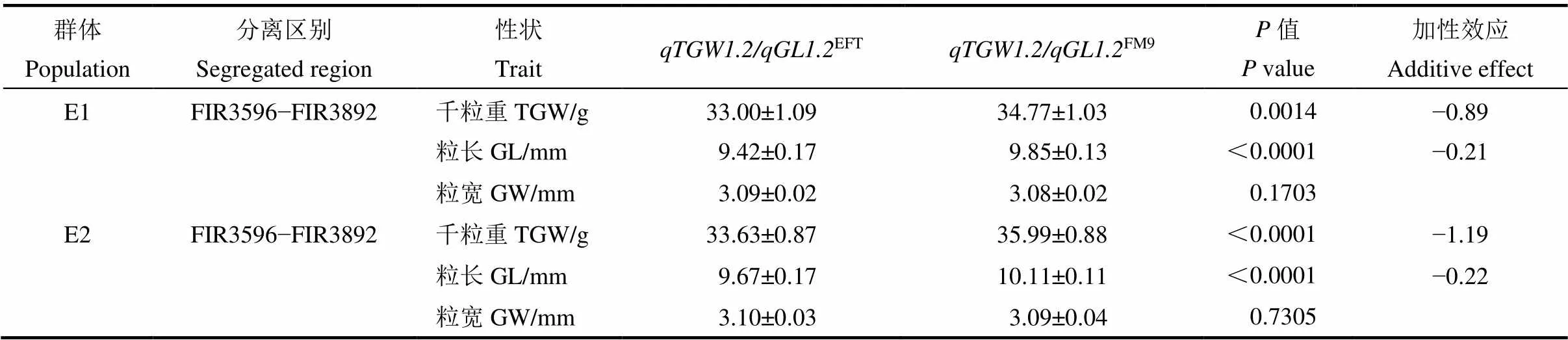
表3 高世代分离群体E1和E2中qTGW1.2/qGL1.2的效应分析
加性效应是指1个EFT等位基因取代1个FM9等位基因所产生的遗传效应。
Additive effect, Genetic effect when the FM9 allele is replaced by the EFT allele.
3 讨论
我们应用千粒重、粒长、粒宽分别相差20.85 g、3.99 mm、0.66 mm的大粒亲本FM9与小粒亲本EFT配组衍生F2:3和RIL两套初定位群体开展粒重粒形的QTL分析,定位到16个千粒重QTL(两套群体共定位的5个),13个粒长QTL(共定位4个)和6个粒宽QTL(共定位2个)。相较于RIL群体,F2:3群体检测到的QTL更多,其累计解释的群体千粒重变异可达77.97%,粒长变异73.80%,粒宽变异64.66%;当进一步整合RIL群体独立检测到的QTL后,更充分地揭示出大粒亲本FM9与小粒亲本EFT之间粒重粒形差异的遗传控制基础。但是,我们注意到,在前人的QTL初定位研究中,与相同组合衍生的F2等低世代临时群体相比,RIL等高世代永久群体定位到的QTL个数普遍更多[40-46];而本研究的QTL定位结果却相反,虽然两套群体对主效QTL的检测都很稳定,但是RIL群体对微效QTL的检测能力明显不足;这其中的一个重要原因可能是微效QTL容易受到环境和遗传背景的双重影响,使得这类QTL在RIL群体中不能有效表达,但具体的机制还需要进一步的研究加以验证。
三个性状也都检测到贡献率>30%的主效QTL,如调控千粒重的、粒长的和粒宽;这些结果与粒重粒形的性状遗传力高,是由少数主效和多数微效QTL共同调控的典型数量性状的普遍认识相符[17, 47-48]。另一方面,在千粒重与和粒长QTL的检测中,我们观察到包括主效QTL在内的绝大多数位点的增效等位基因均来自大粒亲本FM9,可能是造成F2:3和RIL群体中这两个性状没有表现超亲分离的主要原因;而相比在粒宽的QTL检测中,增效等位基因的分布平均,且两个主效位点和的加性效应方向相反,就使得F2:3和RIL群体的粒宽频率分布出现了明显的超亲现象。
针对我们验证的第1染色体长臂区间,发现在其两侧已分解出7个粒重粒形QTL,比如位于着丝粒方向的QTL、和,靠端粒方向的、、-和-[6, 49-54]。我们注意到,其中的6个QTL来源于同一组合“珍汕97/密阳46”,它们的增效等位基因既有来自珍汕97也有来自密阳46,但这些QTL的效应普遍较小,千粒重加性效应变幅仅为0.12~0.39 g,粒长仅为0.004~0.037 mm,粒宽仅为0.006~0.023 mm。我们验证的并未在珍汕97/密阳46的组合中得到鉴定,且千粒重和粒长加性效应较大,分别达到1.11 g和0.22 mm;此外,Zhang等[41]在日本晴/93-11群体中检测的也未在珍汕97/密阳46的组合中起显著作用,其粒长效应也可达0.26 mm。从中可以看出,粒重粒形QTL可通过连锁排列的方式成簇分布于染色体某一特定区段,并以不同的等位基因组合出现在不同的水稻品种中,因此,通过配组不同类型亲本,进一步分解、鉴定QTL簇中潜在的调控基因,有助于全面解析粒重粒形的调控机制,可更有效地借助分子技术进行水稻粒重粒形的改良。
在本研究检测的35个QTL中,13个包含或邻近已克隆粒重粒形QTL,而且多效性区间均有主效QTL的报道,比如与,与,与,与,以及9与(表2)。针对其中3个主效水稻粒重粒形基因、和,我们初步分析了大粒亲本FM9与小粒亲本EFT间的基因型差异。首先,借助功能分子标记-Ⅰ,N1212del分别对和位点进行基因分型[56],结果显示,大粒亲本FM9携带不能被Ⅰ内切酶酶切的长粒基因型,以及不含950 bp缺失的宽粒基因型,小粒亲本EFT则相反,该结果与预期相符(附图1)。然而,在位点上,我们未发现前人报道的在第4个外显子上的1 bp移码突变;并且通过对包括928 bp启动子在内的基因区间进行测序分析[56],也未能鉴定到可能引起其编码蛋白环型E3泛素连接酶功能缺失的关键序列差异(附图2),该结果一方面表明可能以其他遗传机制调控FM9与EFT之间粒重粒形变异,另一方面也暗示可能存在新的调控基因,毕竟区间除对粒重和粒宽起作用外,还显著影响粒长。
在后续的研究中,除了深入开展的精细定位与克隆研究,针对尚未克隆的且能在不同群体中稳定表达的的进一步分析鉴定,将有助于完善我们对水稻粒重和粒形遗传基础的认识。
辅助信息:有1个辅助性表格和2个辅助性图片放在中国水稻科学网站(http://www.ricesci.cn)上。
[1] Wang A H, Hou Q Q, Si L Z, Huang X H, Luo J H, Lu D F, Zhu J J, Shangguan Y Y, Miao J S, Xie Y F, Wang Y C, Zhao Q, Feng Q, Zhou C C, Li Y, Fan D L, Lu Y Q, Tian Q L, Wang Z X, Han B.The PLATZ transcriptionfactoraffects grain length and number in rice[J]., 2019, 180(4): 2077-2090.
[2] 罗玉坤, 朱智伟, 陈能, 段彬伍, 章林平.中国主要稻米的粒型及其品质特性[J].中国水稻科学, 2004(2): 49-53.
Luo Y K, Zhu Z W, Chen N, Duan B W, Zhang L P.Grain types and related quality characteristics of rice in China[J]., 2004(2): 49-53.(in Chinese with English abstract)
[3] 杨联松, 白一松, 张培江, 许传万, 胡兴明, 王伍梅, 佘德红, 陈桂芝.谷粒形状与稻米品质相关性研究[J].杂交水稻, 2001(4): 51-53, 57.
Yang L S, Bai Y S, Zhang P J, Xu C W, Hu X M, Wang W M.Studies on the correlation between grain shape and grain quality in rice[J].2001(4): 51-53, 57.(in Chinese with English abstract)
[4] Huang R Y, Jiang L R, Zheng J S, Wang T S, Wang H C, Huang Y M, Hong Z L.Genetic bases of rice grain shape: So many genes, so little known[J]., 2013, 18(4): 218-226.
[5] Xing Y Z, Zhang Q F.Genetic and molecular bases of rice yield[J]., 2010, 61(1): 421-442.
[6] Chan A N, Wang L L, Zhu Y J, Fan Y Y, Zhuang J Y, Zhang Z H.Identification through fine mapping and verification using CRISPR/Cas9-targeted mutagenesis for a minor QTL controlling grain weight in rice[J]., 2021, 134: 327-337.https://doi.org/10.1007/s00122-020-03699-6.
[7] Li N, Xu R, Duan P, Li Y H.Control of grain size in rice[J]., 2018, 31: 237-251.
[8] 康艺维, 陈玉宇, 张迎信.水稻粒型基因克隆研究进展及育种应用展望[J].中国水稻学, 2020, 34(6): 479-490.
Kang Y W, Chen Y Y, Zhang Y X.Research progress and breeding prospects of grain size associated genes in rice[J]., 2020, 34(6): 479-490.(in Chinese with English abstract)
[9] Yamamoto T, Yonemaru J, Yano M.Towards the understanding of complex traits in rice: Substantially or superficially[J]., 2009, 16: 141-154.
[10] Takai T, Ikka T, Kondo K, Nonoue Y, Ono N, Arai-Sanoh Y, Yoshinaga S, Nakano H, Yano M, Kondo M, Yamamoto T.Genetic mechanisms underlying yield potential in the rice high-yielding cultivar Takanari, based on reciprocal chromosome segment substitution lines[J]., 2014, 14(1): 295.
[11] Nagata K, Ando T, Nonoue Y, Mizubayashi T, Kitazawa N, Shomura A, Matsubara K, Ono N, Mizobuchi R, Shabaya T, Ogisotanaka E, Hori K, Yano M, Fukuoka S.Advanced backcross QTL analysis reveals complicated genetic control of rice grain shape in a×cross[J]., 2015, 65(4): 308-318.
[12] 王琳琳, 陈玉宇, 郭梁, 张宏伟, 樊叶杨, 庄杰云.水稻第1染色体区域粒重组分性状QTL的剖析[J].中国水稻科学, 2015, 29(3): 232-240.
Wang L L, Chen Y Y, Guo L, Zhang H W, FanY Y, Zhuang J Y.Dissection of quantitative trait loci for grain weight and its component traits in theregion on chromosome 1 of rice[J]., 2015, 29(3): 232-240.(in Chinese with English abstract)
[13] 朱安东, 孙志超, 朱玉君, 张荟, 牛小军, 樊叶杨, 张振华, 庄杰云.应用剩余杂合体衍生群体定位水稻粒重粒形QTL[J].中国水稻科学, 2019, 33(2): 144-151.
Zhu A D, Sun Z C, Zhu Y J, Zhang H, N X J, Fan Y Y, Zhang Z H, Zhuang J Y.Identification of QTL for grain weight and grain shape using populations derived from residual heterozygous lines of indica rice[J]., 2019, 33(2): 144-151.(in Chinese with English abstract)
[14] Ye H, Foley M E, Gu X Y.New seed dormancy loci detected from weedy rice-derived advanced populations with major QTL alleles removed from the background[J]., 2010, 179(6): 612-619.
[15] Bai X, Wu B, Xing Y.Yield-related QTLs and their applications in rice genetic improvement[J]., 2012, 54: 300-311.
[16] Wang Z, Chen J Y, Zhu Y J, Fan Y Y, Zhuang J Y.Validation of, a quantitative trait locus for grain size on the long arm of chromosome 10 in rice (L.)[J]., 2017, 16(1): 16-26.
[17] Zhang H W, Fan Y Y, Zhu Y J, Chen J Y, Yu S B, Zhuang J Y.Dissection of theregion into twotightly-linked minor QTLs having stable effects for grain weight in rice[J]., 2016, 17: 98.
[18] 王建康.数量性状基因的完备区间作图方法[J].作物学报,2009, 35(2): 239-245.
WangJ K.Inclusive composite interval mapping of quantitative trait genes[J]., 2009, 35(2): 239-245.(in Chinese with English abstract)
[19] McCouch S R, CGSNL (Committee on Gene Symbolization, Nomenclature and Linkage, Rice Genetics Cooperative).Gene nomenclature system for rice[J]., 2008, 1(1): 72-84.
[20] Ruan B P, Shang L S, Zhang B, Hu J, Wang Y X, LIN H, Zhang A P, Liu C L, P Y L, Zhu L, Ren D Y, Shen L, Dong G J, Zhang G H, Zeng D L, Guo L B, Qian Q, Gao Z Y.Natural variation in the promoter ofdetermines grain width and weight in rice[J]., 2020, 227(2): 629-640.
[21] Song X J, Huang W, Shi M, Zhu M Z, Lin H X.A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase[J].2007, 39: 623-630.
[22] Fan C C, Xing Y Z, Mao H L, Lu T T, Han B, Xu C G, Li X H, Zhang Q F., a major QTL for grain length and weight and minor QTL for grain width and thickness in rice, encodes a putative transmembrane protein[J]., 2006, 112: 1164-1171.
[23] Mao H L, Sun S Y, Yao J L, Wang C R, Yu S B, Xu C G, Li X H, Zhang Q F.Linking differential domain functions of theprotein to natural variation of grain size in rice[J]., 2010, 107: 19579-19584.
[24] Dong N Q, Sun Y W, Guo T, Shi C L, Zhang Y M, Kan Y, Xiang Y H, Zhang H, Yang Y B, Li Y C, Zhao H Y, Yu H X, Lu Z Q, Wang Y, Ye W W, Shan J X, Lin H X.UDP-glucosyltransferase regulates grain size and abiotic stress tolerance associated with metabolic fux redirection in rice[J]., 2020, 11: 2629.
[25] Li Y B, Fan CC, Xing Y Z, Jiang Y H, Luo L J, Sun L, Shao D, Xu C J, Li X H, XiaoJ H, He Y Q, Zhang Q F.Natural variation inplays an important role in regulating grain size and yield in rice[J].2011, 43(12): 1266-1269.
[26] Xu C J, LiuY, Li Y B, Xu X B, Xu C J, Li X H, Xiao J H, Zhang Q F.Differential expression ofregulates grain size in rice[J]., 2015, 66(9): 2611-2623.
[27] Duan P G, Xu J S, Zeng D L, Zhang B L, Geng M F, Zhang G Z, Huang K, Huang L J, Xu R, Ge S, Qian Q, Li Y H.Natural variation in the promoter ofcontributes to grain size diversity in rice[J]., 2017, 10: 685-694.
[28] Liu J, Chen J, Zheng X, Wu F, Lin Q, Heng Y, Cheng Z, Zhou K, Lin Q, Zhang X, Guo X, Wang J, Wang H, Wan J.acts in the brassinosteroid signaling pathway to regulate grain width and weight in rice[J]., 2017, 3: 17043.
[29] Shomura A, Izawa T, Ebana K, Ebitani T, Kanegae H, Konishi S, Yano M.Deletion in a gene associated with grain size increased yields during rice domestication[J]., 2008, 40: 1023-1028.
[30] Wan X Y, Weng J F, Zhai H Q, Wang J K, Lei C L, Liu X L, Guo T, Jiang L, Su N, Wan J M.Quantitative trait loci (QTL) analysis for rice grain width and fine mapping of an identified QTL allelein a recombination hotspot region on chromosome 5[J]., 2008, 179: 2239-2252.
[31] Weng J, Gu S, Wan X, Gao H, Guo T, Su N, Lei C, Zhang X, Cheng Z, Guo X, Wang J, Jiang L, Zhai H, Wan J.Isolation and initial characterization of, a major QTL associated with rice grain width and weight[J]., 2008, 18: 1199-1209.
[32] Yan C J, Zhou J H, Yan S, Chen F, Yeboah M, Tang S Z, Liang G H, Gu M H.Identification and characterization of a major QTL responsible for erect panicle trait inrice (L.)[J]., 2007, 115(8): 1093-1100.
[33] Huang X, Qian Q, Liu Z, Sun H, He S, Luo D, Xia G, Chu C, Li J, Fu X.Natural variation at thelocus enhances grain yield in rice[J]., 2009, 41(4): 494-497.
[34] Fumio T S, Yasushi K, Hiroshi K, Haruko O, Akemi T, Naho H, Akio M, Hirohiko H, Hidemi K, Masahiro Y, Seiichi T.A loss-of-function mutation of ricecauses semi-dwarfness and slightly increased number of spikelets[J]., 2011, 61(1): 17-25.
[35] Sun H, Qian Q, Wu K, Luo J, Wang S, Zhang C, Ma Y, Liu Q, Huang X, Yuan Q, Han R, Zhao M, Dong G, Guo L, Zhu X, Gou Z, Wang W, Wu Y, Lin H, Fu X.Heterotrimeric G proteins regulate nitrogen-use efficiency in rice[J]., 2014, 46(4): 652-656.
[36] Li M, Li X, Zhou Z, Wu P, Fang M, Pan X, Lin Q, Luo W, Wu G, Li H.Reassessment of the four yield-related genes,,, andin rice using a CRISPR/Cas9 system[J]., 2016, 7: 377.
[37] Liu Q, Han R, Wu K, Zhang J, Ye Y, Wang S, Chen J, Pan Y, Li Q, Xu X, Zhou J, Tao D, Wu Y, Fu X.G-protein βγ subunits determine grain size through interaction with MADS-domain transcription factors in rice[J]., 2018, 9: 852.
[38] Sun S, Wang L, Mao H, Shao L, Li X, Xiao J, Ouyang Y, Zhang Q.A G-protein pathway determines grain size in rice[J]., 2018, 9: 851.
[39] Li, N, Li Y H.Signaling pathways of seed size control in plants[J]., 2016, 33: 23-32.
[40] Yu S.B, Li J.X, Xu C.G, Tan Y.F, Gao Y.J, Li X.H, Zhang Q F, Maroof S.Importance of epistasis as the genetic basis of heterosis in an elite rice hybrid[J]., 1997, 94: 9226-9231.
[41] Zeng Y X, Ji Z J, Wen Z H, Liang Y, Yang C D.Combination of eight alleles at four quantitative trait loci determines grain length in rice[J]., 2016, 11:3.
[42] Odonkor S, Choi S, Chakraborty D, Martinez-Bello L, Wang X, Bahri BA, Tenaillon MI, Panaud O, Devos KM.QTL mapping combined with comparative analyses identified candidate genes for reduced shattering in[J]., 2018, 9: 918.
[43] Li J X, Yu S B, Xu C G, Xu C G, Tan Y F, Gao Y J, Li X H, Zhang Q F.Analyzing quantitative trait loci for yield using a vegetatively replicated F2population from a cross between the parents of an elite rice hybrid[J]., 2000, 101: 248-254.
[44] 庄杰云, 樊叶杨, 吴建利, 夏英武, 郑康乐.应用二种定位法比较不同世代水稻产量性状QTL的检测结果[J].遗传学报, 2001(5): 458-464.
Zhuang J Y, Fan Y Y, Wu J L, Xia Y W, Zheng K L.Comparison of the detection of QTL for yield traits in different generations of a rice cross using two mapping approaches[J]., 2001(5): 458-464.
[45] 姜树坤, 张喜娟, 黄成, 邢亚南, 郑旭, 徐正进, 陈温福.基于粳稻F2和F2:6群体的连锁图谱及剑叶性状QTL比较分析[J].中国水稻科学, 2010, 24(4): 372-378.
Jiang S K, Zhang X J, Huang C, Xing Y N, Zheng X, Xu Z J, Chen W F.Comparison of genetic linkage map and QTLs controlling flag leaf traits based on F2and F2:6populations derived fromrice[J].,2010, 24(4): 372-378.(in Chinese with English abstract)
[46] 郭小蛟, 张涛, 蒋开锋, 杨莉, 曹应江, 杨乾华, 游书梅, 万先齐, 罗婧, 李昭祥, 高磊, 郑家奎.水稻籼粳交F8、F2群体穗长QTL比较分析[J].中国农业科学, 2013, 46(23): 4849-4857.
Guo X J, Zhang T, Jiang K F, Yang L, Cao Y J, Yang Q H, You S M, Wan X Q, Luo J, Li ZX, Gao L, Zheng J K.Comparison of panicle length QTL based on F2and F8populations derived from rice subspecies cross[J]., 2013, 46(23): 4849-4857.(in Chinese with English abstract)
[47] Mackay T F C, Stone E A, Ayroles J F.The genetics of quantitative traits: Challenges and prospects[J].2009, 10: 565-577.
[48] Kumar J, Gupta D S, Gupta S, Dubey S, Gupta P, Kumar S.Quantitative trait loci from identification to exploitation for crop improvement[J].2017, 36: 1187-1213.
[49] 李盼盼, 朱玉君, 郭梁, 庄杰云, 樊叶杨.利用剩余杂合体衍生的近等基因系精细定位水稻粒长微效QTL[J].中国水稻科学,2020,34(2):125-134.
Li P P, Zhu Y J, Guo L, Zhuang J Y, Fan Y Y.Fine mapping of, a minor QTL for grain length, using near isogenic lines derived from residual heterozygotes in rice[J].2020, 34(2): 125-134.(in Chinese with English abstract)
[50] Zhang B, Shang L G, Ruan B P, Zhang A P, Yang S L, Jiang H Z, Liu C L, Hong K, Lin H, Gao Z Y, Hu J, Zeng D L, Guo L B, Qian Q.Development of three sets of high-throughput genotyped rice chromosome segment substitution lines and QTL mapping for eleven traits[J]., 2019, 12:33.
[51] Ma F Y, Du J, Wang D C, Wang H, Zhao B B.He G H, Yang Z L, Zhang T, Wu R H, Zhao F M.Identification of long-grain chromosome segment substitution line Z744 and QTL analysis for agronomic traits in rice[J]., 2020, 19(5): 1163-1169.
[52] Wang L L, Chen Y Y, Guo L, Zhang H W, Fan Y Y, Zhuang J Y.Dissection ofto three QTLs for grain weight and grain size in rice (L.) [J]., 2015, 202: 119-127.
[53] Wang W H, Wang L L, Zhu Y J, Fan Y Y, Zhuang J Y.Fine-mapping of, a quantitative trait locus for 1000-grain weight in rice[J]., 2019, 26(4): 220-228.
[54] Dong Q, Zhang Z H, Wang L L, Zhu Y J, Fan Y Y, Mou T M, Ma L Y, Zhuang J Y.Dissection and fine-mapping of two QTL for grain size linked in a 460-kb region on chromosome 1 of rice[J]., 2018, 11: 44.
[55] Yan S, Zou G H, Li S J, WangH, Li·H Q, Zhai G W, GuoP, Song H M, Yan C H, Tao Y Z.Seed size is determined by the combinations of the genes controlling different seed characteristics in rice[J].2011 123: 1173-1181.
[56] Lu L, Shao D, Qiu X J, Sun L, Yan W H, Zhou X C, Yang L, He Y Q, Yu S B, Xing W Z.Natural variation and artificial selection in four genes determine grain shape in rice[J]., 2013, 200(4):1269-1280.
QTL Analysis for Grain Weight and Shape and Validation of
DU Chengxing#, ZHANG Huali#, DAI Dongqing, WU Mingyue, LIANG Minmin, CHEN Junyu*, MA Liangyong*
(State Key Laboratory of Rice Biology and Chinese National Center for Rice Improvement, China National Rice Research Institute, Hangzhou 310006, China;#These authors contributed equally to this work;*Corresponding author, E-mail: chenjunyu@caas.cn, maliangyong@caas.cn)
【Objective】 Grain weight and shape have an important impact on the yield and quality of rice.The purpose is to further reveal the genetic mechanism of grain weight and shape in rice by primary mapping of QTLs for grain weight and shape and validating of the newly identifiedon the long arm of chromosome 1.【Method】 An F2:3population consisting of 277 individuals and a recombinant inbred line (RIL) population with 211 individuals were derived from the cross between the large-grain male parent FM9, and the small-grain female parent EFT.The1000-grain weight (TGW), grain length (GL) and grain width (GW) were measured.QTL mapping was performed by the inclusive composite interval mapping.As for theregion, two residual heterozygotes were screened and self-fertilized to produce segregated populations for QTL validation.【Results】 A total of 35 QTLs for TGW, GL and GW were detected by QTL primary mapping.Among them, 11 QTLs were detected in both populations, 18 QTLs were detected only in the F2:3population, and six QTLs only in the RIL population.The effects of the newly identifiedon TGW and GL were validated by using the segregated populations derived from the two residual heterozygotes.Additionally, significant variations in the length of glume cells were observed, and qPCR results demonstrated that the expression levels of genes related to cell cycle, auxin metabolism and grain shape were up- or down-regulated significantly.【Conclusion】 The primary mapping of 35 QTLs and the validating ofwill contribute to a deep insight into the genetic basis underlying grain weight andshape of rice, and also lay a foundation for gene cloning and marker assisted selection.
rice; grain weight; grain shape; QTL; residual heterozygote
10.16819/j.1001-7216.2021.201205
2020-12-08;
2021-01-26。
国家自然科学基金青年科学基金资助项目(31701398);国家863计划资助项目(2014AA10A604-15);浙江省农业(粮食)新品种选育重大科技专项(2016C02050-4);国家重点研发计划资助项目(2016YFD0101104)。
