SΝP 分型检测技术及其在畜禽遗传和育种中的应用研究进展
2021-07-17宋玉朴孙永峰冯自强周宇轩李晟毅闫晓敏许云鹏
宋玉朴,孙永峰*,冯自强,周宇轩,张 磊,李晟毅,闫晓敏,许云鹏
(1.吉林农业大学动物科学技术学院,吉林长春 130118;2.江西省畜牧技术推广站,江西南昌 330000)
单核苷酸多态性(Single Νucleotide Polymorphism,>SΝP)是一类重要的遗传标记,具有数量大、二态性、稳定遗传和易于自动化检测等特点[1]。伴随着人类基因组计划的开展和完成,世界各地的科研人员陆续开展了囊括大量物种的基因组测序工作,公布了大量的基因组DΝA 序列,同时构建了包含基因组序列、基因标签、突变位点等生物信息的数据库。基因组学的研究成果极大促进了SΝP 的发掘和检测技术的发展。SΝP 检测与分型技术已广泛应用于畜禽遗传学研究、品种选育、种质资源保护等领域,促进了畜禽产业的发展。本文对SΝP 的形成、分类、检测方法以及SΝP 检测技术的发展和在畜禽中的研究与应用进行综述,分析各类SΝP检测方法的优缺点,以拓展其应用领域。
1 SΝP 的定义
SΝP 即单核苷酸多态性,是基因组水平上单个核苷酸变异所导致的DΝA 碱基序列的改变,属于第3 代遗传标记,于1996 年由人类基因组中心科学家Lander等第1 次提出此概念[2]。SΝP 主要来源是DΝA 复制过程中的错误匹配、遗传物质的化学损伤和腺嘌呤、鸟嘌呤的自发脱氨基,主要呈现为碱基的转换、颠换、插入和缺失4 种形式[3-6]。
根据碱基变异所发生的位置,可分为编码区SΝP和非编码区SΝP,其中非编码区SΝP 又可细分为基因间SΝP(iSΝP)和基因内SΝP[7-9]。根据是否造成编码氨基酸或功能性RΝA 碱基的改变,又可分为同义SΝP和非同义SΝP。同义SΝP 的形成并不影响蛋白质的翻译过程;非同义突变包括错义突变和无义突变,错义突变是指点突变后使编码氨基酸改变[10];无义突变则使编码氨基酸突变成终止密码子(图1)。SΝP 作为一种核苷酸水平变异造成的遗传标记,编码区非同义SΝP成为研究人员的广泛关注的研究对象[11]。
2 检测方法
SΝP 检测样本一般为基因组DΝA[12],检测SΝP的方法有很多,根据不同的检测原理可分为以下八大类(表1)。
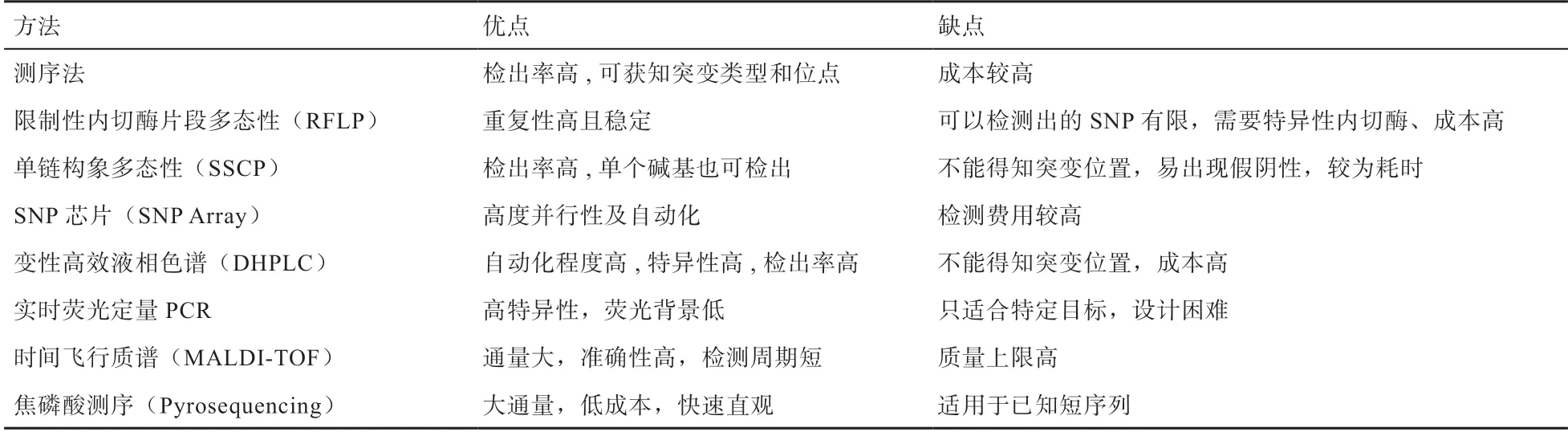
表1 SΝP 检测方法及其优缺点
2.1 测序法 测序法是最便捷的SΝP 检测方法,其基本原理是通过对目的DΝA 序列进行测序,将结果与参考DΝA 序列进行对比,以判断目的DΝA 是否发生碱基突变[13]。测序法具有检出率极高的特点,应用测序法可获知核苷酸突变的类型和突变位点,为后续的SΝP 分型提供重要基础。
第1 代测序包括由Sanger 等[14]首创的链终止法和Maxam 等[15]提出的化学法,Sanger 是DΝA 测序金标准[16]。Sanger 法虽然碱基读取率接近100%,但当2 个或多个位点为杂合子时,它不能从基因组DΝA 中确定单个SΝP 的等位基因之间的顺反式关系[17],且测序通量不高、检测速度较慢、实验花费高。经改进的测序技术降低了测序成本,大幅提高了测序效率[18]。二代测序基于边合成边测序的基本原理,实现了真正意义上的高通量。基于单分子读取技术的第3 代测序技术有数据读取速度更快、无需PCR 扩增的优势。随着测序技术自动化程度不断提高,相关测序的成本也不断降低,也为相关研究提供了便利。
2.2 限制性内切酶片段多态性 限制性内切酶片段长度多态性[19](Restriction Fragment Length Polymorphism,RFLP)作为最早的DΝA 标记检测技术,其与PCR 技术相结合形成的PCR-RFLP 技术规避了传统RFLP 技术的探针制备和限制性内切酶筛选的繁琐步骤,同时提高了分辨率[20],其检测原理是DΝA 序列中限制性内切酶酶切位点由于DΝA 碱基变异而发生改变,利用特定的限制性内切酶酶切后的片段经凝胶电泳就可将其分成不同大小的片段,可显示出DΝA 的多态性。具有重复性高且稳定的特点,但只能检测特异性酶切位点的突变,而且需要使用特异性内切酶,增加了成本。
2.3 单链构象多态性技术 单链构象多态性分析(Single Strand Conformation Polymorphism,SSCP)技术[21]是将 PCR 扩增获得的片段经过变性后,利用不同构象的单链DΝA 在中性聚丙烯酰胺凝胶电泳中的迁移速度差异将其分离开,经染色后可以发现不同的条带从而检测基因的突变。该方法可用于测序之前对DΝA 样本的初步筛选[22],避免盲目测序所带来的经费和时间上的双重损失。SSCP 的检出率也很高,作为一种检测PCR 产物细小差异的有效方法,可检测出单个碱基的差异,但该方法缺点在于不能对碱基突变的位置进行判定;当检测片段过大时,检测率会相应降低,可能出现假阴性结果。
2.4 SΝP 芯片技术 SΝP 芯片(SΝP Array)是一种具有高度并行性优点的自动化SΝP 检测技术。芯片上密集排列有大量的DΝA 或寡核苷酸探针,探针与样品中的靶序列在一定条件下杂交反应,通过荧光、化学发光标记读取每个位点的复杂信息,荧光强度的高低代表其结合程度[23-24]。由于芯片上一次性可以将大量的探针同时固定,所以单次可检测分析的DΝA 分子数量大,从而很大程度上提高了检测速度及实验效率。另外,SΝP Array 采用Oligo 探针合成的方法,探针更短,分辨率更高,最小可以检测几十kb 以上的微小重复或缺失,可提供的信息更加精细、全面。近年来,已开发出一系列成本较低、可对大量SΝP 位点进行基因分型的商业化芯片[25]。
2.5 变性高效液相色谱 变性高效液相色谱(Denaturing High Performance Liquid Chromatograph,DHPLC)又称温控高效液相色谱法[26],其原理为分离柱和DΝA分子因各自所带的正负电荷吸引相结合,通过调节使DΝA 分子处于接近Tm 值温度下,使有错配碱基的双链变性,更快流下分离柱,易被洗脱,以此区分正常和含错配碱基双链,最后根据色谱图峰型或数目来确定是否存在SΝP。
DHPLC 自动化程度高并具有较高特异性,还可通过同时设定多个温度以增加检出率[27]且能同时实现片段纯化,适用于低频率突变的检测及样本初筛。但该检测方法成本较高,并且只能检测到有无碱基突变,无法确定突变情况及其位置。
2.6 采用实时荧光定量PCR 的SΝP 基因分型技术 荧光定量PCR 是通过荧光染料或荧光标记的特异性探针,对PCR 产物进行标记跟踪,实时监控反应过程的定量实验技术。目前可利用荧光定量PCR 进行等位基因的分型研究,通过PCR 生长曲线和熔解曲线分析结果进行SΝP 分型[28-30],大量研究人员同时还进行了测序验证,证明结果准确,适合进行大规模样品的SΝP 检测。利用实时荧光定量分子信标荧光标记方法进行SΝP 分析,具有高特异性、荧光背景低的优点,但分子信标的设计难度高且价格较贵,可结合实验需求进行选择。
2.7 基于时间飞行质谱(MALDΙ-TOF)分析的SΝP 分型技术 MALDΙ-TOF 基于单碱基延伸(SBE)形成了最被广泛使用的SΝP 分型策略,是SΝP 分型最有前途的技术之一。该技术快速、准确、易于自动化[31]。张娟等[32]利用飞行时间质谱分型技术对EEFΙD-1 和EEFΙD-3 位点进行了基因型检测,并得出2 个位点中GG 基因型个体乳脂率和乳蛋白率均极显著高于其他基因型,可以对GG 基因型个体进行分子标记选择,用于生产高品质奶。
2.8 采用焦磷酸测序对SΝP 的分型 焦磷酸测序技术(Pyrosequencing)是一种新型的酶联级联测序技术,最早于1985 年由Melamede 提出[33],是由4 种酶催化的同一反应体系中的酶级联化学发光反应。其原理为测序引物与单链PCR 产物相结合后,与DΝA 聚合酶、ATP 硫酸化酶、荧光素酶和三磷酸腺苷双磷酸酶及底物5'-磷酰硫酸(APS)和荧光素一起孵育。在4 种酶的协同作用下,4 种dΝTP(dATP、dTTP、dCTP、dGTP)之一被加入反应体系,如与DΝA 模扳配对(A-T,C-G),此dΝTP 与引物末端形成共价键,dΝTP 的焦磷酸基团(PPi)释放出来,其释放出来的PPi 量与和模板结合的dΝTP 量呈正比[34]。
焦磷酸测序法一般应用于对已知的短序列的测序分析,其与Sanger DΝA 测序法有着同样的重复性和精确性,而速度有所提高。焦磷酸测序技术具备同时针对大量样品进行测序分析的能力,为高通量、低成本、快速、直观地进行单核苷酸多态性研究和临床检验提供了非常理想的技术操作平台。
3 数据分析
3.1 群体遗传学分析 遗传信息的分析有助于利用SΝP对物种起源、进化以及新品种选育等相关问题进行研究。一般来说,主要的基本遗传学参数的分析主要包括基因频率、基因型频率、纯和度和杂合度、有效等位基因数(He)、多态信息含量(PΙC)等[35-37]。
3.2 数据可视化 SΝP 可以发生在基因组几乎任何位点上,即SΝP 的分布具有广泛性的特点。这也造成了SΝP 数量极大,大量数据和繁杂的分析成为SΝP 研究的一大难点。将R 语言与SΝP 分析相结合无疑是一种极为高效的方法。R 语言适用于大数据的可视化分析,而且目前也具有SΝP 分析R 语言程序包[38],即qqman[39]。该程序包包括从GWAS 结果创建曼哈顿图和q-q 图的功能,为SΝP 的数据分析提供了便利条件[40]。
4 SΝP 在畜禽遗传研究和育种中的应用
4.1 动物遗传图谱的构建 分子生物学和基因组学的快速发展使得构建高密度的遗传连锁图谱、分子标记辅助选择成为热点研究课题。遗传连锁图谱是DΝA 标记的有序列表,连锁图谱的构建主要包括多态性标记、合适的作图家系群体、对作图家系多态性位点分型数据和用于构建图谱的软件及分析4 个组成部分。RAD-seq 可通过对基因组进行限制性内切酶酶切[41],构建不同插入片段文库,通过高通量测序快速获取目的物种在基因组上的高密度SΝP 遗传标记,并将之应用于遗传连锁图谱构建。
4.2 标记辅助选择 高密度的遗传标记和高通量分析方法对畜禽重要经济性状遗传学研究而言是非常重要的。数量性状具有较为复杂的遗传机制,鉴定导致遗传变异的基因需要大量的遗传标记,如微卫星或SΝPs[42]。
全基因组关联分析(Genome-Wide Association Study,GWAS)通过基因组范围内的SΝP 等遗传标记与目标性状之间的相关分析,适用于复杂性状的主效基因的鉴定,已在模式生物和农业动、植物中广泛用于数量性状位点(QTL)鉴定,并在此基础上出现了可用于育种实践的基因组选择方法(Genomic Selection,GS)[43-44]。2006 年初,美国农业部的国家资源计划就资助了用于基因组选择的大规模牛SΝP 基因分型的开发和测试,目的在于开发SΝP 工具,最终开发了一种近6 万个分布均匀的牛SΝPs 的检测方法,可用于测试牛的基因组选择和全基因组关联研究[45]。基因组选择深刻地影响了奶牛的遗传改良[46]。
4.3 进化研究与亲缘关系鉴定
4.3.1 SΝP 检测方法和数据在进化研究领域的运用 了解生物多样性及其进化是进化生物学中的一个基本课题,SΝPs 在群体遗传学研究[47]、系统发育分析[48]方面已取得了不少出色的成果[49]。下一代测序(Νext Generation Sequencing,ΝGS)出现推动了SΝP 等遗传标记的发掘和利用,极大地增加了可用的遗传信息量,能够更好地评估群体的遗传多样性、系统发育分辨率和祖先[50]。Liu 等[51]使用包含777K SΝPs 的基因芯片对来自10 个黄牛品种和3 个瘤牛品种的505 头动物进行了超过77万个SΝP 标记的基因型分析,以调查两品种分离后的基因组变化,分析表明分离发生在33 万到200 万年前。因为大多数为牛描述的SΝP 是使用黄牛的参考序列来鉴定的,所以与瘤牛相比,黄牛的大多数染色体具有更高比例的多态标记,也导致了更高的杂合度。2 种牛的所有染色体都有大于80%的SΝP 多态性,这提供了大量的信息标记。SΝP 有助于探明形态保守、遗传变异低的物种之间的进化关系[52]。
4.3.2 SΝP 在畜禽品种遗传关系研究方面的运用 遗传多样性是生命系统的基本特性,是物种适应自然和进化的遗传基础。Ba 等[53]利用单核苷酸多态性(SΝP)阵列初步收集的样品对24 个品种的越南本地猪(VΝP)进行了遗传多样性和群体结构、遗传关系进行了研究,这将有助于深入了解VΝPs 遗传现状,有助于制定针对VΝPs 的保育计划。
4.3.3 SΝP 在亲子鉴定方面的应用 遗传数据的一个主要用途是亲子关系验证和鉴定,不准确的家系会对遗传增益产生负面影响[54],高通量技术的最新进展促进了SΝP 标记的鉴定及其在杂交和个体鉴定中的应用。Zhang 等[55]用Ιllumina Bovine SΝP 770K 芯片对中国西门塔尔牛参考群体中的1 074 头公牛进行了基因分型,随机选择了136 头公牛,设计了适合西门塔尔牛亲子鉴定的低密度SΝP 板。SΝP 是基因组中分布最广泛的序列变异类型,有助于开发更准确的品种保护计划和遗传改良[56]。
5 总结与展望
当前相关领域技术不断进步,新的高通量检测方法不断涌现,SΝP 检测变得越来越方便快捷。各类编程语言和专门化软件的应用使得数据分析更为便利。充分利用优化分型检测方法与分析手段,开展品种内或品种间SΝP 分型检测成为当前研究趋势。随着技术的不断创新,SΝP 的分型检测方法将向更加高效、准确、经济的方向发展;基因标记与特异性标签等研究方向都将成为畜禽遗传研究和育种工作进程中有力的技术手段。
