甘薯基因组bHLH转录因子鉴定与逆境胁迫表达分析
2021-07-14黄小芳毕楚韵王和寿陈其俊胡韵卓黄碧芳杨志坚陈选阳林世强
黄小芳, 毕楚韵, 王和寿, 陈其俊, 胡韵卓, 黄碧芳, 许 明, 杨志坚, 陈选阳,5, 林世强,5
(1.福建农林大学生命科学学院,福建 福州 350002;2.福建农林大学作物生物技术福建省高校重点实验室, 福建 福州 350002;3.宁德市农业农村局,福建 宁德 352100;4.福建省种子总站 福建 福州 350003; 5.福建农林大学教育部作物遗传育种与综合利用重点实验室,福建 福州 350002)
转录因子是真核生物中重要的一类调节因子,通过与顺式元件相互作用来调节特定基因的表达以适应环境的胁迫[1].bHLH转录因子是植物中最大的转录因子家族之一,因其结构域由Basic-Helix-Loop-Helix组成而得名.bHLH结构域通常约由60个氨基酸组成,分为碱性氨基酸区和α螺旋-环-α螺旋区,碱性氨基酸区位于结构域N端,主要负责识别和结合特定DNA,根据其中所含的碱性氨基酸数目和特异位点氨基酸的类型,可分为E盒结合(5′-CANNTG-3′)和G盒结合(5′-CACGTG-3′);HLH区通过疏水性氨基酸的互作,促进蛋白形成同源/异源二聚体,从而使bHLH蛋白可以发挥功能作用[2].近年来,越来越多的研究发现bHLH转录因子在植物的生长发育、生物合成与信号转导、生物与非生物胁迫以及一些次生代谢物的合成等方面均有参与.例如bHLH转录因子MYC2可通过整合各种决定植物生长发育的内源性和外源性信号,在多个信号传导途径中充当调节中心,而且还可以通过调节拟南芥中脯氨酸的生物合成来调节盐胁迫下的耐受性[3];研究表明[4],拟南芥中bHLH121的功能缺失突变会引起严重的铁缺乏症状,减少铁的积累,并破坏与铁稳态相关基因的表达,bHLH121可与bHLH IVc共同正向调节FIT(铁缺乏症的转录因子)的表达以维持拟南芥中的铁稳态.bHLH转录因子还参与调节植物的免疫反应,在稻瘟病菌感染期间,水稻OsbHLH6基因表达上调,OsbHLH6通过动态调节水杨酸和茉莉酸的信号传导来调节水稻在稻瘟病菌胁迫下的防御机制[5].目前对甘薯中bHLH转录因子的功能研究还未见报道.
本研究以甘薯泰中6号基因组测序数据为基础[6],利用生物信息学方法对甘薯bHLH转录因子进行检索和鉴定,对其基因结构、结构域保守性、基因在染色体的位置分布、系统进化树以及真菌病害胁迫下和低温胁迫下的基因差异表达进行分析,为甘薯bHLH转录因子在抗性育种方面的应用提供参考,同时丰富bHLH转录因子在不同物种中的研究.
1 材料与方法
1.1 材料
从NCBI基因组数据库下载甘薯全基因组序列,利用snap程序中附带的HMM模型(At.hmm, Ce.hmm, Os.hmm)检索甘薯15条染色体序列,得到178 458个CDS序列[7].
1.2 甘薯bHLH基因家族鉴定
从拟南芥数据库网站https://www.arabidopsis.org/index.jsp下载拟南芥bHLH序列,以此为基础建立bHLH转录因子数据库.利用blastp将甘薯全基因组蛋白序列与数据库中的序列进行比对(阈值设置为默认值),比对的输出文件中比对成功的甘薯蛋白序列,统计每一条序列比对上的拟南芥bHLH序列数,选取其中与一半以上的拟南芥bHLH序列比对成功的甘薯蛋白序列为基础,利用hmmbuild程序构建甘薯特异的bHLH HMM模型.构建好的HMM模型重新搜索甘薯所有的蛋白序列获得含有bHLH结构域的甘薯蛋白序列.利用CDD、Interproscan和Clustal Omega对获得的序列进行验证分析,去除不含bHLH结构域的序列;利用augustus程序[8]对结构域不完整的序列进行二次CDS区检索以修补序列,并结合IGV软件比较查看转录组测序数据[9]的reads对应bHLH序列在基因组区间的比对情况,最终得到甘薯bHLH转录因子基因家族序列.
1.3 bHLH结构域保守性分析
利用Clustal Omega[10]对甘薯bHLH序列进行多重比对.将比对后的甘薯bHLH结构域与水稻bHLH结构域的19个保守位点相比较,根据各个位点氨基酸的类型、数目、位置等信息确定甘薯bHLH结构域的19个保守位点,并统计各个位点的氨基酸组成特征.
1.4 甘薯bHLH基因染色体定位
统计甘薯15条染色体的序列长度,根据snap程序获得到ZFF文件中甘薯所有CDS序列的基因位置信息,对每个bHLH序列的位置信息进行查找并统计,利用circos程序[11]绘制圈图,标出bHLH基因在甘薯各染色体上的位置.为了寻找具有潜在复制关系的bHLH基因对,利用blastn程序两两比对甘薯bHLH基因序列,具有潜在复制关系的基因对在圈图中用线条进行连接.其中,含有潜在复制关系的基因需满足以下两个条件[12]:两条序列比对后其覆盖度超过较长序列的75%;序列比对后的相似度不小于75%.
1.5 甘薯bHLH motif分析
利用MEME程序[13]预测甘薯bHLH蛋白序列的保守基序,查找的保守基序最大数目为5,其它参数设为默认值.
1.6 甘薯bHLH系统进化树分析
以多重比对后的bHLH蛋白序列为基础,利用MEGA X[14]生成甘薯bHLH转录因子系统进化树.采用邻接法(N-J method)P-distance模型,空位选项为pairwise deletion,检验参数Bootstrap method值取1 000.
1.7 甘薯bHLH转录因子基因表达分析
从EBI(https://www.ebi.ac.uk/)数据库下载甘薯新种花(高感)和金山57(高抗)在尖孢镰刀菌F04、F07胁迫下的转录组数据[9]以及徐薯15-1和徐薯15-4低温胁迫下的转录组数据[15],其中,尖孢镰刀菌胁迫下的甘薯转录组数据,试验组用F07/F04接种甘薯幼苗,分别为JS57_F07、JS57_F04和XZH_F07;对照组用水接种甘薯幼苗,分别为JS57_CK和ZXH_CK,共5个处理,每个处理3个重复.Ji et al[15]将徐薯15-1和徐薯15-4的薯块在4 ℃条件下储存0周、2周、6周后进行测序,获得甘薯在低温胁迫下的转录组数据,6个处理组分别为Xushu15_1_Cold_0w、Xushu15_1_Cold_2w、Xushu15_1_Cold_6w、Xushu15_4_Cold_0w、Xushu15_4_Cold_2w和Xushu15_4_Cold_6w,每个处理重复3次.以143个甘薯bHLH转录因子的DNA序列为基础,通过Hisat2[16]建立bHLH索引文件,与下载的转录组测序数据比对后,得到包含每个read与甘薯bHLH家族成员比对信息的sam文件.手动统计每个bHLH转录因子的read数,DESeq2标准化read数.比较试验组与对照组的标准化read数获得log2FoldChange值(padj<0.05)利用pheatmap(https://cran.r-project.org/web/packages/pheatmap/index.html)绘制甘薯bHLH转录因子的差异表达聚类图.
2 结果与分析
2.1 甘薯基因组中bHLH转录因子鉴定和分类
以144条拟南芥bHLH蛋白序列为基础建立bHLH转录因子数据库,用于检索甘薯全基因组蛋白质序列,得到276条候选蛋白.选取其中特异性较高的bHLH序列建立甘薯特异的HMM模型,hmmsearch程序检索得到191条bHLH候选蛋白.将blastp程序检索得到的276条甘薯bHLH候选蛋白和hmmsearch程序检索得到的191条bHLH候选蛋白合并、去除重复,共得到285条甘薯bHLH候选蛋白序列.经过比对筛选,去除不含有bHLH结构域或结构域缺失严重的序列,最终得到143个甘薯bHLH转录因子.研究表明,bHLH转录因子的保守结构域中含有19个保守位点[17](碱性氨基酸区5个,第一螺旋区5个,环区1个,第二螺旋区8个),参考拟南芥bHLH结构域的19个保守位点[18],对经序列比对后的甘薯bHLH转录因子结构域保守位点进行定位,统计获得各个位点氨基酸的组成及其出现的频率(表1).
bHLH结构域的碱性氨基酸区是特定DNA的结合位点,与bHLH转录因子的功能相关.参考水稻和拟南芥的分类结果[19],根据bHLH蛋白DNA结合能力的不同对甘薯bHLH转录因子进行分类(表2).以Toledo-Ortiz et al[18]的分类为参考:若bHLH转录因子中bHLH结构域N端17个氨基酸中碱性氨基酸(Lys、Arg、His)数不少于5个,则认为该蛋白具有DNA结合活性,少于5个的蛋白(HLH蛋白)不具有DNA结合活性,缺失碱性区的HLH蛋白虽然没有DNA结合能力,但是可以与bHLH蛋白形成二聚体,bHLH转录因子通常以二聚体形式发挥作用.具有DNA结合活性的蛋白可分为E-盒结合和非E-盒结合,具有E-盒结合活性的蛋白第13位和16位氨基酸分别为谷氨酸(Glu)和精氨酸(Arg).另外,一些E-盒结合活性的蛋白具有与G-盒结合的能力,根据bHLH蛋白第9位和17位是否为赖氨酸/组氨酸(Lys/His)和精氨酸(Arg)进一步将E-盒结合分为G-盒结合和非G-盒结合.
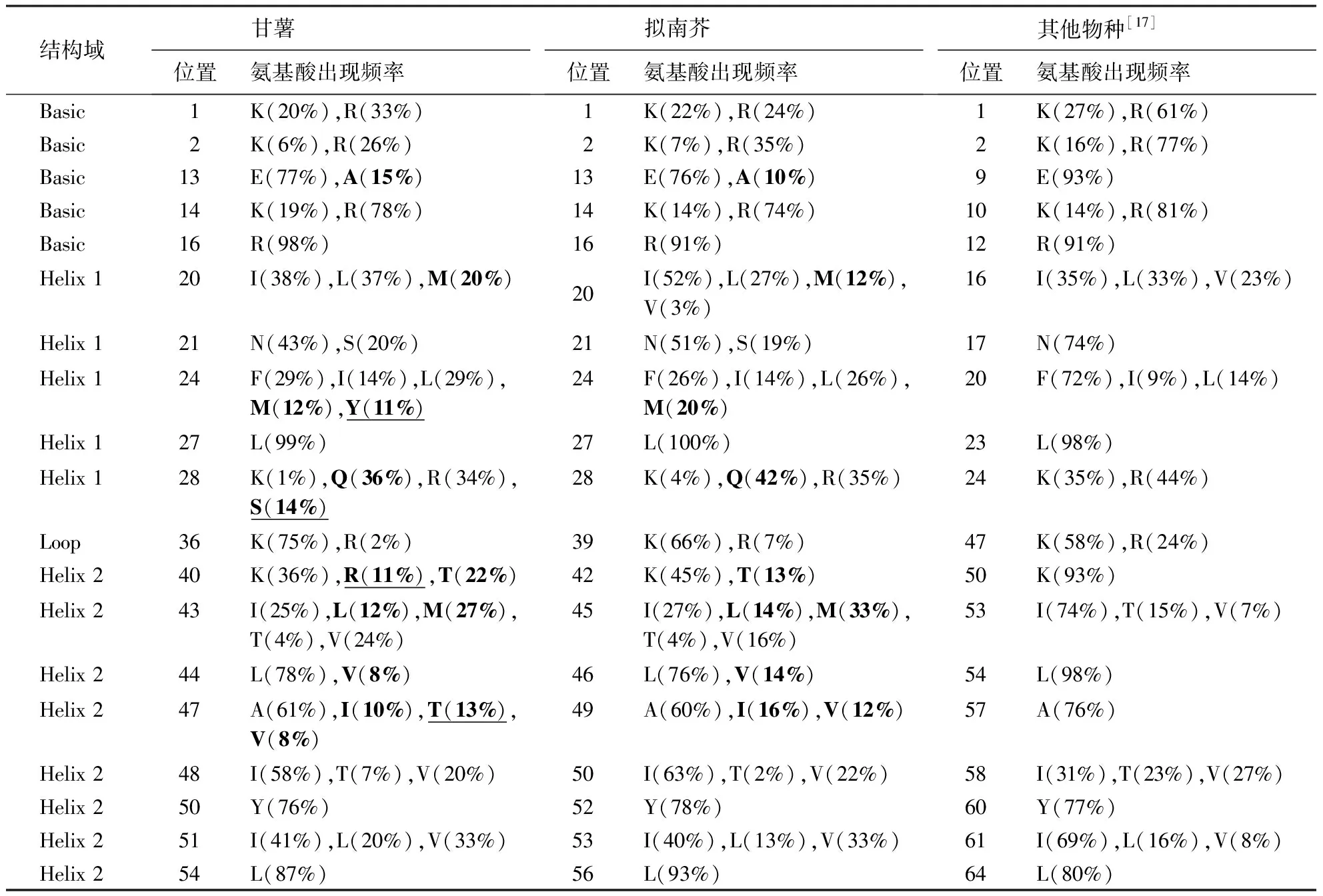
表1 bHLH结构域保守位点1)Table 1 Conservative sites of the bHLH transcription factors domains
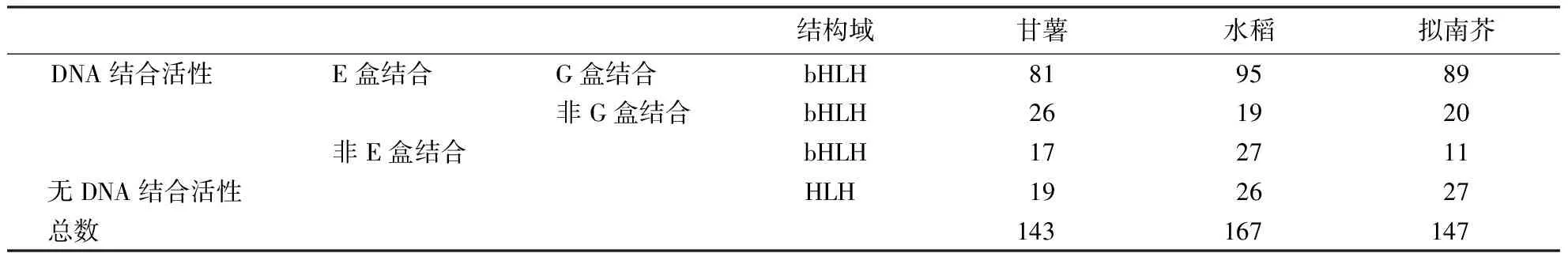
表2 bHLH转录因子DNA结合活性Table 2 DNA binding activity of the bHLH transcription factors
2.2 甘薯bHLH家族成员在染色体上的分布
利用circos程序将甘薯143条bHLH蛋白序列对应的基因定位到15条染色体上(图1).从图中可以看出,甘薯bHLH家族成员在15条染色体上分布并不均匀,在某些染色体上分布数量较多,如1号(15个)、2号(18个)、5号(15个)、6号(12个)以及14号(13个)染色体;而在其它染色体上分布的bHLH家族成员则较少,如第9和12号染色体,均只有4个bHLH转录因子.
为了判断甘薯bHLH各家族成员之间的潜在复制关系,本研究利用blastn计算得到39个含有复制关系的基因对,用连线对其进行连接,其中存在于染色体内的具复制关系基因对有31对(蓝色),根据基因在染色体上的位置和序列之间的相似性可判断基因簇,而具有潜在复制关系的基因对之间的序列相似性均大于75%,因此,存在于染色体内的紧密排列的具有重复关系的基因可认为是基因簇,例如CM008331.1-snap.11624和CM008331.1-snap.11743在1号染色体上成簇状分布,且两序列之间的相似度为98%.从图中可以发现,大多数染色体内成簇状分布的基因均存在潜在的复制关系(图1).另外,染色体间的具有潜在复制关系的基因有8对(橙色),如2号染色体上CM008332.1-snap.11316及CM008332.1-snap.11318分别与11号染色体上的CM008341.1-snap.4864和CM008341.1-snap.1582、CM008341.1-snap.4863和CM008341.1-snap.1581成潜在复制关系.
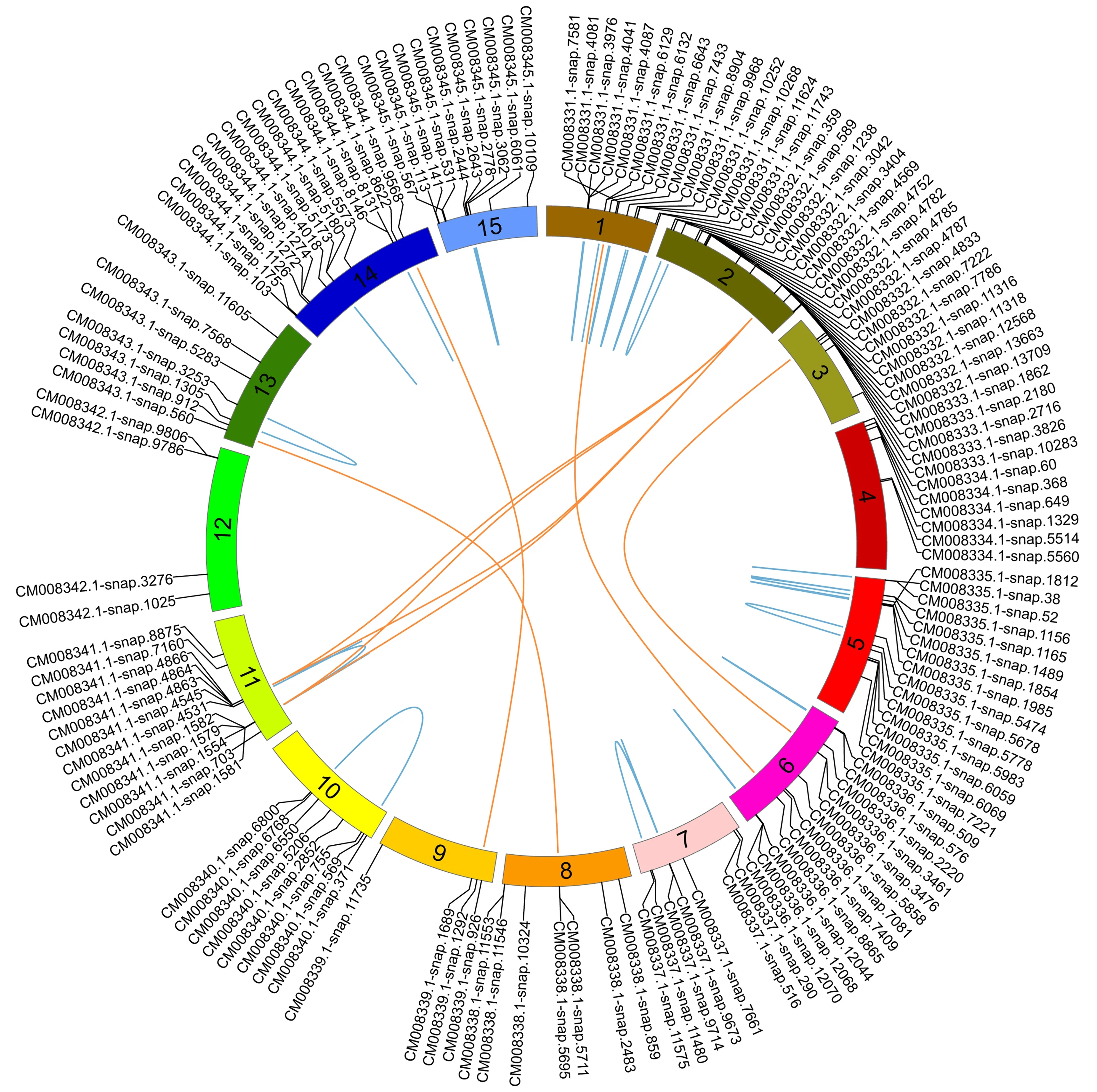
图中圆的不同颜色部分表示不同的染色体;蓝色连接线条表示染色体内重复基因对,橙色连接线条表示染色体间重复基因对.图1 甘薯bHLH基因的染色体定位和潜在复制关系Fig.1 Chromosomal localization and potential duplication relationship of I.batatas bHLH genes
2.3 bHLH结构域保守性分析及MEME分析
为了研究甘薯bHLH转录因子的结构域保守性,对得到的143条bHLH蛋白序列进行多重比对(图2).从比对结果中可以发现,甘薯bHLH结构域大约由54个氨基酸组成,其中有21个保守性大于50%的位点(图中阴影部分),这些保守位点有6个位于碱性氨基酸区(Basic)(1-17),5个位于第一螺旋区(Helix 1)(18-32),2个位于环区(Loop)(33-39),8个位于第二螺旋区(Helix 2)(40-54).21个保守位点中有10个位点的氨基酸保守性在70%以上,分别为13-E(76.9%)、14-R(78.3%)、16-R(97.9%)、17-R(90.9%)、27-L(99.3%)、32-P(86.0%)、36-K(74.8%)、44-L(77.6%)、50-Y(75.5%)和54-L(87.4%).碱性氨基酸区含有3个高度保守的精氨酸(Arg)残基位点,作为碱性氨基酸在长期进化中在该区域保持着高度的保守性,说明这3个保守位点在碱性氨基酸区与特定DNA结合时有着重要的作用.两个螺旋区均有高度保守的亮氨酸(Leu)残基,其中第一螺旋区的27-Leu的保守性高达99.3%,已知动物中的Max蛋白bHLH结构域27-Leu在形成二聚体时发挥重要作用,推测植物中该位点的Leu也具有重要功能[20].
利用MEME分析甘薯bHLH转录因子,得到5个保守基序(图3),分别为motif 1(HSLAERRRREKINERLKALQSLVPGGSKM)、motif 2(DKASMLDEAIKYIKELQEQVE)、motif 3(EAPVPPEYVHVRARRGQATDS)、motif 4(LPEIEVRISGDDVLIRIHCSK)和motif 5(GLLLKIFSELEELGLEVVSASVSTFGDLA),括号内划横线部分表示该位点氨基酸高度保守.motif 1和motif 2共同组成甘薯bHLH转录因子的bHLH结构域,且这两个基序在所有143条甘薯bHLH转录因子中出现的频率均在90%以上,其中142个甘薯bHLH家族成员中均含有motif 1,134个成员中均含有motif 2,表明甘薯bHLH转录因子拥有较保守的bHLH结构域.另外,有50个家族成员含有motif 5,而motif 3和motif 4则分别仅存在于23和34个家族成员中.

图2 甘薯bHLH转录因子保守性分析Fig.2 Conservation analysis of I.batatas bHLH transcription factors
2.4 甘薯bHLH转录因子系统进化树分析
利用N-J法生成甘薯bHLH转录因子系统进化树(图4),树状图分枝上的Bootstrap值仅显示大于50%,并以此作为甘薯bHLH亚家族的划分标准[19],将甘薯143个bHLH家族成员划分为22个亚组,命名为S1~S22,各亚家族之间用不同颜色加以区分.分支上显示的Bootstrap值大多在90%以上,说明构建的进化树可靠性较高[14,21].另外,图中22个亚组所包含的bHLH转录因子数目并不相同,囊括家族成员最多的是S12亚组(20个);S5亚组次之(15个);S4、S6、S13、S15以及S21均仅有2个成员.
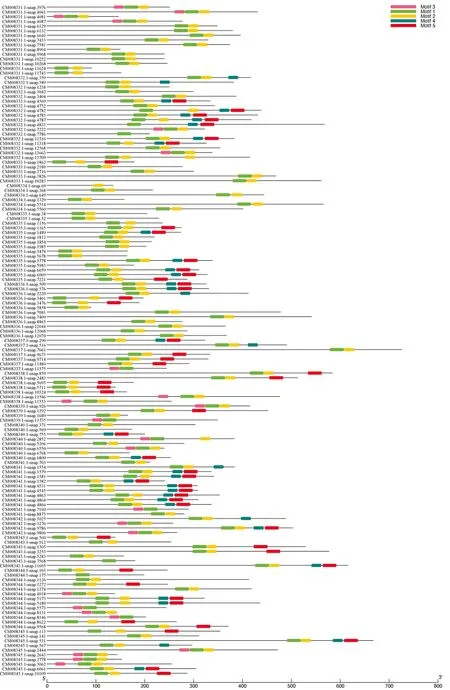
图3 甘薯bHLH转录因子的基序分布Fig.3 Motif distribution of I.batatas bHLH transcription factors

图中各亚家族之间用不同的颜色加以区分,即颜色相同的成员为同一亚家族.图4 甘薯bHLH转录因子系统进化树Fig.4 Phylogenetic tree of I.batatas bHLH transcription factors
2.5 甘薯bHLH转录因子在生物胁迫和非生物胁迫下的差异表达分析
Lin et al[9]将分离得到的甘薯尖孢镰刀菌菌株F04和F07作为致病菌分别侵染甘薯高抗品种金山57(JS57)和高感品种新种花(XZH),24 h后取其幼苗茎段进行转录组测序,我们利用以上转录组数据计算和绘制了甘薯bHLH转录因子响应尖孢镰刀菌胁迫时的基因表达变化图(图5).筛选得到6个bHLH转录因子在受到尖孢镰刀菌胁迫时表达量发生显著变化,分别为CM008332.1-snap.4569、CM008332.1-snap.13709、CM008333.1-snap.2180、CM008335.1-snap.7221、CM008340.1-snap.755和CM008340.1-snap.6800,筛选标准遵循log2FoldChange的绝对值≥1且padj<0.05.
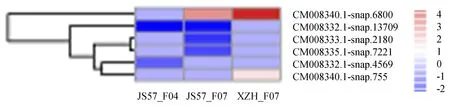
图5 甘薯bHLH基因在尖孢镰刀菌Fob胁迫下的差异表达Fig.5 Differential expression of I.batatas bHLH genes under Fusarium oxysporum stress
从图5中可以看出,6个响应尖孢镰刀菌胁迫的基因中,4个表达下调,2个表达上调.用不同强度的致病菌F04和F07侵染同一甘薯品种金山57时,同一基因的表达量变化并不完全一致,如CM008332.1-snap.4569在F04侵染时,表达量下调,在F07侵染时,无明显变化;CM008333.1-snap.2180、CM008335.1-snap.7221以及CM008340.1-snap.6800则仅在F07侵染时,表达量有所变化.其中,不同于CM008333.1-snap.2180和CM008335.1-snap.7221表达量呈现下调的趋势,CM008340.1-snap.6800在金山57响应F07胁迫时,表达量呈现上调趋势,且在感病品种新种花中,在同样受到F07致病菌的胁迫时,CM008340.1-snap.6800表达量的上调幅度更大,log2FoldChange值高达4.23.
Ji et al[15]将甘薯品系Xushu15-1和Xushu15-4的块根在4 ℃下冷藏0、2和6周后,对其进行了转录组分析.本研究利用该转录组数据,分析bHLH基因的差异表达(图6),结果表明,甘薯bHLH转录因子中有44个基因在低温胁迫下表达量发生变化,且大部分甘薯bHLH基因在低温胁迫下表达量呈现下降的趋势,有13个基因在4个处理组中的表达量均有变化,在4个处理组中均下调的基因为CM008332.1-snap.4569、CM008336.1-snap.7409、CM008339.1-snap.11735、CM008340.1-snap.2852、CM008341.1-snap.703、CM008341.1-snap.1579、CM008342.1-snap.9806和CM008344.1-snap.175;均上调的基因为CM008336.1-snap.12068、CM008336.1-snap.12070、CM008338.1-snap.11546、CM008338.1-snap.11553和CM008342.1-snap.9786,且CM008336.1-snap.12068和CM008336.1-snap.12070表达量上调幅度变化较大(log2FoldChange值>7.0).

图6 甘薯bHLH基因低温胁迫下的差异表达Fig.6 Differential expression of I.batatas bHLH genes under low temperature stress
2.6 甘薯部分bHLH基因序列位置信息
为了更直观的表示甘薯bHLH转录因子的序列信息,根据差异表达的分析结果,分别筛选出尖孢镰刀菌胁迫下表达量变化最大的2个基因和冷胁迫下表达量变化最大的8个基因,其基因的序列位置信息如表3中所示.表中的基因名称由染色体序列文件名和基因编号两部分组成,用“-”符号分隔开,例如CM008332.1-snap.13709表示NCBI数据库“泰中6号”基因组的染色体序列文件CM008332.1.fasta(第2号染色体)上根据snap预测的编号为snap.13709的基因.基因由1个或多个外显子组成,表中外显子的位置区间均以正义链坐标为准,链为“+”的基因取正义链的序列,链为“-”的基因取正义链反向互补的序列;外显子排列顺序按照其在基因中出现的顺序,拼接外显子即基因全长序列.

表3 甘薯部分bHLH基因序列信息Table 3 Sequence information of several bHLH genes of I.batatas
3 讨论
bHLH转录因子是植物中最大的转录因子家族之一,在植物生长发育、生物合成及抗逆性等方面发挥重要作用.本研究基于生物信息学分析方法,从甘薯全基因组CDS序列中鉴定得到143个bHLH转录因子,与水稻(至少165个),拟南芥(162个)及烟草(190个)相比数目略少[2].143个甘薯bHLH蛋白中的107(74.8%)个具有E-盒结合活性,其中81个(56.6%)具G-盒结合活性;水稻bHLH转录因子中具E-盒结合活性的蛋白占68.5%,具G-盒结合活性的占57.0%;拟南芥中为74.1%(E-盒结合),60.5%(G-盒结合).从bHLH转录因子中具有E-盒/G-盒结合活性的蛋白所占的比例来看,甘薯与拟南芥和水稻的结果相似.
Atchley et al[22]运用统计学的方法,分析了242个生物中的bHLH蛋白,在bHLH结构域中找到了19个保守位点,这19个保守位点可作为bHLH基因的判断依据,即如果一个序列含有这19个保守位点的13个及以上,则可认为这个蛋白为bHLH蛋白,Toledo-Ortiz等同样从147个拟南芥bHLH序列中找到了19个保守位点.本研究根据这19个位点在bHLH结构域中的位置及氨基酸组成特征,从联配后的甘薯bHLH转录因子中找到19个相类似的保守位点.这19个保守位点与Atchley等统计的19个位点并不完全相同,在个别位点在结构域中所处的位置有些差异,氨基酸组成也有所不同.但与拟南芥的19个位点相比,甘薯bHLH转录因子19个保守位点的氨基酸组成及所处位置均与其相似.另外,根据序列联配结果中各位点的氨基酸保守性是否大于50%,从甘薯的比对结果中找到了21个保守性大于50%的位点,其中有10个位点与上述判断bHLH蛋白的19个位点相重叠,分别为Glu-13、Arg-14、Arg-16、Leu-27、Lys-36、Leu-44、Ala-47、Ile-48、Tyr-50和Leu-54;拟南芥的相关研究中同样也找到了17个保守性在50%以上的位点,其中同样有10个位点与上述的19个位点相重叠,分别为Glu-13、Arg-14、Arg-16、Leu-27、Lys-39、Leu-46、Ala-49、Ile-50、Tyr-52和Leu-56;从中可以发现两者的这10个保守位点氨基酸类型完全相同,但位置上略有差异,这主要是因为bHLH结构域中的Loop区在不同物种中长度不一,Loop区的长度范围约在5~21之间[17].
对甘薯bHLH蛋白构建进化树,根据Bootstrap值将其分为22个亚组,每个亚组所包含的家族成员个数有所差异,最多的有20个,最少的仅2个.动物中,根据bHLH转录因子的碱性DNA结合模式不同,可将bHLH蛋白分为A、B、C、D、E和F 6个组[2],其中A组和B组都能与E-box结合,B组中还包括Myc家族;而在植物中bHLH转录因子的划分还未有系统的分类.已知拟南芥bHLH转录因子分为21个亚组,其中18亚组家族成员数最多,有18个,最少的亚组成员有2个.而在167个水稻(Oryzasativa)[19]和159个小麦(TriticumaestivumL.)[23]bHLH转录因子中,分别被划分为22和19个亚组.
利用转录组测序数据分析了甘薯在尖孢镰刀菌和低温胁迫下bHLH转录因子的表达量变化,分别从中筛选到了6个和44个bHLH家族成员,甘薯bHLH转录因子中在低温胁迫下所响应的基因数远多于在尖孢镰刀菌胁迫下响应的基因数,这可能是因为两者在不同的逆境条件下的响应机制有所不同.
