鄱阳湖流域蚌类环境DNA宏条形码引物的筛选验证*
2021-07-07陈金萍周春花欧阳珊黄晓晨吴小平
陈金萍,周春花,欧阳珊,黄晓晨,吴小平
(南昌大学生命科学学院,南昌 330031)
淡水生态系统是世界上最脆弱的生态系统[1],蚌类是世界上最易受威胁的动物类群之一[2].蚌类是一种重要的底栖动物, 在淡水生态系统中占有非常重要的地位, 它们不但是食物链中不可缺少的环节, 而且对水质净化起着重要的作用,许多种类具有较高的经济价值[3-5].蚌类资源的调查具有挑战性,因为它们存在于高浊度或低能见度的生境,并且蚌类种群密度低、斑片状分布、与相近物种杂交以及存在潜在的隐藏种.传统调查方法即直接捕捉会破坏它们的生活环境,使种群规模变小,造成二次伤害,耗费大量人力物力,且与现行的禁渔政策相悖.此外,由于蚌类鉴别特征少,高变异性所以难以鉴定,需要专门的分类学知识[6].因此,亟需一种不违背目前政策的、高效率、低成本和高灵敏度的监测方法来评估蚌类生物多样性.
环境DNA技术是最近开发的一种非侵入、高灵敏、高效率且无损伤的调查工具.环境DNA是从环境样本(例如土壤、水样、空气)中提取的总DNA, 不需要对任何目标生物体进行分离[7].利用环境DNA技术可以从环境样品中直接提取DNA片段后利用测序技术对生物进行定性或定量分析.最早将环境DNA技术用于淡水生态系统的是监测一种入侵物种——美国牛蛙[8].此后,很多水生物种开始使用环境DNA技术研究,但在蚌类中的研究起步稍晚.Carlsson等利用环境DNA技术检测到了欧洲淡水濒危种珠母珍珠蚌的存在,并且认为环境DNA在物种检测中可作为一种非常重要的工具[9].随后环境DNA作为一种检测工具在濒危蚌中的有效性得到Currier等学者的确认[10].Pearrubia等将环境DNA技术用来检测两种入侵蚌的位置,并认为其在入侵种早期的预防扩散中发挥着至关重要的作用[11].Shogren等对入侵蚌的环境DNA在河流中转运方式及浓度变化作了进一步的研究[12].Prié等利用环境DNA宏条形码评估了西古北界淡水双壳类2个科的生物多样性[6].环境DNA技术用于蚌类的研究多限于监测入侵种和濒危种,而对于生物多样性的研究鲜见报导.研究环境DNA方法应用于检测生物多样性的研究催生了宏条形码的产生[13].宏条形码技术不仅提供了准确、快速的物种多样性监测手段,还提供了认识和解决环境问题的新方式.在使用环境DNA宏条形码技术监测生物多样性时,首先需要进行的就是针对不同的目标种选取DNA识别片段来设计引物.环境DNA宏条形码引物在监测、定量过程中存在较大偏好性,选择适合的引物可识别生态群落中不同的物种.但环境中细胞外源的DNA会不断降解,在保证物种分辨率的前提下,选择的分子标记越短越好.所以使用宏条形码技术分析环境DNA的研究中,为了避免在不同分类种群中放大偏差,选择设计较短的、高度保守序列片段的引物是极其重要的.如果实验设计的引物保守度低和特异性不足的话,容易引起碱基错配甚至引起假阳性和假阴性的实验结果.
蚌的分布呈明显的地域性特征,北美蚌类特有种多集中于美国,而亚洲蚌类最丰富的国家则是中国,且集中分布在长江中下游地区[14].鄱阳湖是长江中下游主要支流之一.为了筛选出适合蚌类环境DNA生物多样性研究的宏条形码引物,本研究设计了多对通用引物,通过普通PCR扩增和高通量测序筛选这些引物,并用鄱阳湖流域水样验证所筛选引物的效果,以期为蚌类环境DNA生物多样性监测提供参考.
1 材料和方法
1.1 选择序列及设计引物
NCBI上下载中国蚌科27种蚌的母系线粒体基因组序列.选择线粒体序列是因为每个细胞的线粒体DNA拷贝比核DNA拷贝多得多,当环境DNA浓度很低或降解时,线粒体DNA更为靶向[15].本研究所在的课题组得出线粒体cox1和nad1基因可作为蚌科DNA条形码,用于蚌科物种的快速鉴定[16],所以我们选取线粒体cox1和nad1基因全序列.在前期实验中发现蚌科通用引物16S rRNA扩增效果较好,对蚌科物种有较好的辨识度,且产物大小也符合我们设计的原则[17].在对水样进行高通量测序时发现,用于鱼类环境DNA多样性研究的引物cytb能鉴定出蚌科动物[18].因此,我们把这2对引物也列入了筛选对象,详见表1.设计引物的具体步骤如下,首先,在下载的27种蚌的线粒体全序列中找到cox1、nad1和cytb基因的保守区域,在mega中比对(图1);然后,使用软件Primer Primier 5设计通用引物,设置参数如下:引物长度(21±5)bp,扩增产物长度在100~400 bp之间,优先选择不含发夹结构、无引物二聚体和错配较少的引物序列;最后,在mega中输入设计的引物,查找其保守区,变异位点位置.引物在靶序列的位置如图2.

表1 本研究设计及引用的引物信息

图1 cyt b 234下游引物的位置

图2 引物在靶序列中的位置
1.2 普通PCR扩增筛选引物
提取单个蚌科物种(24种,详见表2)的组织DNA:取酒精固定或-80℃保存的蚌类闭壳肌,使用蛋白酶K消化,试剂盒(TIANamp® Marine Aniamal DNA Kit)提取,最后溶解于70 μL TE缓冲液中,-20℃保存备用.

表2 用于DNA提取、PCR扩增所用的24个蚌科物种及高通量测序比对结果
用本研究设计的通用引物及引用的引物分别对这24个物种进行PCR扩增,PCR反应条件:94℃ 预变性2 min; 94℃ 变性 30 s,退火(47~56℃)40 s,72℃ 延伸 1 min,35个循环; 71℃ 延伸 10 min;最后4℃保存.PCR反应体系(25 μL):10×buffer 2.5 μL,dNTP 2 μL,上下游引物(10 μmol/L)各1.25 μL,Taq酶0.2 μL,DNA模板2 μL, ddH2O 15.8 μL.PCR反应所用的酶为TaKaRa Premix.筛选出可以同时扩增出这24个蚌类物种,且凝胶电泳条带清晰单一的引物对,并对PCR产物加以测序验证,确保为目的物种的序列.
1.3 构建文库及高通量测序筛选引物
随机将这24个蚌科物种的DNA混合,即每个蚌的DNA各1 μL装至1个管中,用上一步筛选出的引物进行高通量测序进一步筛选.对原液进行PCR扩增,通过2%琼脂糖凝胶电泳检测PCR扩增产物,PCR反应体系为25 μL,5×reaction buffer 5 μL, 5×GC buffer 5 μL, dNTP(2.5 mmol/L)2 μL, 上下游引物(10 μmol/L)各1 μL, DNA模板3 μL,ddH2O 7.75 μL, Q5 DNA酶0.25 μL.PCR程序:98℃ 预变性5 min;98℃变性30 s、55℃退火30 s、72℃延伸45 s(35个循环);72℃再延伸5 min;12℃保存.对目标片段进行切胶回收,然后采用Illumina MiSeq测序平台进行测序,之后采用Illumina公司的TruSeq Nano DNA LT Library Prep Kit制备测序文库.最后对原始reads数进行整理过滤及质量评估,剔除嵌合体序列,如:1)5’端引物错配碱基数> 1的序列;2)含有连续相同碱基数>8的序列等.产生的可操作性分类单元(operational taxonomic units,OTUs)通过BLAST在NCBI数据库中查找蚌科物种同源序列,以97%的序列相似度作为OTU划分阈值,该阈值大致相当于分类学中物种(Species)水平的序列差异.且e_value(e值)<1×10-5作为该序列的物种注释信息.对于NCBI数据库中没有序列的棘裂脊蚌(Schistodesmusspinosus)、微红楔蚌(Cuneopsisrufescens)、矛形楔蚌(Cuneopsisceltiformis),我们补测了其线粒体基因组全序列.
1.4 水样采集、传统数据采集、DNA提取及测序
于2019年4月在鄱阳湖子湖青岚湖进行水样采集,设置了6个取样点,每个样点的坐标如下:1#(28.536°N,116.166°E)、2#(28.471°N,116.164°E)、3#(28.928°N,116.157°E)、4#(28.513°N,116.142°E)、5#(29.472°N,116.110°E)、6#(29.164°N,115.934°E).6个采样点水体总体较浑浊,水流较湍急,水面浮有水生植物,有渔船,人工养殖带,时有鸟栖居,岸边有堤坝或沙带,水草灌木茂盛,水体连通性较好.每个点用采水器采集1 L中下层水样,做3次重复,并在相应采样点同时进行传统方法采集蚌(蚌耙采蚌),水样和蚌的采集都在渔船上进行,采集活体蚌之后,当天返回实验室进行鉴定.选取6个点的水样用高通量筛选出的引物进行验证.水样于采集后24 h内用0.45 μm的混合纤维素滤膜过滤.使用DNeasy Blood & Tissue Kit(Qiagen)提取环境DNA.Qubit(Thermo Fisher Scientific)测试DNA浓度.3个重复样本混合后提取DNA,并测浓度,然后由上海派森诺生物科技股份有限公司进行高通量测序.PCR反应体系和步骤如下,PCR反应体系为25 μL,5×reaction buffer 5 μL, 5×GC buffer 5 μL, dNTP(2.5 mmol/L)2 μL, 上下游引物(10 μmol/L)各1 μL, DNA模板2 μL,ddH2O 8.75 μL, Q5 DNA酶0.25 μL,PCR程序:98℃预变性2 min;98℃变性15 s、55℃退火30 s、72℃延伸30 s(30个循环);72℃再延伸5 min;10℃保存(注:因引物的退火温度不同,每个引物会进行部分微调).质控步骤与上文一致.测序结果在NCBI数据库中进行比对验证,以97%的序列相似度,且e_value(e值<1×10-5)最小的,作为该序列的物种注释信息(因为e值越小准确度越高,可信度也越高).在物种注释中,对于reads<3的OTU不作统计.
2 结果
2.1 普通PCR扩增筛选引物结果
用表1所列的11对引物分别对提取的24个蚌科物种的DNA进行PCR扩增,有3对引物(cytb、cox1233和16S rRNA)能将这24个蚌科物种都扩增出来,扩增条带清晰明亮,且扩增产物大小符合预期大小,如图3~图5.其余8对引物均不能完全地扩增出这24个蚌科物种,或者是扩增效果不理想.
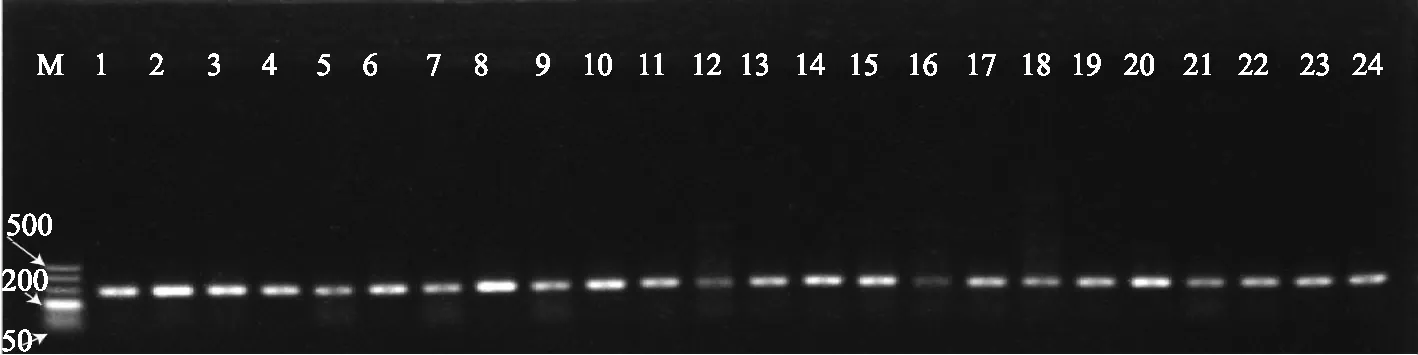
图3 使用引物cyt b 扩增蚌科动物的结果(M: DNA Marker 500, 1~24编号对应的蚌类见表1)
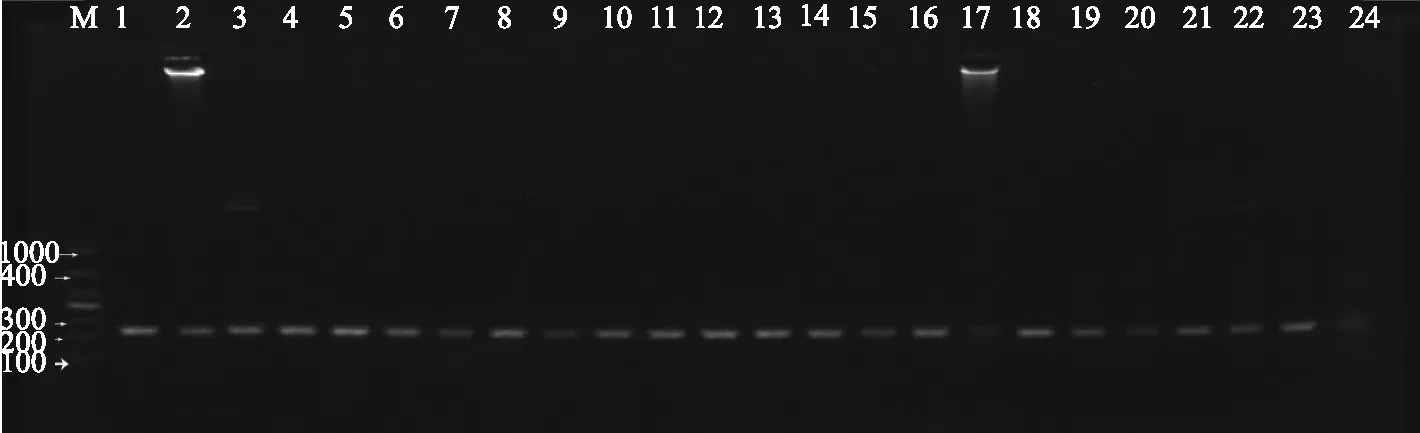
图4 使用引物cox1233扩增蚌科动物的结果(M: DNA Marker 1000, 1~24编号对应的蚌类见表1)
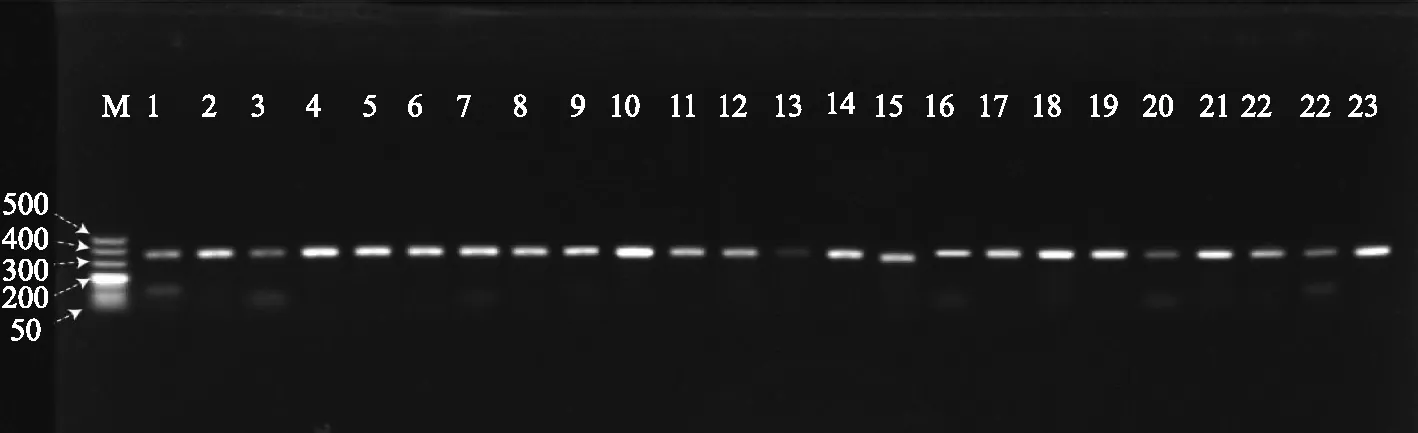
图5 使用引物16S rRNA扩增蚌科动物的结果(M: DNA Marker 500, 1~24编号对应的蚌类见表1)
2.2 高通量测序筛选引物
用普通PCR扩增筛选出的cytb、cox1233和16S rRNA三对引物对混合24个蚌科物种的DNA进行高通量测序,16S rRNA扩增产物测序共获得202148条reads,蚌201683条reads;cytb扩增产物测序共获得55096条reads,其中蚌类有55038条;cox1233扩增产物测序共获得179354条reads,其中蚌类有179277条.通过BLAST 对OTUs进行物种注释,引物cytb的扩增产物注释到24种蚌中的23种蚌,只有棘裂脊蚌没有注释到,该引物辨识度达96%;引物cox1233的扩增产物注释到18种蚌,而没有注释到卵形尖嵴蚌、蚶形无齿蚌、高顶鳞皮蚌、射线裂脊蚌、棘裂脊蚌和龙骨蛏蚌,引物辨识度只有75%;引物16S rRNA的扩增产物能注释到除卵形尖嵴蚌之外的所有物种,该引物辨识度也达96%.高通量测序比对结果详见表2.
2.3 水样环境DNA高通量测序
为验证引物效果,对6个样点的水环境DNA使用引物cytb和16S rRNA扩增后进行高通量测序.16S rRNA扩增产物测序共获得461185条reads,其中蚌类的序列有123104条,占26.69%,共注释到蚌科6属8种(表3).cytb扩增产物共获得97263条reads,其中蚌类共6416条,约占6.60%,共注释到蚌科4属6种(表3).2对引物共检出蚌科7属11种,均注释到圆顶珠蚌、洞穴丽蚌、背瘤丽蚌.传统方法在6个点采集到的活体蚌9属13种(表3).环境DNA技术和传统方法共同鉴定出圆顶珠蚌、背角无齿蚌、洞穴丽蚌、扭蚌、短褶矛蚌.
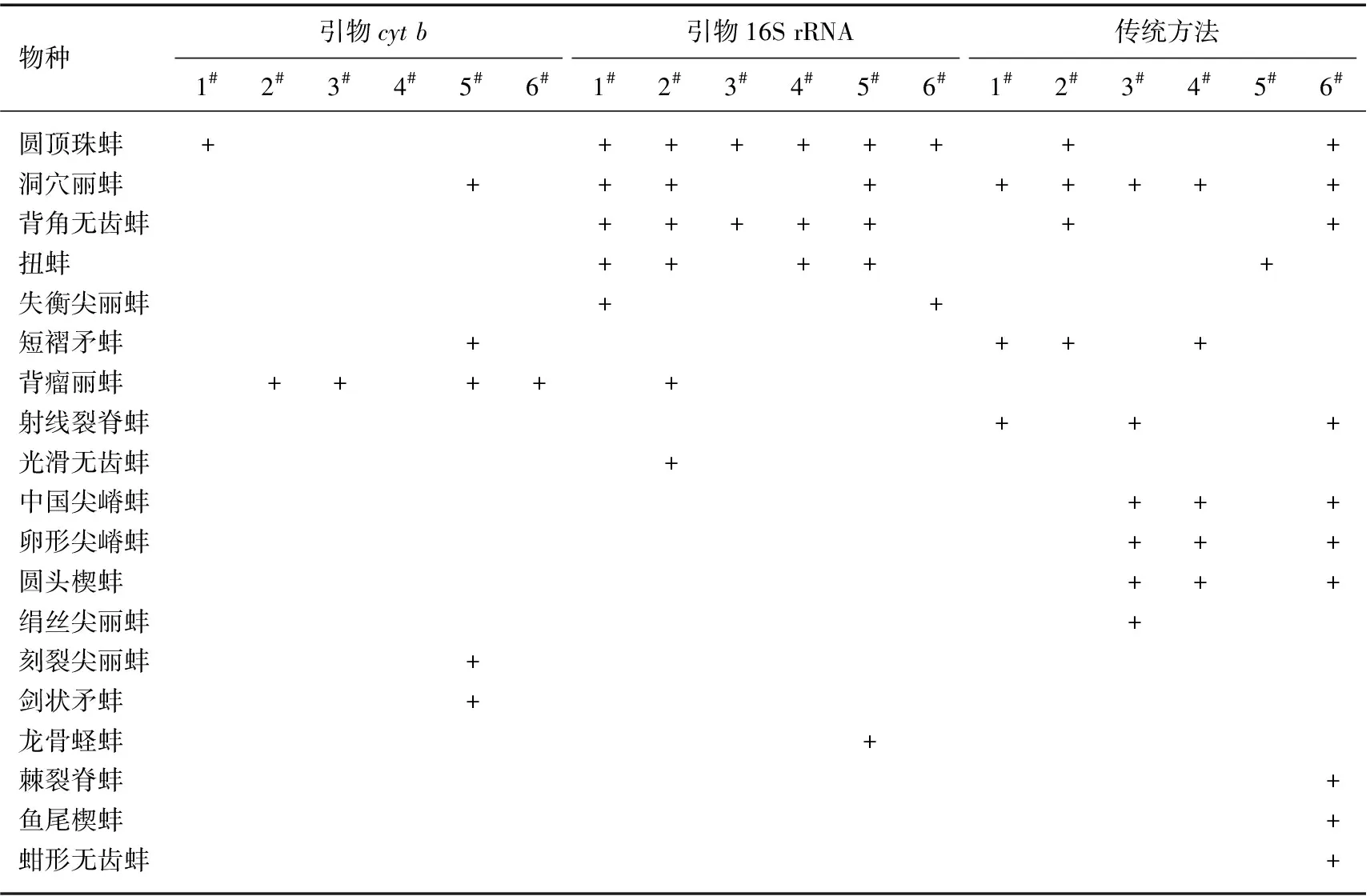
表3 环境DNA的鉴定结果及传统方法采集的蚌科物种信息
2.4 序列相对丰度
两对引物注释到的蚌类物种的序列数及占所有蚌的序列数的比例见表4.引物cytb注释到的背瘤丽蚌和剑状矛蚌的序列数较多,分别占蚌总序列数的54.27%和34.13%;引物16S rRNA注释到的8个物种中,序列数占比最多的为圆顶珠蚌(84.76%),其次是背角无齿蚌(10.86%),最少的为龙骨蛏蚌(0.01%).传统方法捕捉到的活体蚌类物种个数见表4,最多的蚌是洞穴丽蚌(41.18%),其次是中国尖嵴蚌(16.67%).

表4 水样环境DNA检测物种注释序列数和传统方法捕捞活体数
3 讨论
3.1 引物的偏好性
理想的环境DNA宏条形码引物应该具有高度保守、高辨别度没有物种偏好性等优点[19].本研究中引物cytb、16S rRNA和cox1233都具有高保守度,即都能将待测物种稳定扩增出.在环境DNA的引物筛选研究中,有研究者用水样DNA来进行引物筛选[20-21],不同的引物对相同的水样注释到的物种存在差异,缺乏一个已知的物种信息名录,无法判断引物的辨别度情况.而本研究用已知的多个物种DNA混合后进行高通量测序,来进一步了解引物的辨别度,引物cytb和16S rRNA辨别度都达96%,均高于引物cox1233引物的辨别度.我们认为影响引物辨别度的因素主要有2个:一是在单物种扩增时,虽然都能很好地扩增出来,但多个物种在引物结合位点没有差异;二是在高通量测序时引物存在偏好性.因此,我们建议在筛选环境DNA宏条形码引物时,先用一代测序验证引物的辨别度,再用二代测序了解引物的偏好性.
环境DNA宏条形码引物在物种多样性和丰富度研究中存在较大偏好性,即对单个物种的扩增效果很好,但当混和多个物种的DNA后,有些物种无法扩增出,导致后续高通量测序注释不到该物种[22-23].本研究结果表明,高通量测序结果中引物cytb没有检测到棘裂脊蚌,推断PCR扩增中由于引物的偏好性导致棘裂脊蚌没有扩增出来.引物cox1233有多个物种没有检测出来,表明该引物有多种蚌没有扩增出,存在较大的引物偏好性.同理16S rRNA没有检测到卵形尖嵴蚌,暗示PCR扩增中由于引物的偏好性导致卵形尖嵴蚌没有扩增出来.引物存在偏好性的原因是通用引物在设计时为了能同时扩增出尽可能多的物种,简并度较高,也导致引物对物种的扩增能力存在较大差异.因此使用通用引物对混合物种DNA进行PCR时,不可避免地会存在物种扩增效率差异.因此,我们建议在进行蚌科环境DNA宏条形码研究时,可以用多对通用引物进行高通量测序鉴定物种,可以有效地避免由于引物偏好性引起的物种遗漏.且使用多重标记即多个引物对,在水生环境DNA方法中已经得到大量应用,且可以增加可信度、准确度以及检测率[24-26].
3.2 环境DNA方法与传统方法在蚌科物种多样性研究中的比较
本研究使用环境DNA方法共检测到蚌科7属11种,传统方法共采集到蚌科9属13种,有5种蚌为两种方法共同鉴定出,2种方法在6个样点中鉴定的物种存在一些差异,一是因为环境DNA会随着水流移动,它并不能实时地反映该采样点的物种信息;二是因为环境DNA容易降解,在不同条件(温度、紫外线强度、微生物群落等)下降解速率不同[27];三是采样策略不同,传统方法用蚌耙采集,采集的是底层的蚌活体,环境DNA方法采集的是中下层水样;四是受PCR引物偏好性的影响,利用通用引物进行混合物种 DNA扩增,通常会导致较大的物种扩增效率差异[24].环境DNA方法在3个(样点1#、2#、5#)点中检测到了比传统方法更多的蚌科物种数,且环境DNA方法检测到了濒危物种龙骨蛏蚌,而这个物种在传统方法中没有采集到.传统方法采集到的优势种为洞穴丽蚌、中国尖嵴蚌、圆顶珠蚌,这和以前的研究结果相一致[28],而环境DNA方法中,引物cytb检测到序列占优势的为背瘤丽蚌、剑状矛蚌、洞穴丽蚌,引物16S rRNA检测到序列占优势的为圆顶珠蚌、背角无齿蚌、扭蚌.这表明序列的相对丰度和物种的相对丰度并没有得到很好的体现.虽然理论上来说,序列的相对丰度可能会体现物种的相对丰度[29],但两者的相关性没有得到很好的证明[30-31].很多研究表明PCR的偏好性是造成环境DNA序列的相对丰度无法体现物种的相对丰度的最主要因素[32].造成PCR偏好性最重要的原因是引物和模板之间存在不同碱基数量的错配,错配数量少的更易扩增,导致其在PCR过程中过量扩增[33].其次,碱基组成也影响引物结合效率,比如GC含量过高的模板不易扩增[34].现有环境DNA分析大多依赖PCR扩增,因此控制和减轻PCR的偏好性是环境DNA对水生生物多样性监测的主要挑战之一.
在传统方法采集到的13种蚌中,绢丝尖丽蚌在NCBI中无线粒体基因片段16S rRNA和cytb,这直接影响环境DNA方法对绢丝尖丽蚌的注释,导致环境DNA方法检测不到该物种.目前NCBI上记录了蚌科60个种的线粒体全基因组序列,其中中国蚌有27种,还有很多中国蚌科物种的线粒体基因组信息有待进一步完善.使用引物16S rRNA共注释到蚌科6属8种,蚌科物种序列占总序列数的26.69%,而使用引物cytb共注释到蚌科4属6种,蚌科物种序列占总序列数的6.60%,且引物16S rRNA在每个样点鉴定的物种数均多于引物cytb.本次水样环境DNA高通量测序结果表明在蚌科环境DNA生物多样性鉴定方面引物16S rRNA优于cytb,最新报道的欧洲淡水双壳类的生物多样性评估中用的也是16S rRNA基因的引物[6].
4 结论
本研究筛选了11对引物,在对蚌类进行普通PCR筛选引物时,8对引物不能稳定扩增出所有蚌,3对(cox1233、cytb和16S rRNA)能稳定扩增出所有蚌.对这3对引物进行混合蚌DNA高通量测序进一步筛选,cox1233辨别度不高,而cytb和16S rRNA有较高的辨别度.同时用这2对引物对水样进行高通量测序显示,引物16S rRNA注释到的蚌科物种序列占总序列数的百分比高,且能注释到更多的物种.引物16S rRNA更适合用于蚌类环境DNA生物多样性研究的宏条形码引物.环境DNA调查用约占传统捕捞调查1/100~1/10的调查次数,1/100000~1/100的调查时间,检测到了80%的蚌类种数,表明环境DNA方法调查效率更高.并且用分子生物学知识来鉴定,准确度更高.所以在蚌科生物多样性研究方面,环境DNA方法可以作为传统方法的有效补充,但适用于蚌科环境DNA技术的数据库还有待于完善,更多适合于蚌科环境DNA宏条形码生物多样性检测的分子标记有待于开发,多个分子标记联合使用可增加环境DNA技术的准确度和检测率.
致谢: 感谢刘雄军博士在传统方法采蚌方面给予的帮助,感谢王维开硕士在水样采集方面的帮助,感谢裘雪梅老师在数据处理方面的帮助.
