罗布麻CesA基因家族的生物信息学分析
2021-06-29解盛李国旗宋立肖谢博勋王雅芳刘星
解盛 李国旗 宋立肖 谢博勋 王雅芳 刘星

摘 要: 植物体中纤维素是细胞壁形成的主要成分,不仅参与细胞形态的建成,调控细胞发育,还参与细胞内多种细胞信号转导途径,进而影响植物体的生长发育。纤维素合酶是植物体合成纤维素的主要酶类。为了探究CesA基因家族对罗布麻生长发育及纤维素合成的调控机理,该文通过生物信息学分析方法,从基因家族鉴定、结构分析、蛋白理化性质与多级结构预测、亚细胞定位、信号肽、进化关系和顺式作用元件等方面,对罗布麻CesA基因家族进行系统鉴定和分子特征分析。结果表明:基于全基因组测序,罗布麻CesA基因家族鉴定含有15个成员,分布在罗布麻11条染色体中的8条上,其编码蛋白的氨基酸数量为730~1 158,相对分子质量81 280.81~130 123.18 kDa,理论等电点6.18~8.83。除了AvCesA3、AvCesA5、AvCesA7、AvCesA10和AvCesA11蛋白为稳定蛋白,其余成员均为不稳定蛋白;除了AvCesA12蛋白为疏水性蛋白,其余成员均为亲水性蛋白。该家族成员包含3~14个外显子,8~15个保守基序。编码蛋白主要分布于质膜与高尔基体上,无信号肽,二级结构以无规则卷曲与α-螺旋为主要构成元件。AvCesA15蛋白的跨膜结构域和三级结构与其他成员存在显著不同。罗布麻CesA基因进化时主要受纯化選择作用。对上游1 500 bp区域顺式作用元件分析,结果显示罗布麻CesA基因受到光、温度、水分、氧气等环境因子及生长素、赤霉素、脱落酸、乙烯、水杨酸等植物激素调控。该研究为进一步探究罗布麻CesA基因家族的生物学功能、提高纤维品质与品种改良奠定理论基础。
关键词: 罗布麻, 纤维发育, 生物信息学分析, CesA基因家族, 生长发育
中图分类号: Q943
文献标识码: A
文章编号: 1000-3142(2021)04-0522-13
Abstract: Cellulose is the main component of the cell wall in plants. It is not only participates in cell morphology and development, but also participates in various cellular signaling transduction pathways in cells, so as to affect the growth and development of plants. Cellulose synthase is the main enzyme used to synthesize cellulose in plants. Here, we explore the regulatory mechanism of the CesA gene family for cellulose synthesis, growth and development in Apocynum venetum. Using bioinformatic methods, we conducted systematic identification and molecular characterization of A. venetum CesA gene family which include gene family member identification, structural analysis, protein physicochemical properties and multi-level structure prediction, subcellular localization, signal peptides, evolutionary relationships, cis-acting elements. The results were as follows: Based on whole-genome sequencing, the A. venetum CesA gene family contained 15 members, which were distributed on 8 of the 11 A. venetum chromosomes. These family proteins were encoded by 730-1 158 amino acids, with the molecular weight at 81 280.81-130 123.18 kDa and theoretical isoelectric point at 6.18-8.83. Among these proteins, AvCesA3, AvCesA5, AvCesA7, AvCesA10 and AvCesA11 proteins were stable proteins, the rest of the members were unstable proteins. Except for AvCesA12 protein as a hydrophobic protein, the remaining members were hydropathicity proteins. Members of this family contained 3-14 exons and 8-15 conserved motifs. The encoded proteins were mainly distributed on the plasma membrane and the Golgi apparatus, without a clear signal peptide. The secondary structure was mainly composed of random coils and α-helices. The transmembrane domained and tertiary structure of AvCesA15 protein were significantly different from other members. Evolutionary selection of A. venetum CesA gene was mainly affected by purification selection. The analysis of cis-acting elements in the upstream 1 500 bp region showed that these gene might be regulated by environmental factors such as light, temperature, water, oxygen and plant hormones such as auxin, gibberellin, abscisic acid, ethylene, salicylic acid, and so on. This study lays a theoretical foundation for further exploring the biological function of CesA gene family and improving fiber quality and variety of A. venetum.
Key words: Apocynum venetum, fiber development, bioinformatics analysis, CesA gene family, growth and development
羅布麻(Apocynum venetum)属夹竹桃科(Apocynaceae)罗布麻属(Apocynum)多年生宿根草本植物,具有耐旱、耐寒、耐碱、适应性强等特点(王东清等,2012;刘克彪和姜生秀,2018)。随着研究的深入,罗布麻的多种经济价值被挖掘,主要体现在纤维的纺织利用(徐宗昌等,2018),黄酮类、槲皮素等化学成分的药用价值(侯晋军等,2006),茶饮保健功能(钱学射等,2005)等。自20世纪50年代,对拥有“野生纤维之王”美誉的罗布麻的研究从未间断,但其分子层次的研究还比较薄弱。纤维素合酶基因(CesA)编码纤维素合酶亚基,是广泛参与陆地植物纤维素生物合成的多基因家族(Yin et al., 2014)。因此,对纤维素合酶的研究是一个有价值的研究课题。通过在罗布麻基因组中系统鉴定CesA基因家族成员,并对其基因结构、蛋白理化性质、结构预测、进化关系进行分析,对进一步认识罗布麻纤维发育过程及培育改良罗布麻品种具有重要意义。
纤维素由24~36个经β-1,4糖苷键连接的葡聚糖残基(500~14 000个单体)链组成,这些残基通过氢键相互连接,形成规则的晶体结构,变成稳定体。纤维素是植物细胞壁的主要组成成分,也是地球上最丰富的生物聚合体和可再生碳源(Andrew & Specht, 2011)。Cantarel et al.(2009)研究显示,2-型糖基转移酶(GT2)广泛分布在植物界,而纤维素合成酶属于2-型糖基转移酶合成酶家族中的一个超基因家族。纤维素合成酶CesA基因编码的蛋白能够在质膜上以复合体的形式合成纤维素( Taylor et al., 2000;Robyn, 2001)。纤维素做为植物细胞壁的重要组成之一,不仅参与细胞形态的建成,控制细胞生长,而且还能参与到多种生物、非生物胁迫的信号应答调控网络(Burton et al., 2010)。因此,CesA基因在植物的生长发育过程中起着非常重要的作用。第一个在植物中报道的CesA基因是从棉花(陆地棉)纤维中根据其与细菌CesA基因序列的高相似性鉴定出来的(Pear et al., 1996),后续学者在模式生物拟南芥中鉴定出了10个CesA基因,命名为CesA1、CesA2……CesA10(Farrokhi et al., 2006),其中CesA1、CesA3和CesA6-like(CesA2,CesA5,CesA6或CesA9)成员之一的蛋白质产物形成具有18~24个CesA异构体的异三聚体构型的CesA复合物(CSCs),并合成初生细胞壁纤维素的成分(Desprez et al., 2007;Persson et al., 2007;Mcfarlane et al., 2014)。通过免疫沉淀和突变体分析,CesA4、CesA7和CesA8在次生细胞壁形成过程中直接相互作用,是合成纤维素所必须的(Somerville, 1997;Taylor et al., 2003)。此外,在植物生长过程中,每个重要基因的突变,造成的结果都是致命性的(例如CesA1、CesA3基因)(Persson et al., 2007)。有研究结果显示,每个CesA6-like基因之间存在部分功能冗余,在此基础上有学者提议将CesA6-like基因列为一个单独的亚家族,虽然一个CesA基因可以替代另一个,但又不能完全取代,所以推断每一个CesA6-like基因可能具有专门的功能(Ruprecht et al., 2017; Hu et al., 2018)。除模式生物外,CesA基因家族也在其他植物中被鉴定,如大麦(Hordeum vulgare)(Burton et al., 2004)、火炬松(Pinus taeda)(Naim & Haselkorn, 2005)、玉米(Zea mays)(Appenzeller et al., 2004;张晓榕等,2019)、水稻(Oryza sativa)(Yin et al., 2009)等。CesA基因家族成员在不同植物中具有相似功能,但由于环境和物种的差异,不同植物在进化过程中的不同选择,CesA基因家族成员在不同物种中均有不同程度的分化。本实验室通过对罗布麻进行全基因组测序(结果未发表),为其进化和研究相关基因的功能关系奠定理论基础,而CesA基因家族在罗布麻基因组中并未进行深入分析和比较分析。本研究利用生物信息学技术鉴定罗布麻CesA基因家族,并系统分析其在基因组中的基因结构、分布、进化特征,为阐述罗布麻CesA基因家族分化历程及生物学功能奠定基础。
1 材料与方法
材料为采自宁夏石嘴山市平罗县罗布麻实验基地的罗布麻。实验于2018—2019年在宁夏大学西北土地退化与生态恢复国家重点实验室培育基地实验室进行。
1.1 罗布麻CesA基因家族成员的鉴定
本研究主要基于罗布麻全基因组测序进行系统分析,同时利用引自美国华盛顿州艾伦斯堡(Washington Ellensburg)的大麻状罗布麻基因组测序结果进行比较分析。研究中使用的罗布麻基因组数据来自实验室前期测序所得。利用现有罗布麻注释基因的蛋白序列,与从拟南芥数据库(TAIR)(https://www.arabidopsis.org/)中检索得到的10个CesA基因家族成员蛋白序列进行BLASTP比对,E-value<1e-10,Identity>40%,输出最优比对结果,筛选罗布麻候选CesA基因。利用Pfam(http://pfam.sanger.ac.uk/search)及 SMART(http://smart.emblheidelberg. de/)进行验证分析,删除缺失结构域的序列,最终获得罗布麻CesA基因家族候选基因。
1.2 罗布麻CesA基因家族成员染色体定位
由羅布麻全基因组注释文件中,获取CesA基因在染色体上的位置信息,利用在线工具MG2C(http://mg2c.iask.in/mg2c_v2.0/)绘制染色体定位图。
1.3 罗布麻CesA基因家族成员分析
利用 ExPASy中的ProtParam(https://web.expasy.org/protparam/)工具对罗布麻CesA蛋白基本理化性质进行分析,包含氨基酸长度、理论等电点、相对分子质量、亲水性总平均值、不稳定系数和脂溶指数等指标。
1.4 罗布麻CesA基因家族成员结构和保守结构域分析
CesA基因的外显子和内含子位置信息参考罗布麻基因组注释信息GFF3文件,利用在线工具GSDS(Hu et al., 2015)(http://gsds.cbi.pku.edu.cn/)对罗布麻CesA基因家族成员进行结构分析。应用在线工具MEME(http://meme-suite.org/tools/meme)预测分析CesA蛋白序列的结构域,搜寻 motif 值设置为15,其他设定为默认参数。通过 TBtools软件绘制出 MEME 结构。
1.5 罗布麻CesA基因家族成员蛋白结构与亚细胞定位分析
利用在线平台SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/ npsa_seccons.html)分析AvCesA蛋白二级结构。同时利用SignalP-5.0 Server(http://www.cbs.dtu.dk/services/SignalP/)对罗布麻CesA蛋白进行信号肽分析。应用在线工具ProtComp Version 9.0(http://linux1.softberry.com/berry.phtml?group=programs&subgroup= proloc&topic=protcomppl)对罗布麻CesA蛋白进行亚细胞定位分析。
1.6 罗布麻CesA基因家族成员蛋白跨膜螺旋与三级结构预测
利用TMHMM Server v. 2.0(http://www.cbs.dtu.dk/services/TMHMM/)工具对罗布麻CesA蛋白跨膜螺旋进行预测。利用Phyre2(Kelley et al., 2015)(http://www.sbg.bio.ic.ac.uk/ phyre2/ html/page.cgi?id=index)对罗布麻CesA蛋白进行三级结构预测,并通过Jmol进行展示。
1.7 罗布麻CesA基因家族成员共线性与进化选择压力分析
利用MCScanX软件对罗布麻CesA基因家族与双子叶模式生物拟南芥和单子叶模式生物水稻进行共线性分析。通过BLAST建库和KaKs_ Calculator 2.0(Wang et al., 2010)对罗布麻CesA基因核苷酸的非同义替换率(Ka)与同义替换率(Ks)进行计算,获取基因的Ka/Ks比率,进行选择压力分析。
1.8 罗布麻CesA基因家族成员系统进化分析
将检索得到的拟南芥(Arabidopsis thaliana)、亚麻(Linum usitatissimum)、水稻(Oryza sativa)与罗布麻(Apocynum venetum)的CesA蛋白的氨基酸序列通过Clustal X进行蛋白序列比对,并通过邻接法(Neighbour-Joining,NJ)在MEGA-X(Kumar et al., 2018)中构建CesA基因家族系统进化树,自展值(Bootstrap)设定为1 000,其他参数为系统默认值。使用EvolView(https:// evolgenius.info//evolview-v2/#login)工具展示系统进化树。
1.9 罗布麻CesA基因家族成员顺式作用元件分析
利用TBtools软件(Chen et al., 2018)获得CesA基因上游1 500 bp序列,利用在线网站PlantCARE(http://bioinformatics. psb.ugent.be/webtools/plantcare/html/)对获得的基因上游序列进行预测,利用在线工具GSDS进行绘图展示,并对主要的顺势作用元件进行探讨。
2 结果与分析
2.1 罗布麻CesA基因家族成员的鉴定
从罗布麻全基因组中共筛选到35个拟南芥CesA的同源蛋白序列,将筛选结果输入到Pfam 数据库与SMART数据库进行比对,删除Cellulose-synt保守结构域缺失的序列,最终获得15条罗布麻CesA序列,依次命名为AvCesA1、AvCesA2……AvCesA15。
2.2 罗布麻CesA基因家族成员染色体定位与基因复制分析
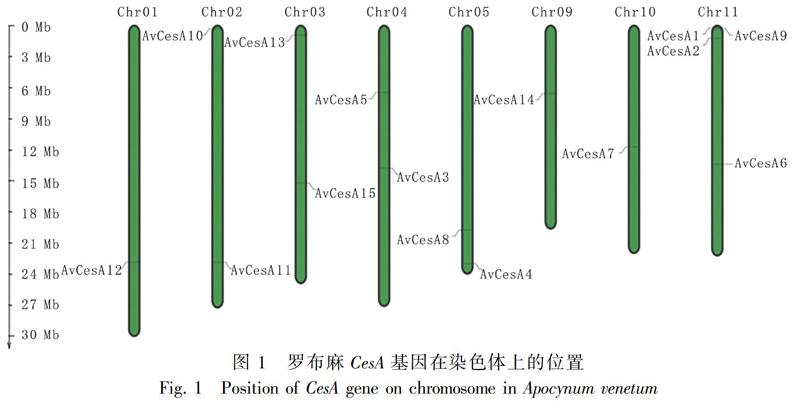
根据CesA基因染色体定位分析,15条CesA基因不均匀地分布在8条染色体上(图1)。其中,11号染色体上分布的基因最多,达到4条,1、9和10号染色体上只分布1条基因,其余染色体上各含有2条基因。基因间无串联重复现象,推断罗布麻CesA基因家族成员间存在功能分化。
2.3 罗布麻CesA基因家族成员蛋白理化性质分析
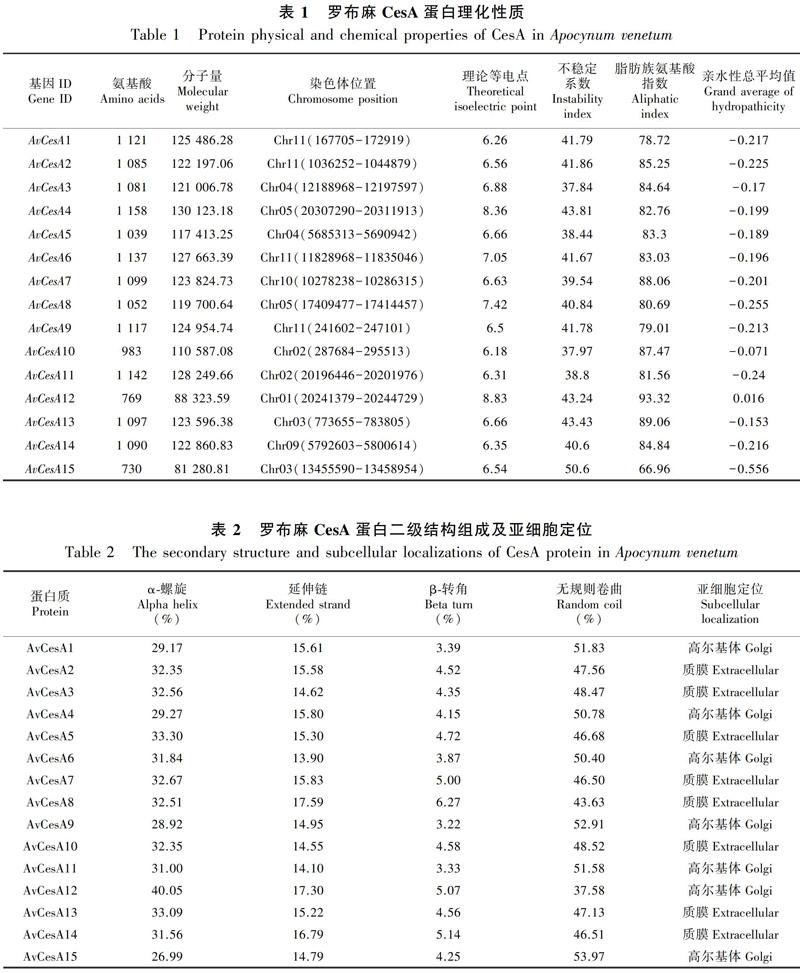
对罗布麻CesA家族蛋白的理化性质进行分析,结果表明罗布麻CesA蛋白长度在730~1 158 aa之间,相对分子质量在81 280.81~130 123.18 kDa之间,理论等电点在6.18~8.83之间,编码氨基酸序列最长的是AvCesA4,其相对分子质量最大,为130 123.18 kDa;编码氨基酸序列最小的是AvCesA15,其相对分子质量也最小,为81 280.81 kDa。当蛋白不稳定系数>40判断其为不稳定蛋白,AvCesA3、AvCesA5、AvCesA7、AvCesA10和AvCesA11蛋白不稳定系数均小于40,为稳定蛋白,AvCesA3蛋白的不稳定系数最小,为37.84;其余家族成员蛋白不稳定系数均大于40,为不稳定蛋白,AvCesA15蛋白的不稳定系数最大,为50.6。罗布麻CesA蛋白脂肪族氨基酸指数为66.96~93.32,亲水性总平均值在-0.556~0.016之间。除AvCesA12蛋白亲水性总平均值为0.016(大于0),为疏水性蛋白;其余成员蛋白亲水性总平均值均小于0,为亲水性蛋白。
2.4 罗布麻CesA基因家族成员基因结构和保守结构域分析
利用在线工具GSDS对15条罗布麻CesA基因家族成员进行基因结构分析,结果见图2。从图2可以看出,CesA基因家族外显子数量主要分布在3~14之间。利用MEME对15条罗布麻CesA蛋白序列进行分析,结果见图3。由图3可知, 共鉴定获得15个保守基序(motif), 除AvCesA12与AvCesA15外,其他家族成员均含有15个motif,且分布相对比较均匀,其分布的数量与位置也基本相同,说明具有高度的保守性,由此推断这部分罗布麻CesA基因家族成员的结构域和功能单位基本相似。AvCesA15所含基序最少,仅含有8个,AvCesA12所含基序为12个,说明AvCesA12和AvCesA15与其他罗布麻CesA家族成员可能存在功能上的差异。
2.5 罗布麻CesA基因家族成员蛋白结构特点、亚细胞定位与信号肽分析
对15条罗布麻CesA蛋白的二级结构的预测结果见表2。由表2可知,CesA蛋白均由α-螺旋、延伸链、β-转角、无规则卷曲四部分组成。其中CesA蛋白的无规则卷曲比例最高,α-螺旋次之,而β-转角比例最小。根据亚细胞定位分析,结果显示罗布麻CesA基因家族主要定位在质膜与高尔基体上,这可能与基因的生物学功能相关联。信号肽分析结果显示,罗布麻CesA基因家族成员均不含有信号肽。
2.6 罗布麻CesA基因家族成员蛋白跨膜结构域与三级结构预测
跨膜结构一般由20个左右的疏水氨基酸残基形成α-螺旋,是蛋白质与膜内蛋白的静电相互作用和氢键键合作用与膜结合的一段氨基酸片段。跨膜结构域是膜中蛋白与生物膜脂质双分子相结合的主要部位, 固着于细胞膜上起“锚定”作用。通过对跨膜结构域的预测和分析,有助于了解蛋白质的结构、功能以及在细胞中的作用位点等。通过前人研究结果可知,当前的基因组数据中,有20%~30%的基因产物被预测为膜蛋白(Taylor et al., 2000),具备多种生物学功能。利用TMHMM对罗布麻CesA基因家族成员蛋白的跨膜结构进行预测,结果见图4。从图4可以看出,除AvCesA12与AvCesA15蛋白外,其余成员蛋白在C端均含有6个完整的跨膜结构域,且每个组成跨膜结构域的氨基酸数量与位置均相似,推断其可能有相同的结构和功能。AvCesA12与AvCesA15可能因为发生突变,相应功能发生变化,从而导致跨膜结构缺失。除AvCesA2、AvCesA3、AvCesA4、AvCesA10、AvCesA13与AvCesA14蛋白外,其余成員蛋白在近N端均含有2个完整的跨膜结构域。
通过Phyre2对罗布麻CesA蛋白进行三级结构预测,结果见图5。由图5可知,除AvCesA15蛋白外,其余成员蛋白三级结构在空间结构上相似度很高,推断其可能具有相似的生物学功能,而AvCesA15结构差异较大,可能发生变异,导致其结构与功能发生改变。
2.7 罗布麻CesA基因家族成员共线性与进化选择压力分析
利用MCScanX将罗布麻分别与双子叶模式生物拟南芥和单子叶模式生物水稻建立2组CesA基因家族的共线性比较图谱(图 6)。共线性关系(同源基因对)在拟南芥CesA中有11对,水稻CesA中有4对。其中AvCesA4与AvCesA8在两个图谱中都存在共线性关系。相较于双子叶植物,罗布麻CesA基因家族与单子叶植物存在共线性关系的基因较少,其原因可能是植物起源基因组不同而造成的。根据Abrouk et al.(2010)的研究,双子叶植物基因组可能源于7条古染色体,单子叶植物基因组可能源于5条古染色体。由此,我们推断AvCesA4与AvCesA8在进化过程中高度保守,其功能有待发掘。
非同义替换率(Ka)与同义替换率(Ks)的比值可以判断是否有选择压力作用于这个蛋白质编码基因,从而反映生物进化的过程中所受到的选择压力。对来源于罗布麻CesA基因家族成员间亲缘关系最近的8条基因序列进行Ka/Ks分析,结果见表3。由表3可知,基因对之间的Ka/Ks的比值均小于1,显示罗布麻CesA基因在进化时主要受纯化选择作用。
2.8 罗布麻CesA基因家族成员系统进化分析
为进一步了解罗布麻CesA基因家族进化关系,通过MEGA-X对罗布麻(15条)、拟南芥(10条)、亚麻(14条)、水稻(11条)共50条CesA蛋白序列构建NJ系统进化树(图7)。由图7可知,聚类树分为5个亚族,罗布麻CesA基因在每一亚族中均有分布,表明各物种间CesA基因家族具有明显的同源关系。
2.9 罗布麻CesA基因家族成员顺式作用元件分析
通过对罗布麻CesA基因上游1 500 bp序列进行分析,预测基因的顺式作用元件,结果见图8。从图8可以看出,将CesA基因家族顺式作用元件划分为基础元件、植物激素调控元件、逆境胁迫相关元件、光反应元件、生长发育调控元件等5大类。基础元件包括TATA-box和CAAT-box等,数量最多。光反应元件包括Sp1、MRE、GT1-motif、G-Box、G-box、3-AF1 binding site、ACE、ATC-motif、ACA-motif、TCT-motif等,数量排第2位。植物激素调控元件包含生长素响应元件TGA-element、AuxRR-core,赤霉素响应元件P-box、F-Box、GARE-motif、CARE,乙烯应答元件ERE,脱落酸响应元件ABRE、ABRE4,水杨酸响应元件TCA-element,参与到茉莉酸甲酯信号转导途径的TGACG-motif、CGTCA-motif,茉莉酮酸酯和激发子应答元件JERE。逆境胁迫相关元件包含损伤诱导元件WRE3、WUN-motif,损伤与病原响应元件W-box,防御与应激反应元件TC-rich repeats,低温调控元件TCA、LTR,胁迫响应元件STRE,干旱响应元件MBS,缺氧特异性诱导GC-motif,干旱和渗透胁迫诱导元件DRE1,厌氧诱导调控元件ARE。生长发育调控元件包含胚乳表达所需元件GCN4_motif,玉米醇溶蛋白代谢调控中的调控元件O2-site,参与种子特异性调控的顺式作用调控元件RY-element,叶肉细胞分化的相关元件HD-Zip1,参与细胞周期调控的顺式作用元件MSA-like,与分生组织特异性激活相关的顺式调控元件CCGTCC-box,与分生组织表达相关的顺式作用调控元件CCGTCC motif 、CAT-box,参与昼夜节律调控的顺式调控元件circadian,根特异表达顺式作用调控元件as-1。罗布麻CesA基因可能受到光、温度、水分、氧气等环境因子及内源激素调控,并且这些顺式作用元件有可能参与到罗布麻抗逆机制中,在植物生长发育过程中起到防御与保护作用。除此之外,该段序列富含参与植株生长发育的顺式作用元件,在植株的生长与纤维素合成中有重大作用。
3 讨论与结论
对罗布麻、亚麻、拟南芥、水稻CesA基因家族构建进化树,由聚类分析可知, AvCesA2与AvCesA3与拟南芥中参与初生细胞壁纤维素合成基因AtCesA1、AtCesA3(Desprez et al., 2007)聚类在同一分支,推断这些罗布麻CesA基因可能参与初生细胞壁纤维素的合成。AvCesA5、AvCesA8、AvCesA10分别与拟南芥中参与次生细胞壁纤维素合成基因AtCesA7、AtCesA4、AtCesA8(Arioli et al., 1998)的亲缘关系较近,因此推断这些AvCesA基因也可能参与次生细胞壁纤维素的合成。AvCesA1、AvCesA4、AvCesA6、AvCesA9、AvCesA11、AvCesA12、AvCesA15聚类在同一分支,同缘关系显著,可知这些基因可能具有类似的生物功能。
拟南芥CesA基因家族蛋白在N端都含有一个保守的锌指蛋白结构域,该保守域能在CesA蛋白形成蛋白复合体时进行蛋白之间的相互识别(周晓馥等,2002)。通过对罗布麻CesA基因家族蛋白保守基序分析显示,多数成员包含的保守结构域基本一致,具有高度的保守性,这说明蛋白功能表达方面存在相似性。AvCesA12 与AvCesA15相比较,缺失部分保守结构域,可能是基因序列发生突变引起,从而使蛋白执行功能发生变化,这一现象在跨膜结构预测和蛋白三级结构中均有体现。AvCesA12蛋白的跨膜结构预测中C端第六个跨膜结构缺失,AvCesA15蛋白的跨膜结构预测中C端无跨膜结构,并且三级构象发生明显变化。在罗布麻CesA蛋白跨膜结构预测中发现最多包含8个跨膜结构,与Nawaz et al.(2019)的总结一致,在植物CesA蛋白中最多存在8个跨膜结构域。徐昌宗等(2017)的研究结果发现,普通烟草CesA蛋白跨膜结构数量差异与该基因参与初生细胞壁或者次生细胞壁纤维素合成间存在一定关系。罗布麻CesA蛋白跨膜结构数量存在差异,以此为思路有助于探究罗布麻CesA基因的功能。
顺式作用元件能被特定的转录因子识别,对下游基因的特异性表达起调控作用。对罗布麻CesA基因上游1 500 bp区域进行顺式作用元件分析,該区域包含多种顺式作用元件,在植物生长发育过程中可能存在重要作用。水杨酸是参与植物光敏反应和系统获得抗性反应的重要信号分子(陈冲等, 2018),茉莉酸甲酯在植物受到病原侵害、创伤或者胁迫时诱导防御基因表达的信号分子,且在植株生命周期的各个阶段也发挥着调节作用(Titarenko et al., 1997)。除此之外,多种激素与环境因素可以影响CesA基因的表达。魏凯莉等(2017)的研究显示,赤霉素处理下杨树次生壁CesA基因表达量明显上调,而生长素与乙烯处理下杨树次生壁CesA基因表达量下调,影响细胞壁中纤维素含量。油菜素甾醇和光照可以促进拟南芥下胚轴伸长和初生壁CesA基因转录,促使拟南芥株形增高(Xie et al., 2011;Leivar & Quail, 2011)。在水稻中,赤霉素信号转导可以促进纤维素的合成,影响水稻植株节间的发育(Huang et al., 2015)。拟南芥AtCesA8突变体植株,抗旱性显著增强(Chen et al., 2005)。龙眼DiCesA基因被脱落酸处理表达量下调,可能参与脱落酸对干旱胁迫的调控,通过对MeJA处理下DiCesA基因表达量下调,证实DiCesA参与龙眼对干旱胁迫的调控(朱永静等,2020)。玉米CesA基因在种子生长阶段参与抵御干旱胁迫的调控(张晓榕等,2019)。在罗布麻AvCesA基因家族成员的启动子区域都包含多种激素响应元件,并且80%以上包含防御与逆境响应元件,60%以上包含了干旱胁迫以及光响应MYB结合位点。由此可见,CesA基因家族包含的大量光响应、胁迫响应、激素响应等元件,在植株生长发育与抗逆防御方面发挥着巨大作用。
在罗布麻纤维发育有关的CesA类基因家族中,被报道的基因数目很少,并且大多数基因的功能还不清楚。本研究通过生物信息学方法分析了罗布麻基因组中的CesA基因家族的鉴定、基因结构、蛋白特性及顺式作用元件分析等,对于发掘新的与植物纤维发育相关的基因,加快罗布麻纤维发育分子机制的研究具有十分重要的意义,也为罗布麻培育高质量纤维育种研究奠定了基础。
参考文献:
ABROUK M, MURAT F, PONT C, et al.,2010. Palaeogenomics of plants: synteny-based modelling of extinct ancestors[J]. Trend Plant Sci, 15(9): 479-487.
ANDREW C, SPECHT CD, 2011. Understanding plant cellulose synthases through a comprehensive investigation of the cellulose synthase family sequences[J]. Front Plant Sci, 2(5): 2-5.
APPENZELLER L, DOBLIN M, BARREIRO R, et al.,2004. Cellulose synthesis in maize: Isolation and expression analysis of the cellulose synthase (CesA) gene family[J]. Cellulose, 11(3-4): 287-299.
ARIOLI T, PENG LC, BETZNER AS, et al.,1998. Molecular analysis of cellulose biosynthesis in Arabidopsis[J]. Science, 279(5351): 717-720.
BURTON RA, 2004. The CesA gene family of Barley. Quantitative analysis of transcripts reveals two groups of co-expressed genes[J]. Plant Physiol, 134(1): 224-236.
BURTON RA, GIDLEY MJ, FINCHER GB, 2010. Heterogeneity in the chemistry, structure and function of plant cell walls[J]. Nat Chem Biol, 6(10): 724-732.
CANTAREL BL, COUTINHO PM, RANCUREL C, et al.,2009. The carbohydrate-active enzymes database (CAZy): an expert resource for glycogenomics[J]. Nucl Acid Res, 37(Suppl. 1): D233-D238.
CHEN C, LIU S, WANG DD, et al.,2018. Identification of PCD induced by salicylic acid and expression analysis of genes related with the PCD in cucumber[J]. Acta Agric Boreal-Sin, 33(6): 60-67.[陳冲,刘双,王丹丹,等,2018. 水杨酸诱导黄瓜PCD的鉴定及相关基因的表达分析[J]. 华北农学报,33(6):60-67.]
CHEN CJ, CHEN H, ZHANG Y, et al.,2020. TBtools: An integrative toolkit developed for interactive analyses of big biological data[J]. Mol Plant, 13(8): 1194-1202.
CHEN ZZ, HONG XH, ZHANG HR, et al., 2005. Disruption of the cellulose synthase gene, AtCesA8/IRX1, enhances drought and osmotic stress tolerance in Arabidopsis[J]. Plant J, 43(2): 273-283.
DESPREZ T, JURANIEC M, CROWELL EF, et al.,2007. Organization of cellulose synthase complexes involved in primary cell wall synthesis in Arabidopsis thaliana[J]. Proc Natl Acad Sci, 104(39): 15572-15577.
FARROKHI N,BURTON RA, BROWNFIELD L, et al.,2006. Plant cell wall biosynthesis: Genetic, biochemical and functional genomics approaches to the identification of key genes[J]. Plant Biotechnol J, 4(2): 145-167.
HOU JJ, HAN LW, YANG GE, et al.,2006. Advances in studies on chemical constituents and pharmacological activities of Apocatharum venetum[J]. Chin Trad Herb Drugs, 37(10): 1603-1605.[侯晋军, 韩利文, 杨官娥, 等, 2006. 罗布麻叶化学成分和药理活性研究进展[J]. 中草药, 37(10):1603-1605.]
HU B, JIN JP, GUO AY, et al., 2015. GSDS 2.0: An upgraded gene feature visualization server. Bioinformatics, 31(8): 1296-1297.
HU HZ, ZHANG R, FENG SQ, et al., 2018. Three AtCesA6-like members enhance biomass production by distinctively promoting cell growth in Arabidopsis[J]. Plant Biotechnol J, 16(5):976-988.
HUANG D, WANG S, ZHANG B, et al., 2015. A gibberellin-mediated DELLA-NAC signaling cascade regulates cellulose synthesis in rice[J]. Plant Cell, 27(6): 1681-1696.
KELLEY LA, MEZULIS S, YATES CM, et al., 2015. The Phyre2 web portal for protein modeling, prediction and analysis[J]. Nat Protoc, 10(6): 845-858.
KROGH A, LARSSON B, VON HG, et al., 2001. Predicting transmembrane protein topology with a hidden Markov model: Application to complete genomes[J]. J Mol Biol, 305(3): 567-580.
KUMAR S, STECHER G, LI M, et al., 2018. MEGA X: Molecular evolutionary genetics analysis across computing platforms[J]. Mol Biol Evol, 35(6): 1547-1549.
LEIVAR P, QUAIL PH, 2011. PIFs: Pivotal components in a cellular signaling hub[J]. Trends Plant Sci, 16(1): 19-28.
LIU KB, JIANG SX, 2016. Responses of Apocynum venetum seed germination to drought and salt stress[J]. Acta Pratac Sin, 25(5): 214-221.[劉克彪, 姜生秀, 2016. 干旱和钠盐胁迫对罗布麻种子萌发的影响[J]. 草业学报, 25(5):214-221.]
MCFARLANE HE, DRING A, PERSSON S, 2014. The cell biology of cellulose synthesis[J]. Ann Rev Plant Biol, 65(1): 69-94.
NAIRN CJ, HASELKORN T, 2005. Three loblolly pine CesA genes expressed in developing xylem are orthologous to secondary cell wall CesA genes of angiosperms[J]. New Phytol, 166(3): 907-915.
NAWAZ MA, LIN XA, CHAN TF, et al., 2019. Characterization of cellulose synthase A (CESA) gene family in eudicots[J]. Biochem Genet, 57(2): 248-272.
PEAR JR, KAWAGOE Y, SCHRECKENGOST WE, et al., 1996. Higher plants contain homologs of the bacterial cela genes encoding the catalytic subunit of cellulose synthase[J]. Proc Natl Acad Sci, 93(22): 12637-12642.
PERSSON S, PAREDEZ A, CARROLL A, et al., 2007. Genetic evidence for three unique components in primary cell-wall cellulose synthase complexes in Arabidopsis[J]. Proc Natl Acad Sci, 104(39): 15566-15571.
QIAN XS, ZHANG WM, GU GP, et al., 2005. Health tea development of Apocynum venetum L. and its medicinal diet[J]. Chin Wild Plant Resour, 24(6): 21-25.[钱学射, 张卫明, 顾龚平, 等, 2005. 罗布麻保健茶的开发与药膳[J]. 中国野生植物资源, 24(6):21-25.]
ROBYN M, PERRIN, 2001. Cellulose: how many cellulose synthases to make a plant?[J]. Curr Biol, 11(6): R213-R214.
RUPRECHT C, PROOST S, HERNANDEZ-CORONADO M, et al., 2017. Phylogenomic analysis of gene co-expression networks reveals the evolution of functional modules[J]. Plant J, 90(3): 447-465.
SOMERVILLE TCR, 1997. Collapsed xylem phenotype of Arabidopsis identifies mutants deficient in cellulose deposition in the secondary cell wall[J]. Plant Cell, 9(5): 689-701.
TAYLOR NG, HOWELLS RM, HUTTLY AK, et al., 2003. Interactions among three distinct CesA proteins essential for cellulose synthesis[J]. Proc Natl Acad Sci USA, 100: 1450-1455.
TAYLOR NG, LAURIE S, TURNER SR, 2000. Multiple cellulose synthase catalytic subunits are required for cellulose synthesis in Arabidopsis[J]. Plant cell, 12(12): 2529-2540.
TITARENKO E, ROJO E, LEN J, et al., 1997. Jasmonic acid-dependent and independent signaling pathways control wound-induced gene activation in Arabidopsis thaliana [J]. Plant Physiol, 115(2): 817-826.
WANG DP, ZHANG YB, ZHANG Z, et al., 2010. KaKs_Calculator 2.0: A toolkit incorporating gamma-series methods and sliding window strategies[J]. Genom Proteom Bioinform, 8(1): 77-80.
WANG DQ, LI GQ, SU DX, 2012. Effect of drought stress on osmotic adjustment substances and activity of protective enzymes in two species of Apocynum[J]. J Arid Land Resour Environ, 26(12): 177-181.[王东清, 李国旗, 苏德喜, 2012. 干旱胁迫对两种罗布麻渗透调节物质积累和保护酶活性的影响[J]. 干旱区资源与环境, 26(12): 177-181.]
WEI KL, ZHOU HJ, JIANG C,et al., 2017. Interaction and expression of secondary wall CESAs in Populus [J]. For Res, 30(2): 245-253.[魏凯莉, 周厚君, 江成, 等, 2017. 杨树次生壁纤维素合酶的表达与互作模式分析[J]. 林业科学研究, 30(2): 245-253.]
XIE L, YANG C, WANG X, 2011. Brassinosteroids can regulate cellulose biosynthesis by controlling the expression of CESA genes in Arabidopsis[J]. J Exp Bot, 62(13): 4495-4506.
XU ZC, KONG YZ, 2017. Genome-wide identification, subcellular localization and gene expression analysis of the members of CESA gene family in common tobacco[J]. Hereditas, 39(6): 512-524.[徐宗昌, 孔英珍,
2017. 普通烟草CESA基因家族成员的鉴定、亚细胞定位及表达分析[J]. 遗传, 39(6): 512-524.]
XU ZC, ZHOU JH, ZHANG CS, et al., 2018. Review of current research and utilization status of Apocynum venetum germplasm in China[J]. Bull Bot, 53(3): 382-390.[徐宗昌, 周金辉, 张成省, 等, 2018. 我国罗布麻种质资源研究利用现状[J]. 植物学报, 53(3): 382-390.]
YIN Y, HUANG J, XU Y, 2009. The cellulose synthase superfamily in fully sequenced plants and algae[J]. Bmc Plant Biol, 9(1): 99-101
YINY, JOHNS MA, CAO H, et al., 2014. A survey of plant and algal genomes and transcriptomes reveals new insights into the evolution and function of the cellulose synthase superfamily[J]. BMC Genom, 15(1): 260.
ZHOU XF, WANG JY, WANG XZ, 2002. Research progress of cellulose synthase genes in higher plant[J]. Hereditas, 24(3): 376-378.[周曉馥, 王景余, 王兴智, 2002. 植物纤维素合成酶基因的研究进展[J]. 遗传, 24(3): 376-378.]
ZHANG XR, TAN JF, WEN MQ, et al., 2019. Systematic identification and functional study of CesA family in maize[J]. J NW A & F Univ (Nat Sci Ed), 47(2): 45-53.[张晓榕, 谭俊峰, 温曼晴, 等, 2019. 玉米CesA家族的系统鉴定及功能研究[J]. 西北农林科技大学学报(自然科学版), 47(2): 45-53.]
ZHU YJ, LU BS, ZHANG ST, et al., 2020. Identification and expression patterns of the cellulose synthase gene family in Dimocarpus longan LOUR[J]. Chin J Appl Environ Biol, 26(5): 1235-1243.[朱永静, 路保顺, 张舒婷, 等, 2020. 龙眼纤维素合成酶基因家族成员鉴定及表达模式[J]. 应用与环境生物学报, 26(5): 1235-1243.]
(责任编辑 何永艳)
