基于条件能量对抗网络的肝脏和肝肿瘤分割
2021-06-11王卫卫
闫 谙,王卫卫
西安电子科技大学 数学与统计学院,西安710071
肝癌是最致命的癌症之一,其致死率在恶性肿瘤中位居第三[1]。肝脏和肝肿瘤影像的分割在临床诊断中具有重要意义。传统肝脏和肝肿瘤分割方法包括图割[2]、区域增长[3]、水平集[4]、形变模型[5]等。这些方法大多依赖于人工设计特征,由于肝脏区域结构或纹理等特征的复杂性,人工设计的特征不能完全描述医学图像特征,导致分割精度不佳。卷积神经网络(Convolutional Neural Network,CNN)[6]可以从数据中自动学习图像特征,取得了很好的分割效果,是目前用于肝脏和肝肿瘤分割的主要神经网络结构。2015年,Long等人提出了全卷积网络(Fully Convolutional Network,FCN)[7],FCN将VGG网络[8]的全连接层替换为卷积层,首次将CNN应用于语义分割中。2016年,Ben-Cohen等人[9]首次使用FCN解决肝脏和肝肿瘤分割问题。Unet[10]以FCN为主干充分挖掘了图像的多尺度特征。Seo等人[11]在Unet的基础上增加了残差路径提升了肝脏和肝肿瘤的分割精度。上述神经网络在分割过程中通过对像素独立分类来实现肝脏和肝肿瘤的自动分割,但是存在一定缺陷,主要表现为目标边界和小体积目标分割精度不佳。造成这一缺陷的原因是没有充分考虑像素类别之间的高阶一致性[12]。为充分利用邻域内像素类别的相关性,Dense CRF[13]将条件随机场(Conditional Random Field,CRF)作为FCN的后处理,将FCN的输出作为一阶势函数,利用二阶势函数描述像素之间的类别关系,提升了分割效果。DenseCRF的结果表明利用高阶关系可以提升分割精度。
然而使用数学方法描述像素类别之间的二阶以上关系十分困难,并且难以求解。因此,本文使用一个卷积网络用于像素分类实现肝脏和肝肿瘤分割,使用另外一个网络学习像素类别的高阶关系,并将两个网络形成生成对抗网络(Generative Adversarial Networks,GANs)[14]结构,通过对抗训练的方法从样本中学习特征,提升肝脏和肝肿瘤的分割精度。为将GANs结构用于肝脏和肝肿瘤分割,并减少GANs训练中出现的梯度消失和梯度爆炸等问题,本文基于能量生成对抗网络(Energy-Based Generative Adversarial Network,EBGAN)[15]和条件生成对抗网络(Conditional Generative Adversarial Network,CGAN)[16]提出了条件能量对抗网络。具体地,将Unet[10]作为EBGAN框架中的生成器,将自编码器(Auto-Encoder,AE)作为判别器。Unet用于生成肝脏和肝肿瘤的分割结果,自编码器用于计算分割结果与真实标注的能量差异值,将该差异值作为损失值对生成器进行训练。在生成器与判别器的对抗训练过程中,判别器作为一种损失函数可以从图像中学习像素类别之间的高阶相关性,从而提升小目标和目标边界分割精度[12,17]。在EBGAN的基础上使用原始医学图像作为条件约束,以提升Unet输出的分割精度。本文提出的方法实现了端到端训练,不需要后处理。在LiTS 2017数据集和3DIRCADb公开数据集上对提出的方法进行验证,实验结果表明该方法在肝脏和肝肿瘤分割任务上具有一定的有效性和一般性。
1 相关算法
本章主要介绍一些相关算法,包括GANs[14]、EBGAN[15]和CGAN[16]。
1.1 GANs
GANs由生成器和判别器组成,可通过求解如下最大最小博弈问题来训练GANs网络:
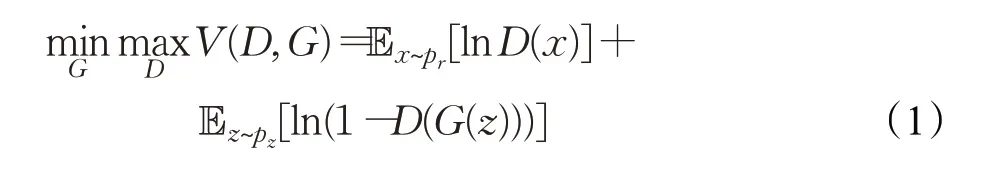
其中,D(⋅)和G(⋅)表示判别器和生成器,z表示从噪声分布pz中随机采样的噪声,x表示从真实分布pr中采样得到的真实样本。由生成器生成的图像输入到判别器中接受真假判别,判别器输出概率值越接近1说明生成器的生成效果越好。当且仅当生成分布pg等于真实分布pr时,最大最小博弈问题达到最优。GANs存在以下不足之处,训练不稳定,判别器不能预训练以及生成数据内容不可控。
1.2 EBGAN
GANs是基于概率的模型,生成分布与真实分布的支撑集之间可能没有交集,使得KL散度没有定义,最终导致梯度消失或梯度爆炸,因此GANs存在一定训练难度。而EBGAN以能量理论为模型,不再考虑生成分布、真实分布以及分布之间的距离,而是将判别器视为能量函数。能量函数将输入映射为一个能量值,并通过能量值的高低来评估生成器的生成能力,若生成数据能量值较低,说明生成器生成的样本接近真实数据。
EBGAN将能量函数作为判别器,为判别器结构和损失函数的设计提供了更大的灵活性。基于边际损失,EBGAN的判别器和生成器的目标函数如下:

其中,LD和LG分别表示判别器和生成器的目标函数,m为大于0的超参数,[]⋅+=max(0,⋅)。与传统GANs不同,EBGAN的判别器可以使用真实数据预训练。
1.3 CGAN
为控制生成器的生成内容,CGAN将类别标签等额外信息作为约束条件引入到生成器和判别器中。CGAN的目标函数为:

其中,y表示条件变量。在某种程度上,CGAN可被视为一个有监督模型,条件变量可被视为监督信息。
2 本文方法
Unet等基于CNN的分割网络通过独立预测每个像素点的类别标签来实现对肝脏和肝肿瘤的自动分割。然而,这些分割网络忽略了相邻像素类别之间的高度相关性。尽管Dense CRF[13]通过二阶势函数来考虑像素类别之间的一致性,但是高阶一致性更有利于提高分割精度。与定义复杂的高阶势函数不同,对抗网络可以直接从数据中学习像素类别之间的高阶一致性[12,17]。
鉴于EBGAN的优点,本文采用EBGAN进行肝脏和肝肿瘤分割。EBGAN的生成器和判别器均采用基于CNN的网络结构。对于生成器,为充分利用输入图像的多尺度特征,本文使用Unet[10];对于判别模型,本文使用自编码器。本文的网络与EBGAN的主要区别在于使用原始医学图像作为条件约束来控制预测分割结果的内容;此外,将交叉熵损失和dice损失[18]加入到目标函数中,使得预测分割结果与真实标注尽可能接近,同时避免样本不均衡问题。本文方法不需任何后处理。
2.1 模型结构
本文使用Unet和一个改进的自编码器作为生成器和判别器,网络结构如图1所示。以原始医学图像作为输入,生成器用于产生对应的预测分割结果。将真实标注和预测分割结果输入到判别器中,计算重构图像与真实标注之间的重构误差。此外,将原始医学图像作为条件约束输入到判别器中。

图1 提出的网络结构
2.2 生成器
本文使用的Unet网络结构如图2所示。该结构由一个收缩路径和一个扩张路径构成,这种结构可以充分利用图像的多尺度特征来获取分割结果。收缩路径用于提取不同尺度的特征,扩张路径用于将这些特征恢复至与原始图像相同的分辨率。

图2 Unet的模型结构
Unet以原始医学图像作为输入,输出对应的预测分割结果。在特征提取阶段,通过最大池化对原始图像进行4次下采样以提取不同尺度特征。每个尺度上的特征提取均由两次重复卷积,批归一化和LeakyReLU实现。此外,通道的数量在每一次最大池化后加倍。Unet对收缩路径最后一个尺度上的特征进行了4次上采样。通道数量在每次上采样过程中减半。由于在每一次下采样过程中都会丢失图像上下文信息,因此引入跳跃连接来连接扩张路径和收缩路径对应尺度上的特征,以保留图像上下文信息。为了限制数值范围,Unet的最后一层添加了softmax激活函数。
为训练Unet,最小化如下目标函数:

其中,xraw和xlab分别表示原始医学图像及其对应的真实标注。xpre表示Unet对原始医学图像的分割结果。x͂pre表示Unet输出的分割结果经判别器产生的重构图像。式(4)中的目标函数采用LCE、Ldice和Lrec的加权形式。μ、λ和β为对应项的权重。LCE表示交叉熵,用于减少Unet的分割结果与真实标注之间的差异。Ldice表示dice损失,用于避免样本不均衡问题。Ldice的具体形式为:

Lrec是来自判别器的重构误差。在原始医学图像的条件约束下,将Unet的分割结果输入到判别器中,使用判别器输出与真实标注计算Lrec。在Unet与判别器的对抗训练过程中,通过减小Lrec使Unet从数据中学习像素类别之间的高阶关系。
2.3 判别器
本文使用与自编码器相似的结构作为判别器,如图3所示。自编码器由编码器和解码器组成,编码器用于提取特征,解码器用于从提取的特征中重构出图像。特征提取部分主要由4个重复的卷积组成,每个卷积后接一个批归一化和LeakyReLU。此外,使用4次最大池化进行下采样。当图像特征提取完成后,对应地使用4次上采样从提取的特征中重构出图像。

图3 AE的模型结构
为训练判别器,最小化如下目标函数:

其中,xlab表示与原始输入图像对应的真实标注,表示将真实标注输入到自编码器后得到的重构图像,Lrec为xlab与之间的重构误差。在原始医学图像的条件约束下,本文中的判别器仅使用真实标注进行训练,目的是令判别器只充分学习真实标注的特征。在此情况下,判别器为真实样本分配较低能量值,为生成样本分配较高能量值。
3 实验
3.1 数据集和预处理
本文在MICCAI 2017肝脏肿瘤分割(LiTS)数据集[19]和3DIRCADb公开数据集[20]上验证提出的方法。LiTS 2017数据集包含131个训练样本和70个测试样本。3DIRCADb数据集包含20个样本。随机从LiTS 2017的训练样本中抽取90%作为训练集,剩余的10%作为验证集。为进一步证明提出方法的有效性和一般性,在3DIRCADb数据集上验证提出的方法。所有样本的分辨率均为512×512,但通道数目各不相同。
训练模型之前需要对数据进行预处理。数据的预处理包括通道筛选、CT值截取、数据增广以及归一化。为避免样本不均衡,从训练集中筛选出17 227个带有标注的通道作为最终训练集。CT值通常用来判断病理组织的性质和类型。为去除干扰细节,将所有CT值截断至[−200,250]HU范围内。为避免训练样本不足造成的过拟合,使用仿射变换和对比度变换对数据集进行了扩充。将输入医学图像的每个像素值归一化至[0,1]范围内,方法如下:

其中,Pij表示任意原始像素点的像素值,Pmin和Pmax表示一个输入图像内所有像素值的最小值和最大值,P̂ij表示归一化后的像素值。
3.2 评估方法
本文采用全局Dice、平均Dice、体积重叠误差(VOE)以及相对体积差异(RVD)作为评估指标。全局Dice是通过将所有样本合并为一个单独的样本来计算的,平均Dice是对所有样本的Dice求平均来计算的。给定2个二值分割图A和B,Dice,VOE和RVD的计算方式如下:

全局Dice和平均Dice的值均在[0,1]之间,并且值越接近1越说明分割效果好。VOE与RVD的值为0时说明分割效果最理想。
3.3 实验设置
本文使用Pytorch框架在单台NVDIA 2080ti GPU上对提出的方法进行训练和验证。生成器使用的是原始Unet。在本文中,自编码器由4个重复的卷积组成,卷积核大小为3×3,通道数量为32,64,128,256,256,128,64,32。此外,将所有Leaky ReLU的负斜率设置为0.1。公式(4)和公式(6)分别作为训练Unet和自编码器的损失函数。本文施加条件约束的方式为将真实标注与预测分割结果分别与原始医学图像拼接,然后输入到判别器中。
3.4 对比实验与分析
为证明提出方法的有效性,设置3组对比实验分别用于验证本文方法对于提升Unet分割性能的有效性,施加条件约束的有效性以及本文方法相比于其他改进GANs的优势。以上3组实验均在相同实验设置下完成。所有对比实验的训练轮数均为100轮,利用LiTS 2017的训练集对模型进行训练,训练使用的样本数量均为17 227个,初始学习率为1E−4,优化器为Adam。本文的损失函数采用了交叉熵损失、dice损失和重构误差的加权形式。各损失项的计算方法是将肝脏器官、肝脏肿瘤和背景视作3个类别分别计算损失值,然后将各损失值进行加权求和作为该损失项整体损失值。对样本中肿瘤、器官区域和背景区域占据图像体积的比例进行统计作为确定超参数的依据,并结合实验过程最终将肝脏、肝肿瘤和背景的加权系数分别设置为0.09、0.9和0.01。为便于在[0,1]范围内观察并对比各项损失值,将损失函数中损失项的超参设置为μ=λ=β=1。图4和图5分别为3组对比实验的dice损失曲线和交叉熵损失曲线。

图4 dice损失曲线

图5 交叉熵损失曲线
3.4.1 条件EBGAN的有效性
本文将Unet嵌入到改进的EBGAN中,通过对抗训练,提升模型的分割性能。为了与未使用对抗的Unet对比,采用交叉熵损失和dice损失训练独立的Unet。根据公式(5)和公式(8)可以看出,dice损失曲线不仅可以反映训练过程中损失值的变换规律,还可以体现出Dice指标的提升情况。由图4中的dice损失曲线观察发现,本文方法的损失曲线低于Unet,说明本文方法的分割精度高于Unet。交叉熵损失曲线反映了像素分类情况,由图5可知本文方法的交叉熵损失值低于Unet,说明本文方法的分类效果略有提升。图6展示了本文方法和Unet在LiTS验证集上的分割结果。通过观察可知,Unet有时无法分割出输入图像中的小体积肿瘤,并且对于目标边缘处的分割效果不佳。从表1中的评估指标也可看出,本文方法与Unet相比对于肝脏分割的平均Dice和全局Dice分别提升了0.8个百分点和0.6个百分点,对于肝肿瘤分割的平均Dice和全局Dice均提升了2.5个百分点。本文将Unet神经网络嵌入到条件能量生成对抗网络的框架下,通过对抗训练学习像素类别之间的高阶一致性,有效提升了小体积目标和目标边界的分割精度。

图6 原始Unet及本文方法在LiTS验证集上的分割结果(灰色区域表示器官,白色区域表示肿瘤)

表1 本文方法以及对比方法在LiTS验证集上的分割结果%
3.4.2 条件约束的有效性
为证明条件约束在本文方法中的重要性,将本文方法与无条件约束的EBGAN进行对比。由于原始图像包含较为精确的位置信息,因此本文将原始图像与对应的Unet输出相拼接后作为判别器输入,加强判别器学习图像高阶结构信息的能力。由图4和图5可知,本文方法的dice损失曲线和交叉熵损失曲线均优于无条件约束的EBGAN。对比表1中的验证指标可知,本文方法的分割性能优于无条件约束的EBGAN。
3.4.3 与其他改进GANs的对比
将相同设置的Unet分别嵌入到CGAN[16]和WGANGP[21]的框架下以与本文方法进行对比。以Unet作为模型的生成器,并将原始输入图像作为CGAN和WGAN-GP判别器的约束条件。将条件约束下的WGAN-GP记为WGAN-GP-C。由图4和图5可以看出,本文方法的损失曲线始终低于CGAN和WGAN-GP-C,并且CGAN和WGAN-GP-C的损失曲线均出现了波动,而本文方法的损失曲线平滑下降。EBGAN的判别器使用能量模型代替传统GANs的概率模型,能量模型相对于概率模型的优势是判别器可以预训练[15],并且训练过程比较稳定。对比表1中的评估指标可知,本文方法的分割性能优于CGAN和WGAN-GP-C。
3.5 方法的一般性验证
3DIRCADb数据集相比于LiTS 2017数据集具有更高的复杂性和多样性。3DIRCADb数据集中包含20个样本,其中15个样本中同时包含肝脏和肿瘤。为证明本文方法的一般性,利用3DIRCADb数据集中的这15个样本对使用LiTS数据集训练好的模型进行验证。3DIRCADb数据集采用的评估指标是Dice、VOE和RVD,其中Dice是指LiTS数据集中使用的平均Dice。图7展示了本文方法在3DIRCADb数据集上的分割效果。从图7中可以看出,当原始输入图像中肿瘤的体积较小或者与肝脏的对比度较低时,Unet出现肿瘤漏检的情况。对于体积较大的肿瘤,Unet分割出的肿瘤体积小于真实标注。肝脏分割的结果显示,Unet出现了小体积空洞。此外,Unet在目标边界的分割效果不佳。相比于Unet,本文提出的基于条件能量对抗网络的分割方法可以完整分割出输入图像中所包含的肿瘤,并且分割边界接近真实标注。为进一步展现本文方法的分割性能,表2中列出了其他方法在3DIRCADb上取得的分割指标。

图7 3DIRCADb数据集上的分割结果
4 结束语
肝脏和肝肿瘤分割在肝癌临床诊断中具有重要意义。为解决肝脏和肝肿瘤分割问题,本文提出了条件能量对抗网络结构,将Unet嵌入到EBGAN的架构中作为模型的生成器,用于学习像素特征得到每个像素的类别信息;基于能量理论构建判别器并将原始图像作为条件约束输入到判别器中,通过学习像素类别之间的高阶一致性,提升小体积目标和目标边界的分割精度,并通过实验验证了本文提出方法的有效性。

表2 本文方法与其他方法在3DIRCADb数据集上的对比%
