花生根际土壤细菌群落多样性对盐胁迫的响应
2021-06-09戴良香张冠初史晓龙秦斐斐张智猛
戴良香 徐 扬 张冠初 史晓龙 秦斐斐 丁 红,* 张智猛,*
花生根际土壤细菌群落多样性对盐胁迫的响应
戴良香1,**徐 扬1,**张冠初1史晓龙2秦斐斐1丁 红1,*张智猛1,*
1山东省花生研究所, 山东青岛 266100;2沈阳农业大学农学院, 辽宁沈阳 110866
为明确盐胁迫条件下花生根际土壤细菌群落结构的变化, 采用盆栽试验设置不同盐胁迫强度处理, 以开花下针期和成熟期花生根际土壤为研究对象, 提取其总DNA并构建细菌16S rRNA基因文库, 利用高通量测序技术进行测序, 并对测序结果进行生物信息学分析。结果显示, 各处理样本根际土壤优势菌门均为变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、Patescibacteria、酸杆菌门(Acidobacteria)、绿弯菌门(Chloroflexi)。优势菌目分别为(Saccharimonadales)、β-变形菌目(Betaproteobacteria)、鞘脂单胞菌目(sphingomonadales)、芽单胞菌目(Gemmatimonadales)和根瘤菌目(Rhizobiales)。盐胁迫提高了变形菌门(Proteobacteria)的相对丰度, 但降低了放线菌门(Actinobacteria)的相对丰度, 且均随盐胁迫强度提高其升降幅度增大。盐胁迫下基施钙肥使β-变形菌目(Betaproteobacteria)、芽单胞菌目(Gemmatimonadales)和鞘脂单胞菌目(Sphingomonadales)相对丰度显著升高, 并均受盐胁迫强度、生育时期和外源施钙肥的影响。聚类分析结果表明, 较高浓度盐胁迫处理样本的优势菌属组成相似而聚为一组, 非盐胁迫处理样本属水平丰度依生育时期相同而相近, 各聚为一组。盐胁迫强度、生育时期对花生根际土壤微生物菌群类型影响较大, 基施钙肥影响较小。土壤微生物功能预测分析显示, 高盐胁迫可明显降低次生代谢产物、聚糖的生物合成与代谢, 以及氨基酸和脂肪酸代谢等功能基因在根际土壤中的富集。花生旺盛生长期、低盐胁迫和基施钙肥处理使得功能基因丰度大幅提高, 可能对花生生长及胁迫应答具有重要功能。根际土壤微生物菌群多样性的研究将为通过改良土壤微生物环境来提高植物胁迫耐受性提供重要途径。
花生; 盐胁迫; 根际; 土壤微生物群落; 16S rRNA基因
土地盐渍化已经成为世界性的资源环境问题之一, 是制约农业发展的巨大环境压力, 每年造成数十亿农业经济损失[1]。我国土壤盐碱化现象日趋严重, 盐碱地面积约9.91×107hm2, 主要分布于气候比较干旱的山东、河北、河南、新疆等省区, 大部分处于待开发状态, 是制约该区农业可持续发展的首要因素[2]。花生是我国重要的油料作物和经济作物, 具有耐瘠、固氮、培肥地力和中度耐盐碱等特性, 可作为盐碱土区种植业结构调整中较为适宜的替代作物; 同时, 限制盐碱地花生高产的关键是缺苗、早衰和养分失衡。因此, 扩大盐碱地花生种植面积, 提高产量, 加强花生抗盐生理机制研究显得更为迫切。盐碱土壤性质的改善主要通过减少土壤中盐离子含量、改善土壤结构、调节土壤酸碱度、增加土壤中可被植物利用营养元素含量等途径, 提高土壤肥力, 通常以石膏、磷石膏、硅酸钙和工业生产中含钙废弃物等为主要原料, 或配施有机物料为改良剂进行盐碱地改良。微生物菌剂与磷石膏联合施用改良盐碱土能够增强盐碱土的改良效果[3]。目前国内外改良盐碱土技术主要包括物理改良、水利工程改良、化学改良和生物改良等, 但费用高、见效慢、推广难, 严重制约了盐碱土区种植业结构的抗风险能力和生态环境的可持续发展。
植物根际微生境是土壤中活性最强的微生境, 也是植物获取养分的主要区域。在此微域里, 植物—微生物—土壤—环境间相互作用, 共同维持着根际微生态系统的平衡, 影响作物生产[4]。研究表明, 盐胁迫使土壤微生物群落结构和组成发生变化, 植物根际微生态区系失衡, 可能涉及到某些功能菌群、有益菌群和有害菌群比例的平衡失调[5]。根际微生物类型和其群落组成受盐胁迫及程度的影响, 其数量和群落组成与环境和植物种类有关[6], 施用钙肥可改善盐碱土壤结构、降低Na+含量、调节pH[3]。关于盐碱土壤微生物群落的研究主要集中在不同植物群落土壤微生物的时空动态和区系特征[7-8]、微生物量与土壤酶[9]、不同改良措施及植被类型对盐碱土壤微生物的影响效果等[9-12], 有关不同盐胁迫强度下配施钙肥对花生根际微生物多样性、种群结构及其生态关系的研究鲜见报道, 为此, 本试验以不同盐胁迫强度下配施钙肥处理的花生根际土壤为研究对象, 利用高通量测序技术对土壤中细菌种类及多样性进行系统分析, 探讨盐胁迫下不同生育时期花生根际土壤细菌群落组成和多样性的变化, 有助于认识盐碱地花生根际土壤微生物群落功能调节并发掘新的功能类群, 对改良盐碱土壤和开发利用盐碱土区资源、提高盐碱土区农业种植结构抗风险能力及盐碱地花生高产优质栽培均具重要意义。
1 材料与方法
1.1 试验材料
采用山东省花生研究所莱西试验站耕层土壤(0~30 cm)作为供试土壤, 含土壤有机质含量13.23 g kg-1、全磷(P2O5) 0.84 g kg-1、全钾(K2O) 11.53 g kg-1、全氮1.70 g kg-1、水解氮(N) 92.1 mg kg-1、速效磷(P2O5) 11.7 mg kg-1、速效钾(K2O) 112.2 mg kg-1, 土壤pH 7.1, 土壤交换性Na+0.82 cmol kg-1, Cl–28.5 mg kg-1, 土壤含水量9.72%。选择花生品种花育25号(HY25)。
1.2 试验设计
试验于山东省花生研究所试验站防雨棚中进行,土壤风干、过筛(1 cm)后装入同批次、同规格塑料盆中(底部直径为36 cm, 高为26 cm), 每盆装土量为18 kg。设置0 (Y0)、1.5 (Y1)、3.0 g kg-1(Y2) 3个盐胁迫梯度, 采用分析纯NaCl重量法控制盐胁迫梯度。各处理均配合基施钙肥(分析纯CaO) 150 kg hm-2(C, 150 kg hm-2CaO), 以过筛后不施钙肥的土壤为对照, 随机排列, 6次重复。播种前将供试肥料按N∶P2O5∶K2O = 1.0∶1.5∶1.5 (15.0、22.5、22.5 kg hm-2)比例连同CaO以基肥形式均匀施入盆中。随后各处理均浇入2 L清水沉实, 待土壤墒情适宜时再次混匀每盆中的土壤, 以使土壤含盐量和施入的钙肥均匀分布。选取饱满均匀的种子, 每盆播6粒, 播深均为3 cm, 留4株长势一致的幼苗。
分别于开花下针期(播后75 d)和收获期(播后125 d)以处理和重复为单位采集花生植株根际土壤样本。根际土壤样本的采集以处理为单元进行, 采用多点混合样本采集方法, 以盆为单位分别小心地从盆中取出植株, 去除根际附着不紧密的土壤, 然后用无菌刷收集附着紧密的土壤, 混合每盆中植株根际土壤, 每3重复根际土壤样品混合为1个生物学样本重复, 每处理均获取2个生物学重复, 封入无菌袋, 置于冰盒带回实验室, 保存于-80℃冰箱备用。于收获期, 各处理收获实测计产, 室内考察花生出米率、百果质量、百仁质量和荚果产量。
开花下针期各处理土壤样本分别表示为CK、CY0、CY1、CY2, 收获期各处理土壤样本分别表示为HCK、HCY0、HCY1、HCY2。由北京诺赛基因公司进行相关测试。
1.3 土壤DNA提取
收集所得土壤样品, 利用OMEGA土壤总DNA提取试剂(OMEGA soil DNA kit)盒提取DNA。采用1.5%琼脂糖凝胶电泳和Nanodrop 2000分光光度计检测DNA的纯度和浓度。
1.4 16srRNA文库构建及高通量测序
以提取的土壤样本DNA为模板, 利用引物340F: 5¢-CCTACGGGNBGCASCAG-3¢以及805R: 5¢-GACTACNVGGGTATCTAATCC-3¢, 对土壤细菌16s rRNA基因V3~V4区进行PCR扩增。扩增程序如下: 95℃预变性3 min; 95℃30 s, 50℃30 s, 72℃60 s, 30个循环; 72℃7 min。使用1.5%浓度的琼脂糖凝胶电泳检测PCR产物; 根据PCR产物浓度进行等浓度混样, 充分混匀后, 使用0.5×TBE浓度1.5%的琼脂糖胶电泳纯化PCR产物, 割胶回收目标条带。产物纯化试剂盒使用QIAGEN公司的MinElute胶回收试剂盒。最后使用HiSeq2500进行250PE测序。
1.5 生物信息学分析
对花生根际土壤微生物的多样性和丰富度进行样本内物种多样性即Alpha多样性和样本间Beta多样性分析, 包括绘制稀释性曲线(Rarefaction curve)和分析Alpha多样性指数(包括Chao、Shannon、ACE、Simpson、coverage和sobs等指数)。同时进行群落结构统计分析和基于OTUs的物种组成聚类分析(heatmap分析), 最后, 进行反映样本间的多样性距离关系和生物群落间分化程度的Beta多样性分析(PCA统计分析), 挖掘样本间物种组成差异和生物功能分析。
2 结果与分析
2.1 对荚果性状和产量的影响
由表1可知, 盐胁迫处理明显影响花生出米率、百果质量、百仁质量和荚果产量, 并随盐胁迫程度的提高作用显著, 施用钙肥可显著提高无盐胁迫处理下的百果质量和荚果产量, 对出米率和百仁质量有促进作用但不显著。盐胁迫处理下百仁质量较CK和CY0分别降低12.94%、20.44%和38.70%、43.98%, 较高盐胁迫处理的荚果产量仅为CK处理的1/2, CY0处理下产量是CY2处理2.5倍。表明盐胁迫严重抑制花生籽仁发育和产量形成。

表1 施钙对盐胁迫下花生产量及构成因素的影响
CK、CY0、CY1、CY2分别表示开花下针期对照和各盐胁迫梯度下施钙肥各处理根际样本; HCK、HCY0、HCY1、HCY2分别表示收获期相应盐胁迫下各处理土壤样本。同列不同小写字母表示处理间差异显著(< 0.05)。
CK, CY0, CY1, and CY2 represent the rhizosphere samples of each treatment with calcium fertilizer under each salt stress gradient at the needle stage at the flowering and needle stage, respectively; HCK, HCY0, HCY1, and HCY2 represent the soil samples of each treatment under the corresponding salt stress at the harvest stage, respectively. Values within a column followed by different lowercase letters mean significant differences among the treatments at the 0.05 probability level.
2.2 花生根际微生物群落测序数据分析
供试的16个根际土壤样本共检测出细菌30个门、73个纲、204个目、351个科、650个属和1279个种。获得有效序列1,187,782.5条, 其中HCY2样本有效序列条数最小为127,455.5条, CY0样本最大为174,409.0条, 多数样本序列平均长度在445.0~452.0 bp之间, 平均长度482.0 bp (表2)。通过维恩图分析, 各处理根际土壤样本共有2446个OTUs, 存在于CY1、CK、CY2中的OTUs数分别为29、25和23个, 而HCK、HCY0和HCY2处理样本中均不足10个, 表明各处理样本微生物组成相似, 盐胁迫强度、外源施钙肥和生育时期对根际土壤细菌群落结构均无显著影响(图1)。
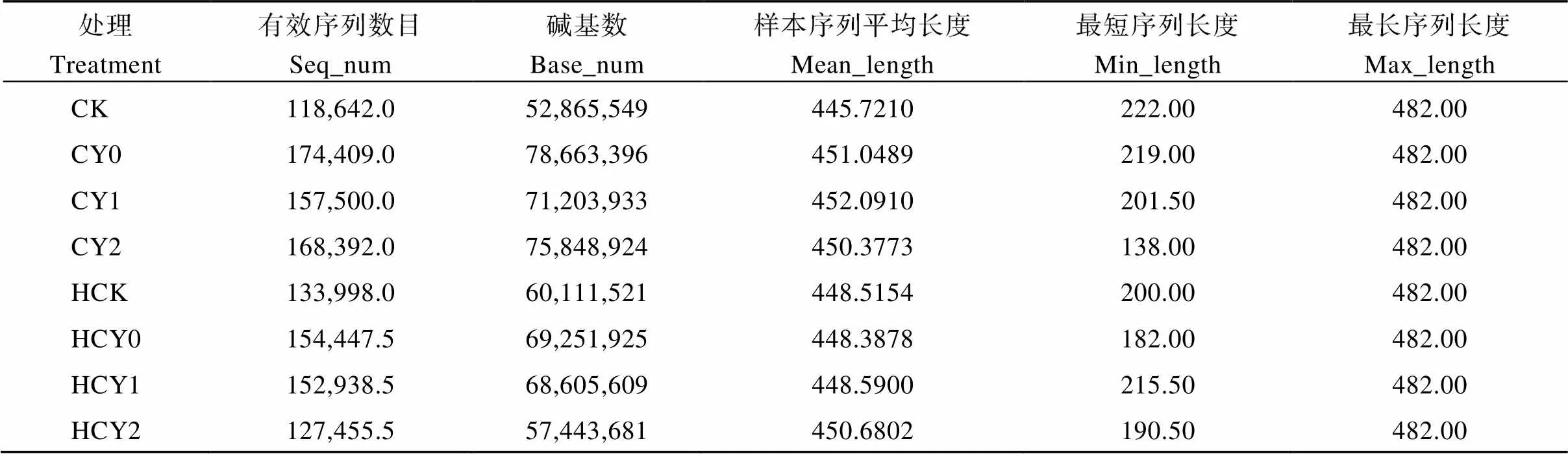
表2 花生根际微生物群落测序质量
处理同表1。Treatments are the same as those given in Table 1.
处理同表1。Treatments are the same as those given in Table 1.
2.3 Alpha多样性分析
2.3.1 稀释曲线 由图2可知, 随测序数据量的增加, 物种丰富度呈现前期迅速增加的趋势, 各处理样本在测序量达到30,000条以上时, 稀释曲线趋近平缓, 各样本微生物群落测序数据达到饱和, 测序量能够覆盖花生根际微生物组群落的绝大部分物种。各处理样本重复间取其均值进行比较发现, 对照、盐胁迫强度和生育时期及外源施用钙肥各处理OTUs数量基本一致, 相互间均无显著差异。
2.3.2 多样性指数 由表3可知, 供试样本的coverage测序深度指数均在98.90%以上, 样本微生物群落中OTUs的chao和ACE菌群丰富度指数分别在3522.40~3762.58和3448.57~3685.00之间, Shannon多样性指数为6.57~7.11, Simpson和sobs指数分别在0.001,946~0.005,682和3113.00~3428.50之间。ACE、chao和sobs指数均以CK处理最小, ACE、chao丰富度指数以CY2处理最大, 而Shannon和sobs指数则均以CY0最高。可见, 配施钙肥可提高盐胁迫下花生根际微生物群落丰富度和多样性, 且对开花下针期的影响作用优于收获期, 无盐胁迫下施用钙肥处理根际土壤细菌多样性更为丰富。
2.4 物种群落组成分析
2.4.1 全样本门水平菌落结构分析 所有样本细菌群落组成的30个门中, 其Top10 (others合并< 0.025)相对丰度之和达93.40%。优势菌门均为变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、Patescibacteria、酸杆菌门(Acidobacteria)、绿弯菌门(Chloroflexi)、疣微菌门(Verrucomicrobia)、芽单胞菌门(Gemmatimonadetes)和拟杆菌门(Bacteroidetes)等8种, 其相对丰度均在5.17%以上, 其中以变形菌门(Proteobacteria)平均相对丰度最高(31.70%), 放线菌门(Actinobacteria)次之(16.12%), 绿弯菌门(Chloroflexi)和酸杆菌门(Acidobacteria)分别为8.23%、8.65%。芽单胞菌门(Gemmatimonadetes)仅为变形菌门(Proteobacteria)的16.32% (图3-A)。盐胁迫提高根际变形菌门(Proteobacteria)的相对丰度, 降低放线菌门(Actinobacteria)丰度且均随盐胁迫强度提高其升降幅度增大, 尤以开花下针期作用明显, 并均受外源配施钙肥的正向调节, 开花下针期和收获期不施钙肥和施钙肥高盐胁迫处理的变形菌门(Proteobacteria)相对丰度较其对照分别提高27.00%、10.46%和32.44%%、50.09%, 而放线菌门(Actinobacteria)丰度较其对照分别降低55.58%、48.83%和40.90%、28.87%。较高盐胁迫处理的拟杆菌门丰度明显升高, 并以收获期较为明显。
处理同表1。Treatments are the same as those given in Table 1.
处理同表1。Treatments are the same as those given in Table 1.
对各处理根际样本进行优势门(Top10) (图3-B)组间差异显著性检验分析表明, 各处理间均呈显著或极显著差异。生育时期、施用钙肥和盐胁迫各因子均使得花生根际微生物群落分异明显。
2.4.2 全样本目水平菌落结构分析 所有样本共包含204个菌目, 其中Top16 (others合并<0.03)相对丰度之和达60.40%, 相对丰度在5.0%以上的优势菌目分别为Saccharimonadales (7.94%)、β-变形菌目(Betaproteobacteria, 7.42%)、鞘脂单胞菌目(Sphingomonadales, 6.41%)、芽单胞菌目(Gemmatimonadales, 5.08%)和根瘤菌目(Rhizobiales, 5.00%)。除优势菌目β-变形菌目(Betaproteobacteria)和外, 其他优势菌目开花下针期样品的相对丰度均明显低于收获期。盐胁迫下基施钙肥使β-变形菌目(Betaproteobacteria)、芽单胞菌目(Gemmatimonadales)和鞘脂单胞菌目(Sphingomonadales)丰度显著升高, 而Saccharimonadales、微球菌目()菌目相对丰度显著降低, 并随盐胁迫强度升高其相对丰度呈降低趋势, 且收获期降幅较大(图4-A)。
处理同表1。*、**、***分别表示处理间达0.05、0.01和0.001显著性水平。
Treatments are the same as those given in Table 1. *, **, and *** indicate significant differencesatthe 0.05, 0.01, and 0.001 levels, respectively.
处理同表1。*、**、***分别表示处理间达0.05、0.01和0.001显著性水平。
Treatments are the same as those given in Table 1. *, **, and *** indicatesignificant differences at the 0.05, 0.01, and 0.001levels, respectively.
各样本细菌群落目水平物种组成heatmap图显示, 不同处理根际样本细菌目水平丰度高低有异, CY1处理的β-变形菌目、土圈菌目(Pedosphaerales)和unclassified_k__norank_d__Bacteria菌目丰度均显著升高, CY2、HCY2两处理均使β-变形菌目和鞘脂单胞菌目(Sphingomonadales)丰度明显升高, 而HCY0和HCK则使Saccharimonadales菌目丰度升幅极为明显。聚类树分析结果表明, 同一生育时期根际样本优势菌目组成较为相似聚为一类, 盐胁迫处理与非盐胁迫处理根际样本中的优势菌目组成存在差异各聚为一类(图4-B)。对各处理根际样本进行优势目(Top10) (图4-C)组间差异显著性检验分析表明, 除根瘤菌目(Rhizobiales)外各处理间均呈显著或极显著差异。
2.4.3 全样本属水平菌落结构分析 各样本共包含650个菌属, 其中、鞘氨醇单胞菌属)、、、芽单胞杆菌属()、等6种为优势菌属。各处理样本中的和两优势菌属丰度随盐胁迫强度升高而升高,、、和非优势菌属菌属则均随胁迫强度升高而降低,和两生育时期分别较其相应对照降低72.27%、67.08%和81.20%、51.75%, 非优势菌属罗河杆菌属()和丰祐菌属()相对丰度则均随盐胁迫强度增加显著升高, 分别是相应对照的1.31、4.23倍和3.02、2.55倍。盐胁迫显著影响不同生育时期根际土壤细菌群落组成, 尤以收获期优势菌群丰度降低显著, 而非优势菌群丰度往往升幅明显(图5-A)。
各样本细菌群落属水平物种组成heatmap图和聚类树分析结果表明, 较高浓度盐胁迫处理样本优势菌属组成相似聚为一组, 非盐胁迫处理样本属丰度依生育时期相同而相近, 各聚为一组, 1.5 g kg-1较低盐胁迫处理根际土壤细菌优势菌属较为丰富(图5-B)。
2.5 样本间Beta多样性分析
通过PCA分析发现, 各处理样本分布于不同象限, 其细菌群落组成具有明显差异, PC1和PC2对结果的解释度分别为25.88%和15.87%, 二者分别由生育时期因子和盐胁迫因子贡献。HCK和HCY0与第1、第2主成分均呈正相关关系, CY2和CY1则与第1、第2主成分均呈负相关关系, HCY1和HCY2与第1主成分为正相关关系而与第2主成分为负相关关系, 而CY0和CK则与之相反(图6-A)。说明盐胁迫、生育时期和施钙肥与否对根际土壤微生物菌群类型影响较大。层级聚类分析表明, 依生育时期聚为两大类的同时, 又以盐胁迫及其强度各聚一簇, 开花下针期受盐胁迫强度影响较小, 成熟期根际土壤微生物菌群类型影响较大(图6-B)。对种水平上菌群分型分析结果亦表明, 盐胁迫强度、生育时期对花生根际土壤微生物菌群类型影响较大, 基施钙肥影响较小(图6-C)。
处理同表1。Treatments are the same as those given in Table 1.
处理同表1。Treatments are the same as those given in Table 1.
2.6 16S功能预测分析
通过KEGG代谢途径的预测差异分析发现, CK、HCK、HCY2处理样本中微生物群落的功能丰度均明显降低, 尤以次生代谢产物的代谢与合成(丙酸酯代谢、2-氧代羧酸代谢、乙醛酸和二羧酸酯代谢、氨酰基-tRNA的生物合成)、聚糖的生物合成与代谢(糖酵解/糖异生、氨基糖和核苷酸糖代谢)、氨基酸代谢(半胱氨酸和蛋氨酸的代谢、甘氨酸, 丝氨酸和苏氨酸的代谢)、脂肪酸代谢等功能基因的丰度降低显著。CY0、CY1、CY2、HCY0和HCY1样本中各功能基因丰度均相对升高, 尤以CY1和HCY0升高明显(图7)。表明无盐胁迫和全生育期高强度盐胁迫可明显降低次生代谢产物的代谢与合成、聚糖的生物合成与代谢、氨基酸代谢和脂肪酸代谢等功能基因在根际土壤中的富集, 花生旺盛生长期、低盐胁迫和基施钙肥处理使得功能基因丰度大幅提高。
处理同表1。Treatments are the same as those given in Table 1.
3 讨论
根际微生物是土壤—根系间养分转化和转运的调节器, 土壤微生物群落多样性反映了群落总体的动态变化。植被对土壤微生物群落的数量具有显著正向的影响, 盐胁迫下, 除土壤微生物生长受到抑制, 植物本身的生理机能也迅速遭到破坏, 根际土壤细菌群落的丰富度显著降低[13-14], 随盐胁迫强度增加微生物种群数量、群落结构和功能稳定性明显降低[15-17]。耐盐植物根际土壤微生物量随种植时间的延长而增加并显著高于非根际土壤[18-19]。但不同盐碱地类型花生根际土壤微生物种类、优势种群数量和群落功能多样性存在差异, 含盐量较高土壤更为丰富。黄河三角洲滨海盐碱土和不同含盐量的盐碱土花生根际土壤微生物优势菌门均以变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、绿弯菌门(Chloroflexi)和酸杆菌门(Acidobacteria)等4种菌群为优势菌群[20]。盐碱土花生根际芽单胞菌门(Gemmatimonadetes)和拟杆菌门(Bacteroidetes)的丰度显著高于非盐碱土壤约2倍[21]。本试验条件下, 盐胁迫强度和花生生长期不影响花生根际细菌群落多样性和丰富度, 各处理微生物种群数量和优势菌群无差异, 但对细菌群落结构组成有影响。各处理优势菌门均为变形菌门、放线菌门、Patescibacteria、酸杆菌门、绿弯菌门、疣微菌门(Verrucomicrobia)、芽单胞菌门和拟杆菌门。优势菌目为Saccharimonadales、β-变形菌目、鞘脂单胞菌目、芽单胞菌目和根瘤菌目。盐胁迫使根际微生物优势菌群数量明显增加, 旺盛生长的开花下针期较为明显。
耐盐植物根际环境中细菌群落组成与耐盐性有关, 盐生植物根际细菌群落受土壤盐分的影响较小[22]。盐胁迫显著提高可加快碳氮循环的蓝藻菌门的含量, 而益于花生抵御逆境胁迫[20]。中度耐盐植物根际环境中耐盐性较高的变形菌门和厚壁菌门丰度较高, 轻度耐盐植物根际环境中酸杆菌门和芽单胞菌门较丰富, 说明酸杆菌门和芽单胞菌门不具耐盐性或更易受高盐影响。本试验条件下, 花生根际土壤优势菌群除变形菌门和酸杆菌门外, 还包括放线菌门、绿弯菌门、拟杆菌门和疣微菌门。酸杆菌门在土壤生态过程中起着重要作用, 变形菌门的增多可更有效固定氮源。放线菌门具有共生固氮和解磷作用[23-26], 绿弯菌存在于根际土壤样本中, 通过光合作用产生能量但不能固氮, 但具有较好的生物解磷作用[27-28]。拟杆菌门具有非常强的营养物质代谢能力, 如复杂有机物、蛋白质和脂类等[29]。解磷菌悬液可显著增加盐碱土壤酶活性和土壤微生物数量, 促进与土壤营养元素循环相关的菌属酸杆菌(Acidobacteria)、绿弯菌(Chloroflexi)和浮霉菌门(Planctomycetes)等微生物群落富集, 全面改善盐碱土性质, 促进植株生长[3]。本试验结果表明, 盐胁迫显著影响不同生育时期根际土壤细菌群落组成, 尤以收获期优势菌属丰度降低显著, 而非优势菌属丰度往往升幅明显。开花下针期β-变形菌目(Betaproteobacteria)和芽单胞菌目(Gemmatimonadales)明显高于收获期。表明花生不同生长期和外源施钙对盐胁迫下花生根际微生物群落具有适应性和调节功能, 根际土壤较高的变形菌门、放线菌门、拟杆菌门和酸杆菌门可能有助于维持根系养分吸收和微环境的平衡, 以改善土壤环境, 增强抵御盐胁迫的能力, 是盐碱土区生物改良盐胁迫的宝贵资源。同时, 盐胁迫可大幅提高变形菌门、拟杆菌门的相对丰度, 且配施钙肥的调节作用明显增强并保持花生全生育期, 从而改善了花生根际氮磷及营养物质代谢的动态平衡而增强了对盐胁迫的抵御作用。
土壤类型和花生根系分布密集程度对土壤微生物菌群类型影响较大, 盐碱土壤花生根际微生物菌群类型依据土壤含盐量高低和根系分布深度不同而聚为不同簇, 非盐碱土壤、盐碱土壤根系分布不同密集程度层0~20 cm、20~40 cm各归为一类。非根际土壤与根际微生物群落分离明显, 非根际土壤群落之间, 以及根际土壤群落之间各具有较高的相似性[20,30-31]。本试验条件下层级聚类分析表明, 生育时期、盐胁迫及强度各聚为一簇, 花生根际土壤微生物菌群类型受盐胁迫强度和生育时期影响较大, 基施钙肥影响较小, 并以花生生育末期根际微生物菌群类型受影响较大。
16S功能预测显示, 盐胁迫影响花生根际微生物群落的功能丰度谱, 尤以某些代谢与合成相关功能如次生代谢产物的代谢与合成、聚糖生物合成与代谢、氨基酸代谢等功能基因丰度变化明显。在非盐胁迫和全生育期高强度盐胁迫处理下, 这些次生代谢产物的代谢与合成、聚糖生物合成与代谢、氨基酸代谢等功能基因丰度显著降低, 而在花生旺盛生长期、低盐胁迫和基施钙肥处理均使得这些功能基因丰度大幅提高。说明花生旺盛生长时期、较低的盐胁迫强度和外源钙肥均可提高花生根际微生物与代谢与合成相关功能基因丰度, 以抵御盐胁迫逆境。然而, 花生根际微生物的富集与代谢和合成相关的功能基因不足以抵抗全生育期内高强度盐胁迫逆境, 致使花生产量显著降低。
4 结论
花生根际土壤微生物优势菌群组成结构不受盐胁迫和生育时期影响, 但其相对丰度与盐胁迫强度有关, 随盐胁迫强度的提高, 变形菌门(Proteobacteria)的相对丰度和放线菌门(Actinobacteria)丰度升降幅度增大。盐胁迫强度、生育时期对花生根际土壤微生物菌群类型影响程度大于基施钙肥处理。花生旺盛生长时期和基施钙肥使得盐胁迫下花生根际微生物菌群功能基因丰度大幅提高, 高盐胁迫明显降低次生代谢产物、聚糖的生物合成与代谢, 以及氨基酸和脂肪酸代谢等功能基因在根际土壤中的富集。通过改良根际土壤微生物环境提高植物耐盐胁迫能力是生物改良盐碱胁迫的可行途径。
[1] Jamil A, Riaz S, Ashraf M, Foolad M R. Gene expression profiling of plants under salts tress., 2011, 30: 435–458.
[2] 贾敬敦, 张富. 依靠科技创新推进我国盐碱地资源可持续利用. 中国农业科技导报, 2014, 16(5): 1–7. Jia J D, Zhang F. Sustainable utilization of saline-alkali land resources through scientific and technological innovation in China., 2014, 16(5): 1–7 (in Chinese with English abstract).
[3] 姜焕焕. 耐盐碱解磷菌与磷石膏联用改良盐碱土的效果与机制. 哈尔滨工业大学博士学位论文, 黑龙江哈尔滨, 2019. Jiang H H. Saline-alkali Soil Remediation by the Combined Application of Halotolerant Phosphate Solubilizing Microorganism and Rock Phosphate. PhD Dissertation of Harbin Institute of Technology, Harbin, Heilongjiang, China, 2019 (in Chinese with English abstract).
[4] 张旭龙, 马淼, 吴振振, 张志政, 高睿, 石灵玉. 不同油葵品种对盐碱地根际土壤酶活性及微生物群落功能多样性的影响. 生态学报, 2017, 37: 1659–1666. Zhang X L, Ma M, Wu Z Z, Zhang Z Z, Gao R, Shi L Y. Effects of Helianthus annuus varieties on rhizosphere soil enzyme activities and microbial community functional diversity of saline-alkali land in Xinjiang., 2017, 37: 1659–1666 (in Chinese with English abstract).
[5] Zhang Q, Yu X. Allelopathy in replant problem in forest soil., 2001, 8: 51–64.
[6] Kielak A, Pijl A S, Veen J A, Kowalchuk G A. Differences in vegetation composition and plant species identity lead to only minor changes in soil-borne microbial communities in a former arable field., 2008, 63: 372–382.
[7] Nelson E B. Microbial dynamics and interactions in the spermosphere., 2004, 42: 271–309.
[8] Buyer J S, Roberts D P, Russek-Cohen E. Microbial community structure and function in the spermosphere as affected by soil and seed type., 1999, 45: 138–144.
[9] 叶淑红, 王艳, 万惠萍, 吉云秀, 林学政, 丁德文. 辽东湾湿地微生物量与土壤酶的研究. 土壤通报, 2006, 37: 897–900. Ye S H, Wang Y, Wan H P, Ji Y X, Lin X Z, Ding D W. Wetland microbial biomass and soil enzyme activities in Liaodong bay., 2006, 37: 897–900 (in Chinese with English abstract).
[10] 李凤霞, 王学琴, 郭永忠, 许兴, 杨建国, 季艳清. 宁夏不同类型盐渍化土壤微生物区系及多样性. 水土保持学报, 2011, 25(5): 107–111. Li F X, Wang X Q, Guo Y Z, Xu X, Yang J G, Ji Y Q. Microbial flora and diversity in different types of saline-alkali soilin Ningxia., 2011, 25(5): 107–111 (in Chinese with English abstract).
[11] 田家怡, 李甲亮, 陈印平, 李建庆, 于祥. 黄河三角洲外来入侵物种米草对底泥微生物群落的影响. 海洋湖沼通报, 2009, (4): 157–162. Tian J Y, Li J L, Chen Y P, Li J Q, Yu X. Effect on the sludge microbial communities by alien invasive species Spartina spp. in yellow river delta., 2009, (4): 157–162 (in Chinese with English abstract).
[12] 王震宇, 辛远征, 李锋民, 高冬梅. 黄河三角洲退化湿地微生物特性的研究. 中国海洋大学学报, 2009, 39: 1005–1012. Wang Z Y, Xin Y Z, Li F M, Gao D M. Microbial community characteristics in a degraded wetland of the Yellow River Delta., 2009, 39: 1005–1012 (in Chinese with English abstract).
[13] 王文铜. 花生根际土壤细菌群落结构分析. 山东农业大学博士学位论文, 山东泰安, 2012. Wang W T. Analysis of Bacterial Community Structure in Rhizosphere Soil of Peanut. PhD Dissertation of Shandong Agricultural University, Tai’an, Shandong, China, 2012 (in Chinese with English abstract).
[14] 吴凤芝, 包静, 刘淑芹. 盐胁迫对黄瓜根际土壤细菌群落结构和生长发育的影响. 园艺学报, 2010, 37: 741–748. Wu F Z, Bao J, Liu S Q. Effects of salt stress on rhizospheric soil bacterial community structure and cucumber yield., 2010, 37: 741–748 (in Chinese with English abstract).
[15] 朱泓, 王小敏, 黄涛, 吴文龙, 李维林. NaCl胁迫对滨梅根际细菌群落多样性及优势菌群的影响. 南京林业大学学报(自然科学版), 2017, 41(4): 49–54. Zhu H, Wang X M, Huang T, Wu W L, Li W L. Effect of NaCl stress on bacterial community diversity and core microbiome in rhizosphere and bulk soil of beach plum (Marshall).(Nat Sci Edn), 2017, 41(4): 49–54 (in Chinese with English abstract).
[16] Baumann K, Dignac M F, Rumpel C, Bardoux G, Sarr A, Steffens M, Maron P A. Soil microbial diversity affects soil organic matter decomposition in a silty grass land soil., 2013, 114: 201–212.
[17] Philippot L, Spor A, Hénaul T C, Bru D, Bizouard F, Jones C M, Sarr A, Maron P. Loss in microbial diversity affects nitrogen cycling in soil., 2013, 7: 1609–1619.
[18] 马晓梅, 尹林克, 陈理. 塔里木河干流胡杨和柽柳根际土壤微生物及其垂直分布. 干旱区研究, 2008, 25(2): 183–189. Ma X M, Yin L K, Chen L. Study on vertical distribution of microorganiss in rhizosphere ofandsp. in the lower reaches of the Tarim river, Xinjiang., 2008, 25(2): 183–189 (in Chinese with English abstract).
[19] 林学政, 陈靠山, 何培青, 沈继红, 黄晓航. 种植盐地碱蓬改良滨海盐渍土对土壤微生物区系的影响. 生态学报, 2006, 26: 801–807. Lin X Z, Chen K S, He P Q, Shen J H, Huang X H. The effects of Suaeda salsa L. planting on the soil microflora in coastal saline soil., 2006, 26: 801–807 (in Chinese with English abstract).
[20] 张智猛, 慈敦伟, 张冠初, 丁红, 杨吉顺, 戴良香, 张岱. 山东地区盐碱土花生种子际土壤微生物群落结构的研究. 微生物学报, 2017, 57: 582–596. Zhang Z M, Ci D W, Zhang G C, Ding H, Yang J S, Dai L X, Zhang D. Diversity of microbial community structure in the spermosphere of saline-alkali soil in Shandong area., 2017, 57: 582–596 (in Chinese with English abstract)
[21] 戴良香, 康涛, 慈敦伟, 丁红, 徐扬, 张智猛, 张岱, 李文金. 黄河三角洲盐碱地花生根层土壤菌群结构多样性. 生态学报, 2019, 39: 7169–7178. Dai L X, Kang T, Ci D W, Ding H, Xu Y, Zhang Z M, Zhang D, Li W J. Comparison of the microbial community in the rhizosphere of peanuts between salinealkali and non-saline soil at different soil depths and intercropping cultivation in the Yellow River Delta., 2019, 39: 7169–7178.
[22] Yongge Y, Brune C, Kleunen M, Li J M, Jin Z X. Salinity-induced changes in the rhizosphere microbiome improve salt tolerance of Hibiscus hamabo., 2019, 443: 525–537.
[23] 赵娇, 谢慧君, 张建. 黄河三角洲盐碱土根际微环境的微生物多样性及理化性质分析. 环境科学, 2020, 41: 1449–1455. Zhao J, Xie H J, Zhang J. Microbial diversity and physicochemical properties of rhizosphere microenvironment in saline-alkali soils of the Yellow River Delta., 2020, 41: 1449–1455 (in Chinese with English abstract).
[24] 郑微. 天津不同盐度土壤微生物群落结构特征及其适应机理研究. 天津师范大学博士学位论文, 天津, 2018. Zheng W. Study on the Structural Characteristics and Adaptation Mechanism of Microbial Community in Different Salinity Soils in Tianjin. PhD Dissertation of Tianjin Normal University, Tianjin, China, 2018 (in Chinese with English abstract).
[25] 涂勇, 杨文钰, 刘卫国, 雍太文, 江连强, 王小春. 大豆与烤烟不同套作年限对根际土壤微生物数量的影响. 作物学报, 2015, 41: 733–742. Tu Y, Yang W Y, Liu W G, Yong T W, Jiang L Q, Wang X C. Effects of relay strip intercropping years between flue-cured tobacco and soybean on rhizospheric microbes quantities., 2015, 41: 733–742 (in Chinese with English abstract).
[26] Cui J, Li Y, Wang C, Kim K S, Wang T, Liu S. Characteristics of the rhizosphere bacterial community across different cultivation years in saline-alkaline paddy soils of Songnen Plain of China., 2018, 64: 925–936.
[27] Yu H, Si P, Shao W, Qiao X, Yang X, Gao D, Wang Z. Response of enzyme activities and microbial communities to soil amendment with sugar alcohols., 2016, 5: 604–615.
[28] Kragelund C, Levantesi C, Borger A. Identity abundance and ecophysiology of filamentous Chloroflexi species present in activated sludge treatment plants., 2007, 59: 671–682.
[29] Hill V R, Kahler A M, Jothikumar N, Johnson T B, Hahn D, Cromeans T L. Multistate evaluation of an ultrafiltration-based procedure for simultaneous recovery of enteric microbes in 100-liter tap water sample., 2007, 73: 4218–4225.
[30] 葛应兰, 孙廷. 马铃薯根际与非根际土壤微生物群落结构及多样性特征. 生态环境学报, 2020, 29: 141–148. Ge Y L, Sun T. Soil microbial community structure and diversity of potato in rhizosphere and non-rhizosphere soil., 2020, 29: 141–148 (in Chinese with English abstract).
[31] 覃潇敏, 郑毅, 汤利, 龙光强. 玉米与马铃薯间作对根际微生物群落结构和多样性的影响. 作物学报, 2015, 41: 919–928. Qin X M, Zheng Y, Tang L, Long G Q. Effects of maize and potato intercropping on rhizosphere microbial community structure and diversity., 2015, 41: 919-928 (in Chinese with English abstract).
Response of rhizosphere bacterial community diversity to salt stress in peanut
DAI Liang-Xiang1,**, XU Yang1,**, ZHANG Guan-Chu1, SHI Xiao-Long2, QIN Fei-Fei1, DING Hong1,*, and ZHANG Zhi-Meng1,*
1Shandong Peanut Research Institute, Qingdao 266100, Shandong, China;2College of Agronomy, Shenyang Agricultural University, Shenyang 110866, Liaoning, China
To characterize the peanut rhizosphere bacteria community in response to salt stress, a pot experiment was performed with different salt concentrations. The peanut rhizosphere soils at flowering and mature stages were sampled to extract DNA for constructing bacterial 16S rRNA gene library, and then high-throughput sequencing was performed for sequencing and bioinformatics analysis. The results showed thatProteobacteria, Actinobacteria, Patescibacteria, Acidobacteria, and Chloroflexi were the dominant phyla, and the orders Saccharimonadales, Betaproteobacteria, Sphingomonadales, Gemmatimonadales, and Rhizobiales were dominated in the peanut rhizosphere soils. Comparisons of the bacterial community structure of peanuts revealed that the relative abundance of Proteobacteria dramatically increased, while that of Actinobacteria decreased in salt-treated soils, and the fluctuation increased with the increase of the salt concentration. Moreover, applying calcium fertilizer under salt stress increased the abundance of Betaproteobacteria, Gemmatimonadales, and Sphingomonadales, which were affected by salt stress, growth stages, and exogenous calcium application. Cluster analysis revealed that the dominant bacteria of soil groups with high salt concentration were similar and clustered together, while the soil samples of the same growth period were similar and clustered together according to the bacterial structure at the genus level under non-salt stress conditions. Bacterial community structure differed in the growth stages and soil salt concentrations, whereas the differences of soil groups with or without calcium application were relatively small. Function prediction analysis indicated that the sequences related to secondary metabolites, glycan biosynthesis and metabolism, and amino acid and lipid metabolism were enriched in high salt-treated soils. The functional groups increased significantly during the fast-growth period, low salt stress, and basal calcium fertilizer treatments, which may play an important role on the growth and stress response in peanut. This study of microbial communities could lay the foundation for future improvement of stress tolerance of peanuts via modification of the soil microbes.
peanut (); salt stress; rhizosphere; soil microbial community; 16S rRNA gene
10.3724/SP.J.1006.2021.04160
本研究由国家自然科学基金项目(31971856,31971854, 31901574), 山东省现代农业产业技术体系创新团队(花生)项目(SDAIT-04-06), 山东省农业科学院创新工程项目(CXGC2018B05)资助。
This study was supported by the National Natural Science Foundation of China (31971856, 31971854, 31901574), the Modern Agricultural Industry Technical System of Shandong Province (SDAIT-04-06), and the Agricultural Scientific and Technological Innovation Project of Shandong Academy of Agricultural Sciences (CXGC2018B05).
张智猛, E-mail: qinhdao@126.com; 丁红, E-mail: dingpeanut@163.com
**同等贡献(Contributed equally to this work)
戴良香, E-mail: liangxiangd@163.com
2020-07-17;
2021-01-13;
2021-02-18.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20210216.1438.004.html
