小麦lncRNA27195及其靶基因TaRTS克隆及表达分析
2021-06-09白建芳马有志郭昊宇王永波陈兆波赵昌平张立平
王 娜 白建芳 马有志 郭昊宇 王永波 陈兆波 赵昌平* 张立平*
小麦lncRNA27195及其靶基因TaRTS克隆及表达分析
王 娜1,2,**白建芳2,**马有志1郭昊宇2王永波2陈兆波2赵昌平2,*张立平2,*
1中国农业科学院作物科学研究所, 北京 100087;2北京市农林科学院北京杂交小麦工程技术研究中心/ 杂交小麦分子遗传北京市重点实验室, 北京 100097
长链非编码RNA (long non-coding RNA, lncRNA)是一种大于200 bp的非编码RNA, 大量存在于植物体中, 其可通过调节基因表达或蛋白功能, 在植物生长发育与胁迫应答中发挥重要作用。前期在研究光照和温度对小麦光温敏核雄性不育系BS366育性诱导中, 利用转录组测序筛选获得与育性相关的lncRNA ()为研究小麦的功能, 本研究在BS366中克隆及其靶基因, 并对进行生物信息学分析, 结果显示,基因全长315 bp, 编码104个氨基酸, 且发现该RTS蛋白仅存在于禾本科植物中。通过实时荧光定量PCR, 对及基因在不同组织不同光温处理及茉莉酸甲酯处理下进行表达模式分析, 发现和均在雄蕊中高表达, 呈显著的正相关, 且两者在不同光温条件下呈现出不同的表达模式。光照和温度均对和有调控作用, 适当浓度的MeJA促进两者的表达, SA抑制两者表达。以上结果表明, 在光周期、温度和植物激素的诱导下,正向调节基因表达, 进而影响花粉育性, 本研究结果有助于促进对小麦光温敏核型雄性不育系的机理研究和生产应用。
lncRNA; RTS蛋白; 花药; 雄性不育
长链非编码RNA (long non-cording RNA), 是指长度超过200个核苷酸的非编码RNA, 通过RNA聚合酶Ⅱ转录[1]。研究表明lncRNA可与DNA、RNA或蛋白质分子相互作用, 从而调节基因表达, 并通过多种机制发挥功能。在转录水平上, lncRNA可以招募转录因子, 调控相邻基因的表达[2-3]; 在表观遗传水平上, lncRNA可以通过影响染色质结构域调节目的基因的表达[4-5]; 在转录后水平上, lncRNA可通过与靶mRNA的互补碱基配对调控mRNA各方面的功能, 类似于微小RNA, 如microRNA和snoRNA[6]。目前, 在拟南芥[7]、小麦[8]、水稻[9-10]、玉米[11]、番茄[12]等植物体内均发现了lncRNA, 并发现他们具有重要的作用, 可调控一系列重要生物途径, 包括生长代谢、激素信号、逆境胁迫响应、生殖发育和维护基因组完整性等。在水稻中, 研究人员发现可调控光敏雄性不育水稻的lncRNA (), 在长日条件下高表达可保持花粉正常发育。而突变体中,的二级结构发生改变, 导致启动子区域的甲基化程度提高, 从而降低转录水平, 导致长日条件下花药绒毡层细胞提早发生程序性死亡, 进而引起光敏雄性不育[9-10]。二系杂交小麦主要是通过环境条件的改变可以调控植物的雄性育性转换[13]。光温敏核雄性不育小麦既可作为雄性不育系, 也可发挥保持系的作用, 是目前杂种优势利用的主要方式, 因此小麦光温敏核雄性不育基因的挖掘和研究具有重要意义。
RTS蛋白, 是一个花药特异性蛋白, 只在绒毡层细胞中特异性表达。研究发现水稻基因的下调表达会导致花粉败育, 从而引起水稻雄性不育[14]。绒毡层位于四层花药细胞的最内层, 对花粉的发育至关重要[15]。在绒毡层发育过程中绒毡细胞的程序性死亡对原染色体的形成、孢粉素的合成至关重要[16]。绒毡层结构破损会导致花粉失活, 引起雄性不育。例如, 拟南芥的、双突变体的花药缺乏绒毡层的生成, 导致小孢子母细胞无法在减数分裂后正常发育, 导致小孢子败育和雄性不育[17]。前期通过对光照和温度诱导的可育环境和不育环境的BS366光温敏核雄性不育小麦的花药进行lncRNA测序, 并对育性相关差异表达的lncRNA进行筛选。转录组结果发现及其靶基因()在可育和不育环境中表达差异较明显。因此, 本研究克隆及其靶基因通过对其进行生信分析、组织特异性和时空表达分析, 并分析其在不同育性条件下和非生物胁迫下的表达模式, 为进一步阐明长链非编码RNA在光温敏雄性不育小麦育性转换过程中的调控模式提供一定的理论基础。
1 材料与方法
1.1 试验材料
试验材料为光温敏核雄性不育系冬小麦BS366, 由北京市农林科学院北京杂交小麦工程技术研究中心提供。BS366表现出光温敏感性育性, 在不育环境(花粉发育阶段日平均温度12℃, 12 h光照/12 h黑暗)下, 花粉失活, 结实率<5%; 在可育环境(花粉发育阶段日平均温度20℃, 14 h光照/10 h黑暗)下, 花粉发育正常, 育性恢复至40%~55%。所有植物均种植于花盆, 放置在北京的实验田, 并进行常规管理, 至四叶一心期放入人工气候箱。
1.2 试验方法
1.2.1基因及基因的克隆
各组织RNA提取按照TRIzol Reagent (Invitrogen)说明书进行。RNA经浓度和纯度鉴定后合格放置–80℃备用。利用TaKaRa PrimeScript RT reagent Kit with gDNA Eraser (Perfect Real Time)试剂盒将总RNA反转录为cDNA。根据前期转录组学的分析结果, 根据基因序列和基因()序列利用Primer premier 5软件设计引物(表1), 以BS366花药组织样品cDNA为模板克隆及基因全长。扩增体系为: 2× Phanta Max Buffera 25 μL, dNTP Mix (10 mmol L–1each) 1 μL, 上游引物(10 μmol L–1) 1 μL, 下游引物(10 μmol L–1) 1 μL, Phanta Max Super-Fidelity DNA Polymerase 1 μL, cDNA 2 μL, ddH2O 19 μL。PCR扩增程序为: 95℃预变性3 min; 95℃变性15 s, 55℃退火15 s, 72℃延伸15 s; 72℃延伸10 min, 4℃保存。按照pBackZero-T Vector Cloning Kit说明书, 将全长序列与基因的CDS序列克隆至pBackZero-T载体上。转化大肠杆菌DH-5α, 挑取阳性单克隆菌, 对目标片段进行测序, 并将甘油菌保存于–80℃冰箱。

表1 引物设计
1.2.2基因生物信息学分析 利用ExPASy ProtParam (https://web.expasy.org/protparam/)在线预测软件对基因进行一级结构和理化性质分析。利用SOPMA (https://npsa-prabi.ibcp.fr/cgi- bin/npsa_automat.pl?page=npsa_sopma.html)在线预测软件对基因进行二级结构预测分析。利用SWISS-MODEL (https://swissmodel.expasy.org/)在线预测软件对基因进行三级结构预测分析。利用Prot Scale (http://web.expasy.org/cgi-bin/protscale/ protscale.pl)在线预测软件对基因进行亲疏水性分析。利用TargetP 1.1 Server (http://www. cbs.dtu.dk/services/TargetP-1.1/index.php)在线预测软件进行亚细胞定位的预测。利用PlantCARE在线预测软件(http://bioinformatics.psb.ugent.be/ webtools/plantcare/html/)预测其启动子元件。利用NCBI网站(https://blast.ncbi.nlm.nih.gov/Blast.cgi)和Ensembl Plant网站(http://plants.ensembl.org/Multi/ Tools/Blast)进行同源基因的查找。通过DNAMAN软件进行多序列比对。利用MEGA6.0软件进行系统进化树的构建, 采用邻接法(neighbor joining method), 其中, Bootstrap值设置为1000, 其余均为默认参数。
1.2.3基因亚细胞定位 将的ORF序列克隆到载体上, 构建融合表达载体。将融合载体转化到新制备的小麦原生质体中, 以转化的小麦原生质体作为对照, 将融合载体转化的小麦原生质体细胞在激光共聚焦显微镜(Leica, 德国)下观察绿色荧光信号在原生质体中的分布。
1.2.4 表达模式分析 组织特异性分析: 在抽穗期采集BS366小麦的根、茎、叶、颖壳、雄蕊和雌蕊样品, 置于–80℃冰箱保存备用, 进行组织特异性表达分析。不同育性环境对基因表达模式影响分析: 在四叶期结束后(育性敏感期), 将长势良好且一致BS366植株分为两组, 分别在可育环境(20℃, 14 h L/10 h D; L/D为光照/黑暗)和不育环境(12℃, 12 h L/12 h D)条件下, 相对湿度为60%~80%的人工气候培养箱(CLC-BIV-M/CLC404-TV, MMM, 德国)中培养。根据叶龄和花药长度, 采集BS366在两个不同生育条件下的孢子母细胞时期(S1)、二分体时期(S2)、四分体时期(S3)和单核期(S4)的花药组织, 置于–80℃冰箱保存备用。不同光温对基因表达模式影响分析: 将育性敏感期长势良好且一致BS366植株分为4组, 分别在低温长日环境(12℃, 14 h L/10 h D)、低温短日环境环境(12℃, 12 h L/12 h D)、高温长日环境(20℃, 14 h L/10 h D)、和高温短日环境(20℃, 12 h L/12 h D)条件下中培养约2周, 至抽穗期, 采集花药组织, 置于–80℃冰箱保存备用。茉莉酸甲酯对基因表达模式调控分析: BS366正常生长20℃ (12 h L/12 h D)至抽穗期, 每天喷施茉莉酸甲酯(MeJA), 浓度分别为0、0.5、2和4 mmol L‒1, 连续5 d后, 同时用10 mmol L‒1水杨酸(SA)喷施MeJA处理后的小穗, 采集花药组织, 置于–80℃冰箱保存备用。根据基因和基因的序列设计qPCR引物(表1), 以上述样品RNA反转录的cDNA为模板, 以为内参基因, 在(CFX96; Bio-Rad, Hercules, CA, USA)平台使用TB Green Premix Ex(Tli RNaseH Plus)试剂盒进行qPCR分析。反应体系为: TB GreenPremix Ex(Tli RNaseH Plus) (2´) 5 μL, cDNA 1 μL, 上游引物(10 μmol L‒1) 0.5 μL, 下游引物(10 μmol L‒1)0.5 μL, ddH2O 3 μL。反应程序为: 预变性95℃5 s; 95℃ 5 s, 60℃ 30 s, 45个循环; 溶解曲线分析95℃15 s, 55℃ 1 min, 95℃15 s。数据按照2–ΔΔCt法计算相对表达量, 生物学重复和技术重复均为3次。
2 结果与分析
2.1 lncRNA27195及TaRTS基因的克隆
以BS366的花药cDNA为模板克隆与基因的CDS序列, 电泳结果与网上公布的同序列大小基本一致,分别为396 bp和315 bp (图1)。将克隆至pBackZero-T载体上, 转化大肠杆菌DH5α, 挑取阳性单克隆菌, 并进行测序,结果同前期转录组测序结果(图2)。
2.2 TaRTS基因的蛋白质结构分析
小麦的基因位于3D染色体上, CDS序列长315 bp, 编码104个氨基酸。通过Ensambl搜索比对, 发现其在3A和3B上存在等位基因和。TaRTS蛋白分子量为10.08 kD; 等电点为7.54, 为碱性蛋白质; 共1389个原子; 半衰期为30 h; 不稳定指数为30.16, 为稳定蛋白; 脂肪族指数为77.31; 总平均疏水指数(grand average of hydropathicity)为0.497; 有4个带负电荷的氨基酸残基, 包括3个天冬氨酸(Asp)和1个谷氨酸(Glu), 有5个带正电荷的氨基酸残基, 包括4个精氨酸(Arg)和1个赖氨酸(Lys)。利用Prot Scale在线软件测得TaRTS蛋白的氨基酸残基整体为疏水性, 同理化性质分析一致, 推测为疏水性蛋白(图3-A)。蛋白二级结构分析显示, 该蛋白由41个α-螺旋(39.42%)、12个延伸链(11.25%)、9个β-转角(8.65%)和42个无规则卷曲(40.38%)组成(图3-B)。蛋白三级结构结果显示主要结构为α-螺旋(图3-C)。亚细胞定位预测结果显示TaRTS蛋白为分泌蛋白。此外, 小麦原生质体亚细胞定位显示, 绿色荧光蛋白GFP在小麦原生质体中瞬时高效表达, 表明TaRTS蛋白定位于原生质体的细胞质部分, 符合预测结果(图4)。
2.3 TaRTS基因编码蛋白的系统发育分析
研究结果显示仅在水稻(Group)、粟()、山羊草()、二穗短柄草()、哈氏黍()和狗尾草()这些禾本科植物中发现同源基因, 表明基因家族是一种禾本科特有基因。多序列比对结果显示,基因家族的保守性较差, 但具有保守的半胱氨酸位点(图5-A)。利用MEGA 6.0软件用邻接法构建系统进化树, 显示基因与二穗短柄草的同源性较高, 同粟、狗尾草同源性较低(图5-B)。
蓝色为α-螺旋, 绿色为β-转角, 红色为延伸链, 紫色为无规则卷曲。
Blue line means alpha helix, green line means beta turn, red line means extended strand, and purple line means random coil.
2.4 TaRTS基因启动子元件分析
对基因启动子区前2000 bp序列的转录因子的预测分析发现, 基因的启动子序列中包含: 光响应元件(ACE, G-box, GT1-motif, T-box, Sp1)、分生组织表达有关(CAT-box)、MYBHv1结合位点(CCAAT-box)、茉莉酸甲酯响应元件(CGTCA-motif, TGACG-motif)、低温响应元件(LTR)、细胞周期调控元件(MSA-like)等元件。推测基因与光、温胁迫, 调节细胞周期相关(表2)。
2.5 lncRNA27195及TaRTS组织特异性分析
本实验研究了与基因在小麦孕穗期(挑旗)根、茎、旗叶、颖壳、雄蕊和雌蕊不同组织的表达谱。结果显示,与基因在小麦雄蕊中表达量最高, 显著高于其他组织(<0.01), 而其他组织中的表达量极低, 表明与在雄蕊上特异性表达, 主要参与花药发育的调控(图6)。
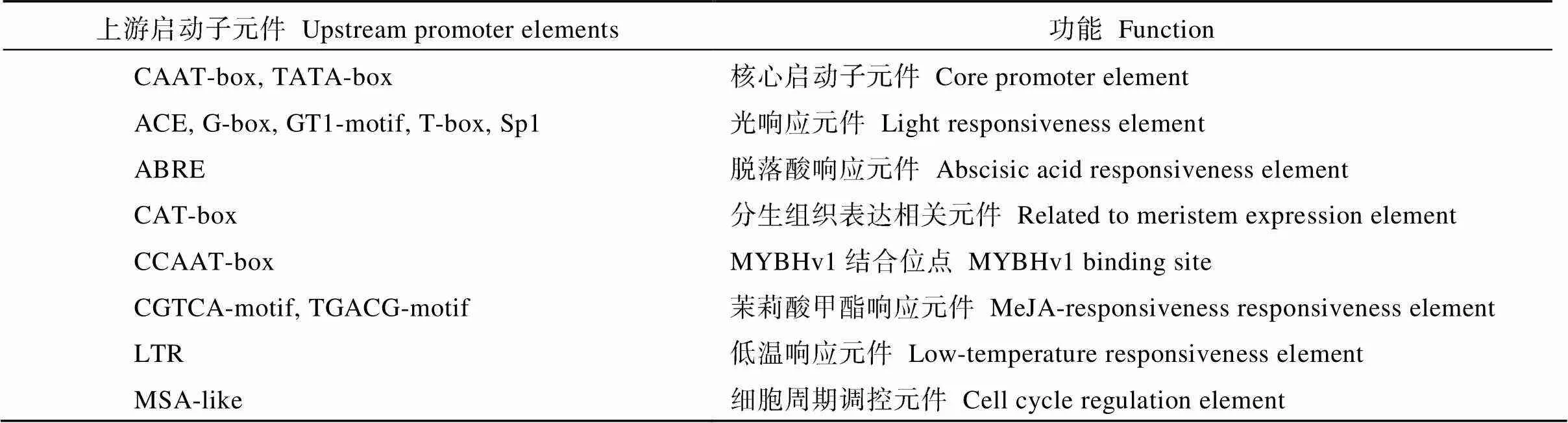
表2 启动子元件分析
2.6 lncRNA27195及TaRTS不同花药发育时期表达分析
本研究对与基因在不同育性条件下的4个发育时期的花药中的表达模式进行了分析。结果表明,基因在二分体时期、四分体时期和单核期, 可育环境下的表达量均极显著高于不育环境(<0.01)。在二分体时期可育环境下的表达量极显著高于不育环境(<0.01); 在四分体时期和单核期, 可育环境下的表达量均显著高于不育环境(<0.05); 在小孢子母细胞时期, 不育环境的表达量显著高于可育环境(<0.05) (图7-A)。皮尔逊相关性分析结果显示,同基因的表达量在小麦花药中呈显著正相关(2=0.8724) (图7-B)。
2.7 lncRNA27195及TaRTS在不同光温处理下的表达模式分析
前期发现BS366的育性受光照和温度的影响, 且生信分析发现基因启动子区包含光和低温响应元件(表2), 因此对不同光照和温度条件下,与基因在花药中的表达水平进行分析。结果显示, 在高温长日条件(20℃, 14 h L/10 h D)、高温短日条件(20℃, 12 h L/12 h D)、低温长日条件(12℃, 14 h L/10 h D)和低温短日(12℃, 12 h L/12 h D)条件下, 小麦的相对表达量依次下降, 四个条件下存在显著性差异; 小麦基因的表达量依次下降, 高温长日条件、高温短日和低温长日条件、低温短日条件三组间存在显著性差异(图8)。此外, 小麦与基因在不同温度(相同光照)和不同光照(相同温度)的组别间的数据分析中显示, 两者皆存在显著性差异, 且受温度影响较大。结果表明, 光照和温度均对小麦与基因的表达存在显著影响, 高温和长日照可以促进与基因的表达, 进而促进育性恢复。
2.8 lncRNA27195及TaRTS在MeJA及SA处理下的表达模式分析
前期研究表明, MeJA参与小麦雄性不育, 可促进花药开裂, 同时SA对该功能有一定的拮抗作用; 本文前期的生物信息学分析中发现基因启动子区存在MeJA响应元件。为研究MeJA与之间的调控方式, 以及对的调控作用, 本研究利用不同浓度的MeJA对抽穗后开花前的花药进行处理, 同时研究SA喷施MeJA处理后的花药中,及的表达模式。在MeJA处理后, 随着浓度的上升,及的表达量先上升后下降, 在MeJA浓度为0.5 mmol L‒1时达到峰值; 通过SA喷施处理后, 发现及的表达量较正常植株显著下降, 且随着MeJA浓度的上升,及的表达量不断下降(图9)。实验表明, 适量的MeJA可以促进及的表达, 但过高浓度的MeJA反而会抑制其表达; SA可以抑制MeJA的功能, 从而抑制及的表达。
*代表显著差异(<0.05), **代表极显著差异(<0.01)。
* means significantly different at<0.05, and ** means highly significant difference at<0.01.
T1: 温长日环境(20℃, 14 h L/10 h D); T2: 高温短日环境(20℃, 12 h L/12 h D); T3: 低温长日环境(12℃, 14 h L/10 h D); T4为低温短日(12℃, 12 h L/12 h D)环境。*代表各组间存在显著差异(< 0.05)。
T1: long-light environment with high temperature (20℃, 14 h L/10 h D); T2: short-light environment with high temperature (20℃, 12 h L/12 h D); T3: long-light environment with low temperature (12℃, 14 h L/10 h D); T4: short-light environment with low temperature (12℃, 12 h L/12 h D). * means significantly different at< 0.05.
3 讨论
在水稻中, 研究表明基因仅在绒毡层中表达, 对花粉发育至关重要, 且抑制其表达会导致雄性不育[14]。TaRTS蛋白结构分析发现, 其丙氨酸含量较高(图1-A), 其他植物中发现的一些功能未知的花药特异性表达蛋白也具有这一特征, 如水稻中的RA8蛋白[18], 拟南芥[19]和油菜花[20]中的Bcp1蛋白。拟南芥基因, 在绒毡层和小孢子中都有表达, 也被证明是花粉发育所必需的, 其在二倍体绒毡层或单倍体小孢子中的低表达或不表达会导致花粉发育停滞[19]。本文研究发现TaRTS蛋白中富含半胱氨酸, 同家族蛋白也同样如此(图5)。半胱氨酸残基可以通过二硫键提供分子内和分子间的交联, 从而形成蛋白质的三级和四级结构, 表明该特征对蛋白质的功能的发挥具有重要作用。此外, RTS蛋白的N端存在较强的疏水结构域(图3), 这种结构域主要与蛋白靶向到膜部分或细胞外空间过程相关。这些特征也在其他一些花药特异性蛋白中被观察到, 如水稻的PS1[21]、玉米的Zmc13[22]和番茄的LAT52[23]。绒毡层主要参与孢粉素的合成和沉积进而释放到花粉中[24], 本研究中TaRTS蛋白被预测为分泌蛋白, 亚细胞定位到细胞质中(图4), 因而TaRTS蛋白可能被绒毡层细胞分泌到花粉中, 为花粉壁的形成提供必要的组分, 或者为花粉的生长发育提供营养。本研究克隆的基因, 其开放阅读框(open reading frame, ORF) GC含量较高(72.7%)。Tsuchiya等[25]分离得到的花药特异性的水稻基因的ORF中显示出较高的GC含量(70.3%和69.6%)。ORF在其他花药特异性基因中是否存在GC含量高的现象尚不清楚。然而, 可以推测高GC含量提高植物对环境胁迫的耐受性, 因为GC之间的氢键比AT更稳定, 而花药是环境胁迫下最脆弱的器官之一。
BS366属于光温敏型核雄性不育, 在低温短日照条件下不育, 高温长日下可育。本研究表明的启动子序列同时存在光响应元件和低温响应元件(表1), 而及在不同光温处理下的表达模式显示,及的表达量存在显著性差异, 高温和长日照可以促进与基因的表达(图8), 可见其同BS366的育性转换存在着密切的联系。
茉莉酸甲酯(MeJA)广泛分布在植物中, 影响果实成熟、失活花粉的产生、根的生长、卷须卷曲、植物对伤害和非生物胁迫的反应, 以及对昆虫和病原体的防御等过程[26]。Mandaokor等[27]的研究显示, 控制拟南芥的雄蕊发育相关转录因子MYB21和MYB24受茉莉酸甲酯调控。在拟南芥突变体中发现茉莉酸甲酯含量低, 花药开裂异常, 花丝伸长未能正常进行、花粉发育延迟, 导致雄性不育[28], 表明茉莉酸与花药育性直接相关。同时, 水杨酸(SA)和JA途径相互影响, SA的积累可以抑制JA的表达[29], WIPK蛋白的转录和表达可以激活JA的生成, 且抑制SA通路的信号传导[30]。本研究中利用MeJA和SA对抽穗期的小麦进行喷施处理的实验结果发现, 0.5 mmol L‒1的MeJA可以促进及的表达, SA的喷施可以抑制MeJA的功能, 从而抑制及的表达, 表明了及受到MeJA介导的JA信号通路调控(图9)。
本研究通过系统进化树分析发现, RTS蛋白目前仅在禾本科植物中有报道, 其他的植物中无相关序列报道。其原因可能是由于, 禾本科的进化路线是由虫媒传粉向风媒传粉演化并且高度特化,花被和雄蕊简化, 因而推测RTS蛋白的出现是为了保证雄蕊的正常发育, 但其具体功能机制尚不可知[31]。此外,与基因均在雄蕊中特异性表达(图6), 且两者间存在显著的正相关关系(图7-B), 说明两者参与花粉的生长发育。另外与基因在可育环境的表达量二分体时期, 达到峰值后表达量下调; 在不育环境则是在小孢子母细胞时期表达量达到峰值, 之后表达量下调(图7-A), 表达峰值较正常植株提前一个时期。另外前人研究表明, 绒毡层程序性死亡的提前和推迟会导致花粉失活, 植株发生雄性不育[32-33]。光周期敏感基因雄性不育水稻(农研58)的花粉退化过程与绒毡层的异常行为密切相关, 光敏雄性不育(PGMS)植株较正常植株提前进行绒毡层程序性死亡, 但绒毡层退化进展缓慢, 直到花粉分解才完全退化[34]。本研究表明, TaRTS蛋白作为分泌蛋白, 其基因表达峰值较正常植株提前, 从而可能导致绒毡层提前降解, 导致花粉败育。
4 结论
和其靶基因是小麦花药特异性基因, 通过生物信息学分析及表达水平分析显示, 两者表达量呈正相关关系, 参与花粉育性调节, 为分泌蛋白, 且受MeJA调控, 可伴随绒毡层凋亡分泌到花粉中, 为花粉壁的形成提供必要的组分, 为花粉的生长发育提供营养。
[1] Espinoza C A, Goodrich J A, Kugel J F. Characterization of the structure, function, and mechanism of B2 RNA, an ncRNA repressor of RNA polymerase II transcription., 2007, 13:583–596.
[2] Sigova A A, Mullen A C, Molinie B, Gupta S, Orlando D A, Guenther M G, Almada A E, Lin C, Sharp P A, Giallourakis C C, Young R A. Divergent transcription of long noncoding RNA/mRNA gene pairs in embryonic stem cells., 2013, 110:2876–2881.
[3] Miao S, Lee K W. From discovery to function: the expanding roles of long non-coding RNAs in physiology and disease., 2015, 36:25–64.
[4] Rinn J L, Kertesz M, Wang J K, Squazzo S L, Xu X, Brugmann S A, Henry Goodnough L, Helms J A, Farnham P J, Segal E, Chang H Y. Functional demarcation of active and silent chromatin domains in humanloci by noncoding RNAs., 2007, 129:1311–1323.
[5] Kristina P S, Thomassen M, Tan Q, Martin B, Torben A K. Long non-coding RNAis an independent prognostic marker of metastasis in estrogen receptor-positive primary breast cancer., 2013, 142:529–536.
[6] Reis E M, Nakaya H I, Louro R, Canavez F C, Flatschart A V, Almeida G T, Egidio C M, Paquola A C, Machado A A, Festa F, Yamamoto D, Alvarenga R, da Silva C C, Brito G C, Simon S D, Moreira Filho C A, Leite K R, Camara Lopes L H, Campos F S, Gimba E, Vignal G M E, Dorry H, Sogayar M C, Barcinski M A, da Silva A M, Verjovski Almeida S. Antisense intronic non- coding RNA levels correlate to the degree of tumor differentiation in prostate cancer., 2004, 23: 6684–6692.
[7] Swiezewski S, Liu F, Magusin A, DeanC. Cold-induced silencing by long antisense transcripts of anPolycomb target., 2009, 462: 799–802.
[8] Xin M M, Wang Y, Yao Y Y, Song N, Hu Z, Qin D D, Xie C J, Peng H R, Ni Z F, Sun Q X. Identification and characterization of wheat long non-protein coding RNAs responsive to powdery mildew infection and heat stress by using microarray analysis and SBS sequencing.,2011,11:61.
[9] Ding J, Lu Q, Ou-Yang Y, Mao H, Zhang P, Yao J, Xu C, Li X, Xiao J, Zhang Q. A long noncoding RNA regulates photoperiod-sensitive male sterility, an essential component of hybrid rice., 2012, 109:2654–2659.
[10] Zhou H, Liu Q J, Li J, Jiang D, Zhou L, Wu P, Lu S, Li F, Zhu L, Liu Z, Chen L, Liu Y, Zhuang C. Photoperiod- and thermo- sensitive genic male sterility in rice are caused by a point mutation in a novel noncoding RNA that produces a small RNA., 2012, 22:649–660.
[11] Dai X Y, Yu J J, Zhao Q, Zhu D, Ao G. Non-coding RNA for, a pollen-specific gene of., 2004, 46:497–504.
[12] Liu C M, Muchhal U S, Raghothama K G. Differential expression of, a phosphate starvation-induced gene in tomato., 1997, 33:867–874.
[13] 赵昌平, 王新, 张风廷, 叶志杰, 戴惠君. 杂种小麦的研究现状与光温敏二系法. 北京农业科学,1999, 17(2):3–5. Zhao C P, Wang X, Zhang F T, Ye Z J, Dai H J. Research status of hybrid wheat and the photosensitive two-system method., 1999, 17(2): 3–5 (in Chinese with English abstract).
[14] Luo H, Lee J Y, Hu Q, Nelson-Vasilchik K, Eitas T K, Lickwar C, Kausch A P, Chandlee J M, Hodges T K., a rice anther- specific gene is required for male fertility and its promoter sequence directs tissue-specific gene expression in different plant species., 2006, 62:397–408.
[15] Kelliher T, Walbot V. Hypoxia triggers meiotic fate acquisition in maize., 2012, 337:345–348.
[16] Shi J, Cui M, Yang L, Kim Y J, Zhang D B. Genetic and biochemical mechanisms of pollen wall development., 2015, 20: 741–753
[17] Julian I S.somatic embryogenesis receptor kinases1 and 2 are essential for tapetum development and microspore maturation., 2005, 17:3350–3361.
[18] Jeon J S, Chung Y Y, Lee S, Yi G H, Oh B G, An G. Isolation and characterization of an anther-specific gene,, from rice (L.)., 1999, 39:35–44.
[19] Xu H, Knox R B, Philip E T, Singh M B., a gene required for male fertility in., 1995, 92:2106–2110.
[20] Theerakulpisut P. Isolation and developmental expression of, an anther-specific cDNA clone in., 1991, 3:1073–1084.
[21] Zou J T, Zhan X Y, Wu H M, Wang H, Cheung A Y. Characterization of a rice pollen-specific gene and its expression., 1994, 81:552–561.
[22] Hanson D D, Hamilton A D, Travis L J, Bashe D M, Mascarenhas J P. Characterization of a pollen-specific cDNA cloneand its expression., 1989, 1:173–179.
[23] Twell D, Wing R, Yamaguchi J, McCormick S. Isolation and expression of an anther-specific gene form tomato., 1989, 217:240–245.
[24] McCormick S. Male gametophyte development., 1993, 5:1265–1275.
[25] Tsuchiya T, Toriyama K, Nasrallah M E, Ejiri S. Isolation of genes abundantly expressed in rice anthers at the microspore stage.,1992, 20:1189–1193.
[26] Creelman R A, Mullet J E. Biosynthesis and action of jasmonates in plants., 1997, 48: 355–381.
[27] Mandaokar A, Thines B, Shin B, Lange B M, Choi G, Koo Y J, Yoo Y J, Choi Y D, Choi G, Browse J. Transcriptional regulators of stamen development inidentified by transcriptional profiling., 2006, 46: 984–1008.
[28] Muruáis G, Lalioti V, Sandoval I V. The Cdk5 inhibitor roscovitine strongly inhibits glucose uptake in 3T3-L1 adipocytes without altering GLUT4 translocation from internal pools to the cell surface., 2009, 220: 238–244.
[29] Seo S, Okamoto N, Seto H, Ishizuka K, Sano H, Ohashi Y. Tobacco map kinase: a possible mediator in wound signal- transduction pathways., 1995, 270:1988–1992.
[30] Seo S, Sano H, Ohashi Y. Jasmonate-based wound signal transduction requires activation of WIPK, a tobacco mitogen-activated protein kinase., 1999, 11:289–298.
[31] 韩建国, 樊奋成, 李枫. 禾本科植物的起源、进化及分布. 植物学报,1996,1:10–14. Han J G, Fan F C, Li F. Origin, evolution and distribution of the Gramineae., 1996,1:10–14 (in Chinese with English abstract).
[32] Yu J, Meng Z, Liang W, Behera S, Jörg K, Tucker M R, Luo Z, Chen M, Xu D, Zhao G, Wang J, Zhang S, Kim Y J, Zhang D. A rice Ca2+binding protein is required for tapetum function and pollen formation., 2016, 176:1772–1786.
[33] Kapoor S. Silencing of the tapetum-specific zinc finger genecauses premature degeneration of tapetum and pollen abortion in Petunia., 2002, 14: 2353–2367.
[34] Shi Y, Zhao S, Yao J. Premature tapetum degeneration: a major cause of abortive pollen development in photoperiod sensitive genic male sterility in rice., 2010, 51: 774–781.
Cloning and expression analysis ofand its target genein wheat (L.)
WANG Na1,2,**, BAI Jian-Fang2,**, MA You-Zhi1, GUO Hao-Yu2, WANG Yong-Bo2, CHEN Zhao-Bo2, ZHAO Chang-Ping2,*, and ZHANG Ling-Ping2,*
1Institute of Crop Sciences, Chinese Academy of Agricultural Sciences, Beijing 100087, China;2Beijing Engineering and Technique Research Center for Hybrid Wheat, Beijing Academy of Agriculture and Forestry Sciences / Municipal Key Laboratory of the Molecular Genetics of Hybrid Wheat, Beijing 100097, China
Long non-coding RNA (lncRNA) is a non-coding RNA length over 200 bp, which is abundant in plants. It plays important roles in plant growth, development, and stress response by regulating gene expression or protein function. In the previous study, a fertility-related lncRNA namedwas screened and obtained by transcriptome sequencing from the anther of wheat Photoperiod-thermo Sensitive Genic Male Sterility (PTGMS) line BS366. To investigate the function ofin wheat, thegene and its target genewere cloned from BS366. Bioinformatics analysis were performed on. The expressions ofandin different tissues and their expression correlation between them were analyzed by qRT-PCR. Meanwhile, the expression patterns ofandunder different light and temperature treatments, and methyl jasmonate (MeJA) treatments were investigated. The results showed that thegene with 315 bp length, encoded 104 amino acids. Additionally, RTS proteins were only found as anther-specific proteins in gramineae plants. Bothandwith a significantly positive correlation were highly expressed in stamens, and revealed different expression patterns in different fertility environments. The results demonstrated that the expression ofandwere also regulated by light and temperature. In addition, we found that the appropriate concentration of MeJA could promote the expression ofandwhile SA could inhibit the expression. The results indicated that under the induction of photoperiod, temperature, and plant hormones,positively regulatedgene expression, resulting in affecting pollen development and male fertility. This study contributed to the mechanism research and production application of PTGMS wheat.
lncRNA; RTS protein; anther; male sterility
10.3724/SP.J.1006.2021.01071
本研究由北京市自然科学基金项目(6182014), 北京市农林科学院杰出科学家项目(JKZX201907), 北京市农林科学院青年基金(QNJJ201916), 国家自然科学基金项目(31872881)和北京市农林科学院科技创新能力建设专项(KJCX20180403)资助。
The work was supported by the Natural Science Foundation of Beijing (6182014), the Outstanding Scientist Cultivation Program of BAAFS (JKZX201907), the Foundation for Youths of BAAFS (QNJJ201916), the National Natural Science Foundation of China (31872881), and the Special Project of Science and Technology Innovation Ability Construction of BAAFS (KJCX20180403).
张立平, E-mail: lpzhang8@126.com; 赵昌平, E-mail: cp_zhao@vip.sohu.com
**同等贡献(Contributed equally to this work)
王娜, E-mail: wangna_00@sina.com
2020-09-02;
2021-01-13;
2021-02-22.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20210222.1051.004.html
