基于生物信息学方法的胃癌靶点预测研究
2021-06-07姚远
姚远
天津医科大学肿瘤医院,国家肿瘤临床医学研究中心,天津市“肿瘤防治”重点实验室,天津市恶性肿瘤临床医学研究中心,胃肠肿瘤生物学研究室 300060
0 引言
胃癌是最常见的消化系统恶性肿瘤[1],我国是世界上胃癌发病率最高的国家之一。目前,临床上针对胃癌的早期诊断方案尚未健全,因此开展特异性靶点研究对于了解胃癌发生、发展过程及临床早期诊治尤为重要,而揭示胃癌发生、发展的分子机制也对后期相关治疗具有重要意义[2]。随着生物信息学的发展和在线数据库的不断完善,根据已验证的基因表达芯片数据进行研究已成为癌症研究不可或缺的一部分。本研究中,对胃癌基因芯片GSE63121(miRNA)[3]和GSE2685(mRNA)[4]进行分析,以获得胃癌中明显差异表达的基因,并通过基因肿瘤学(gene ontology,GO)和京都基因和基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)进行富集分析,从而对差异表达基因的作用途径进行预测,在蛋白质交互作用网络(proteinprotein interaction networks,PPI)中对差异基因在蛋白水平的作用进行评估,再使用基因表达谱交互分析(gene expression profiling interactive analysis,GEPIA)数据库和TIMER2.0数据库对差异表达基因进行再一次筛选富集,以寻找胃癌的关键作用靶点。
1 方法
1.1 数据的获取
从高通量基因表达(gene expression omnibus,GEO)数据库(http://www.ncbi.nlm.nih.gov/geo/)中检索到的胃癌相关基因表达芯片为GSE63121(miRNA)和GSE2685(mRNA)。在这些数据中,实验组为胃癌组织样本(样本数均大于15),对照组为正常胃组织样品。所获得的基因表达芯片数据提供了足够的信息进行生物信息学分析。其中,GSE2685数据中包含22例胃癌组织样本和8例正常胃组织样本;GSE63121数据中包含15例胃癌组织样本和15例正常胃组织样本。
1.2 数据处理与分析
利用GEO数据库提供的GEO2R在线工具对筛选到2个胃癌相关的基因表达芯片数据进行初步分析,从中提取出基因表达矩阵。随后,使用R语言提供的DESeq2工具包对基因表达矩阵中的实验组和对照组数据进行差异基因分析,并根据调整P值和筛选阈值(|Log2FC|)对差异基因进行筛选,其中FC(fold change)表示实验组和对照组间表达量的比值。筛选标准:P<0.05、|Log2FC|>1.0。绘制胃癌相关差异表达mRNA和miRNA基因的火山图。
1.3 差异表达miRNA的潜在靶基因预测
使用miRDB(http://mirdb.org/)对基因表达芯片数据中差异表达miRNA的潜在靶基因进行预测,并将预测到的潜在靶基因与筛选到的差异表达基因进行互相映射,得到两者的共有基因,用于富集分析。
1.4 差异表达基因的GO和KEGG富集分析
使用注释、可视化和集成发现(the database for annotation,visualization and integrated discovery,DAVID)(https://david.ncifcrf.gov/)数据库对差异表达基因进行GO和KEGG富集分析。由于DAVID是一套完整的基因功能在线注释工具,能够较为方便地为研究者提供基因背后的生物学意义。将筛选到的差异表达基因的基因符号(gene symbol)输入DAVID数据库中,进行GO和KEGG富集分析,以P<0.05作为筛选标准。
1.5 蛋白-蛋白相互作用以及网络构建
为了进一步分析GSE63121和GSE2685中共有的差异表达基因的功能,使用检索相互作用基因的搜索工具(the search tool for the retrieval of interacting genes,STRING)数据库(https://string-db.org/)对差异表达基因之间可能存在的或已经通过实验验证的蛋白-蛋白相互作用进行评估和预测。将GSE63121和GSE2685中共有的差异表达基因的基因符号(gene symbol)输入STRING数据库进行蛋白-蛋白相互作用分析,然后选取相互作用分值大于0.4的基因进行可视化分析。
1.6 基因表达谱分析
使用GEPIA数据库(http://gepia.cancer-pku.cn/detail.php?gene)分析来自癌症基因组图集(the cancer genome atlas,TCGA)数据库(https://portal.gdc.cancer.gov/)和GTEx数据库中的胃癌相关差异基因和患者总生存期(overall survival,OS)之间的相关性。
1.7 肿瘤免疫浸润水平相关分析
使用TIMER2.0(http://timer.cistrome.org/)分析最终筛选到的基因的表达与肿瘤微环境中调节性T细胞(Tregs)浸润水平的相关性。
2 结果
2.1 差异表达mRNA潜在靶基因预测结果
当以P<0.05、|Log2FC|>1.0为标准时,使用DESeq2工具包共获得了734种差异表达的mRNA基因(下调和上调的基因数分别为473和261)和20个差异表达的miRNA基因(上调和下调的miRNA数分别为1和19)。(图1)

图1 差异表达基因的火山图
2.2 GO和KEGG富集分析结果
富集分析结果如图2和图3所示。从图中可以发现,差异表达基因主要注释到GO terms,功能涉及细胞组成或生物发生、应答刺激反应、免疫系统过程、信号转导及发育过程等。此外,这些差异表达基因可能参与的代谢途径主要有参与肿瘤发生和发展、细胞焦点连接形成、p13K-Akt信号通路、神经活性配体受体连接途径以及药物代谢-细胞色素P450代谢通路。表1和表2种列出了富集分析的详细结果。上述结果表明,这些差异表达基因可能通过多种代谢途径来响应机体应对胃癌造成的损伤。
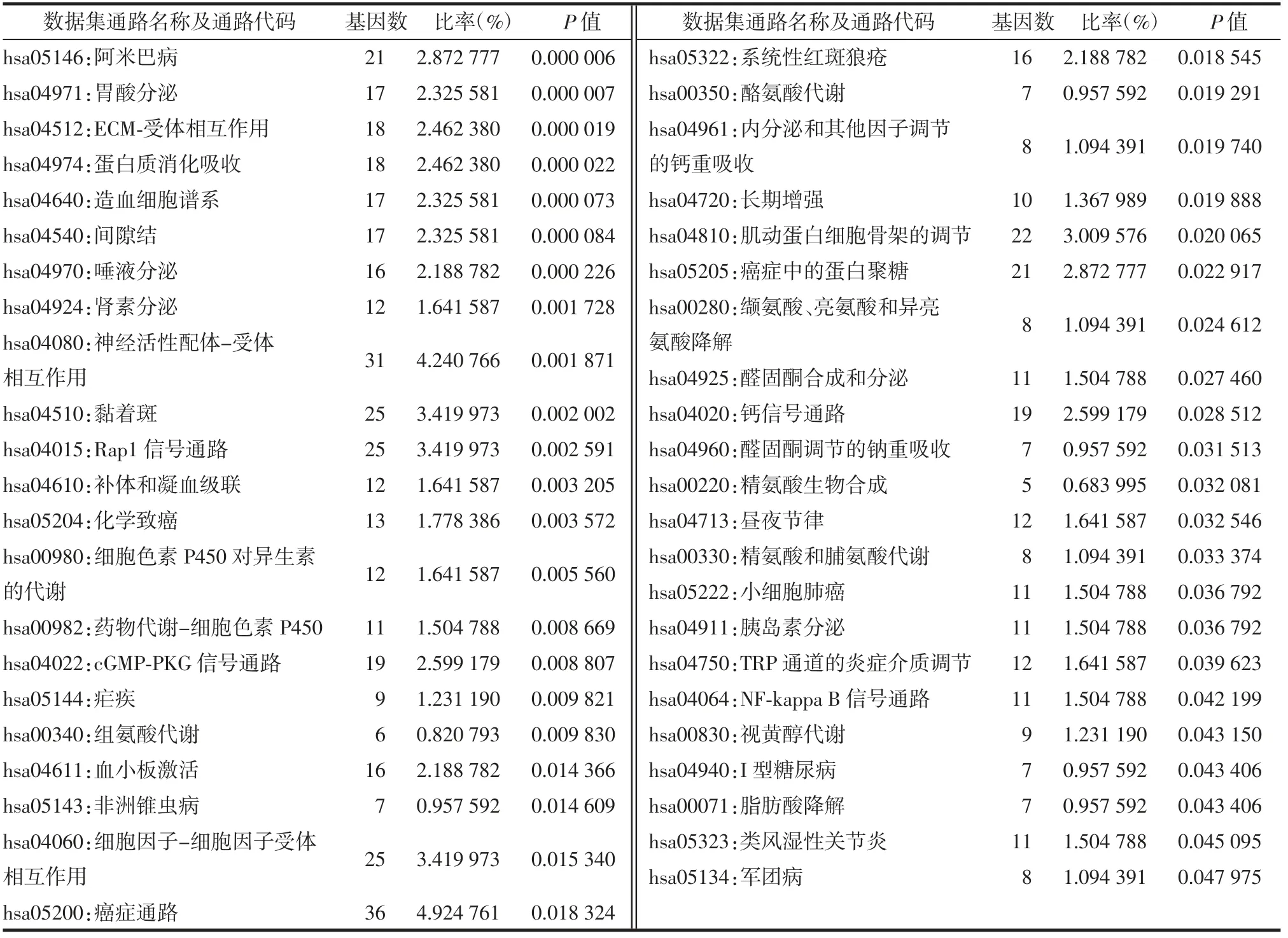
表2 差异表达基因的京都基因和基因组百科全书(KEGG)代谢途径富集分析结果

图3 差异表达基因的京都基因和基因组百科全书(KEGG)通路富集分析

表1 差异表达基因的基因肿瘤学(GO)富集分析结果详情(前20位)

图2 差异表达基因的基因肿瘤学(GO)富集分析
2.3 差异表达miRNA潜在靶基因预测及共有差异表达基因鉴定结果
miRNA作为一类非编码单链小RNA,在转录后水平上调节基因的表达,进而参与多种代谢途径。为寻找胃癌相关的miRNA并构建miRNA-mRNA调控网络,本文中对鉴定到的差异表达miRNA的潜在靶基因进行预测,共预测到2 740种潜在靶基因。进一步,将这些潜在靶基因与之前得到的胃癌相关差异表达mRNA的靶基因(共734种)进行相互映射。共筛选到103种差异表达miRNA和mRNA的潜在靶基因,称为共有差异表达基因,这些基因在胃癌组织均差异表达。上述结果表明,miRNA通过调控mRNA的表达来应答胃癌的发展。
2.4 共有差异表达基因PPI网络构建
将103种共有差异表达基因的基因符号(gene symbol)输入STRING数据库,进行PPI网络构建,总共得到103个节点,221条边,平均每个节点的度为4.29,如图4所示。结果表明,C-X-C基序趋化因子 配 体12(C-X-C motif chemokine ligand 12,CXCL12)、蛋白激酶cAMP依赖性Ⅱ型调节亚基β(protein kinase cAMP-dependent typeⅡregulatory subunit beta,PRKAR2B)、蛋白激酶cAMP依赖性催化β(protein kinase cAMP dependent catalytic beta,PRKACB)、KIT(KIT原癌基因)、C-X-C基序趋化因子配体8(C-X-C motif chemokine ligand 8,CXCL8)、cAMP反应元件结合蛋白1(cAMP responsive element binding protein 1,CREB1)、雌激素受体1(estrogen receptor 1,ESR1)和发动蛋白(Dynamin 1,DNM1)等基因位于调控网络的中心,且和许多差异表达基因存在相互作用关系,表明这些基因在胃癌形成以及机体应对胃癌的过程中扮演重要的角色。进一步分析可知,CREB1和ESR1这两种基因与胃癌具有较强的相关性,且在GSE2685数据中,这两种基因的相对表达量均下调,变化率分别为-1.08%和-1.46%。这两种基因参与多条代谢通路,如代谢类,免疫调节类等。
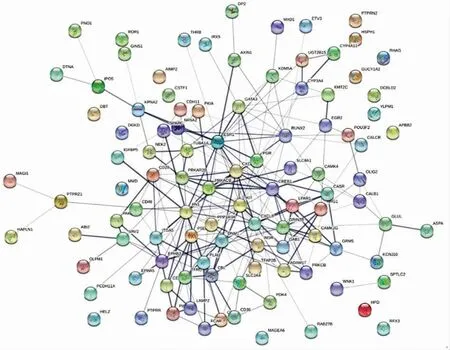
图4 共有差异表达基因的蛋白质交互作用网络(PPI)构建
2.5 差异表达基因与胃癌预后的关系
在GEPIA数据库中,当将截断值(cut off)设置为“median”,以95%CI为筛选标准,并将预后节点设为120个月,此时的生存分析结果如图5所示。结果表明,CREB1和ESR1低表组胃癌患者的预后明显著好于高表达组。
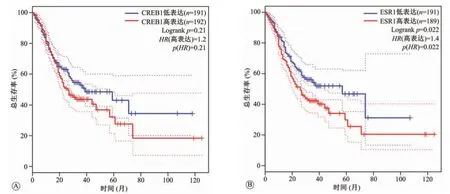
图5 cAMP反应元件结合蛋白1(CREB1)和雌激素受体1(ESR1)表达水平与胃癌患者生存率的关系
2.6 肿瘤免疫浸润相关性分析结果
由于富集分析结果提示差异表达基因涉及的主要途径为免疫过程,因此使用TIMER2.0数据库分析CREB1和ESR1与Tregs细胞丰度的相关性。如图6所示,CREB1(ρ>0,P=7.21×10-18)和ESR1(ρ>0,P=4.46×10-26)与Tregs细胞的丰度均正相关。该结果提示CREB1和ESR1高表达可能会提高肿瘤微环境中免疫抑制性细胞的浸润水平。
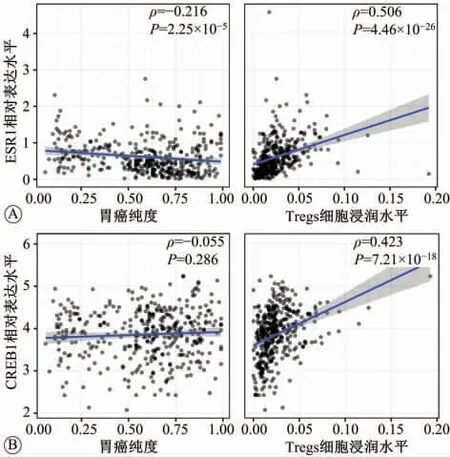
图6 cAMP反应元件结合蛋白1(CRE B1)和雌激素受体1(ESR1)与胃癌的Treg细胞的免疫浸润水平
3 讨论与结论
胃癌是消化系统中常见的恶性肿瘤,其早期诊断和治疗一直是研究热点。本研究中使用生物信息学方法分析与胃癌相关的基因表达芯片数据并成功筛选到CREB1和ESR1基因,进而对这两种基因进行功能富集分析。结果表明,CREB1和ESR1在胃癌患者中表达升高,说明二者参与调节胃癌微环境中免疫系统的构成,且通过调节免疫作用来影响胃癌的发展。
在小鼠结肠炎模型中,alpinetin会上调CREB1与Foxp3启动子区结合,通过调控miRNA-302/DNMT-1/CREB1信号通路促进Tregs细胞分化。与肿瘤环境相反的是,过表达CREB1在炎症中可以有效改善病情[5]。在胰腺癌模型中,靶向CREB1可抑制胰腺肿瘤的发展[6]。已有研究结果表明,CREB1在肿瘤组织中异常高表达且对淋巴结转移有促进作用[7],CREB1可通过调控p-Akt和mTOR信号通路来增强肿瘤细胞的增殖和迁移能力[8]。此外,结直肠癌组织中通常高表达葡萄糖转运蛋白(glucose transporter 3,GLUT3),而葡萄糖代谢降低时会产生低糖应激效应,通过AMPK/CREB1途径显著上调GLUT3[9],同时伴随不良预后。在乳腺癌模型中,突变激活ESR1会对芳香化酶抑制剂产生抗性,这使ESR1成为潜在的作用靶点[10]。ESR1是乳腺癌中常见的驱动基因[11],表达ESR1时会增强突变信号。研究结果表明,ESR1与转移性乳腺癌的不良预后相关[12]。在转移性肿瘤模型中会出现高丰度的基因组突变,这往往与肿瘤的免疫浸润相关[13-14]。然而,目前尚未见到关于上述机制在胃癌中作用的相关报道。但现有研究结果均支持CREB1和ESR1在肿瘤发生过程中可以影响免疫浸润,同时调节相关通路,从而对肿瘤细胞的增殖、迁移、凋亡发挥作用。然而,现有的研究均在胃癌指标选择方面或多或少存在相对局限性,因此开展多种功能交叉联合诊断研究对临床有重要意义。
综上,本研究中使用生物信息学方法找到CREB1和ESR1这两种胃癌相关的差异表达基因,推测二者在胃癌发生、发展过程中影响免疫浸润,同时通过相关调节通路影响胃癌细胞的增殖、迁移、凋亡等。CREB1和ESR1有望成为研究胃癌发病机制和分子机制及临床应用的重要靶点,后续可进一步通过分子生物学实验和免疫学实验对其进行功能验证。
利益冲突所有作者均声明不存在利益冲突
