植物果胶甲酯酶与果胶甲酯酶抑制子研究进展
2021-05-26李佳颖胡利华周海连魏晓双王艳婷王令强
李佳颖 胡利华 周海连 魏晓双 王艳婷 王令强

摘要:果胶是植物细胞壁的主要组分,存在于胞间层与中间层,影响细胞的流变性与黏附性能。果胶的甲酯化程度影响果胶形态,对稳定细胞壁性质以及植物生长发育过程具有重要作用。果胶甲酯酶(PME)是果胶去甲酯化修饰的关键酶类,果胶甲酯酶抑制子(PMEI)可特异地结合PME,调节其活性,共同决定果胶的去甲脂化。本文综述了PME与PMEI的结构及生化作用机制、相关基因家族和表达模式,并进一步对二者介导的果胶去甲酯化过程对细胞壁的影响及其与花粉管发育、根器官形成等植物生长发育过程和逆境响应关系的最新研究进展做了介绍,并进行了展望。
关键词:果胶;果胶甲酯酶;果胶甲酯酶抑制子;细胞壁;逆境响应
中图分类号:S188+.3文献标志码: A
文章编号:1002-1302(2021)08-0049-07
收稿日期:2020-07-27
基金项目:国家自然科学基金(编号:31771775)。
作者简介:李佳颖(1993—),女,河南开封人,博士研究生,主要从事果胶甲酯化修饰对细胞壁结构影响研究。E-mail:jiayingli@webmail.hzau.edu.cn。
通信作者:王令强,博士,教授,主要从事植物细胞壁合成调控机理研究。E-mail:lqwang@gxu.edu.cn。
果胶是由半乳糖醛酸(D-galacturonic acids,D-Gal-A)以α-1,4-糖苷键连接形成的酸性杂多糖[1],它可以改变细胞的流变性与黏附性能,對植物组织形态、器官建成、果实发育以及激素和逆境响应等具有重要作用[2]。在工业中果胶也可作为食品添加剂、化妆品原料等被广泛用于食品、医疗保健化学化工行业。
根据侧链基团不同,果胶主要分为同聚半乳糖醛酸聚糖(HG)、鼠李Ⅰ型半乳糖醛酸聚糖(RG-Ⅰ)和鼠李Ⅱ型半乳糖醛酸聚糖(RG-Ⅱ)。HG是由半乳糖醛酸共价连接的线性长链,RG-Ⅰ是由α-1,2-L-鼠李糖与α-1,4-D-半乳糖醛酸结合的聚合体,RG-Ⅱ的复杂多糖组成则包括D-半乳糖、L-半乳糖、L-鼠李糖和D-木糖等至少12种单糖,并通过硼酯键形成二聚体[3]。
除了单糖组成的差异外,果胶还受到各种修饰。在高尔基体合成的果胶高度甲酯化,通常在HG的C-6位高度甲酯化,O-2、O-3位部分乙酰化[1,4]。果胶的甲酯度可达70%,乙酰化程度为40%~85%,不同组织器官间存在差异。高尔基体合成的高甲酯化的果胶经分泌小泡运输至细胞壁后,被果胶酶修饰,其中包括果胶甲酯酶(pectin methylesterases,PME)[3,5]。PME属于碳水化合物酯酶CE8家族,是果胶发生水解反应的第1个关键酶,目前发现和研究较多[6]。PME的活性可特异地被果胶甲酯酶抑制子(pectin methylesterases inhibitor,PMEI)调控。二者对于果胶的性质、维持细胞形态及中间层的稳定具有重要作用[7-8]。本研究综述PME、PMEI的结构和作用机制及其在植物生长发育中的作用。
1 PME和PMEI蛋白的结构与生化作用机制
1.1 PME和PMEI的序列和结构
根据N端有无PRO结构域(PF04043),PME可分为Group Ⅰ 和Group Ⅱ 2类。Group Ⅰ(type Ⅱ)无PRO,分子量27~45ku。Group Ⅱ (type Ⅰ)有 1~3个PRO,分子量52~105 ku[2]。对来自微生物和植物的127个PME氨基酸序列比对后发现,PME有5个特征序列片段(GxYxE、QAVAL、QDTL、DFIFG和LGRPW)[9]。
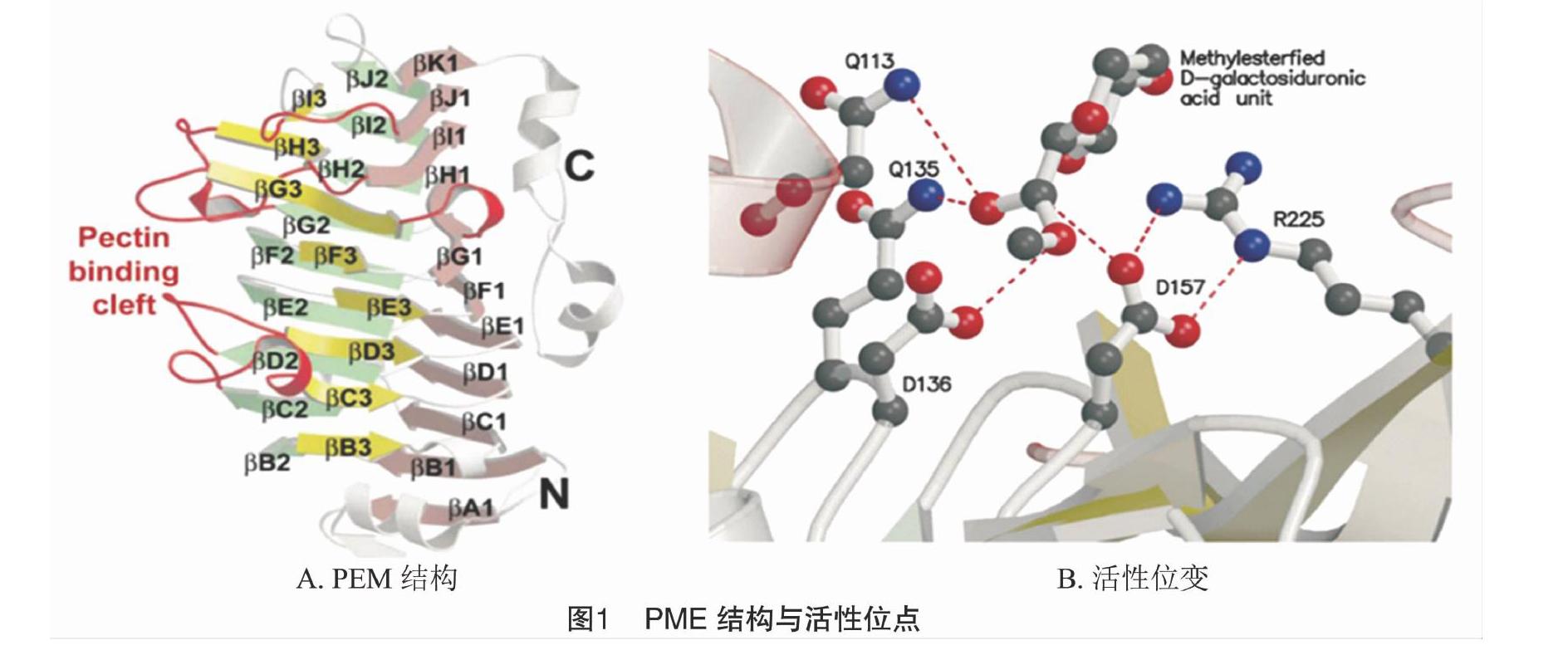
PME蛋白的三维结构在细菌中首次被解析,此后在植物中也被阐明(图1)[10-11]。番茄的PME为右旋β-螺旋蛋白,呈三股螺旋,其N端主要由一个α-螺旋与第1个β-折叠后的β-转角构成,C端由4个α-螺旋从与β-折叠侧面平行的β-转角处伸出,内部芳香族氨基酸残基形成较长的裂缝区域,为PME活性结合区域[12]。活性结合区域内的保守残基预测为Phe80、Tyr135、Phe156、Tyr218、Trp223和Trp248,在植物中具保守性,在菊欧文式菌(Erwinia chrysanthemi)中Tyr135、Phe156、Trp223仍具保守性[11-12]。Dorokhov等也证明了天冬氨酸残基(Asp136、Asp157)与谷氨酰胺残基(Gln113、Gln135)在PME活性位点中的作用[13]。从三维(3D)模型看,植物的PME间活性位点残基和序列相似度高,与细菌的PME有差异[2]。
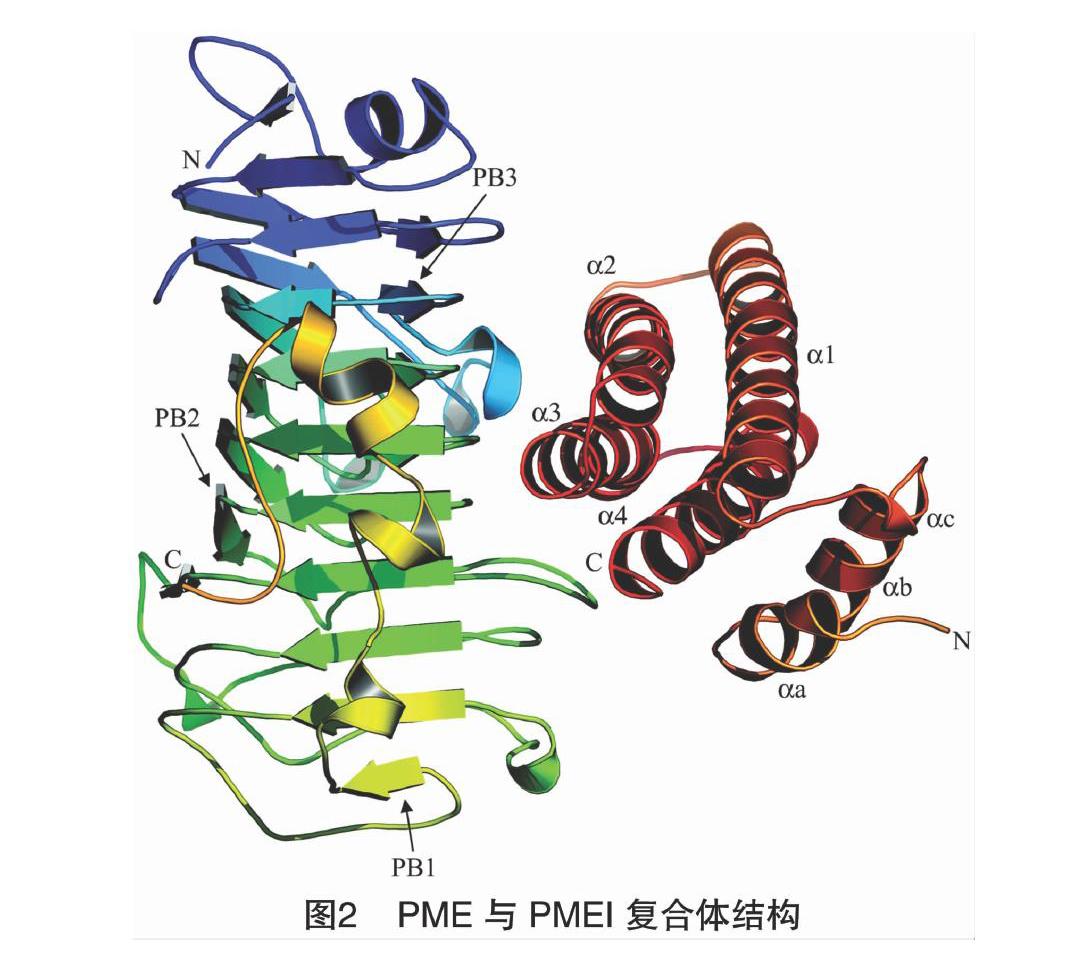
按蛋白的基序分类,PMEI属于隐马尔科夫模型PF04043。typeⅠ型PME中的PRO结构域与PMEI蛋白的序列具有相似性[7]。PMEI中保守性最强的为4个半胱氨酸残基位点,两两结合形成二硫键。猕猴桃PMEI的三维结构中,4个α螺旋(α1、α2、α3、和α4)反向平行排列形成螺旋束,5个半胱氨酸残基中前4个即Cys74、Cys114、Cys9和Cys18两两形成二硫键。其中,第1个二硫键与螺旋束内部的疏水作用对蛋白结构和稳定性起到支撑作用,第2个二硫键连接αa与αb,αa、αb与αc分别是构成N末端的3个短而弯曲的螺旋,与α1、α4构成的平面平行并延伸至外侧,N末端所构成的区域对PMEI蛋白结构的稳定性具有重要作用[14]。与PMEI均具有保守的半胱氨酸残基不同的是,PME 除了部分植物外,真菌和细菌中均未发现有保守性半胱氨酸残基[9]。
1.2 PME和底物的作用机制
PME的去甲脂化催化过程:PME中Asp225与Asp157二者以氢键结合,亲核攻击羧甲基酯键,与Phe160距离较近的Asp136充当质子供体,2个谷氨酰胺可以稳定带有负电荷的中间体,从而释放甲醇,后Asp136与H2O中的质子结合,致使催化底物与Asp157间氢键裂解,活性位点得以恢复(图1)[11]。PME与底物形成多聚嵌合结构,有3种作用方式:(1)多链机制(multiple-chain mechanism)。在多条HG链上,PME催化底物去甲酯化后与底物解离,每次只有1个甲酯化的残基被攻击。(2)单链机制(single-chain mechanism)。在单条HG链上,PME将甲酯化的基团连续去甲酯化。(3)多重攻击机制(multiple-attack mechanism)。PME对底物中多个甲酯化基团去甲酯化[15]。
PMEs的活性受温度、pH值等植物所处内外环境影响[16-17]。Denès等在苹果中发现,在pH值=7.0时,PME的作用机制为单链多重攻击机制,在pH值=4.5时,则为多链多重攻击机制[18]。Bonnin等用荧光标记对PME的扩散进行追踪发现,纤维素存在的凝胶结构中PME的扩散更加明显,且扩散能力受到PME活性调控[19]。
type Ⅰ 型PME中的PRO也和PME活性调节有关。PRO与PMEI蛋白序列相似,可作为分子内抑制子抑制PME活性,避免PME分泌至细胞壁前过早发挥去甲酯化作用;另外PRO还与PME在细胞壁上的锚定有关。研究表明,表达type Ⅰ型PME基因后,在生长发育前期可检测到酸性PRO结构域蛋白的活性,抑制PME活性防止果胶过早去甲酯化。而将PRO表达抑制后,PME活性积累增加,果胶的去甲酯化时期提前。再将PRO超表达后可恢复由于超表达PME引起的花粉管长度的改变[20-21]。
1.3 PMEI对PME的作用机制
PMEI主要结合PME的活性区域,与PME 以 1 ∶1 形成非共价可逆复合体(图2)[12]。PMEI与PME中和底物结合有关残基位点形成接触面,使底物无法进入PME活性区域。PME保守残基位点Phe80、Tyr135和Trp223与底物间的相互作用同时受到阻碍。复合体的结合区域可形成较多氢键,提高其稳定性。
PMEI和PME复合物的稳定性受pH值影响。酸性条件下的解离常数低于中性条件下的10倍。當处于接近生理pH值环境时,PMEI与PME间的亲和力最高。有趣的是,植物的PMEI对于真菌的PME抑制效果却不大。猕猴桃PMEI蛋白对植物PME活性有抑制作用,但对细菌和真菌的PME无抑制作用[7,22-24]。原因包括:一是细菌PME的活性区域裂缝较深,PMEI无法深入覆盖其活性区域;二是植物中的PME序列保守性较强,而真菌中无类似的保守性位点[25-26]。
PMEs的活性受PMEI调控已有较多的分子生物学研究。在拟南芥种皮黏液释放机制中,类枯草杆菌蛋白酶(subtilisin-like Ser protease,SBT)激活PMEI活性从而抑制PME活性[27]。转录因子LEUNIG_HOMOLOG/MUCILAGE MODIFIED1(LUH/MUM1)、SEEDSTICK(STK)与MYB52分别通过调控PMEI6、SBT1.7和PMEI14的表达而调控PME活性,影响种皮黏液释放[28-30]。SBT3.5和PME17表达模式一致,SBT3.5可靶向结合type Ⅰ型PME的2个碱性基序位点,对PME加工进而将其释放到胞质中[27]。
PMEI可同时对多个PME产生抑制作用,具体互作模式仍需深入研究。水稻的PMEI12和PMEI8与小麦的TaPMEI对于外源性的PME活性都有抑制作用[31];棉花中的GhPMEI3对GhPME13和GhPME2均有抑制作用[32]。拟南芥的AtPMEI4与AtPMEI9和猕猴桃的PMEI可对柑橘和番茄的PME表现出活性抑制。植物的PMEI对植物PME有抑制作用,但对真菌PME无影响[28,33-34]。
2 PME和PMEI基因家族和表达模式
植物中PME是一类较大基因家族,在拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)、亚麻(Linum usitatissimum)、杨树(Poplus trichocarpa)、番茄(Lycoperscicon esculentum)、梨(Pyrus bretschneideri)中已发现66、43、105、89、79、81个家族成员[35-37]。PMEI基因家族成员众多,目前在拟南芥、高粱(Sorghum bicolor)、亚麻、短柄草(Brachypodium distachyon)、甘蓝(Brassica oleracea)和芸薹属物种白菜(Brassica campestris ssp. chinensis)中鉴定的PMEI成员分别有79、54、55、95、38、95、100个[38-39]。
PME和PMEI家族成员具有特异表达模式,倾向于在幼嫩组织中表达。拟南芥的66个PME成员中,85%在花蕾发育期有表达,表明PME可能参与花粉发育及花粉管伸长。PMEI基因与PME类似,在花粉中的表达最高,在花药中表达数量最多[21]。部分PMEI基因存在组织特异表达,且大多在花、花药和幼根等幼嫩组织中表达。荠菜(Capsella rubella)与拟南芥等中的PMEI转录本数的比例相对较高,而与禾本科植物较低[35]。亚麻中有77.4%的PME在花蕾中有表达,在成熟组织中分别有19个PME及24个PMEI表达。在水稻中也发现部分PMEI基因的组织表达特异性,如OsPMEI28和OsPMEI49分别在幼根和花中特异表达[33]。PME和PMEI存在互作表达模式,在亚麻早期纤维发育中PMEI表达数量最少,活性最弱,自次生细胞壁合成开始至停止沉积期间PMEI的活性较高,调控PME活性[35]。
逆境可诱导PME与PMEI的表达。在茉莉酸甲酯(MeJA)、水杨酸(SA)、脱落酸(ABA)、过氧化物、干旱、乙烯和冷害等胁迫下,PME与PMEI的表达均变化[32]。Chen等研究发现,AtPMEI13与CbPMEI13受低温诱导,而受盐胁迫和ABA处理抑制[40]。在水稻43个PME基因中,有27个在细胞伸长期如营养生长期以及幼茎中有较高表达,且对逆境胁迫有不同表达响应[36]。水稻中不同的PMEI成员对逆境的响应在转录水平上存在调控差异[33]。
此外,研究发现,过氧化物酶与PMEI有类似的表达模式。Paynel等研究发现,镉诱导下细胞壁中果胶糖醛酸的共价交联增加且过氧化物酶活性和PME均有所增加[41]。在拟南芥中发现的PRX36与PMEI6均定位于细胞壁结构域,且存在共表达。最近,利用免疫荧光共振能量转移-荧光寿命成像,确定拟南芥中PMEI6和与其有共表达关系的过氧化物酶PRX36存在互作,PMEI介导的去甲酯化果胶平台在PRX36锚定细胞壁结构过程中具有重要作用[42]。这些与其他酶之间的互作表明PME与PMEI在植物生长发育过程中发挥作用的复杂性,其中关系值得进一步研究。
3 PME和PMEI对植物生长发育的影响
大量研究表明,PME和PMEI介导的果胶去甲酯化修饰对植物的生长发育有重要影响。果胶的去甲酯化与细胞壁硬度密切相关,去甲酯化的HG产生带负电的羧基基团可结合阳离子(如与Ca2+螯合)形成 “蛋箱结构”,或固定Al3+。细胞快速扩张时需要高甲酯化的果胶,当停止生长后在PMEs作用下,低甲酯化的果胶排列在细胞外壁使细胞壁硬度改变[35,43]。PME和PMEI的调控和互作,介导的果胶甲酯度改变,对植物细胞壁性质有重要影响,在植物种子萌发与花粉管发育、根部发育过程以及逆境响应中均扮演了重要角色(图3)[44]。
3.1 PME与PMEI影响种子萌发与花粉管发育
植物花粉管细胞壁主要由胼胝质和纤维素组成,顶端花粉管壁的组成则几乎完全是呈片层状的单层果胶。在花粉管中,果胶的去甲酯化使细胞壁变硬,在分生组织及叶片中,去甲酯化的果胶分布较多影响细胞壁延展性[45-47]。
PME与PMEI对花粉管发育有重要影响。Bosch等研究发现,在施加外源PME之后,烟草的发芽率与花粉管生长显著降低,生长速度与PME浓度成反比[21]。Zhang等将甘蓝BoPMEI1异源反义表达后发现,拟南芥花粉管伸长受到抑制,且部分表现为雄性不育和结实率下降。体外试验表明,AtPMEI2在花粉管顶端特异表达抑制PME活性,且抑制AtPME1活性,从而调节花粉管外壁稳定性[48]。在拟南芥的基因表达分析中,大量PME和PMEI特异地在花器官中表达,这与其功能是相一致的。
黏液释放是拟南芥种子在萌发过程中的重要过程,与去甲酯化的果胶有较大关联。PME58是首个被确定对种皮黏液释放有直接影响的PME基因。利用Ca2+螯合剂乙二胺四乙酸(EDTA)处理拟南芥种子,发现种皮周围黏液释放层扩大[49]。PMEI6是控制种皮黏液释放的关键基因,拟南芥PMEI6突变体中黏液释放的初生壁与次生壁的交联处变薄,使得种子在吸涨萌发后更易于将种皮黏液异常释放[42]。
3.2 PME和PMEI影响根部和果实发育
PME与PMEI介导的果胶去甲酯化影响细胞壁结构,对果实发育、软化、成熟过程均有影响。苹果果实成熟过程中,乙烯和低温可显著提高PME活性,加速果实软化。在番茄果实中,PME酶活性的降低对果实的果胶代谢产生影响,改变植物果实中可溶性糖和可溶性固形物含量[50]。此外,木质部中高度甲酯化的果胶为蔗糖转运提供运输通道,从而影响光合产物积累与碳源分配[51]。草莓转录组数据表明,FvPME38和FvPME39均在成熟期高度表达,且受到ABA调控,且二者的超表达植株与RNAi植株果实发育均受到影响[52]。果胶去甲酯化会影响根部发育,拟南芥中AtPME3参与不定根的形成,突变体的根部对Zn2+表现出敏感性。同时,萌发提前、根毛减少、种子黏液释放异常,且部分受体激酶以及GA相关基因表达下降,说明AtPME3参与调控了植物生长发育的多个过程[53]。研究发现,在缺磷时,粳稻日本晴中PME活性升高,难溶态磷释放增加,根尖中果胶去甲酯化程度与难溶态磷的活化程度趋势一致[54-55]。
3.3 PME和PMEI影响植物逆境响应
PME介导的去甲酯化过程中释放的甲醇可作为信号分子引起植物对逆境的响应。产生的质子引起胞内pH值的改变为细胞壁提供酸性环境,从而改变果胶降解酶如多聚半乳糖醛酸酶(polygalacturonase,PG)的活性。当植物遭受病原体攻击时,PGs作用HG的裂解产物寡聚半乳糖醛酸(oligogalacturonide,OGA)可作为信号分子诱发植物防御机制(图4)[56]。
在水稻中超表达OsPME14,植株根的生长受到抑制,表现出对铝胁迫的敏感性[57]。将水稻中PME活性降低后发现,细胞壁中铝含量降低[58]。Liu等研究发现,在GhPMEI3的基因沉默植株中,GhPME2、GhPME31与VdPG1的活性有所升高,抗病性减弱;而异源超表达植株下胚轴根毛增多,伸长区细胞形态改变,抗病性增强[32]。类似地,在AtPMEI2与AtPMEI3超表达植株中根长显著增加,半乳糖醛酸总含量未变,但甲酯化水平提高约16%,对灰霉的抗性增强。反之,AtPMEI10、AtPMEI11、AtPMEI12的突变体植株均表现灰霉病抗性降低[48]。将杨树PtoPME35在拟南芥异源超表达后,在甘露醇胁迫处理下可控制叶片气孔开合,进而调控植物抗逆性[59]。
4 展望
果胶影响细胞间的黏附性、流变性,其合成与修饰途径影响着植物根部发育、花粉管萌发、器官建成以及逆境响应。由果胶甲酯酶与果胶甲酯酶抑制子所介导的果胶去甲酯化修饰影响植物生长发育的多个过程,在种子萌发、花器官建成、根部与果实发育、逆境与胁迫响应及细胞壁结构中发挥重要作用。但在植物中,PME与PMEI均为多基因编码家族蛋白,在发挥作用过程中的冗余现象目前仍需进一步探讨。另外,果胶的本身性质受到PME和PMEI作用而改变,但越来越多的研究表明,果胶的改变造成整个细胞壁的性质和结构变化,而这一点在以前并没有得到足够的认识[60-61]。由于去甲酯化的果胶与其他多种物質存在交联,如果胶与细胞壁大分子纤维素间的交联主要与果胶的甲酯度有关。而PME与PMEI是否参与这些交联从而改变细胞壁结构影响植物生长发育值得研究。此外,PMEI可同时对多个PME产生活性抑制作用,对于PME的抑制作用是否皆为广谱性的抑制,其间的关联机制分析可为未来的研究提供方向。
参考文献:
[1]Atmodjo M A,Hao Z,Mohnen D. Evolving views of pectin biosynthesis[J]. Annual Review of Plant Biology,2013,64:747-779.
[2]Pelloux J,Rusterucci C,Mellerowicz E J. New insights into pectin methylesterase structure and function[J]. Trends in Plant Science,2007,12:267-277
[3]Mohnen D. Pectin structure and biosynthesis[J]. Current Opinion in Plant Biology,2008,11(3):266-277.
[4]Harholt J,Suttangkakul A,Scheller H V. Biosynthesis of pectin[J]. Plant Physiology,2010,153(2):384-395.
[5]Wolf S,Mouille G,Pelloux J. Homogalacturonan methyl-esterification and plant development[J]. Molecular Plant,2009,2(5):851-860.
[6]Jolie R P,Duvetter T,van Loey A M,et al. Pectin methylesterase and its proteinaceous inhibitor:a review[J]. Carbohydrate Research,2010,345(18):2583-2595.
[7]Giovane A,Servillo L,Balestrieri C,et al. Pectin methylesterase inhibitor[J]. Biochimica et Biophysica Acta -Proteins and Proteomics,2004,1696(2):245-252.
[8]Lionetti V,Cervone F,Bellincampi D. Methyl esterification of pectin plays a role during plant-pathogen interactions and affects plant resistance to diseases[J]. Journal of Plant Physiology,2012,169(16):1623-1630.
[9]Markovicˇ O,Janecˇek Sˇ. Pectin methylesterases:sequence-structural features and phylogenetic relationships[J]. Carbohydrate Research,2004,339(13):2281-2295.
[10]Kohli P,Kalia M,Gupta R. Pectin methylesterases:a review[J]. Journal of Bioprocessing & Biotechniques,2015,5(5):1.
[11]Johansson K,El-Ahmad M,Friemann R,et al. Crystal structure of plant pectin methylesterase[J]. FEBS Letters,2002,514(2/3):243-249.
[12]di Matteo A,Giovane A,Raiola A,et al. Structural basis for the interaction between pectin methylesterase and a specific inhibitor protein[J]. The Plant Cell,2005,17(3):849-858.
[13]Dorokhov Y L,Skurat E V,Frolova O Y,et al. Role of the leader sequence in tobacco pectin methylesterase secretion[J]. FEBS Letters,2006,580(13):3329-3334.
[14]Hothorn M,Wolf S,Aloy P,et al. Structural insights into the target specificity of plant invertase and pectin methylesterase inhibitory proteins[J]. The Plant Cell,2004,16(12):3437-3447.
[15]Grasdalen H,Andersen A K,Larsen B. NMR spectroscopy studies of the action pattern of tomato pectinesterase:generation of block structure in pectin by a multiple-attack mechanism[J]. Carbohydrate Research,1996,289:105-114.
[16]Cameron R G,Luzio G A,Goodner K,et al. Demethylation of a model homogalacturonan with a salt-independent pectin methylesterase from citrus:Ⅰ. Effect of pH on demethylated block size,block number and enzyme mode of action[J]. Carbohydrate Polymers,2008,71(2):287-299.
[17]Duvetter T,Fraeye I,Sila D N,et al. Mode of de-esterification of alkaline and acidic pectin methyl esterases at different pH conditions[J]. Journal of Agricultural and Food Chemistry,2006,54(20):7825-7831.
[18]Denès J M,Baron A,Renard C M G C,et al. Different action patterns for apple pectin methylesterase at pH 7.0 and 4.5[J]. Carbohydrate Research,2000,327(4):385-393.
[19]Bonnin E,Alvarado C,Crépeau M J,et al. Mobility of pectin methylesterase in pectin/cellulose gels is enhanced by the presence of cellulose and by its catalytic capacity[J]. Scientific Reports,2019,9(1):1-10.
[20]Micheli F. Pectin methylesterases:cell wall enzymes with important roles in plant physiology[J]. Trends in Plant Science,2001,6(9):414-419.
[21]Bosch M,Cheung A Y,Hepler P K. Pectin methylesterase,a regulator of pollen tube growth[J]. Plant Physiology,2005,138(3):1334-1346.
[22]Giovane A,Balestrieri C,Quagliuolo L,et al. A glycoprotein inhibitor of pectin methylesterase in Kiwi fruit:purification by affinity chromatography and evidence of a ripening‐related precursor[J]. European Journal of Biochemistry,1995,233(3):926-929.
[23]Jiang C M,Li C P,Chang J C,et al. Characterization of pectinesterase inhibitor in jelly fig (Ficus awkeotsang Makino) achenes[J]. Journal of Agricultural and Food Chemistry,2002,50(17):4890-4894.
[24]Dedeurwaerder S,Menu-Bouaouiche L,Mareck A,et al. Activity of an atypical Arabidopsis thaliana pectin methylesterase[J]. Planta,2009,229(2):311-321.
[25]梅曉宏,陈燕卉,高红岩,等. 果胶甲酯酶抑制剂的研究进展[J]. 食品科技,2008,33(6):64-68
[26]DAvino R,Camardella L,Christensen T M I E,et al. Tomato pectin methylesterase:modeling,fluorescence,and inhibitor interaction studies-comparison with the bacterial (Erwinia chrysanthemi) enzyme[J]. Proteins(Structure,Function,and Bioinformatics),2003,53(4):830-839.
[27]Sénéchal F,Graff L,Surcouf O,et al. Arabidopsis PECTIN METHYLESTERASE17 is co-expressed with and processed by SBT3. 5,a subtilisin-like serine protease[J]. Annals of Botany,2014,114(6):1161-1175.
[28]Sénéchal F,Mareck A,Marcelo P,et al. Arabidopsis PME17 activity can be controlled by pectin methylesterase inhibitor4[J]. Plant Signaling & Behavior,2015,10(2):e983351.
[29]Turbant A,Fournet F,Lequart M,et al. PME58 plays a role in pectin distribution during seed coat mucilage extrusion through homogalacturonan modification[J]. Journal of Experimental Botany,2016,67(8):2177-2190.
[30]Shi D,Ren A,Tang X F,et al. MYB52 negatively regulates pectin demethylesterification in seed coat mucilage[J]. Plant Physiology,2018,176(4):2737-2749.
[31]Hong M J,Kim D Y,Lee T G,et al. Functional characterization of pectin methylesterase inhibitor (PMEI) in wheat[J]. Genes & Genetic Systems,2010,85(2):97-106.
[32]Liu N N,Sun Y,Pei Y K,et al. A pectin methylesterase inhibitor enhances resistance to Verticillium wilt[J]. Plant Physiology,2018,176(3):2202-2220.
[33]Nguyen H P,Jeong H Y,Kim H,et al. Molecular and biochemical characterization of rice pectin methylesterase inhibitors (OsPMEIs)[J]. Plant Physiology and Biochemistry,2016,101:105-112.
[34]Hocq L,Pelloux J,Lefebvre V. Connecting homogalacturonan-type pectin remodeling to acid growth[J]. Trends in Plant Science,2017,22(1):20-29.
[35]Pinzón-Latorre D,Deyholos M K. Characterization and transcript profiling of the pectin methylesterase (PME) and pectin methylesterase inhibitor (PMEI) gene families in flax (Linum usitatissimum)[J]. BMC Genomics,2013,14(1):742.
[36]Jeong H Y,Nguyen H P,Lee C. Genome-wide identification and expression analysis of rice pectin methylesterases:implication of functional roles of pectin modification in rice physiology[J]. Journal of Plant Physiology,2015,183:23-29.
[37]Tang C,Zhu X X,Qiao X,et al. Characterization of the pectin methyl-esterase gene family and its function in controlling pollen tube growth in pear (Pyrus bretschneideri)[J]. Genomics,2020,112(3):2467-2477.
[38]Ren A,Ahmed R I,Chen H,et al. Genome-wide identification,characterization and expression patterns of the pectin methylesterase inhibitor genes in Sorghum bicolor[J]. Genes,2019,10(10):755.
[39]Liu T,Yu H,Xiong X,et al. Genome-wide identification and characterization of pectin methylesterase inhibitor genes in Brassica oleracea[J]. International Journal of Molecular Sciences,2018,19(11):3338.
[40]Chen J,Chen X H,Zhang Q F,et al. A cold-induced pectin methyl-esterase inhibitor gene contributes negatively to freezing tolerance but positively to salt tolerance in Arabidopsis[J]. Journal of Plant Physiology,2018,222:67-78.
[41]Paynel F,Schaumann A,Arkoun M,et al. Temporal regulation of cell-wall pectin methylesterase and peroxidase isoforms in cadmium-treated flax hypocotyl[J]. Annals of Botany,2009,104(7):1363-1372.
[42]Francoz E,Ranocha P,Le Ru A,et al. Pectin demethylesterification generates platforms that anchor peroxidases to remodel plant cell wall domains[J]. Developmental Cell,2019,48(2):261-276.
[43]Carpin S,Crèvecoeur M,de Meyer M,et al. Identification of a Ca2+-pectate binding site on an apoplastic peroxidase[J]. The Plant Cell,2001,13(3):511-520.
[44]Wormit A,Usadel B. The multifaceted role of pectin methylesterase inhibitors (PMEIs)[J]. International Journal of Molecular Sciences,2018,19(10):2878.
[45]Palin R,Geitmann A. The role of pectin in plant morphogenesis[J]. Biosystems,2012,109(3):397-402.
[46]Hongo S,Sato K,Yokoyama R,et al. Demethylesterification of the primary wall by PECTIN METHYLESTERASE35 provides mechanical support to the Arabidopsis stem[J]. The Plant Cell,2012,24(6):2624-2634.
[47]Qi J,Wu B B,Feng S L,et al. Mechanical regulation of organ asymmetry in leaves[J]. Nature Plants,2017,3(9):724-733.
[48]Lionetti V,Fabri E,de Caroli M,et al. Three pectin methylesterase inhibitors protect cell wall integrity for Arabidopsis immunity to Botrytis[J]. Plant Physiology,2017,173(3):1844-1863.
[49]Turbant A,Fournet F,Lequart M,et al. PME58 plays a role in pectin distribution during seed coat mucilage extrusion through homogalacturonan modification[J]. Journal of Experimental Botany,2016,67(8):2177-2190.
[50]Tieman D M,Harriman R W,Ramamohan G,et al. An antisense pectin methylesterase gene alters pectin chemistry and soluble solids in tomato fruit[J]. The Plant Cell,1992,4(6):667-679.
[51]Xu Y H,Sechet J,Wu Y B,et al. Rice sucrose partitioning mediated by a putative pectin methyltransferase and homogalacturonan methylesterification[J]. Plant Physiology,2017,174(3):1595-1608.
[52]Xue C,Guan S C,Chen J Q,et al. Genome wide identification and functional characterization of strawberry pectin methylesterases related to fruit softening[J]. BMC Plant Biology,2020,20(1):1-17.
[53]Guénin S,Hardouin J,Paynel F,et al. AtPME3,a ubiquitous cell wall pectin methylesterase of Arabidopsis thaliana,alters the metabolism of cruciferin seed storage proteins during post-germinative growth of seedlings[J]. Journal of Experimental Botany,2017,68(5):1083-1095.
[54]趙旭升,朱晓芳,吴 启,等. 水稻根系果胶去甲酯化促进细胞壁磷再利用的机制探究[J]. 土壤学报,2018,55(5):1190-1198.
[55]Zhu X F,Wang Z W,Wan J X,et al. Pectin enhances rice (Oryza sativa) root phosphorus remobilization[J]. Journal of Experimental Botany,2015,66(3):1017-1024.
[56]Duan W K,Huang Z N,Song X M,et al. Comprehensive analysis of the polygalacturonase and pectin methylesterase genes in Brassica rapa shed light on their different evolutionary patterns[J]. Scientific
Reports,2016,6:25107.
[57]Yang X Y,Zeng Z H,Yan J Y,et al. Association of specific pectin methylesterases with Al‐induced root elongation inhibition in rice[J]. Physiologia Plantarum,2013,148(4):502-511.
[58]Zhu C Q,Cao X C,Bai Z G,et al. Putrescine alleviates aluminum toxicity in rice (Oryza sativa) by reducing cell wall Al contents in an ethylene‐dependent manner[J]. Physiologia Plantarum,2019,167(4):471-487.
[59]Yang W,Ruan M,Xiang M,et al. Overexpression of a pectin methylesterase gene PtoPME35 from Populus tomentosa influences stomatal function and drought tolerance in Arabidopsis thaliana[J]. Biochemical and Biophysical Research Communications,2020,523(2):416-422.
[60]Chung D,Pattathil S,Biswal A K,et al. Deletion of a gene cluster encoding pectin degrading enzymes in Caldicellulosiruptor bescii reveals an important role for pectin in plant biomass recalcitrance[J]. Biotechnology for Biofuels,2014,7(1):147.
[61]Biswal A K,Atmodjo M A,Li M,et al. Sugar release and growth of biofuel crops are improved by downregulation of pectin biosynthesis[J]. Nature Biotechnology,2018,36(3):249-257.
