甲状腺乳头状癌预后标志物的研究
2021-05-13罗锦琳张超杰邹联洪范培芝
陈 帅,罗锦琳,罗 霁,张超杰,邹联洪,3,范培芝
(1.湖南师范大学第一附属医院/湖南省人民医院,长沙 410002;2.湘雅常德医院,常德 415003;3.急危重症代谢组学湖南省重点实验室,长沙 410002)
甲状腺癌是内分泌器官最常见的恶性肿瘤,其发病率在过去三十年间增加了三倍多[1][2],虽然甲状腺乳头状癌(thyroid papillary carcinoma,PTC)患者可获得良好的预后[3],但仍有一部分的甲状腺乳头状癌的患者会发生复发和远处转移[4]。目前有许多关于疾病的生物信息学研究,但有关PTC 的生物信息学研究却不多见,固在本研究中,我们期望通过生信的方法来初步探索PTC 在分子水平的机制研究。
1 资料与方法
临床资料:在2016 年9 月~2018 年9 月就诊于湖南省人民医院诊断为PTC 的患者中随机选取3 位患者,在术中收集PTC 患者的甲状腺乳头状癌组织及相对应的癌旁组织标本(距肿瘤大于2cm),我们将其收取的标本用于转录组芯片的相关研究。本研究收集相关标本均得到湖南省人民医院伦理委员会的批准同意,患者或其法定监护人对本次研究方法及内容均知情同意,并签字认可。所有的患者病理和临床相关资料均齐全。

表1 甲状腺乳头状癌患者一般临床资料
1.1 转录组芯片检测 用Trizol 试剂组织RNA 试剂盒分别从冷冻组织中提取总RNA。采用基因芯片技术对基因化学进行LncRNA、mRNA 分析。简单地说,将rRNA 去除后,制备生物素化Arna(CrNA)。得到原始视图人类基因表达阵列(CAT)。再进行杂交和扫描。所有样品均经技术复制件处理。基因芯片扫描仪GeneChip Scanner 3000 7G 用于扫描完成的阵列。图像用基因芯片命令控制台(AGACC)提取,用表达式控制台软件进行分析。
1.2 LncRNAs 和差异基因筛选 将获取的基因芯片数据进行标准化处理,使用差异倍数法筛选出相关差异表达的lncRNAs 和mRNAs,以Fold-chang ≥2,P 值≤0.05,作为筛选符合差异表达的入组条件。
1.3 差异基因的GO 功能富集分析及KEGG 通路富集分析 通过DAVID 在线分析工具对甲状腺乳头状腺癌差异表达基因进行基因本体论(Gene Ontology,GO)功能富集分析识别基因谱的分子功能和京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)通路富集分析探讨这些基因在这些路径中的潜在功能。P<0.05 为差异有统计学意义。
1.4 差异基因蛋白互作网络分析 通过STRING 数据库(http://string-db.org/)对差异基因进行蛋白互作网络分析(protein protein interaction network,PPI),找出在PTC 中作用的关键基因及关键通路,初步探讨PTC的相关分子机制,为PTC 的治疗和预后评估提供理论依据。
1.5 TGCA 数据采集及处理 采用癌症基因组图谱(TGCA)数据库中检索了具有临床信息和基因表达谱的PTC 病例。使用R 包“OIsurv”将基因的表达水平定义为为高表达值(表达值>中值)或低表达值(表达值<中值)。采用Kaplan-Meier 法进行生存曲线分析。采用对数秩检验进行单因素生存分析。
1.6 统计学分析 统计学处理采用SPSS 19.0 软件,采用双面对数秩检验(Kaplan-Meier)来分析15 个基因在TGCA 数据库中PTC 临床数据样本中的总体生存率(OS)及无名生存期(DFS)差异的统计学意义。P 值<0.05 具有统计学意义.
2 结果
2.1 甲状腺乳头状癌中差异表达的基因 为了分析PTC 中差异表达的LncRNAs,我们对3 例PTC 和与之相匹配的癌旁正常甲状腺组织中筛选出的差异mRNA和Lnc RNA 进行了基因组分析。根据筛选出的差异表达基因芯片数据,我们发现228 个Lnc RNA 在PTC和配对非癌组织之间有差异表达(差异倍数≥2.0,P<0.05)。其中207 个LncRNAs 下调,21 个LncRNAs上调。n336302 所对应的lncRNA(差异倍数:26.32)是上调最大的LncRNA,n407932 所对应的lncRNA 是下调最大的lncRNAs(差异倍数:263.1579),结果如图1A、B。
同样,我们采用差异倍数≥2.0,P<0.05 的标准筛选甲状腺乳头状癌的差异表达基因,发现共有364 个mRNA 在PTC 和成对癌旁非癌组织中差异表达。其中,有82 个mRNA 表达上调,282 个mRNA 表达下调。上调的差异基因有FN1、MXRA5、DUSP6、TGFβR1、CTGF,其中纤维连接蛋白1(Fibronectin1,FN1)是上调差异基因中差异倍数最大的基因(FC=94.12)。下调的差异基因有TPO、GOLGA8Q、FCGBP、KIT、PLA2R1等,TPO(甲状腺过氧化物酶)是其中下调最明显的基因(FC=400),结果如图1C、D。
2.2 差异基因的GO 功能富集分析及KEGG 通路分析 差异基因在GO 分析中分为三个功能群:生物过程、分子功能及细胞成分。根据KFGG 通路分析,在筛选的差异表达基因中共有13 条通路富集,分别是“小细胞肺癌”、 “甲状腺激素合成”、胰液分泌”、“TGF-β信号通路”、“补体和凝血级联”、“色氨酸代谢”、“蛋白多糖在癌症中的作用”、“胃酸分泌”、“胰高血糖素信号”、“ECM-受体相互作用”、“矿物吸收”、“焦点粘连”和“金葡菌感染”(图1E)。差异基因在生物过程中主要富集于“负增长调节”。“蛋白酶结合”在分子功能中显著富集。在细胞组成中,差异基因主要富集在“细胞外小体”(图1F)。
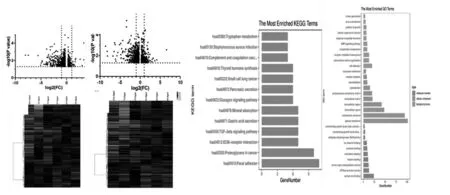
图1 甲状腺乳头状癌中差异表达的基因A表示差异LncRNAs的火山图,B为差异LncRNAs的热图,C表示筛选出的差异基因的火山图,D表示差异基因的热图,火山图显示差异基因的表达,log 2值(折叠变化)>1.5,P<0.05。E表示KEGG通路排名前十的富集图,F表示上调差异基因GO功能分析各条目前5位
2.3 差异基因蛋白互作网络分析 我们使用STRING在线分析软件进一步分析差异表达基因中的关键基因,将364 个差异表达基因进行在线分析,得出如下差异基因的蛋白共表达网络图。筛选条件:score ≥0.4。并且score 得分越高表明两基因间的相互作用越强(图3A)。为进一步筛选出作用的关键基因,我们选取了score 得分最高,即相互作用最强基因,ATP6V1A、TGFβR1、BCL2、SPARC、CFH、ATP6V1F、ITGβ1、FN1、ITGA8、KCNQ1、CFI、SMAD9、BID、ITGAV 及KCNJ2 等15 个关键基因(图2 B)。TGCA 数据库中进行生存分析显示,对DFS 有差异的是FN1(图3 A)、SPARC(图3 B);与OS相关的是TGFβR1(图3 4C)、SMAD9(图3 D)。
3 讨论
PTC 是世界上最常见的内分泌相关癌症之一,其发病率在过去的近30 年里几乎增加两倍[5]。有研究表明,TERT 突变、BRAF V600E 突变和RET/PTC 重排等遗传变异都是肿瘤增殖和转移的原因之一[6-8]。PTC 的确切发病机制仍不清楚。因此,识别和鉴定与PTC 相关的关键基因具有十分重要的意义。在目前的研究中,我们的结果提供了一个全面的生物信息学分析的基因和途径,可能参与了PTC 的进展,我们将筛选出的4 个基因进行简要介绍。
纤维连接蛋白1(FN1)参与细胞的粘附和迁移过程,包括胚胎发生、伤口愈合、凝血、宿主防御、转移,并参与各种生化过程[9]。FN1 在多种肿瘤中表达,既是促癌基因又是抑癌基因。同时研究显示,在甲状腺乳头状癌中,FN1 促进甲状腺乳头状癌的增殖、粘附、迁移和侵袭[7];FN1 的过表达是甲状腺癌侵袭性的重要因素,我们研究同样显示,FN1 在甲状腺乳头状癌组织中表达是上调的,生存分析提示,高表达的FN1 与甲状腺乳头状癌的预后正相关,提示FN1 在甲状腺乳头状癌中发挥致癌基因的作用,FN1 是甲状腺乳头状癌的一个潜在靶点。
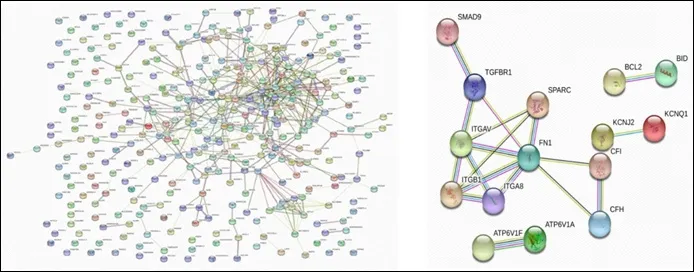
图2 A图为PTC患者中相关差异表达基因蛋白共表达的网络图,B图为STRING在线软件筛选的15个关键基因的蛋白共表达网络图
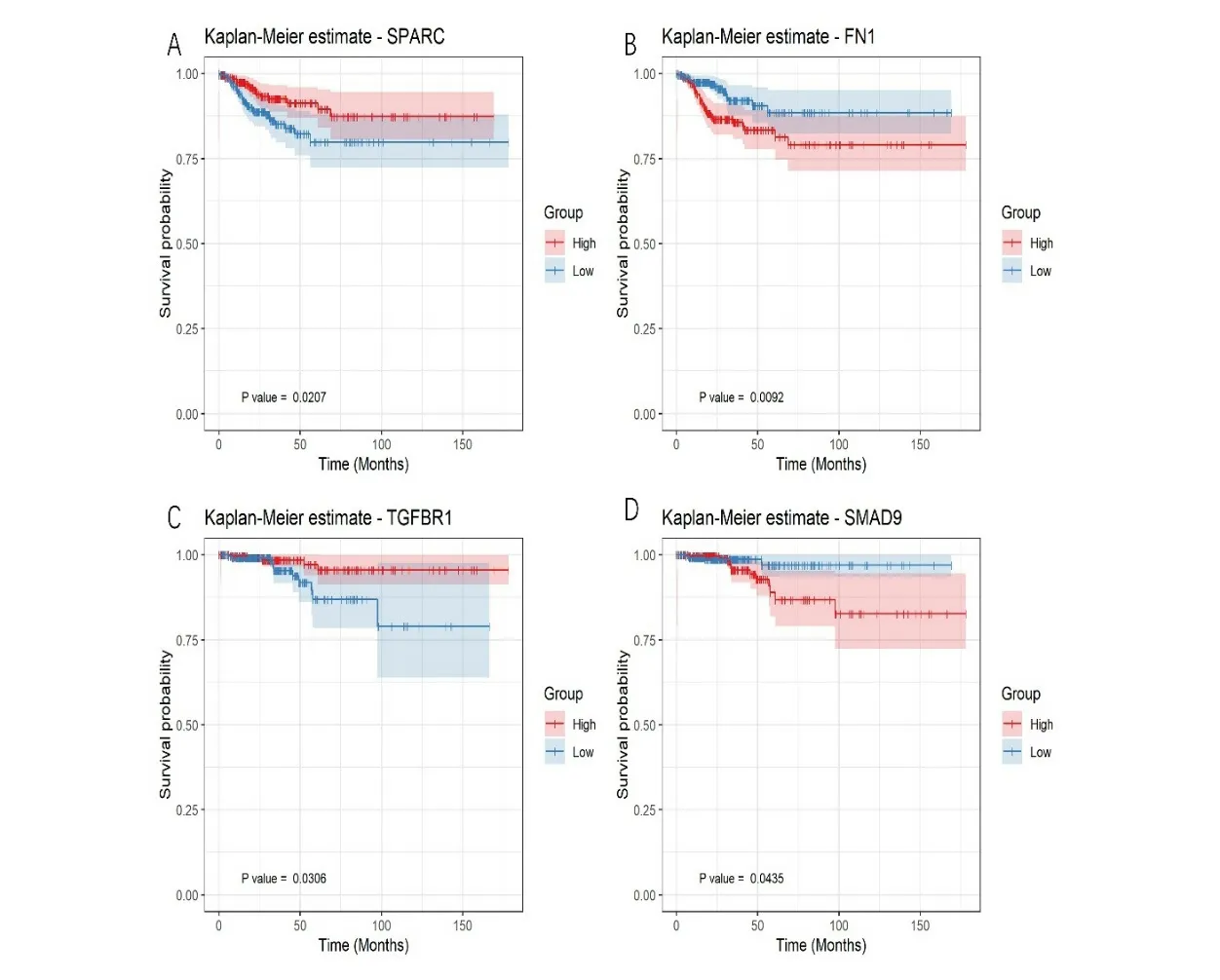
图3 根据Kaplan-Meier估计的TCGA数据
基质细胞蛋白SPARC(分泌蛋白酸性,富含半胱氨酸,又称骨连接蛋白或BM-40)是一种胶原结合蛋白,具有诱导细胞聚集和影响细胞增殖的功能[10]。SPARC在肿瘤细胞的转移和侵袭中发挥着重要作用,是一种多面蛋白。SPARC 在甲状腺癌中的报道较少,本研究生存分析显示高表达SPARC 的甲状腺乳头状癌患者具有更长的生存预后,提示SPARC 在甲状腺乳头状癌中发挥抑癌基因的作用,但其具体机制仍有待进一步验证。
转化生长因子β(Transforming Growth Factor-β,TGF-β)信号通路在调节细胞生长、分化、发育中具有重要的作用,与肿瘤的侵袭和转移密切相关。TGFβR1 在TGFβ信号通路传导中起着关键的作用。在甲状腺癌中,TGF-β1 诱导高迁移率组A(HMGA 1)使得促进甲状腺癌的增殖及侵袭。通过显微解剖中央和浸润区肿瘤组织的寡核苷酸芯片分析发现人乳头状甲状腺癌组织中TGF-β1 的表达高于正常甲状腺组织[11]。
我们对甲状腺乳头状癌差异基因的信号通路富集分析结果同样提示TGF-β信号通路参与了甲状腺腺癌的发生发展。TGF-β1、SMAD9 在甲状腺乳头状癌中表达上调,生存分析提示与SMAD9 与TGF-β1 甲状腺乳头状癌的总生存(OS)相关。
