黑曲霉3.316β-葡萄糖苷酶底物亲和性研究
2021-04-16侯林燕朱凤妹
侯林燕,朱凤妹
(河北科技师范学院食品科技学院,河北秦皇岛066600)
β-葡萄糖苷酶(β-D-glucosidase),因其能将结合于非还原性末端的β-D-葡萄糖苷键水解,所以也被称为β-D-葡萄糖苷葡萄糖水解酶。它属于纤维素酶系的一种,能够水解纤维二糖来减少纤维素水解过程中的可逆限制。该酶可以从微生物和动植物中获得,比如细菌[1]、真菌[2]、霉菌[3]、水果、龙井茶叶[4-5]、昆虫[6]等。来源不同的β-葡萄糖苷酶所呈现的蛋白质结构存在差异,从而导致β-葡萄糖苷酶的酶学性质不同。根据酶作用底物的糖苷键的不同可以将β-葡萄糖苷酶分成只水解烃基-β-葡萄糖苷键的酶、只水解芳香基-β-葡萄糖苷键的酶和既能水解烃基-β-葡萄糖苷又能水解芳香基-β-葡萄糖苷[7-8]的酶。底物种类的不同与β-葡萄糖苷酶发生反应时的连接方式也不同。通常采用分子对接了解小分子配体与大分子受体的连接方式[9-11],分子对接的常用软件有AutoDock、LeDOCK、MOE Dock 等[12-14],可视化软件有MOE[15]、Pymol[16]等。本次试验采用分子对接了解3 种底物与β-葡萄糖苷酶的连接方式,从分子角度找到两者结合稳定的原因,为β-葡萄糖苷酶结构性质的理论研究提供了一定的参考价值。
1 材料与方法
1.1 菌种与试剂
黑曲霉(Aspergillus niger 3.316):中国菌种保藏中心;4-硝基苯基-β-D-吡喃葡萄糖苷(4-nitrophenyl beta-D-glucopyranoside,pNPG):上海华蓝化学科技有限公司;葡萄糖、3,5-二硝基水杨酸(3,5-dinitrosalicylic acid,DNS)试剂:上海羽朵生物科技有限公司;纤维二糖:MERCK 公司;水杨苷:RHAWN 公司;对硝基苯酚(p-nitrophenol,pNP):武汉克米克生物医药技术有限公司;以上试剂均为分析纯。
1.2 底物与酶的分子对接
在能力最小化基础上将二维结构转化为三维结构,利用LigX 优化靶的质子化状态和氢的取向,残基Asp280 附近是结合区域。AMBER10 ∶EHT 的力场和反应场(R 场)的隐式溶剂化模型用于对接前,采用半柔性对接方式,对位置进行约束,但允许受体口袋的侧链根据配体构象移动。侧链原子束缚到原始位置的重量为10,过渡态由London dG 评分,对前30 个复合物过渡态进行力场细化,然后对GBVI/WSA dG 重新评分,结果以MOE 软件可视化。
1.3 酶活测定方法
DNS 法测定酶活性[17]:配制0.02 mmol/L,pH 4.5 的水杨苷溶液,取2 mL 底物溶液与1 mL 酶液于60 ℃水浴,加入DNS 试剂1.5 mL,沸水浴5 min 终止反应,测定OD540值。将灭活的酶液作为空白对照处理。
pNPG 酶活测定方法[18]:50 μL pNPG 的溶液加入1.5 mL 的pH 4.5 的缓冲液,在60 ℃下预热10 min。加入500 μL 的酶液,反应10 min,取410 μL 混合液,加入490 μL 的1 mol/L Na2CO3终止反应,测定OD400值。
纤维二糖测定方法[19]:取pH 4.5,浓度为4 mmol/L的纤维二糖溶液70 μL 加入酶液70 μL,于60 ℃下发生反应,加入2.1 mL 的葡萄糖氧化酶生化分析显色液,反应10 min,测定OD520值。
1.4 标准曲线的测定
1.4.1 DNS 法葡萄糖标准曲线的测定
测定还原糖采用DNS 比色法,将不同浓度的葡萄糖溶液(mg/mL)在540 nm 波长下测量OD 值。
1.4.2 pNP 标准曲线的测定
测定pNP 采用pNPG 法,将不同浓度的pNP(mmol/L)于400 nm 波长处测量OD 值。
1.4.3 葡萄糖氧化酶法葡萄糖标准曲线的测定
采用葡萄糖氧化酶法测定葡萄糖标准曲线,将不同浓度的葡萄糖溶液(mg/mL)在500 nm 波长下测量OD 值。
1.5 动力学参数测定
分别配制不同浓度的底物溶液,其中水杨苷浓度为0.01、0.02、0.03、0.04、0.05 mol/L;pNPG 的浓度为1、2、5、7、10 mmol/L;纤维二糖的浓度为1、2、3、4、5 mmol/L;按1.3 酶活测定方法,酶与底物混合液在pH 4.5,温度为60 ℃条件下反应,每隔2 min,加入各自对应的反应终止剂,分别测定OD540值、OD400值、OD520值。根据Lineweaver-Burk 双倒数作图法求3 种底物的动力学参数。
2 结果与分析
2.1 标准曲线的测定结果
以葡萄糖浓度作为横坐标,以吸光度值为纵坐标做标准曲线图,得到的标准曲线方程为:y=0.431 5x-0.020 8(R2=0.994 4);以pNP 浓度作为横坐标,以吸光度值为纵坐标做标准曲线图,得到的标准曲线方程为:y=6.594 4x-0.002 1(R2=0.998 8)。以葡萄糖浓度为横坐标,以吸光度值为纵坐标,得到标准曲线方程为:y=0.012x+2.213 6(R2=0.998 7)。
2.2 3 种底物与β-葡萄糖苷酶对接结果
2.2.1 纤维二糖与β-葡萄糖苷酶对接结果
纤维二糖与β-葡萄糖苷酶对接结果见图1。

图1 β-葡萄糖苷酶与纤维二糖的对接复合物Fig.1 Complex of β-glucosidase with cellobiose
如图1A 所示,黑色圈内的环状结构物为纤维二糖,其余部分为β-葡萄糖苷酶。结果显示纤维二糖在预定的结构“活性凹槽处”[20]与酶连接。利用MOE 软件得到两分子连接的二维结构图。如图1B 所示,与纤维二糖对接时,存在3 个氢键和3 个H-π 共轭。两个氢键是由纤维二糖作为氢供体与Asp280 侧链形成的,另一个氢键是由纤维二糖作为氢供体与Asp92 形成的。2 个H-π 共轭是Tyr511 同时与纤维二糖形成,另一个由Trp281 与纤维二糖形成。
2.2.2 水杨苷与β-葡萄糖苷酶对接结果
水杨苷与β-葡萄糖苷酶对接结果见图2。
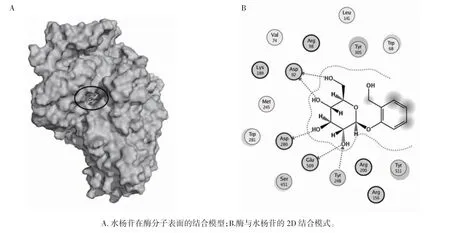
图2 β-葡萄糖苷酶与水杨苷的对接复合物Fig.2 Complex of β-glucosidase with salicin
如图2A 所示,黑色圈内的环状结构物为水杨苷,其余部分为β-葡萄糖苷酶。结果显示水杨苷在预定的结构活性凹槽处与酶连接。二维结构如图2B 可知,在对接时,存在4 个氢键,Asp92 作为氢受体与水杨苷相连2 个氢键,Asp280 作为氢受体与水杨苷相连1 个氢键,Glu509 作为氢受体和Tyr248 作为氢供体与水杨苷上的同一个羟基形成氢键。
2.2.3 pNPG 与β-葡萄糖苷酶对接结果
pNPG 与β-葡萄糖苷酶对接结果见图3。
如图3A 所示,黑色圈内的环状结构物为pNPG,其余部分为β-葡萄糖苷酶。结果显示pNPG 在预定的结构活性凹槽处与酶连接。二维结构如图3B,在对接时存在4 个氢键,Asp92 和Ser451 侧链与底物不同羟基位点处分别形成一个氢键、Glu509 作为氢受体和Tyr248 作为氢供体同时与pNPG 上的羟基形成一个氢键。

图3 β-葡糖苷酶与pNPG 的对接结果Fig.3 Complex of β-glucosidase with pNPG
2.2.4 3 种底物与β-葡萄糖苷酶对接分数
3 种底物与β-葡萄糖苷酶对接分数见表1。
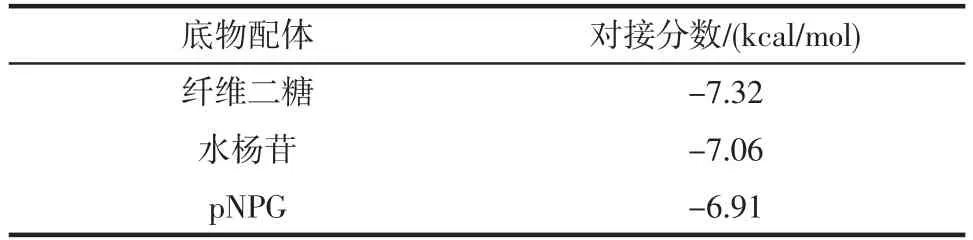
表1 3 种底物与β-葡萄糖苷酶的对接分数Table 1 The docking score of three substrate with β-glucosidase
由表1 可知,纤维二糖的能量分数最小,在3 种底物与酶复合物中属于最稳定的复合状态,也就说明纤维二糖与酶的结合力在三者中最强。结合能量的大小与结合时形成的氢键和H-π 共轭有关,其中H-π 共轭即芳香氢键在对多肽和蛋白质稳定方面、提高底物的亲和力和催化效率起着重要作用[21]。这可能是纤维二糖较水杨苷和pNPG 作为底物时与酶的复合物相对稳定的原因。
2.3 动力学参数测定
2.3.1 纤维二糖与β-葡萄糖苷酶动力学方程
以纤维二糖为底物时,以底物浓度(S)的倒数1/[S]作为横坐标,以反应速度(V)的倒数1/V 为纵坐标,得到纤维二糖为底物时的动力学方程结果如图4 所示。
2.3.2 水杨苷与β-葡萄糖苷酶动力学方程
以水杨苷为底物时,根据Lineweaver-Burk 双倒数作图,计算所得到的动力学方程结果如图5 所示。
2.3.3 pNPG 与β-葡萄糖苷酶动力学方程
以pNPG 为底物时,根据根据Lineweaver-Burk 双倒数作图,计算得到的动力学方程结果如图6 所示。
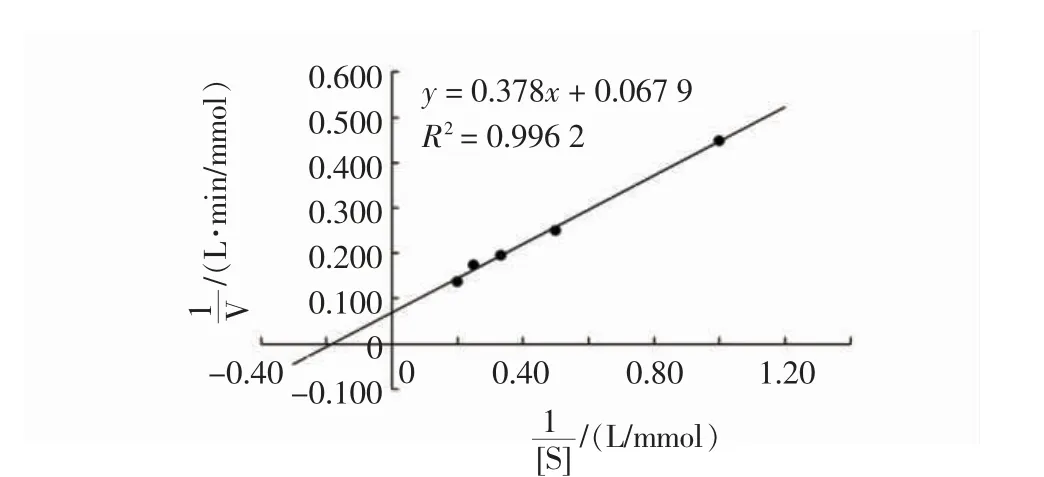
图4 纤维二糖的动力学方程Fig.4 Kinetic equation of cellobiose
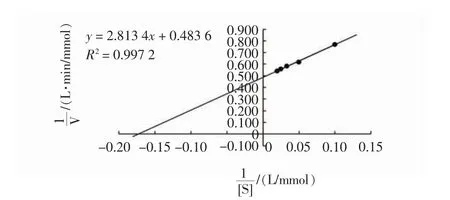
图5 水扬苷的动力学方程Fig.5 Kinetic equation of salicin
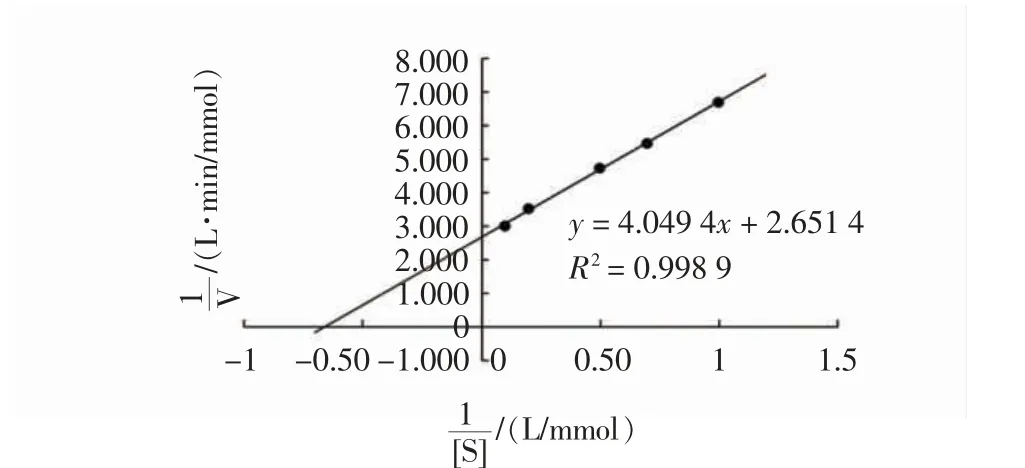
图6 pNPG 的动力学方程Fig.6 Kinetic equation of pNPG
根据3 个方程计算可得,纤维二糖、水杨苷和pNPG 3 种底物所对应的Km分别为5.567、5.820、1.527 mmol/L,则Km最小的为pNPG,这表明酶对pNPG 的亲和力明显高于酶对纤维二糖和水杨苷的亲和力;最大反应速率由高到低依次为66.138、8.886 和1.013 μmol/(min·mg),则酶与纤维二糖为底物时的最大反应速率是酶与水杨苷和酶与pNPG 最大反应速率的7.44 倍和65.28倍;纤维二糖、水杨苷和pNPG 3 种底物所对应的催化效率分别为14.728、2.068 和0.377,这表明当以纤维二糖为底物时有最大的催化效率,且分别是以水杨苷和pNPG 为底物时的7.12 倍和39.07倍。
3 结论与讨论
本研究采用分子对接将3 种β-葡萄糖苷酶的底物与该酶进行对接,从三维结构上初步了解β-葡萄糖苷酶与底物分子接触发生反应时的连接方式,并通过动力学参数进行了试验验证。结果发现3 种底物均在β-葡萄糖苷酶的凹槽处进行了对接,其中在与水杨苷和pNPG 对接时,活性位点相邻的Ser451、Tyr248、Asp92 和Asp98 会与底物形成氢键网络。有的一个氨基酸与底物的两个位点结合生成氢键,有的两个氨基酸侧链与底物的一个位点同时生成氢键,产生这样的结果可能与不同氨基酸所带电荷数以及所形成氢键类型有关[22-25];在与纤维二糖对接时,除氨基酸与底物形成的3 个氢键外,生成了3 个H-π 共轭,由于阳离子-π 能够增强周围形成氢键的氨基酸侧链的成键能力[26],进而影响了酶的催化效率和反应速率。
通过对β-葡萄糖苷酶进行动力学参数分析可知,pNPG 最接近β-葡萄糖苷酶天然底物,纤维二糖为底物时,复合物结构最为稳定,催化效率以及反应速率也最高,这结果符合上述分子对接结果。本次试验结果找到了β-葡萄糖苷酶的最适底物,并从分子方面解释了底物与酶复合物连接的方式,同时通过试验验证了分子对接的可靠性,为以后β-葡萄糖苷酶的分子改造提供了设计方法与试验基础。
