白刺14-3-3基因家族的鉴定及表达分析
2021-04-01段慧荣王丽蓉
易 丹, 王 博, 段慧荣, 李 毅*, 王丽蓉
(1.甘肃农业大学林学院, 甘肃 兰州 730070; 2.青海民族大学, 青海 西宁 810000; 3.中国农业科学院兰州畜牧与兽药研究所, 甘肃 兰州 730050)
1967年,Moore和Perez等从牛脑组织中分离鉴定出一种可溶异源二聚体蛋白,根据迁移率命名为14-3-3蛋白[1]。该蛋白是一个结构高度保守的多功能蛋白家族[2],普遍存在于真核生物中,并且是能够识别蛋白质磷酸化的家族之一[3]。目前为止,14-3-3基因家族在拟南芥(Arabidopsisthaliana)、水稻(Oryzasativa)、玉米(Zeamays)中被广泛研究并发现其在植物的生长发育、信号传导、抵御胁迫等方面发挥着重要作用[4]。14-3-3蛋白是蛋白质之间相互作用的纽带,它可以和信号蛋白直接作用,从而影响这些配体分子在亚细胞中的定位,14-3-3蛋白与H+-ATPase酶结合,可以增强其活性,还可以通过与激素信号途径中的关键分子相互作用,发挥其调节功能,以此来响应非生物胁迫[5]。由于该家族蛋白通常在植物中可形成同源二聚体或异源二聚体,并且是组成G-box蛋白复合体的一部分,因此又被称为G-box factor 14-3-3 homologs(GF14)或G-box regulatory factor or general regulatory factor(GRF)[6-7]。
随着模式植物和多种作物基因组测序的完成,目前已经从拟南芥[8]、蚕豆(Viciafaba)[9]、水稻[10]、烟草(Nicotianatabacum)[11]、大麦(Hordeumvulgare)[12]、大豆(Glycinemax)[13]、番茄(Solanumlycopersicum)[14]、棉花(Gossypiumspp)[15]中分别鉴定出15,4,8,17,5,18,12,25个14-3-3基因家族成员,且这些基因在不同的组织器官、激素及逆境胁迫下呈现出不同的表达规律,这些研究结果对进一步深入探究14-3-3基因家族的功能具有重要意义。
白刺(Nitrariatangutorum)为蒺藜科(Zygophyllaceae)白刺属(Nitraria)的超旱生灌木,主要分布于我国陕西北部、内蒙古西部、宁夏、甘肃河西、青海地区。白刺极耐盐碱和干旱,并且根系发达,有很强的防风固沙能力,是西北荒漠区优良的生态树种[16]。同时它还兼有营养、药用、饲用等价值,具有很高的经济开发潜力。前人在白刺的营养、药用成分、生理生态学等方面进行了诸多研究[17-23],并取得一定成果。在分子生物学方面也有少量的研究报道,且主要集中在相关抗逆基因的克隆及表达分析等方面[24-26]。而有关白刺14-3-3基因家族鉴定及分析的研究尚未见报道。为此,本研究基于白刺转录组数据,对白刺14-3-3基因家族成员进行鉴定,并对其理化性质、亚细胞定位、保守结构域、系统进化关系及其基因在不同浓度盐、干旱处理下的组织器官表达进行分析,以期为揭示白刺14-3-3基因家族的功能奠定基础。
1 材料与方法
1.1 材料
2019年8月23号于青海省诺木洪农场采集新鲜白刺果实,并经过处理获得干净的白刺种子。培养基质为黄河河沙(清洗、高温消毒后)。2019年10月9日将种子种入花盆(花盆口径:7 cm×7 cm,底径:5 cm×5 cm),置于培养间培养,培养条件为:白天光照12 h,夜晚光照6 h,白天温度26℃,夜晚温度22℃,相对湿度60%。每3 d向花盆底部托盘中灌入蒸馏水至盆内沙子完全浸湿。随机选取生长良好且长势一致的白刺幼苗进行胁迫处理。
1.2 方法
1.2.1不同胁迫处理 2019年12月24日将植株移至1/2 Hoagland(霍格兰)溶液中,培养条件同上,水中全天充氧。适应培养3 d后,将材料移至处理溶液中:设定低浓度聚乙二醇(Polyethylene glycol,PEG)(10%)、高浓度PEG(30%)两个浓度,对白刺幼苗进行干旱胁迫处理,并设立对照组(正常培养条件,无处理),在处理后2,12,24 h取样。另外设定低浓度NaCl(200 mM)和高浓度NaCl(450 mM)两个浓度,对白刺幼苗进行盐胁迫处理,并设立对照组(正常培养条件,无处理),在处理后2,12,24,48 h取样。每处理每时间点9株苗,每3株作为一个生物学重复,分别取根、茎、叶后迅速放入液氮中,后放入-80℃冰箱保存待用。
1.2.2白刺14-3-3基因家族鉴定 在本地数据库中,我们利用注释库筛选的方法[27]得到了28条14-3-3基因家族相关序列,除去重复序列后,利用DNAMAN(v6.0)获取氨基酸序列后放入Pfam(http://pfam.xfam.org/)在线软件和NCBI的CDD(https://www.ncbi.nlm.nih.gov/cdd)数据库中进行保守结构域分析,通过与两个数据库的比对,把不含有14-3-3蛋白保守结构域的序列剔除,最终得到的序列推测为白刺14-3-3蛋白序列,并利用白刺转录组数据库中的CDS库、NCBI的Blast(https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=tblastn&PAGE_TYPE=BlastSearch &BLAST_SPEC=&LINK_LOC=blasttab& LAST_PAGE=blastp)和ORF Finder(https://www.ncbi.nlm.nih.gov/orffinder/)在线软件共同预测白刺14-3-3蛋白序列CDS的完整性,对CDS不完整的序列不进行蛋白理化性质分析。
1.2.3蛋白理化性质分析 利用ExPASy在线网站的ProtParam(https://web.expasy.org/protparam/)分析已经鉴定出来的白刺14-3-3蛋白的(具有完整CDS)氨基酸数目、分子量、等电点和平均亲水性。通过CELLO(http://cello.life.nctu.edu.tw/)在线软件对该家族基因进行亚细胞定位预测。利用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)和SWISS-MODEL(https://swissmodel.expasy.org/interactive)在线软件分别预测蛋白的二级与三级结构。
1.2.4多序列比对及保守结构域的分析 利用GeneDoc软件绘制白刺14-3-3蛋白多序列比对图并标注高度保守的结构区域。通过在线软件MEME(http://meme-suite.org/tools/meme)进行motif预测分析,经验证基序最多值设置为7,其余为默认值。通过TBtools工具对导出的MEME文本结果完成保守基序图的绘制与分析。
1.2.5进化树的构建 首先利用ClustalW(http://www.clustal.org/)软件将拟南芥和白刺14-3-3蛋白序列进行多序列比对,再利用Mega 7.0软件的邻接(Neighbor-joining)法,构建两种进化树,一种由拟南芥和白刺14-3-3家族成员共同构成,另外一种由白刺14-3-3家族成员单独构成,设置bootstrap值为1 000,其他参数默认,进一步利用在线软件iTOL(https://itol.embl.de/)美化进化树,依据进化树分枝分组。
1.2.6荧光定量PCR 总RNA用RNAprep Pure Plant Kit RNAprep Pure植物总RNA提取试剂盒(北京天根生化科技有限公司,货号:DP432,产地:北京)按照试剂盒说明在低温无菌条件下提取RNA。用PrimeScriptTMRT reagent Kit with gDNA Eraser(Perfect Real Time)反转录试剂盒(TaKaRa北京宝生物公司,货号:RR047A,产地:北京)反转录成cDNA,反应体系为(20 μL):Master Mix 10 μL,5 x PrimeScript Buffer 2 (for Real Time) 4 μL,PrimeScript RT Enzyme Mix I 1 μL,RT Primer Mix 1 μL,RNase Free dH2O 4 μL,整个过程置于冰上操作。反转录程序为:37℃ 15 min,85℃ 5 s,4℃保存。
qRT-PCR用TB Green Premix Ex TaqTMII(Tli RNaseH Plus)试剂盒(TaKaRa北京宝生物公司,货号:RR820A,产地:北京)。使用Thermo荧光定量PCR仪(美国,型号:Step One Plus)检测cDNA样品的CT值。首先将cDNA稀释10倍备用,qRT-PCR反应体系(20 μL):TB Green Premix Ex TaqⅡ(Tli RNaseH Plus) (2X) 10 μL,cDNA模板2.5 μL,正反向引物(10 μmol·L-1)各0.8 μL,ddH2O 5.5 μL,ROX 0.4 μL。qRT-PCR反应程序:95℃预变性2 min,95℃变性10 s,60℃复性30 s,72℃延伸20 s,40个循环之后采集荧光。设置3个技术重复。引物根据鉴定获得的核苷酸序列,在家族成员非重叠区域,利用Primer在线软件(http://www.primer3plus.com/cgi-bin/dev/primer3 plus.cgi)设计。内参基因采用EF1α基因。特异性引物序列如表1所示。反应结束后分析各基因的溶解曲线及CT值。挑选正常条件、PEG 30% 2 h和NaCl 450 mM 24 h的白刺幼苗对这10个成员做qRT-PCR分析预试验,对表达量相对较高的成员进行后续试验分析。使用公式2-ΔΔCT计算基因的相对表达量[28]。
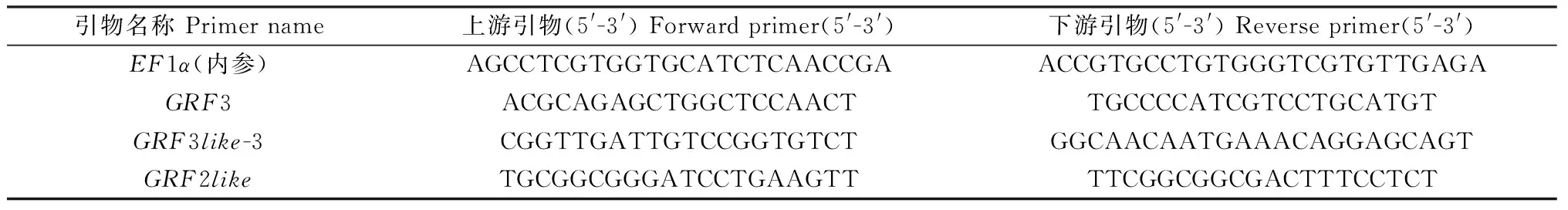
表1 荧光定量PCR引物序列Table 1 The primers used in qPCR
1.2.7数据分析 利用SPSS 21.0进行统计学分析,采用单因素方差(one-way ANOVA)的统计方法对白刺在不同处理下14-3-3基因的相对表达量进行显著性差异分析(P<0.05),利用Origin 9.0绘制柱形图并注明各组间的差异显著性。
2 结果与分析
2.1 白刺14-3-3基因家族的鉴定
从转录组数据库中共得到了28条14-3-3基因家族的转录组序列,剔除重复冗余的序列,进一步利用Pfam和CDD结构域预测软件初步筛选出10条含有14-3-3蛋白保守结构域的序列。命名规则如下:此基因家族统称为“GRF”家族,取白刺拉丁学名的缩写(Nt)作为前缀,编号参考白刺和拟南芥14-3-3成员的同源关系,同源性分析通过NCBI的Blastp数据库检索比对来完成。根据与拟南芥的同源性比对,将10条候选序列按“NtGRF+编号”进行命名。同源性高的命名为NtGRF+编号(编号与拟南芥对应),与拟南芥同源性较高但与其它成员有差别的命名为NtGRF+编号+like(表2、表3),对白刺14-3-3成员CDS的预测结果显示,除NtGRF12不含有完整的CDS,其他成员均含有完整的CDS(表2)。
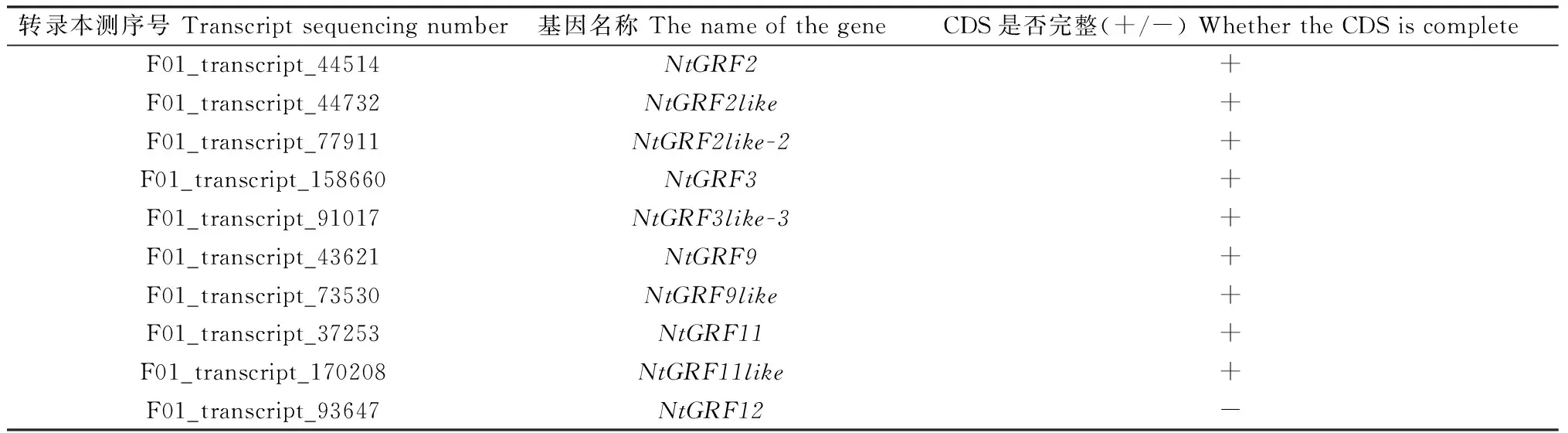
表2 白刺14-3-3基因家族基本信息Table 2 Basic information of 14-3-3 gene family in N. tangutorum

表3 白刺14-3-3基因家族成员理化性质Table 3 The physic-chemical characters of 14-3-3 gene family in N. tangutorum
2.2 理化性质
从表3中可知,9个白刺14-3-3基因所对应的编码氨基酸的数目为212 ~ 297个,编码蛋白理论分子量范围是24.06 ~ 33.51 kDa,蛋白理论等电点为4.73~6.14,平均大小为5.17,呈酸性。蛋白的不稳定系数除NtGRF9小于40,为稳定蛋白,其余均大于40,为不稳定蛋白。蛋白的平均亲水系数小于零,证明这9个蛋白均为亲水性蛋白,亚细胞定位预测显示这9个成员除NtGRF11like定位于质膜,其他成员均定位在细胞质。
白刺14-3-3蛋白二级结构预测结果显示(表4),9个蛋白的组成均有α螺旋、β转角、无规卷曲和延伸链,α螺旋最多,β转角最少。SWISS-MODEL结果显示该家族蛋白的三级结构高度保守,主要由α螺旋构成(图1)。
2.3 14-3-3基因家族多序列比对及保守基序分析
对10个白刺14-3-3蛋白序列进行多重序列比对,结果显示它们的一致性是63.78%,中间区域的位置高度相似,且包含14-3-3基因家族的保守结构域(L G L A L N F S V F Y Y E I),部分序列绝对保守(图2中红色方框),N-端和C-端变异程度大。
对白刺14-3-3蛋白的保守基序进行分析,将预测的最大值设置为20,发现只出现了7个不同的motif,再返回上一步将最大预测值设置为7,以此保证motif的完整性(图3)。如图4(A)所示,被分析的成员中共包含7个保守基序,不同的成员所含基序的数量和种类存在一定的差异,但相同分枝上的成员有相似的基序组合。10个成员皆包含motif1,motif2,motif5和motif6,仅NtGRF3和NtGRF3 like-3包含motif7,除NtGRF2like-2和NtGRF 3like-3以外,其余成员均含有motif4,motif1,motif2,motif5,motif6在这个10个成员中均出现10次,motif3出现9次,motif4出现8次,motif7出现2次。出现频率较高的motif往往在该基因中具有重要的作用[29]。

表4 白刺14-3-3蛋白的二级结构预测Table 4 Prediction of the secondary structure of N. tangutorum 14-3-3 protein

图1 白刺14-3-3蛋白的三级结构预测Fig.1 Prediction of the tertiary structure of N. tangutorum 14-3-3 protein
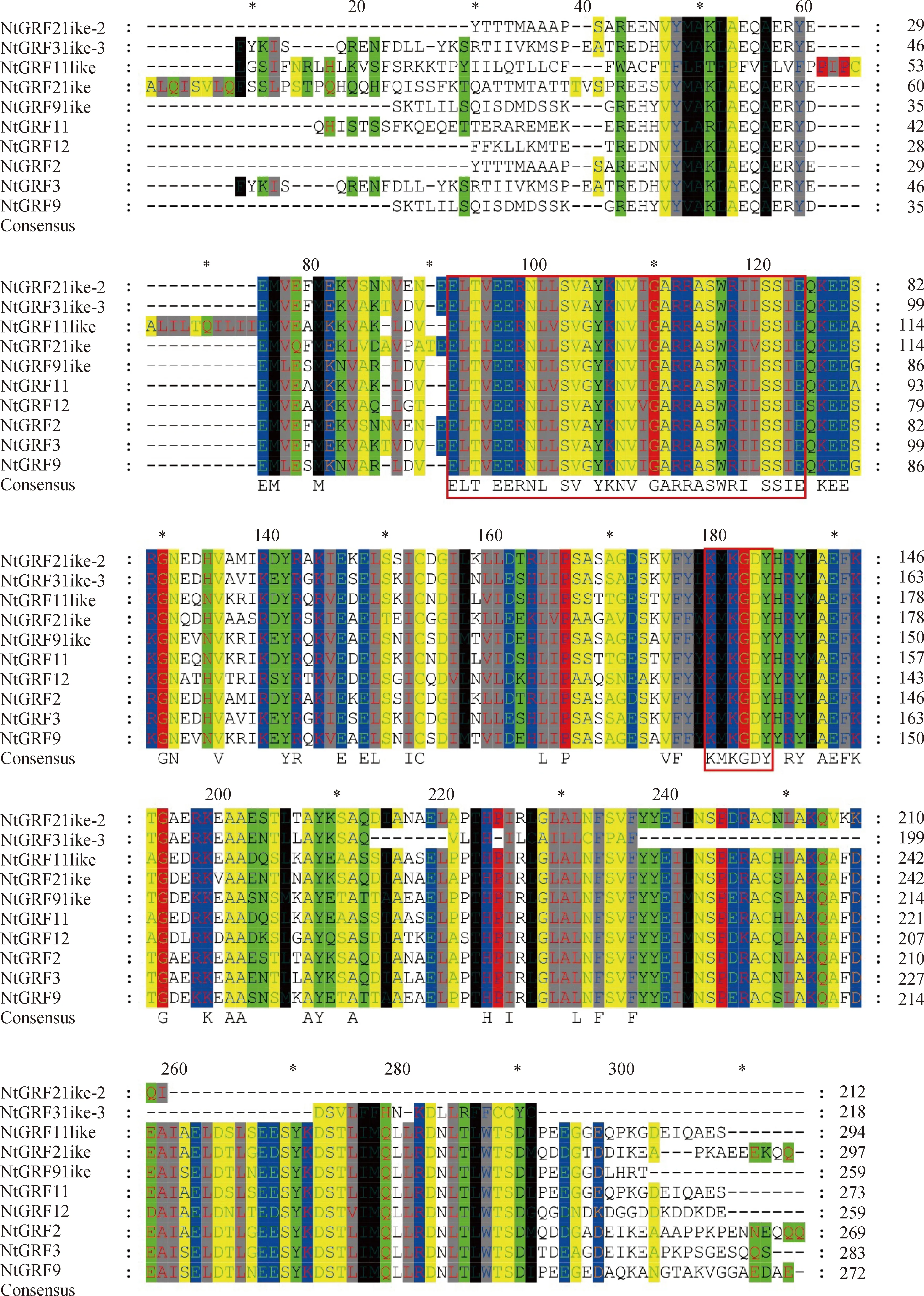
图2 白刺14-3-3蛋白多序列比对Fig.2 Multiple sequences alignment of N. tangutorum 14-3-3 protein
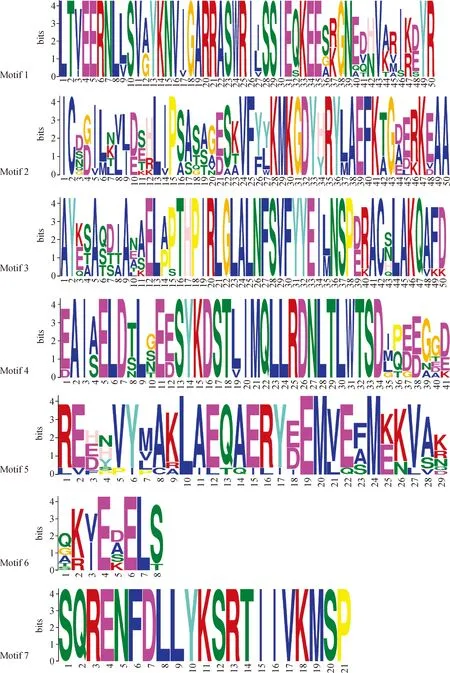
图3 白刺14-3-3蛋白保守基序Fig.3 Conserved motifs of N. tangutorum 14-3-3 protein
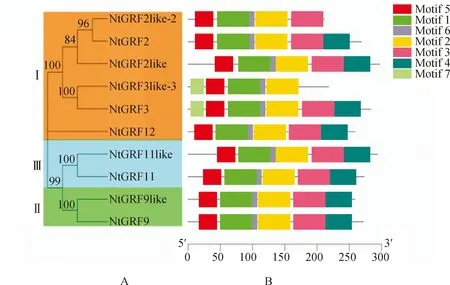
图4 白刺14-3-3蛋白系统进化关系(A)及保守基序分析(B)Fig.4 Phylogenetic relationship of protein 14-3-3 in N. tangutorum (A) and conserved motif analysis (B)
2.4 14-3-3基因家族进化树分析
图4(A)是由白刺14-3-3蛋白序列建立的系统发育树,从图中可以看出白刺14-3-3家族成员的进化关系:最早分化出来的是NtGRF11,NtGRF 11like,NtGRF9和NtGRF9like,之后是NtGRF12,NtGRF3和NtGRF3like-3,最后是NtGRF 2like,NtGRF2以及NtGRF2like-2。用15条拟南芥14-3-3蛋白序列(AtGRF1-AtGRF15)与10条白刺14-3-3蛋白序列构建系统发育树(图5)。由图4可知,NtGRF2like,NtGRF2和NtGRF2like-2与拟南芥中AtGRF2,AtGRF6,AtGRF8亲缘关系较近;NtGRF3和NtGRF3like-3与拟南芥AtGRF3的亲缘关系最近;是AtGRF5和AtGRF7,NtGRF12独成一枝与AtGRF12亲缘关系较近;NtGRF9和NtGRF9like与AtGRF9亲缘关系最近;NtGRF11和NtGRF11like与AtGRF11关系近。这与我们之前在命名时与NCBI的Blastp数据库的比对结果一致。

图5 白刺、拟南芥14-3-3蛋白系统进化关系Fig.5 The evolutionary relationship of 14-3-3 protein system of N. tangutorum and Arabidopsis thaliana
2.5 白刺14-3-3基因家族成员在胁迫处理中的表达分析
为解析白刺14-3-3基因家族成员对逆境的响应模式,利用qRT-PCR检测其在干旱胁迫(PEG)和盐胁迫(NaCl)处理下的表达量。根据前期试验结果,选取表达量相对较高的基因GRF3,GRF3like-3,GRF2like用于后续表达模式分析。
如图6所示,在不同浓度PEG处理下,GRF3,GRF3like-3,GRF2like在各时间段均有表达,其中,GRF3在低浓度PEG(10%)处理下表达量下调,在处理后期表达量有回升,在高浓度PEG(30%)处理下,2 h表达量明显上调且达到最高,是对照组(CK)的5.9倍,随后表达量下调;GRF3like-3在低浓度PEG 24 h和高浓度PEG 24 h表达量分别达到最高,是对照组的1.7倍和1.67倍,在这两个浓度下表达量都呈现先下调后上调的趋势;GRF2like在低浓度PEG 24 h的表达量最高,是对照组的1.67倍,在高浓度PEG 2 h表达量上调达到最高,是对照组的1.9倍,随后下调。这3个基因在低浓度PEG的处理下,表达量都出现先下调后上调的趋势,在高浓度PEG下表达量没有固定的趋势。说明GRF3,GRF3like-3,GRF2like参与白刺的干旱胁迫应答,且具有各自不同的调节功能。
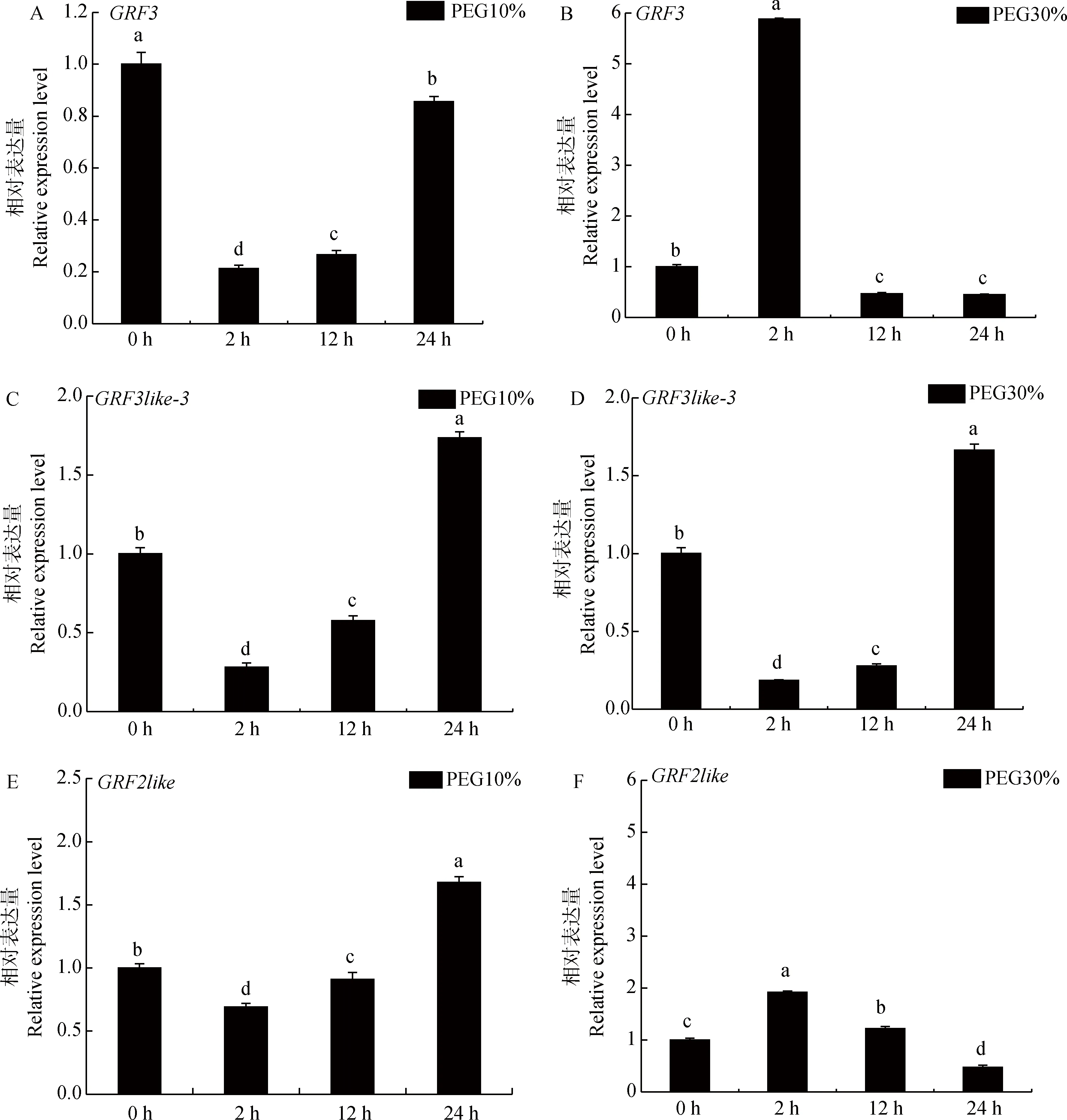
图6 不同浓度PEG处理下白刺14-3-3基因家族3个代表基因的表达量Fig.6 The expression of three representative genes of N. tangutorum 14-3-3 gene family under different concentrations of PEG treatments注:A-B,不同浓度PEG处理下GRF3的表达量;C-D,不同浓度PEG处理下GRF3like-3的表达量;E-F,不同浓度PEG处理下GRF2like的表达量。不同处理之间不同小写字母表示差异性显著(P<0.05)。下同Note:A-B,Expression of GRF3 under different concentrations of PEG treatment;C-D,Expression of GRF3like-3 under different concentrations of PEG treatment;E-F,Expression of GRF2like under different concentrations of PEG treatment.Bars marked with different lowercase letters showed significant difference at the 0.05 level.The same as below
如图7所示,在NaCl不同浓度处理下,GRF3,GRF3like-3,GRF2like在各时间段均有表达。其中,GRF3在低浓度NaCl(200 mM)处理下,整体表达呈现先减少后增加的趋势,48 h的表达量最高与对照组无显著差异,在高浓度NaCl(450 mM)48 h表达量上调到最高,是对照组(CK)的12倍;GRF3like-3在低浓度NaCl 2 h表达量明显上调且达到最高,是对照组的12.9倍,在这之后表达量下调,在高浓度NaCl 12 h表达量最高,是对照组的19倍,在此之后下调;GRF2like在低浓度NaCl 12 h表达量最高是对照组的5.1倍,在高浓度NaCl 12 h表达量明显上调,到24 h表达量达到最大值,是对照组的15.5倍,随后持续下降。说明GRF3,GRF3like-3,GRF2like同样参与白刺的盐胁迫应答,且具有各自不同的调节功能。
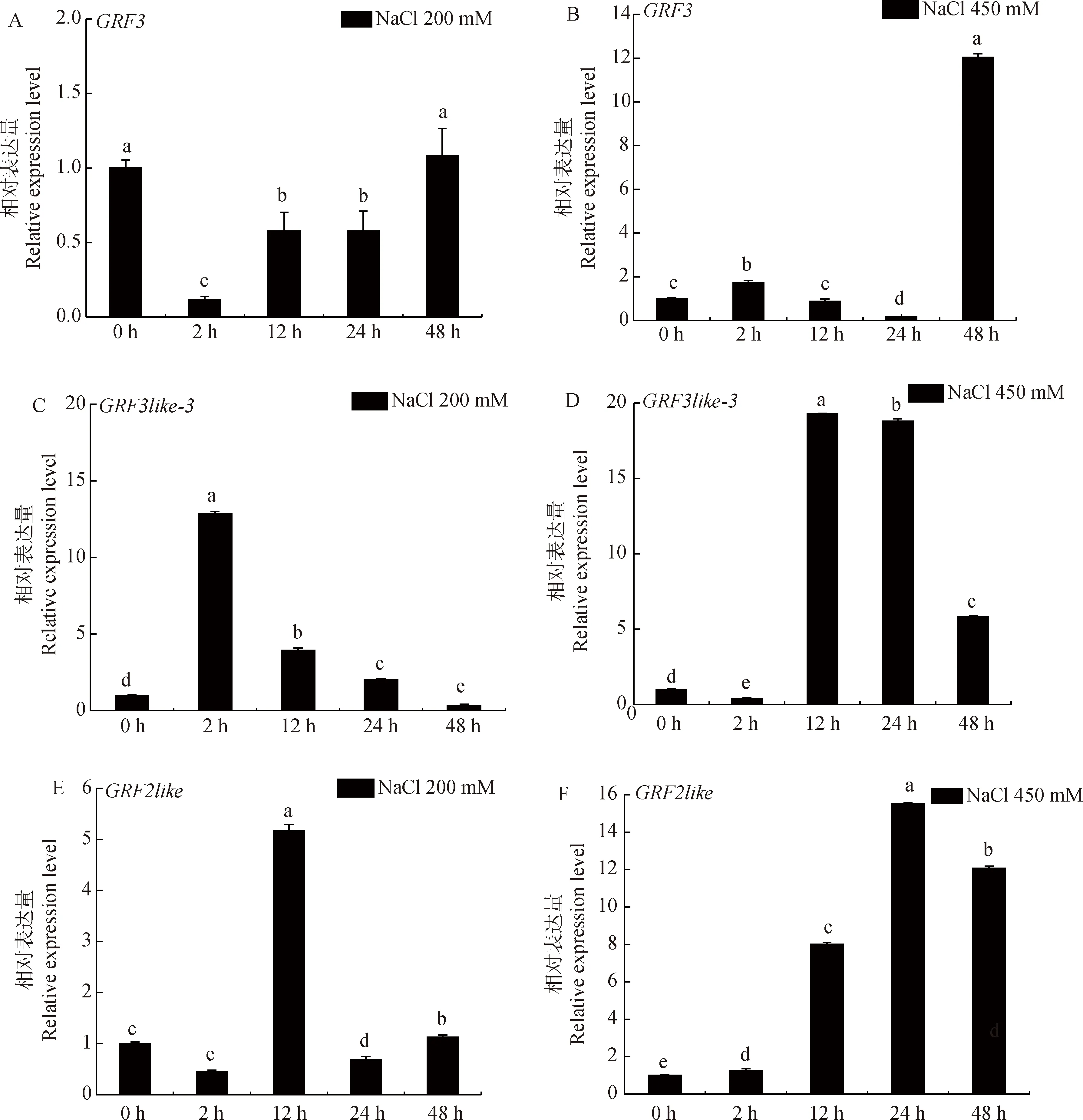
图7 不同浓度NaCl处理下白刺14-3-3基因家族3个代表基因的表达量Fig 7 The expression of three representative genes of N. tangutorum 14-3-3 gene family under different concentrations of NaCl注:A-B,不同浓度NaCl处理下GRF3的表达量;C-D,不同浓度NaCl处理下GRF3like-3的表达量;E-F,不同浓度NaCl处理下GRF2like的表达量Note:A-B,Expression of GRF3 under different concentrations of NaCl;C-D,Expression of GRF3like-3 under different concentrations of NaCl;E-F,Expression of GRF2like under different concentrations of NaCl
2.6 白刺14-3-3基因家族成员的组织器官特异性表达分析
如图8所示,GRF3,GRF3like-3,GRF2like在根、茎、叶中均有表达,但不同处理条件下表达存在差异。其中,GRF3在正常条件和PEG处理下,根中的表达量最高,叶中的最低,在NaCl处理下,根中的表达量和茎中的基本一致,叶中的最低;GRF3like-3在正常条件和PEG处理下,叶中的表达量最高,茎中的表达量最低,在NaCl处理下,根中的表达量最高,叶中的最低;GRF2like在正常条件下,根中的表达量最高,茎中的表达量最低,在PEG和NaCl处理下都表现出根中的表达量最高,叶中的表达量最低。说明该家族基因在组织器官中存在表达差异,并且能对干旱胁迫和盐胁迫做出响应。
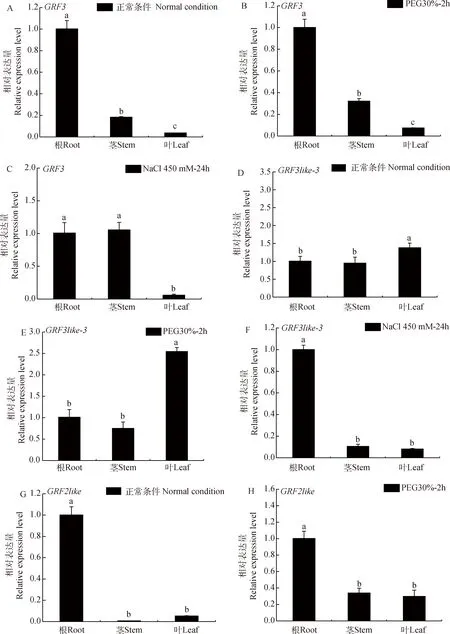

图8 不同处理下白刺14-3-3基因家族3个代表基因组织器官的表达量Fig 8 Expression levels of three representative genes of N. tangutorum 14-3-3 gene family in tissues and organs under different treatments注:A,正常条件下GRF3在不同组织器官中的表达量;B,PEG处理下GRF3在不同组织器官中的表达量;C,NaCl处理下GRF3在不同组织器官中中的表达量;D,正常条件下GRF3like-3在不同组织器官中的表达量;E,PEG处理下GRF3like-3在不同组织器官中的表达量;F,NaCl处理下GRF3like-3在不同组织器官中的表达量;G,正常条件下GRF2like在不同组织器官中的表达量;H,PEG处理下GRF2like在不同组织器官中的表达量;I,NaCl处理下GRF2like在不同组织器官中的表达量Note:A,Expression of GRF3 in different tissues and organs under normal conditions;B,Expression of GRF3 in different tissues and organs under PEG treatment;C,Expression of GRF3 in different tissues and organs under NaCl treatment;D,Expression of GRF3like-3 in different tissues and organs under normal conditions;E,Expression of GRF3like-3 in different tissues and organs under PEG treatment;F,Expression of GRF3like-3 in different tissues and organs under NaCl treatment;G,Expression of GRF2like in different tissues and organs under normal conditions;H,Expression of GRF2like in different tissues and organs under PEG treatment;I,Expression of GRF2like in different tissues and organs under NaCl treatment
3 讨论与结论
蛋白磷酸化作为一个重要的信号传导机制,它存在于所有真核生物中,参与细胞分裂、分化、凋亡等多个基础环节。14-3-3蛋白是一类基础性蛋白,它主要通过调控激酶转导的信号响应与转录因子对植物进行细胞信号转导调控[3]。本研究中,基于转录组数据,初步筛选出10个白刺14-3-3基因家族成员,通过亚细胞定位预测发现白刺14-3-3蛋白主要分布在细胞质上,与在拟南芥中的规律相同[30],说明14-3-3蛋白的功能在不同植物中是保守的。
多序列对比与保守基序分析表明,白刺14-3-3蛋白N-端和C-端区域具有较高的变异性,其余部分均保守,这与拟南芥中的研究一致[31]。motif1,motif2,motif5,motif6在白刺14-3-3家族中高度保守,N-端和C-端的motif3,motif4和motif7产生了变异,motif1包含该家族的保守结构域。这种中心区域高度保守,N-端和C-端的变化使14-3-3蛋白形成很多异构体。N-端会影响14-3-3蛋白与不同的膜结合,C-端直接参与和靶蛋白的相互作用,每一个异构体的N-端和C-端几乎是独一无二的,这是造成不同的异构体功能特异性的基础[11]。有研究表明,N-端和C-端不同的motif是14-3-3蛋白发挥其不同功能时的核心结构[32],本研究中白刺14-3-3蛋白的N-端与C-端有不同的motif,推测该蛋白能与众多靶蛋白相结合而发挥重要作用。
系统进化树显示白刺和拟南芥14-3-3家族成员分为Ⅰ,Ⅱ,Ⅲ,Ⅳ4组,分别包含了6,2,2和0个白刺14-3-3蛋白,表明白刺14-3-3家族成员之间存在广泛的多样性。第Ⅰ组中白刺14-3-3家族成员比Ⅱ,Ⅲ,Ⅳ组大大增加,说明第Ⅰ组的成员在进化过程中较为激烈,发生了基因加倍的情况,使得成员数目增加。基因加倍为白刺提供了更高的进化潜能[33]。在Ⅰ,Ⅱ,Ⅲ组中,均有白刺和拟南芥14-3-3家族成员,推测白刺和拟南芥可能具有相似的进化轨迹。并且同源性较高的基因可能具有相似的功能。
表达模式分析可以在一定程度上预测基因的分子功能和涉及的生命过程,本研究在PEG模拟干旱胁迫的处理下,GRF3,GRF3like-3,GRF2like参与干旱胁迫响应,除GRF3在低浓度处理下较对照组表达量没有上调,其他处理下3个基因的表达量均出现上调,这与水稻14-3-3基因家族中GF14b,GF14c,GF14e,GF14f的研究一致[30]。在NaCl高、低浓度处理下这3个基因的表达量均出现上调,这与番茄14-3-3家族中的TFT1,TFT4,TFT7和TFT10的结论一致[30]。与此同时这3个基因对低浓度PEG胁迫的响应模式相似,表达量都是先下调后上调,均在胁迫后期响应更明显,而对高浓度PEG有不同的响应模式,并且在高浓度NaCl处理下都在胁迫中后期响应更明显,但在低浓度NaCl处理下有不同的响应模式,这可能跟不同基因在胁迫中承担的功能不同有关。14-3-3家族成员在不同物种中的表达存在器官特异性,拟南芥中15个14-3-3基因只有GRF1和GRF2在所有的器官中都有表达,多数14-3-3基因的表达具有明显的组织器官特异性,如GRF14仅在生殖器官中表达[34]。本研究中的3个基因除GRF3like-3在正常条件和PEG处理下叶片中的表达量最高,在NaCl处理下根中的表达量最高以外,剩余2个基因均在正常条件、PEG和NaCl的处理下根中的表达量最高。综合上述结果来看,14-3-3基因家族在白刺不同组织器官中表现出明显不同的表达规律,并可响应外界的干旱和盐胁迫。由此推测,14-3-3基因家族可能在白刺的胁迫防御方面发挥重要功能。
本研究基于白刺转录组数据,初步筛选出10个14-3-3家族成员,通过生物信息学分析,预测了其基因家族的理化性质、亚细胞定位、保守结构域及系统进化关系。同时,通过分析白刺14-3-3家族成员在不同浓度盐、干旱处理下的组织器官表达特性,初步证明了14-3-3基因家族参与白刺抗旱耐盐胁迫防御反应过程,该结果为进一步深入探究揭示白刺14-3-3基因家族的功能奠定基础。
