碳青霉烯耐药肺炎克雷伯菌ST11菌株的基因组学特点分析*
2021-04-01曹小利程莉周万青张之烽张葵沈瀚
曹小利,程莉,周万青,张之烽,张葵,沈瀚
(南京大学附属鼓楼医院检验科,南京210008)
肺炎克雷伯菌是医院和社区获得性感染的主要病原菌,主要引起尿路感染、肺炎、败血症等感染性疾病[1]。目前已知,肺炎克雷伯菌是全球主要流行的碳青霉烯耐药肠杆科细菌(carbapenem resistant enterobacteraceae, CRE)[2]。KPC-2是碳青霉烯耐药肺炎克雷伯菌(carbapenem resistanceKlebsiellapneumoniae, CRKP)中主要的碳青霉烯酶,ST11是CRKP的主要流行克隆[3]。本研究主要对南京地区不同医院分离的3株CRKP-ST11进行基因组测序,并与上海、杭州、美国/印度的相应菌株进行基因组比对,进行基因组学特点分析,报道如下。
1 材料和方法
1.1实验菌株 在前期的研究基础上,挑取南京鼓楼医院、南京明基医院、南京医科大学医学院附属第二医院分离的3株CRKP-ST11菌株[4],样本分别从患者血液、尿液及痰液培养中获得。细菌经基质辅助激光解吸电离飞行时间质谱技术鉴定。本研究中CRKP定义为对亚胺培南耐药,ST11经肺炎克雷伯菌多位点序列分型确定。
1.2仪器和试剂 磁珠法通用型基因组DNA提取试剂盒DP705(北京天根公司);NanoDrop 2000分光光度计(德国Thermo公司);恒温震荡培养仪器ZWY-211B(ZHICHENG公司);全自动快速微生物质谱检测系统(法国生物梅里埃公司)。
1.3基因组提取和全基因组测序 取新鲜培养的单个菌落接种至50 mL LB培养液中,37 ℃ 200 r/min过夜培养后,4 ℃、(4 000~8 000)×g离心10 min,弃上清液,细菌沉渣用无菌水洗2次后,使用磁珠法通用型基因组DNA提取试剂盒DP705,按说明书提取细菌基因组DNA,并用NanoDrop 2000分光光度计检测基因组的浓度和纯度。基因组浓度要求:DNA≥5 ng/μL;纯度要求:A260 nm/A280 nm=1.8~2.0。本项目委托广东美格基因公司进行基因组高通量测序。提取的DNA样品经电泳检测合格后,用Covaris超声波破碎仪随机打断成长度约350 bp的DNA片段。经末端修复、加A尾、加测序接头、纯化、PCR扩增等步骤完成文库制备;构建的文库先使用Qubit 2.0进行初步定量,稀释文库至2 ng/μL,随后使用Agilent 2100对文库的插入片段进行检测,片段符合预期后,使用Q-PCR方法对文库的有效浓度进行准确定量,以保证文库质量。库检合格后,进行Illumina HiSeq 测序,测序平台采用Illumina Hiseq PE150。获得的核苷酸片段在CLC genomics workbench软件(苏州协云基因科技公司)上进行拼接,通过软件去除测序质量较差的片段后,使用de novo拼接成contigs。
1.4细菌单核苷酸多态性(single nucleotide polymorphism,SNP)分析 以中国浙江(JM45)、上海(HS11286)和美国/印度(ATCC BAA-2146)的报道的CRKP-ST11全基因组为对照菌株,进行比对分析。将获得的3株草图基因组的contigs,采用CLC Main Workbench进行首尾串连成一条假的完整DNA,然后将6个基因组的核苷酸序列导入Mauve软件[5],进行多序列比对,比对完成后,选择导出SNP分析结果即可获得所有SNP位点信息。采用Circos软件[6],以每1 000 bp内发生的SNP数量为数据,分别对每个基因组的SNP分布进行可视化。
1.5亲缘关系分析 为获得核心区域的高质量SNP位点信息,采用Perl对Mauve生成的SNP位点信息进行过滤,标准如下:(1)不允许出现“-”;(2)每一个SNP位点只能出现2种不同的碱基。采用自编写的Perl脚本对过滤后的SNP位点进行串联,共获得14 052个SNPs,然后导入RAxML构建最大似然树[7]。
1.6荚膜多糖(capsular polysaccharide,CPS)基因簇组成及GC含量分析 将基因组gbk格式(序列及基因注释)文件导入CLC Main Workbench,对CPS区域基因组成进行共线性分析,并导出基因簇组成图(SVG格式);根据基因注释并进行人工核对,确定基因名。选择导出每个基因组的CPS区域核苷酸序列,然后采用自编写的Perl脚本程序对核苷酸序列进行GC含量步移法(步长设置为120 bp)扫描分析。最后采用Adobe Illustrator CS对图形进行编辑。
1.7基因组的获取号 本实验中测序得到的基因组已上传NCBI,基因组获取号分别依次为:RZKL00000000、RZME00000000和RZMP00000000;本实验中的对照基因组从NCBI中下载,基因组获取号如下:肺炎克雷伯菌HS11286:NC_016845.1;肺炎克雷伯菌JM45:NC_022082.1;肺炎克雷伯菌ATCCBAA-2146:NZ_CP006659.2。
2 结果
2.13株CRKP-ST11的基因组组装结果 3株测序细菌的基因组组装结果见表1。

表1 3株碳青霉烯耐药肺炎克雷伯菌ST11菌株的基因组组装结果
2.2CRKP-ST11菌株的SNP特点 6株细菌基因组的单核苷酸位点多态性见图1。结果显示,在整个肺炎克雷伯菌基因组中,CPS基因簇是CRKP-ST11的主要的SNP位点分布区之一。
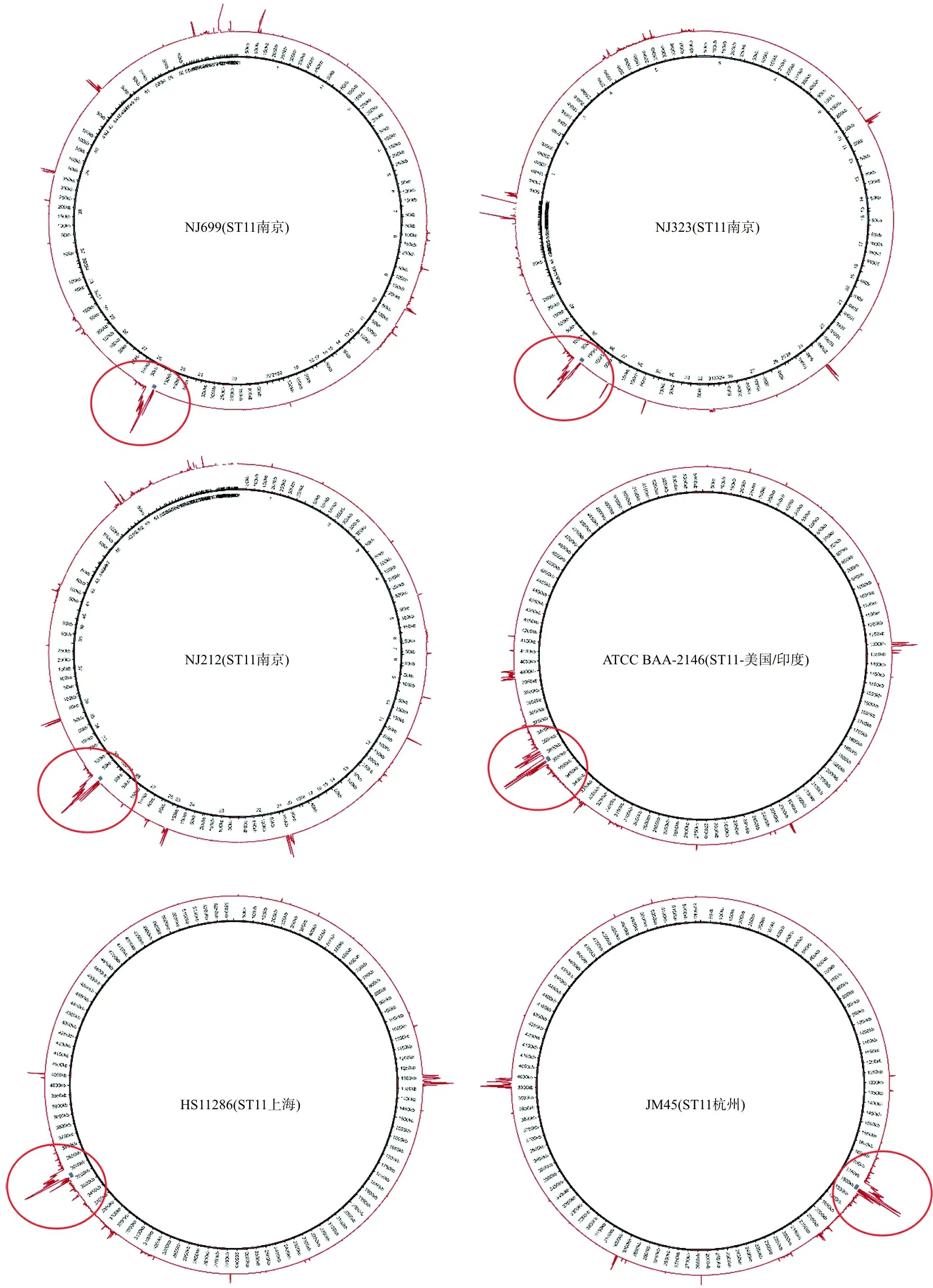
注:红色标记表示CPS基因簇的SNP变化,箭头的幅度越大,显示该区的多态性越大。
2.3CRKP-ST11菌株的亲缘关系特点 CRKP-ST11菌株的群体结构分析见图2。结果显示,6株CRKP-ST11菌株分为4个进化枝,其中,南京地区分离的3株CRKP-ST11菌株在同一进化枝中,亲缘关系最近;其次,这3株细菌与上海、杭州的CRKP-ST11也有较近的亲缘关系,而与美国/印度的CRKP-ST11较远。
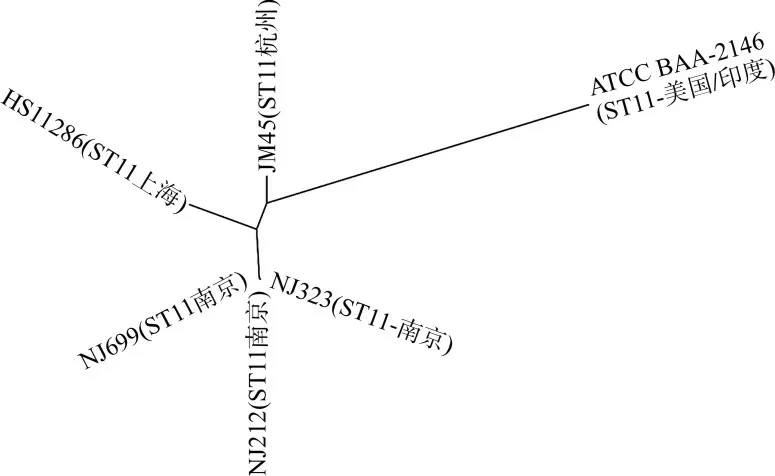
注:NJ699、NJ323和NJ212是在南京分离的3株CRKP-ST11菌;HS11286(中国上海)、JM45(中国杭州)、ATCC BAA-2146(美国/印度)的基因组从NCBI数据库中下载。
2.4CRKP-ST11菌株的CPS基因簇结构特点 CRKP-ST11的CPS基因簇结构分析见图3。结果显示,从南京地区分离的3株CRKP-ST11菌株,其CPS基因簇5′端的galF、orf2、wzi、wza、wzb和wzc及3′端的gnd和ugd基因间的成对核苷酸相似性为99%(galF)到55%(wzc),组成相当一致,具有很好的共线性;但是与杭州、上海及美国/印度的3株CRKP-ST11菌株主要在可变区存在差异。
2.5CRKP-ST11菌株CPS的GC含量特点 南京地区分离的3株CRKP-ST11的CPS区GC含量见图4。结果显示,CPS可变区的GC含量大多<50%,显著低于其保守区域(GC含量>50%)以及基因组平均GC含量(约57%),表明CPS可变区的GC含量明显不同于基因组核心保守区。

注:蓝色区域表示5′端保守区,红色区域表示3′端保守区,绿色区为可变区。粉色阴影部分表示碱基序列相似或相同。
3 讨论
本研究中,我们获取的3株细菌的基因组组装结果与以往的研究结果一致[8],并且提交NCBI时已通过审核,表明我们在基因组提取、测序与组装方面的结果比较可靠,可以深入分析。通过SNP分析,我们发现,CPS基因簇是CRKP-ST11的主要的SNP位点分布区之一。目前已知全球CRKP主要流行克隆组(clonal group,CG)258包括ST11和ST258等,CG258的对比基因组学分析显示cps基因簇是CRKP的热点重组区域,容易发生重组或替换而导致荚膜转换[9],这种荚膜转换在CG258中相当常见,更接近于或在CPS基因簇内,可能是CG258进化的主要特征[10],很可能是CG258全球播散主要的和潜在的共同驱动力[9,11]。此外,CPS基因簇内常存在编码转座酶或噬菌体相关蛋白的基因[3],表明转座以及和转座相关的水平基因转移可能也在CPS基因簇内发生。其内外的DNA交换可能是肺炎克雷伯菌快速多样化和进化的重要机制[12]。
亲缘性进化分析表明,南京地区分离的3株CRKP-ST11的亲缘性相近,这与我们前期的研究结果一致[13],表明CRKP-ST11在南京地区存在播散流行。此外,虽然南京和杭州距离上海很近,但不同地区来源细菌位于不同的进化枝,表明CRKP-ST11菌株的流行可能有地域特点。
目前已知,CPS由染色体基因编码,在基因组CPS位点成簇,大小一般为21 000~30 000 bp,有16~25个开放读码框架(open reading frame,ORF)[14]。其有一个基本结构特征:由两端的保守区和中间的高度可变区组成,5′端高度保守区由6个基因(galF、orf2、wzi、wza、wzb和wzc)组成,参与CPS易位运输和细菌表面蛋白加工;3′端有高度保守的葡萄糖-6-磷酸脱氢酶(gnd)和UDP-葡萄糖脱氢酶(ugd)基因;中心区域(wzc-gnd区)为可变区,随不同的荚膜类型而变化,通常含有编码葡萄糖转移酶(GTs)、翻转酶(wzx)、聚合酶(wzy)和修饰酶(乙酰转移酶、丙酮酰转移酶等)的基因,与特定CPS亚基的聚合和装配有关。研究报道,CPS基因簇中心可变区具有很大的多样性,很可能是进化的起源[15]。然而目前研究仅集中于CPS的保守区域,不足以解释其多样性特征。本研究发现,南京地区分离的CRKP-ST11与杭州、上海及美国/印度的3株CRKP-ST11菌株的差异主要在可变区。Zhou等[16]报道,与非暴发肺炎克雷伯菌菌株相比,暴发菌株KP-ST1427的CPS基因簇可变区多了一段编码GTs的3个ORF。奥地利学者发现,作为全球最为流行的CRKP,ST258的2个进化枝都具有D-半乳聚糖-Ⅲ,其编码基因在CPS可变区;相继的流行病学分析显示,200多个ST258的全基因组序列中,83%的肺炎克雷伯菌ST258的CPS可变区都有这种编码GTs的3个基因的ORF[17],提示CPS可变区在ST258流行播散中的作用。最近的一项研究表明,CRKP-ST258的CPS分型与ST258的流行相关[18]。

注:CPS可变区的G+C含量大多<50%。A,NJ699 CPS可变区的GC含量;B,NJ323 CPS可变区的GC含量;C,NJ212 CPS可变区的GC含量。
此外,CPS区的GC含量分析发现,可变区的碱基含量明显低于保守区域和基因组平均GC含量。有研究发现,细菌的基因组核苷酸含量变化很大,各物种之间的GC含量为13%~75%[19]。微生物进化和环境都会导致微生物群体中的重组,而重组也会影响基因组碱基含量的变化。在大多数原核核心基因组中,GC含量的增加似乎是由系统发育惯性而维持的,而基因组相应的附属和非核心部分中更为多样和丰富的碱基组成可能更多地受到环境和其他宿主碱基组成的影响。在核心基因组中观察到的基因内GC含量越高,GC变异越小,这很可能与选择性限制有关[20],意味着该菌与其他微生物进行胞内重组和遗传交换的频率比其他微生物少[21]。因为细菌的突变主要发生在AT上[22],噬菌体摄取也有AT偏向,所以比起宿主染色体,外源性DNA序列(如噬菌体和质粒)富集更多的AT碱基[23-24]。所以,可变区GC含量越低,表明外源性DNA序列(如噬菌体和质粒等的序列)越多,该区基因重组的可能性越大。
本研究中,不管是基因组GC含量,还是基因组SNP分析均显示,CPS基因簇很可能是CRKP的热点重组区域,这与以往的研究报道一致[25],表明CPS基因簇可变区可能通过同源重组等导致了CPS基因座基因内容和总体长度的差异,这可能在细菌的进化中起着重要的作用。总之,CPS是CRKP-ST11的主要单核苷酸位点,其可变区碱基含量<50%,可能是CRKP-ST11菌株的主要进化区,可能与CRKP-ST11的流行有关。
