胰腺癌血浆循环游离DNA甲基化预测模型的构建及应用
2021-03-24黄一鸣康亚妮
陈 旻,黄一鸣,赵 晖,康亚妮
胰腺癌是高度致死性的恶性肿瘤,是目前第4大癌症死亡原因[1],90%以上为胰腺导管腺癌[2]。由于超过75%的胰腺癌患者确诊时已处于晚期阶段[3],因此仅10%~20%患者有机会接受手术治疗[4]。即便如此,在接受手术治疗的胰腺癌患者中,仍有50%的患者在18个月内出现肿瘤复发[5-6],因此胰腺癌患者5年生存率仅为9%[1]。所以,早期诊断胰腺癌能够有效提高胰腺癌患者的生存率[7]。
基于循环游离DNA(cell-free DNA,cfDNA)的液体活检技术通过检测肿瘤患者体液中肿瘤组织释放的成分以实现肿瘤诊断,在肿瘤的早期诊断中显示出很好的应用前景。相较于目前常规的肿瘤检测技术,液体活检具有微创、负担小、敏感度高[8]以及结果更为全面[9]等优势。DNA甲基化作为一种稳定的肿瘤早期变化事件,具有很强的组织稳定性和不同的个体保守性[9],因此很适合作为有效的肿瘤早期标志物,以实现肿瘤的早期诊断。基于cfDNA甲基化的液体活检技术在实现胰腺癌早期诊断和预后预测方面显示出一定的潜力。
本研究采用胰腺癌患者和健康人群血浆cfDNA样本,结合数据库中胰腺癌与癌旁组织的甲基化450K数据,联合筛选获得胰腺癌血浆cfDNA甲基化候选标志物,构建胰腺癌的诊断和预后预测模型。
1 材料与方法
1.1 标本和材料
胰腺癌患者和健康人群的5份血浆样本由上海交通大学医学院附属瑞金医院提供;QIAamp循环核酸试剂盒购自QIAGEN公司;KAPA Hyper Prep试剂盒购自KAPA Biosystems公司;5-甲基胞嘧啶单克隆抗体购自EpiGentek公司;Q5高保真DNA聚合酶购自New England Biolabs公司。
1.2 方法
1.2.1 血浆样本收集
收集胰腺癌患者血浆样本3份以及健康人群血浆样本2份,所有患者均签署知情同意书,并获得上海交通大学医学院附属瑞金医院伦理委员会的批准。
1.2.2 血浆cfDNA提取
在手术和药物治疗之前,使用EDTA抗凝管收集5 mL外周血。在采集后6 h内,1 500×g离心15 min以纯化血浆。使用QIAamp循环核酸试剂盒从800 μL血浆中提取cfDNA,应用2100 Bioanalyzer系统(购自Agilent Technologies公司)进行定量。
1.2.3 MeDIP-seq建库和测序
使用KAPA Hyper Prep试剂盒,将约20 ng cfDNA与Illumina接头相连接。构建的cfDNA文库与95 ℃变性10 min。使用5-甲基胞嘧啶单克隆抗体,使甲基化cfDNA从文库中免疫沉淀。使用Q5高保真DNA聚合酶进一步扩增MeDIP DNA。采用2100 Bioanalyzer系统进行质量评估,扩增文库由Illumina HiSeq 2000平台进行深度测序。
1.2.4 血浆cfDNA原始数据的处理与差异甲基化区域(differential methylation regions,DMRs)筛选
使 用Trim_Galore(0.4.3版 本)去 除Illumina接头,Cutadapt(1.8.3版本)切除低质量序列。采用Bowtie2软件(2.1.0版本)将处理后的序列比对到人类参考基因组GRCh38/hg38(UCSC)。比对完成后,通过SAMtools(1.3.1版本)过滤唯一比对序列,并通过Picard(1.2版本)去除重复序列。最后,应用MACS2软件(2.1.0版本)对序列进行校峰。预处理完成后,应用DiffBind R软件包(2.8.0版本)筛选胰腺癌患者与健康人群之间的DMRs(P<0.05,log|FC|>1)。对筛选获得的DMRs,应用ChIPseeker R软件包(1.5.1版本)进行注释,进一步筛选出其中位于基因启动子区的DMRs用于后续分析。
1.2.5 胰腺癌与癌旁组织甲基化450K数据的获取与差异甲基化位点(differential methylation positions,DMPs)的筛选
从GEO数据库中获取胰腺癌与癌旁组织样本的甲基化450K数据(GSE49149),共获得胰腺癌组织样本155个,癌旁组织样本19个。应用ChAMP R软件包(2.18.3版本)对数据进行质量控制和预处理,并且筛选出DMPs(P<0.05,|Δβ|>0.2)。选择位于启动子区域的DMPs用于后续分析。
1.2.6 胰腺癌血浆cfDNA与胰腺癌组织甲基化状态的联合分析及模型的建立
筛选出位于启动子区域且与DMRs拥有相同靶基因和甲基化差异趋势的DMPs,将这些DMPs作为胰腺癌血浆cfDNA甲基化候选位点用于模型的构建。将GEO数据库中获得的胰腺癌与癌旁组织的甲基化450K数据按1:1分为训练集与测试集,在训练集中进行模型的构建。采用500次Lasso算法进一步缩减候选位点,选取在500次Lasso算法中出现500次的位点作为具有显著诊断能力的位点。从CFEA数据库中获取14组健康人群的血浆cfDNA甲基化450K数据,将其作为血浆背景的依据,排除选择的位点中血浆背景干扰较大的位点,采用余下的位点,在训练集中采用Lasso算法构建胰腺癌模型。
1.2.7 胰腺癌模型的诊断性能与预后性能的评估
《意见》坚持市场主导、政府引导、精准施策的基本原则,以供给侧结构性改革为主线,提出了推动绿色餐饮发展的主要任务:一是健全绿色餐饮标准体系,二是构建大众化绿色餐饮服务体系,三是促进绿色餐饮产业化发展,四是培育绿色餐饮主体,五是倡导绿色发展理念。
通过绘制受试者工作特征(receiver operating characteristic,ROC)曲线计算曲线下面积(area under the curve,AUC),计算特异度和敏感度以评估模型的诊断效能。除了使用训练集和测试集评估模型的效能以外,为防止模型产生过拟合,另从TCGA数据库中获取一组临床信息齐全的胰腺癌与癌旁组织甲基化450K样本,包括184个胰腺癌组织样本和10个癌旁组织样本,作为独立测试集评估模型的诊断效能。通过绘制Kaplan-Meier法绘制生存曲线以及多重时间依赖性ROC曲线,在独立测试集中评估模型的预后预测能力。此外,采用多因素COX回归分析胰腺癌预后相关因素。
1.2.8 数据处理平台
本研究的测序数据均采用Linux Mint 17.3 Rosa系统进行分析,包括高通量测序数据的质检、预处理、比对、去重和校峰等。下游分析均采用R version 4.0.2软件,包括差异甲基化分析、模型构建和效能评估等。
2 结果
2.1 血浆cfDNA中DMRs的筛选结果
共筛选获得857个差异高甲基化区域和933个差异低甲基化区域(图1A)。经过注释后,发现共有26.13%的DMRs位于启动子区(图1B),其中包括高甲基化区域225个和低甲基化区域243个。

Fig.1 The results of differential methylation regions (DMRs) analysis of plasma cell-free DNA (cfDNA).A:The heatmap of all DMRs (P<0.05,|logFC|>1),in which the blue ones are DMRs hypomethylated in pancreatic cancer patients’ plasma cfDNA and the red ones are DMRs hypermethylated in pancreatic cancer patients’ plasma cfDNA.B:The annotation results of DMRs.图1 血浆cfDNA差异甲基化分析结果(A)DMRs(P<0.05,|logFC|>1)热图和DMRs注释结果(B)
2.2 胰腺癌组织和癌旁组织DMPs筛选结果
从胰腺癌组织和癌旁组织样本中共筛选获得7 630个高甲基化位点以及8 763个低甲基化位点(图2)。其中,位于启动子区的高甲基化位点2 186个,低甲基化位点1 190个。
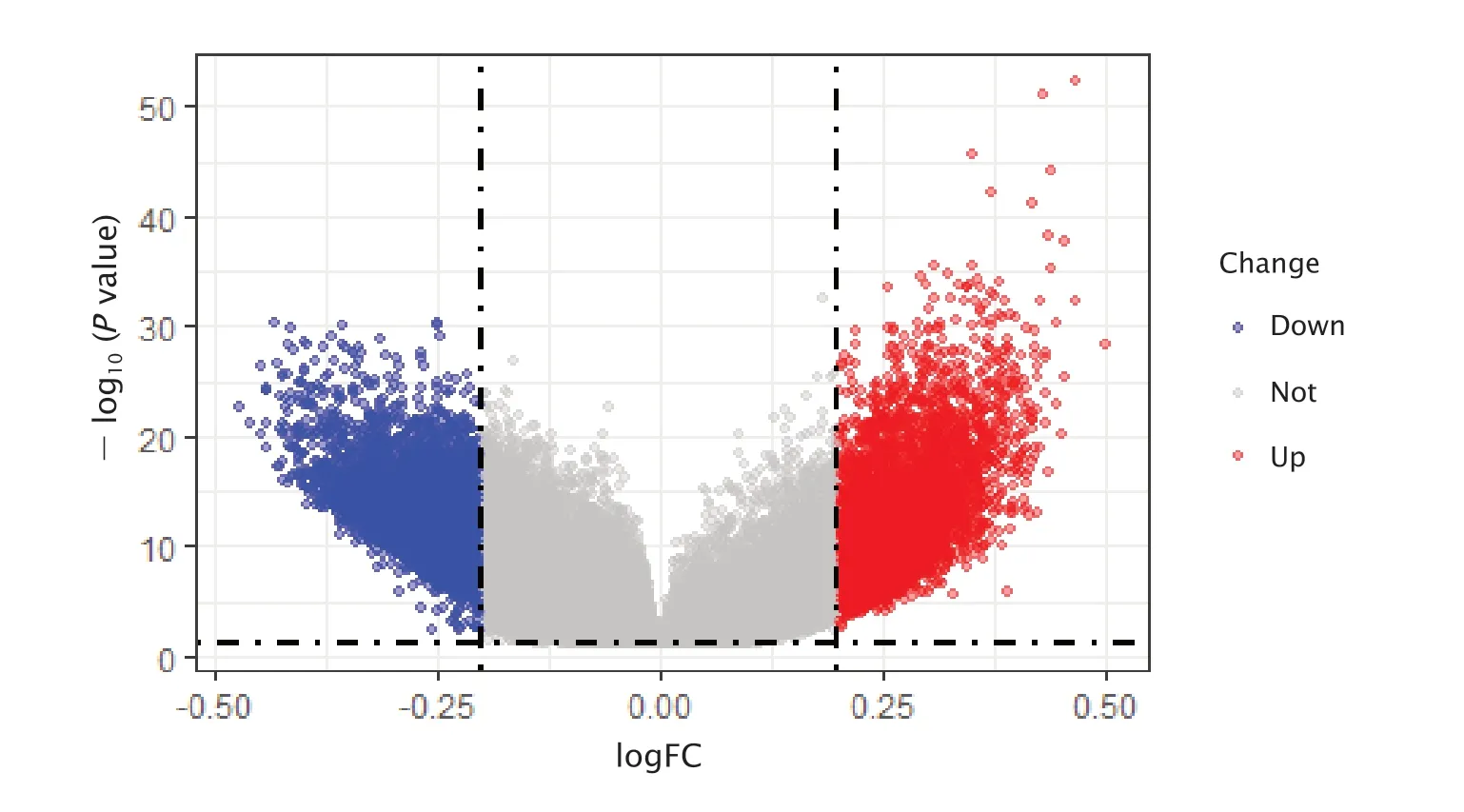
Fig.2 The volcano plot of all differentially methylated positions (DMPs) (P<0.05,|Δβ|>0.2) in pancreatic cancer tissues,where blue dots are those methylation sites hypomethylated in pancreatic cancer tissues,and red dots are those methylation sites hypermethylated in pancreatic cancer tissues.图2 胰腺癌组织DMPs(P<0.05,|Δβ|>0.2)的火山图
2.3 胰腺癌模型的构建
将胰腺癌血浆cfDNA与组织样本联合进行分析,共获得41个具有潜在诊断与预后预测能力的血浆cfDNA候选甲基化位点。通过500次Lasso迭代算法进一步筛选,留下6个候选位点,包括3个高甲基化位点(图3A)和3个低甲基化位点(图3B)。对这6个位点进行血浆背景干扰的排除,其中甲基化位点cg12951282在胰腺癌组织和健康人群血浆cfDNA中都表现出明显的低甲基化状态,存在明显的干扰,因此将其排除(图3D)。使用剩余的5个位点进行胰腺癌模型的构建。基于Lasso算法构建的胰腺癌模型的基本信息见表1。

Fig.3 Screening results of methylation sites.A:The methylation status of 3 hypermethylated sites.B:The methylation status of 3 hypomethylated sites.C:The methylation status in plasma background of 3 hypermethylated sites.D:The methylation status in plasma background of 3 hypomethylated sites.图3 甲基化位点的筛选结果

表1 胰腺癌甲基化分析模型的基本信息Table Basic information of methylation analysis model for pancreatic cancer
2.4 胰腺癌模型诊断效能的评估结果
从箱线图和ROC曲线来看,该诊断模型在训练集和测试集中都可以很好地鉴别胰腺癌患者与健康人群(图4)。在训练集中的AUC值为0.998(图4B),特异度为0.962,敏感度为0.750;在测试集中的AUC为0.997(图4D),特异度为0.987,敏感度为0.875。由此可见,该模型具有很好的诊断效能。在独立测试集中,同样得到令人满意的结果,AUC值为0.963(图4F),特异度为0.900,敏感度为0.788。
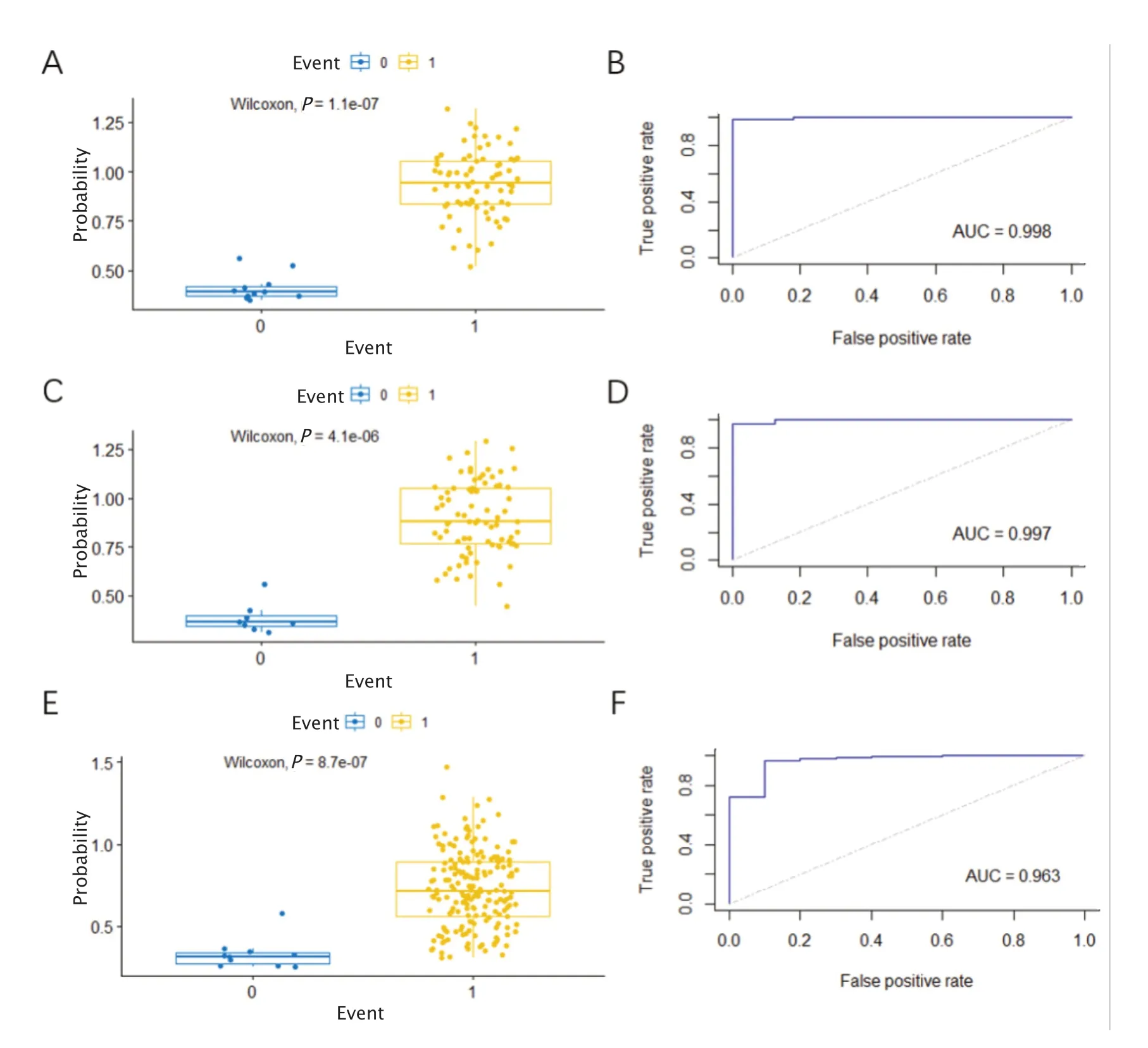
Fig.4 The diagnostic performance of pancreatic cancer model in training,test and independent test sets.A:The box plot of model in the training set.B:The receiver operating characteristic (ROC) curve of model in the training set.C:The box plot of model in the test set.D:The ROC curve of model in the test set.E:The box plot of model in the independent test set.F:The ROC curve of model in the independent test set.AUC:Area under the curve.图4 胰腺癌模型在训练集、测试集和独立测试集中的诊断效能
2.5 胰腺癌模型对预后的预测效能评估结果
Kaplan-Meier曲线显示,胰腺癌模型可以有效地将胰腺癌患者分为高风险组和低风险组(P=0.002)(图5A)。此外,该模型可以较为准确地预测胰腺癌患者1、3和5年的生存情况,AUC值分别为0.61、0.71和0.74(图5B)。多因素COX回归分析显示,种族、年龄、性别和肿瘤分期不是胰腺癌患者预后的独立预测因素(P>0.05,图6)。
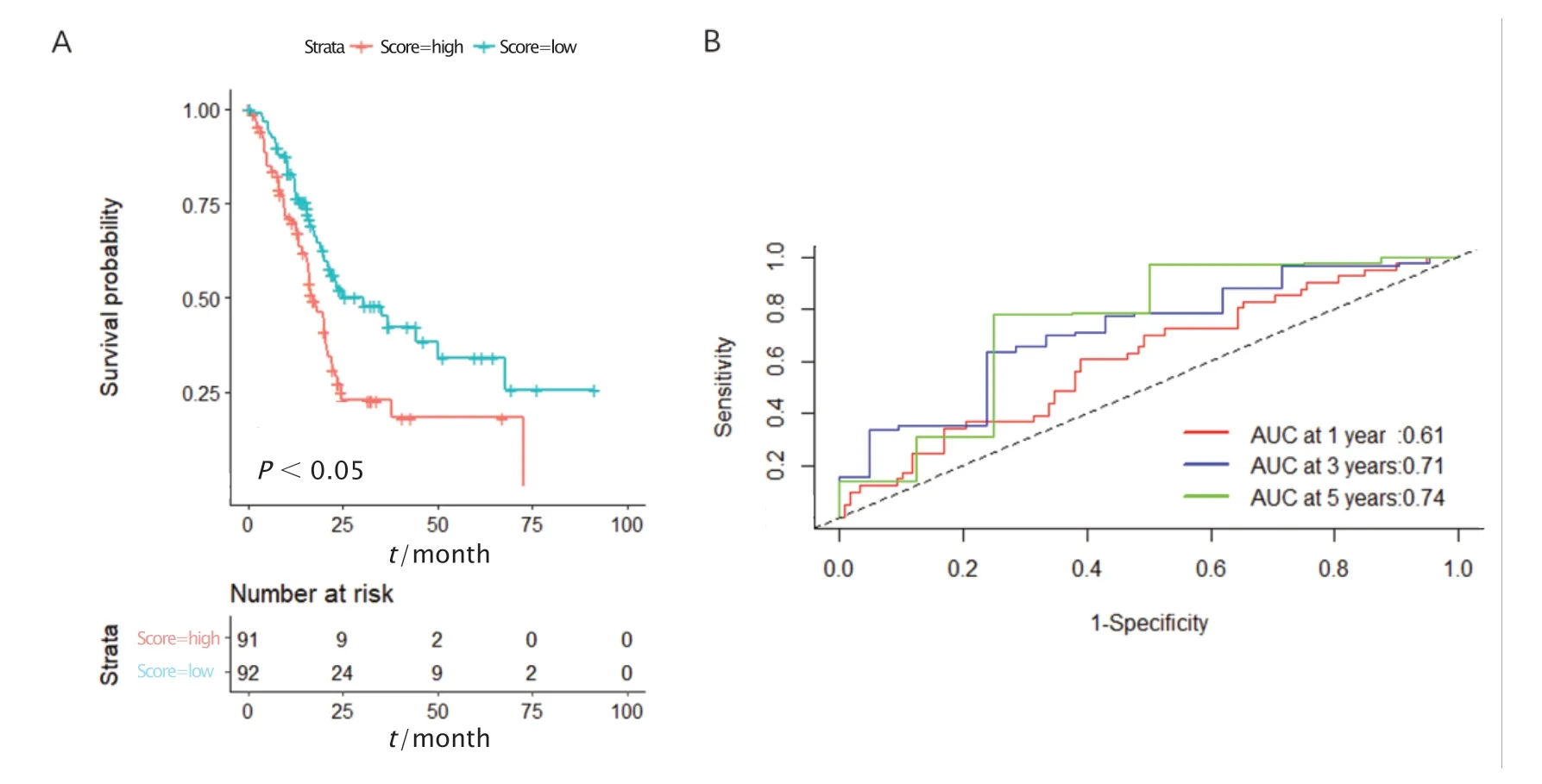
Fig.5 The ability of pancreatic cancer model to predict prognosis in the independent test set.A:The Kaplan-Meier survival curves of the model.B:The time-dependent receiver operating characteristic (ROC)curve of the model.AUC:Area under the curve.

Fig.6 The forest plot of prognostic factors in patients with pancreatic cancer.图6 胰腺癌患者预后预测因素的森林图
3 讨论
液体活检技术是肿瘤早期检测领域最具应用前景的技术之一,可以在微创条件下实现对肿瘤患者进行较为全面的早期筛查。相较于一般的基于基因组特征的液体活检技术,基于cfDNA甲基化的筛查方式能够在更早期鉴别肿瘤患者和健康人群,其表观遗传特征也更为普遍和稳定[10]。本研究使用胰腺癌患者和健康人群的血浆cfDNA甲基化数据,与胰腺癌组织和癌旁组织的甲基化450K数据进行联合分析,构建了具有较好的诊断和预后预测能力的模型。
在构建模型时,综合了胰腺癌患者cfDNA的高甲基化特征和低甲基化特征,更为全面地揭示了胰腺癌患者与健康人群的差异,并且具有更好的诊断和预后预测能力。既往研究在构建胰腺癌诊断模型时,大多数仅考虑胰腺癌患者cfDNA高甲基化特征,或是仅考虑低甲基化特征。MELNIKOV等[11]的模型基于5个基因启动子区的低甲基化,而YI等[12]、HENRIKSEN等[13]和LI等[14]的研究都聚焦于一些基因的高甲基化特征,这可能导致部分特征的丢失,造成诊断和预后预测能力的不足。同时,此前的模型大多仅具有诊断或预测预后的能力,而无法同时实现对胰腺癌患者的诊断和预后预测。本研究构建的模型不仅具有理想的诊断效能,还能对胰腺癌患者的预后进行预测,显著提高了模型的应用价值。
应用胰腺癌血浆cfDNA甲基化预测模型筛选到5个胰腺癌血浆cfDNA甲基化位点,分别位于GALNT9、CACNA1H、ADAMTS2、THBS1和KIAA1671基因的启动子区,其中GALNT9、CACNA1H和ADAMTS2基因的启动子区呈现高甲基化趋势,而THBS1和KIAA1671基因的启动子区呈现低甲基化趋势。在这些基因中,ADAMTS2[15]和THBS1[11]基因在胰腺癌患者中呈现出与既往研报道的相同的甲基化趋势,表明ADAMTS2基因的高甲基化和THBS1基因的低甲基化是胰腺癌的标志物之一,而其他基因在胰腺癌甲基化相关研究中尚未见报道。
本研究构建的模型诊断胰腺癌的敏感度为87.4%,特异度为84.6%,AUC值为0.962。目前临床上应用的胰腺癌生物学标志物是CA19-9。KATHERINE等[16]的研究中,对胰腺癌患者诊断的敏感度为78.2%,特异度为82.8%,都明显低于本模型。而癌胚抗原作为一种常用的癌症生物标志物,在胰腺癌患者中的敏感度和特异度也较低,仅为44.2%和84.8%。MELNIKOV等[11]构建了5个基因的低甲基化特征模型,在尚无独立测试集的情况下,区分胰腺癌患者与健康人群的敏感度和特异度分别仅为76%和59%。YI等[12]发现了2个高甲基化标志物,诊断胰腺癌的敏感度为81%,特异度为85%。HENRIKSEN等[13]基于8个基因的高甲基化特征,诊断胰腺癌的敏感度为76%,特异度为83%,但未经过测试集的验证。
综上所述,本研究构建的cfDNA甲基化诊断模型有望提高胰腺癌的诊断效能,其敏感度高于癌胚抗原(44.2%)和CA19-9(78.2%)。该模型在测试集和独立测试集中均通过验证,显示出较高的敏感度和适用性。该模型还具有较好的预后预测能力。总之,基于cfDNA甲基化的液体活检技术可以有效提高胰腺癌的诊断效能,为胰腺癌的早期诊断提供新的思路和理论依据。
