紫苏叶绿体全基因组特征分析
2021-03-18许冬梅贾小云王文斌
梁 璇,李 鹏,许冬梅,贾小云,王文斌
(山西农业大学生命科学学院,山西太谷 030801)
紫苏(Perilla frutescens(L.)Britton)又名赤苏、红紫苏、香苏,是唇形科紫苏属下的唯一种,一年生草本植物,是我国传统的药食两用植物,具有超过2 000 a 的栽培历史。紫苏的茎、叶和种子均可入药,其叶又称苏叶,具有解表散寒、行气和胃功效;其果实紫苏子具有降气消痰、平喘、润肠功效;紫苏梗有理气宽中、止痛、安胎功效。
叶绿体是植物体内重要的细胞器,不仅通过光合作用为植物的生长发育提供必要能量,还拥有半自主性遗传的基因组,可进行半保留复制。同核基因组相比,叶绿体基因组长度较短,易于提取纯化,且序列高度保守,SSR 位点丰富,在揭示物种起源、进化演化及确定不同物种之间的亲缘关系等方面具有重要的参考价值[1]。因此,对紫苏叶绿体基因组的特征分析是后续系统发育研究和基因工程研究的前提。近年来,关于不同物种叶绿体基因组的研究日益增多,已有1 000 多种叶绿体基因组序列被公布,如蒙古韭[2]、黄芩[3]、紫花苜蓿[4]、竹子[5]等。这些研究均对不同物种的叶绿体基因组进行了SSR 分布规律、密码子使用频率、密码子偏好性及RNA 编辑位点等多方面的分析,但是目前为止关于紫苏叶绿体基因组的研究分析还鲜有报道。
有研究发现,被子植物叶绿体基因组DNA(Chloroplast DNA,cpDNA)多为闭合的双链环状四分体结构,包括一个大单拷贝区(Large Single Copy,LSC)和一个小单拷贝区(Small Single Copy,SSC)以及2 个序列相同但方向相反的反向重复区(Inverted Repeat,IRA 和IRB),且IR 和单拷贝区交替排列[3]。不同植物叶绿体基因组大小差异主要表现在IRA和IRB 区的长度和方向变化上[6]。真核生物中重复序列广泛分布但原核生物中却鲜有出现,这是进化的必然结果,重复序列能够起到保护编码序列、产生进化的动力并形成新基因等作用[7]。重复序列是指在基因组中不同位置出现的相同或对称性片段,基因重复类型有4 种,即F 型(正向重复)、C 型(完全重复)、P 型(回文重复)和R 型(反向重复)。
微卫星序列,又称简单重复序列(Simple Sequence Repeat,SSR),随机均匀分布于基因组的各个区域,由于其高度多态性和共显性,被广泛应用于生物学的多个领域[8-9]。叶绿体SSR 标记技术作为一种新型高效的分子标记技术,不仅具有叶绿体DNA 母系遗传几乎不发生重组的优点,而且还具有SSR 的高突变性,是目前发展前景良好且广受欢迎的标记技术。密码子偏好性(Codon bias)具有种族特异性,能够降低外源基因导入宿主细胞后的表达,提高目的基因在叶绿体中的表达量,使外源基因更加高效稳定地表达[10]。RNA 编辑是发生在大多数有机生物体的线粒体、叶绿体和细胞核的一种转录水平调控,是在DNA 转录为RNA 的过程中核苷酸发生改变,进而改变mRNA 上所携带的遗传信息,从而改变翻译后氨基酸的序列,最终影响蛋白质功能变化的过程。叶绿体RNA 编辑在高等植物中广泛存在,不仅能提高转录的稳定性,而且参与叶绿体基因的表达调控,使一个基因序列产生不同的蛋白产物,是扩展其原有遗传信息的一种重要方式[11]。
本研究分析了紫苏叶绿体基因组的重复序列、SSR、密码子偏好性,并对其RNA 编辑位点进行了预测,旨在为紫苏的种质资源鉴定、遗传育种和品种改良提供重要的理论依据[12],为紫苏叶绿体RNA编辑的生物学功能和作用机制研究奠定基础。
1 材料和方法
1.1 序列数据
紫苏叶绿体全基因组序列下载于NCBI 数据库(GenBank 号为KT220691.1),序列全长152 656 bp,注释信息显示其包含88 个蛋白编码基因,从中筛选出编码区序列长度大于300 bp 的CDS 序列以减小误差[13],最终获得符合条件的59 条CDS 序列。
1.2 叶绿体基因组重复序列和SSR 分析
紫苏叶绿体重复序列和SSR 分析及相关参数设置参照文献[14]的方法进行。
1.3 密码子使用偏好性分析
使用CodonW1.4.2 程序对紫苏叶绿体基因组中59 条CDS 序列的氨基酸使用频率、有效密码子数(Effective Number of Codon,ENC)以及密码子相对使用频率(RelativeSynonymousCodonUsage,RSCU)进行统计及偏好性分析。
1.4 紫苏RNA 编辑位点预测
使用Prep-Cp(http://prep.unl.edu/)对紫苏叶绿体基因组序列进行RNA 编辑位点的预测,设置参数阈值(Cutoff value)为0.8,以保证其预测的准确性。
2 结果与分析
2.1 紫苏叶绿体基因组重复序列分析
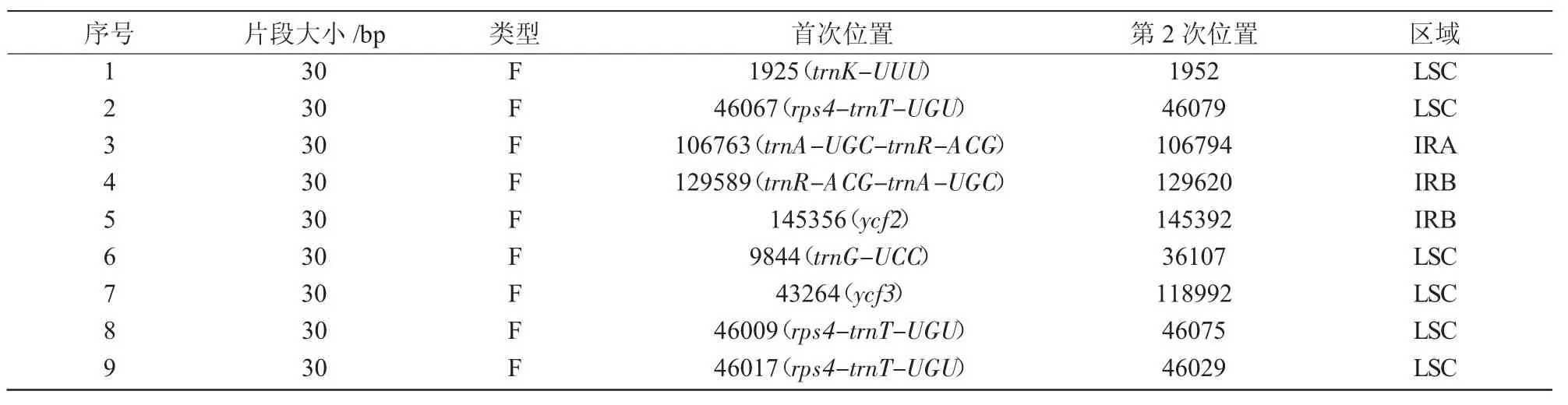
表1 紫苏叶绿体基因组的重复序列

续表1
使用REPuter 程序,在紫苏叶绿体基因组中检测到3 种类型的重复序列共60 个,主要为F(正向重复)和P(回文重复),分别占比50.0%和48.33%,而R(反向重复)占比仅为1.67%(表1)。
由图1 可知,重复序列在LSC 区和IRA 区分布最多,各25 个,占比均为41.67%;在IRB 区分布较少,为8 个,占比为13.33%;在SSC 区分布最少,为2 个,占比为3.33%。正向重复在LSC、IRA 和IRB 区分别有13、9、8 个,在SSC 区无分布;回文重复在LSC区、IRA 区和SSC 区分别有11、16、2 个,在IRB 区无分布;反向重复仅有1 个,分布于LSC 区。由此可见,重复序列在4 个区域均有分布但分布极不均衡。
2.2 紫苏叶绿体基因组SSR 分析
使用MISA 软件,共检测到44 个SSR 位点,由A/T 组成的SSR 占总数的75%;单碱基重复序列28个,占比最多(63.64%),其中,26 个单碱基重复均由A 或T 组成(表2),表明SSRs 的碱基组成偏向使用A/T 碱基。
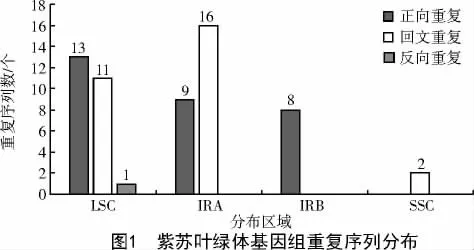
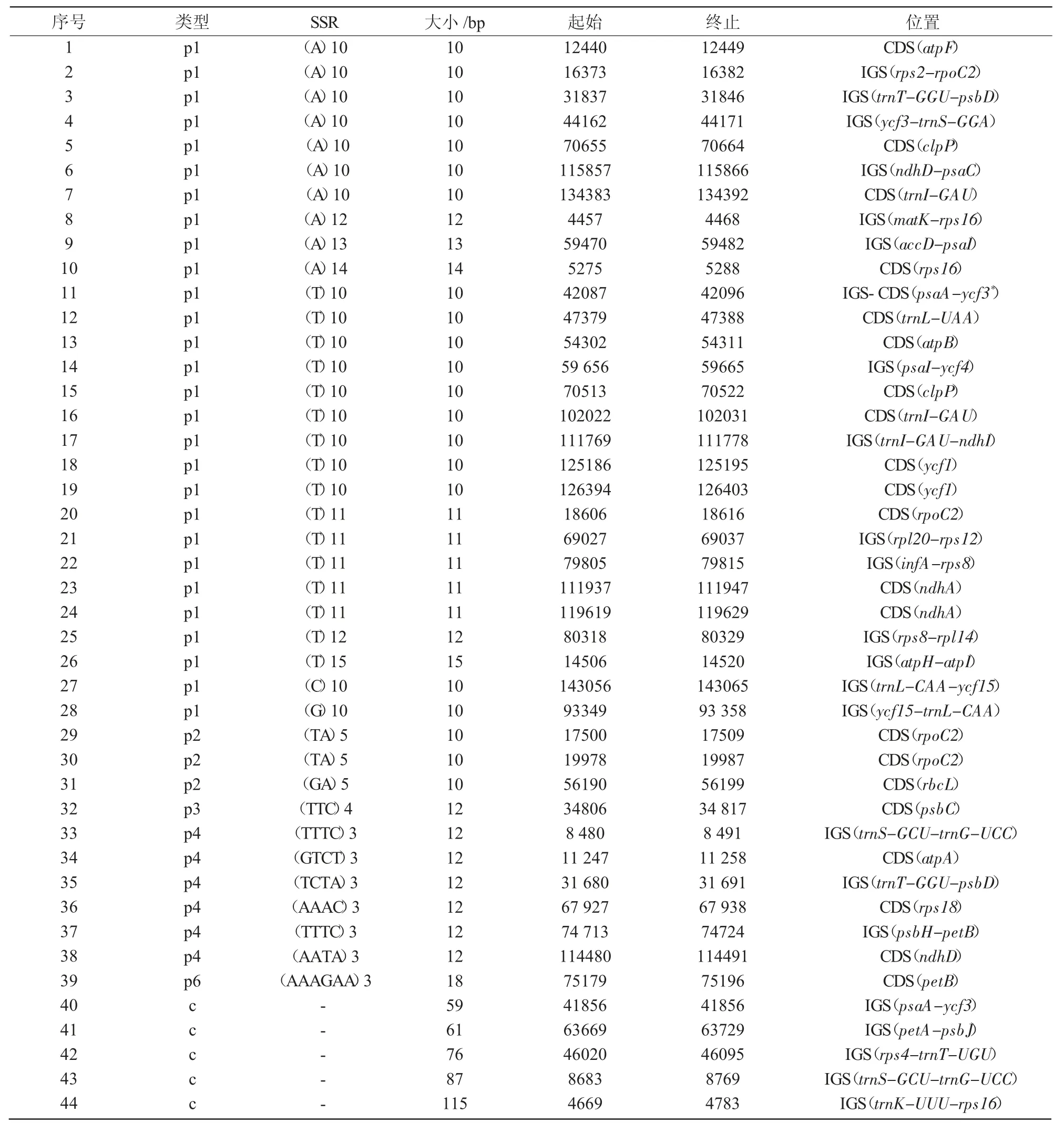
表2 紫苏叶绿体基因组中的SSR
由表3 可知,28 个单碱基重复中,14 个位于IGS 区,13 个位于CDS 区,1 个位于CDS-IGS 区;3 个2 碱基重复、1 个3 碱基重复、1 个6 碱基重复均分布于CDS 区;6 个4 碱基重复均匀分布于IGS区和CDS 区;5 个复合SSR 则均分布于IGS 区,表明SSR 位点以单碱基重复为主。总体来看,44 个SSR 位点中22 个位于IGS 区,21 个位于CDS 区,只有一个单碱基重复的SSR 位点位于CDS-IGS区,表明SSR 分布集中在IGS 区和CDS 区。
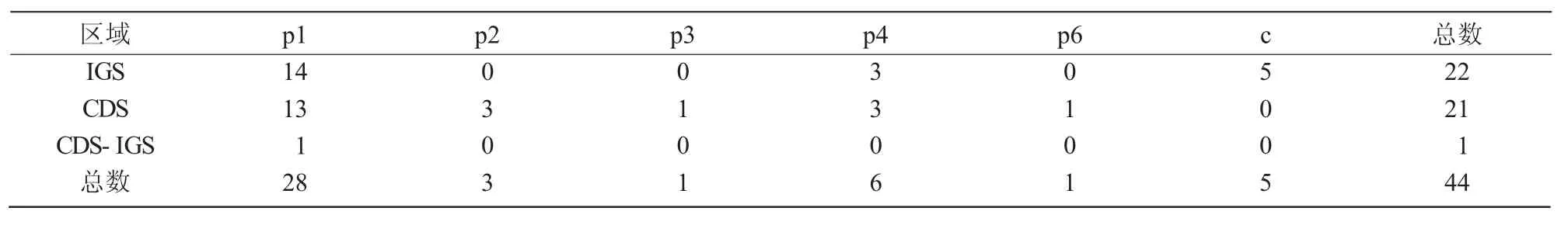
表3 紫苏叶绿体基因组中SSR 分布
2.3 紫苏叶绿体基因组密码子偏好性分析
对紫苏叶绿体基因组中各氨基酸的RSCU 分析结果显示(表4),高频密码子(RSCU>1)共有31个,其中,16 个以U 结尾,13 个以A 结尾,以G和C 结尾的密码子各1 个,说明紫苏叶绿体基因组密码子偏好以A 和U 结尾。编码亮氨酸(L)的密码子使用频率最高,为10.68%;异亮氨酸(I)次之,为8.46%;而编码半胱氨酸(C)的密码子使用频率最低,为1.15%。

表4 紫苏同义密码子使用情况
由表5 可知,紫苏叶绿体59 条CDS 序列的ENC 取值在37.77~59.70,均值为49.97。取44 作为本研究中判定偏好性强弱的值[14],紫苏ENC>44的有48 个,表明其密码子偏好性较弱。

表5 紫苏蛋白编码基因密码子ENC 值比较
2.4 紫苏RNA 编辑位点预测
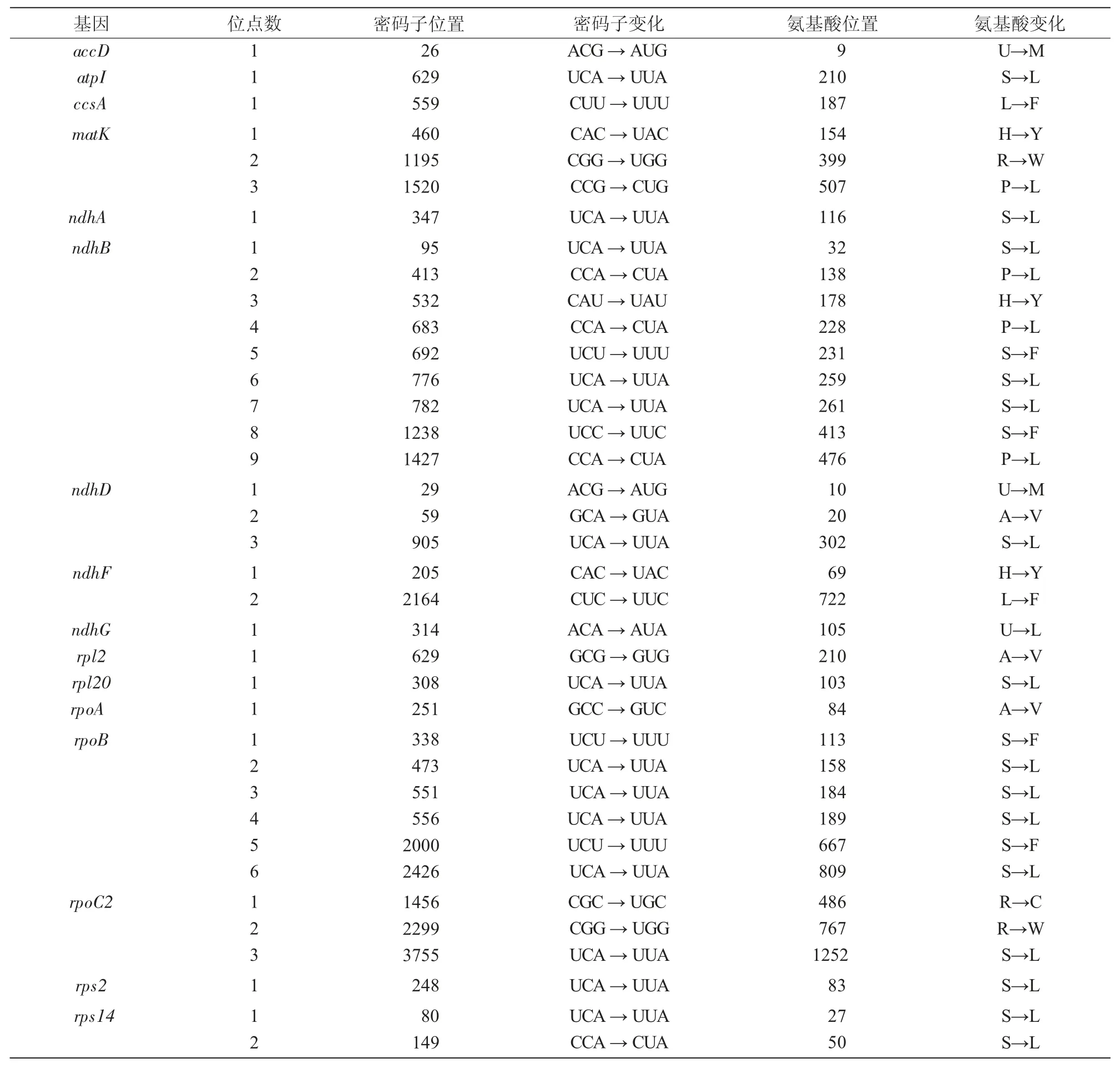
表6 紫苏叶绿体基因组RNA 编辑位点预测
使用Prep-cp 软件对紫苏叶绿体基因组的88 个蛋白编码基因进行分析,共预测到37 个RNA 编辑位点,分布于16 个基因上(表6),且均为特定的碱基C 脱氨基变成U。密码子第1 位碱基上的RNA编辑位点有8 个,第2 位碱基上有29 个,而第3 位碱基上没有发现RNA 编辑位点。RNA 编辑位点最多的是ndhB,有9 个;rpoB 次之,有6 个。此外,这些编辑位点中共有10 种氨基酸转变类型,其中,S→L(15 个)、P→L(4 个)、S→F(4 个)、H→Y(3 个)、R→W(2 个)、U→L(1 个)是亲水性氨基酸变成疏水性氨基酸,有29 个,占比约78.38%;由疏水性变成亲水性的氨基酸是L→F(2 个),占比约5.41%;其他氨基酸转变类型有A→V(3 个)、U→M(2 个)、R→C(1 个)。所以,蛋白质一级结构大部分是由亲水性转变为疏水性,这将增加蛋白质的疏水性。
3 结论与讨论
目前,有关植物叶绿体基因组的研究受到了广大科研工作者的密切关注。重复序列对基因的转录调控、蛋白翻译、染色体的形成和代谢调节等方面均有影响,重复序列对物种的进化、物种内基因的遗传和变异有着深远的影响。基因组中的重复序列大多分布于非编码区,这是由于在物种进化过程中,生物体仅保留尽可能少的遗传信息以提高其遗传效率。叶绿体中光系统Ⅰ(psa)和光系统Ⅱ(psb)等与光合作用有关的基因多数分布于SSC 和LSC区,编码rRNA 的基因(rps)多数分布于IRA 和IRB区[15],因此,位于LSC 区和IRA 区的50 个重复序列可能与紫苏叶绿体的光合作用和rRNA 的编码有关。
紫苏叶绿体44 个SSR 位点中由A/T 组成的占75%,这与玄参叶绿体基因组的研究中,44 个SSR位点中仅有一个含G/C 的结果不相符,推测可能是由于物种差异所导致的[16]。由于A-T 键比G-C 键少一个氢键,导致A-T 键较G-C 键更容易断裂,因此,叶绿体基因组SSR 中A-T 键出现的概率更大[16]。紫苏叶绿体SSR 主要分布于IGS 区和CDS 区,这与烟草叶绿体基因组的研究中,SSR 几乎全部位于非翻译区(UTR),而内含子和外显子区域几乎没有分布的结果相一致[1]。这一结果也和生物自身的进化过程一致,即在物种的进化过程中,核酸所处位置的不同会使其承受的选择压力有很大差异,SSR的分布也会受到影响,进而导致外显子区域出现SSR 的概率减小,而在UTR 及内含子区域出现SSR的概率增加。
密码子偏好性指同义密码子的非均衡使用。密码子偏好性的研究不但可以诠释物种进化问题,而且可以用于优化外源基因的表达、预测基因功能及基因表达水平等。RSCU 指某一同义密码子使用次数的观察值与该密码子出现次数期望值的比例,当RSCU>1 时为高频密码子[17]。通过紫苏叶绿体基因组密码子的RSCU 值以及第3 位密码子上A/U/C/G这4 种碱基使用频率的分析得到31 个高频密码子,且以A/U 结尾的共有29 个。这与苦荞[18]、蒺藜苜蓿[19]、蝴蝶兰[20]以及圆锥南芥[21]等双子叶植物中叶绿体最优密码子偏好以A/U 结尾的结果相一致[22]。ENC 能够反映基因有效使用密码子种类的数目和基因密码子使用的偏好性程度。生物体的ENC 值范围在20.0~61.0,该值越靠近20 表明其偏好性越强[23]。紫苏叶绿体基因组CDS 序列的ENC 平均值为49.97,本研究以44 为区分标准,说明其密码子偏好性较弱。
RNA 编辑是指DNA 转录成mRNA 的过程中发生核苷酸的替换或改变阅读框,从而增加了基因产物的多样性而获得新的结构和功能,有利于提高蛋白质的稳定性[24]。本研究发现,发生在紫苏密码子第1 位和第2 位碱基的RNA 编辑个数分别为8、29 个,这与前人对连翘[14]、青稞[25]、大麦[26]等的研究结果一致;而且大部分由亲水性氨基酸转变为疏水性氨基酸,具有高等植物叶绿体基因组RNA 编辑的典型特性[27]。很多研究发现,这一特性可能与蛋白质中包含大量疏水性残基相关,发生在蛋白质内核中的疏水性突变较亲水性突变形成的结构更稳定,最终可能影响蛋白质的二级结构与功能,还可以扩展其遗传信息[14]。
本研究通过对紫苏叶绿体基因组59 条CDS序列(≥300 bp)的重复序列、SSR、密码子偏好性进行分析,得到60 个重复序列,多以F 和P 为主,主要分布于LSC 区和IRA 区,可能参与光合作用与rRNA 的编码;44 个SSR 位点以单碱基重复为主,主要由A 或T 组成,位点多态性是区分物种差异的基础;31 个高频密码子偏好大多以A 和U 结尾。对88 个蛋白编码基因进行RNA 编辑位点预测可知,RNA 编辑位点发生在前2 位,均为C→U,使亲水性氨基酸变为疏水性氨基酸。本研究结果对紫苏的系统进化、遗传育种和生物工程育种工作具有重要意义。
