GA3介导miR171s及其靶基因VvSCLs调控葡萄种子发育的作用分析
2021-03-08王文然解振强诸葛雅贤白云赫管乐吴伟民张培安郑婷房经贵王晨
王文然,解振强,诸葛雅贤,白云赫,管乐,吴伟民,张培安,郑婷,房经贵,王晨
GA3介导miR171s及其靶基因调控葡萄种子发育的作用分析
王文然1,解振强2,诸葛雅贤1,白云赫1,管乐1,吴伟民3,张培安1,郑婷1,房经贵1,王晨1
1南京农业大学园艺学院,南京 210095;2江苏农林职业技术学院,江苏镇江 212499;3江苏省农业科学院园艺研究所,南京 210014
【】鉴定vvi-miR171s成员及其靶基因,明确vvi-miR171s成员及其靶基因在葡萄种子发育中的重要作用及其响应赤霉素(Gibberellin,GA)调控种子发育的表达模式。以‘魏可’葡萄(‘Wink’)果实为试材,利用miR-RACE技术鉴定vvi-miR171a/b/e/f/g/h的成熟体序列;通过PsRNATarget软件预测vvi-miR171s的靶基因,利用生物信息学软件对其进行染色体定位、系统进化树、基因结构、保守结构域及motif分析;通过启动子作用元件分析预测vvi-miR171s及其靶基因的潜在功能;采用RLM-RACE和PPM-RACE验证vvi-miR171s对靶基因的裂解作用;利用qRT-PCR鉴定其应答外源GA3在葡萄种子区中的表达模式。从‘魏可’葡萄果实中鉴定并克隆了6条葡萄vvi-miR171s成熟体序列,并用RLM-RACE和PPM-RACE鉴定到3条靶基因。靶基因的生物信息学分析显示,2与苹果、桃子和樱桃同源基因遗传距离较近,而则与拟南芥、苹果、桃子和樱桃中的同源基因遗传距离相同,且的基因结构也与进化树中亲缘关系较近的基因相似;SCLs蛋白均具有GRAS结构域,鉴定到5个与GRAS结构域对应的保守motif,且它们含有的motif元件类型及排列顺序均相似,表明SCL家族结构较为保守。vvi-miR171s及靶基因的启动子均具有大量的GA、水杨酸(Salicylic acid,SA)和种子发育相关作用元件,表明它们可能参与响应GA、SA和种子发育过程;qRT-PCR表达分析显示,GA3强烈抑制了种子区vvi-miR171a的表达,但同时显著上调种子区的和表达;vvi-miR171a和在对照组和GA3处理后的种子/种子区均呈强烈的负相关,表明GA3可能在葡萄种子/种子区显著增强vvi-miR171a对的负调控作用,从而介导葡萄种子发育。在‘魏可’葡萄中鉴定到vvi-miR171a/b/e/f/g/h 6个成员;均可裂解3个靶基因;vvi-miR171a-可能作为主要的调控途径响应赤霉素并参与调控葡萄种子发育。
葡萄;vvi-miR171s;;种子发育;赤霉素
0 引言
【研究意义】葡萄(L.)是世界四大水果之一,其在鲜食、加工方面均具有极重要的经济价值。无核是鲜食和制干葡萄重要的优良性状,无核因其食用方便,风味口感俱佳,倍受消费者青睐,目前已成为重要的育种目标,也是能够显著提高葡萄商品价值的首要性状。葡萄种子由种皮、胚乳和胚构成,胚是种子的重要组成结构。细胞学水平的研究发现,胚败育是导致葡萄无核化的重要原因之一[1],胚的各部分都是由胚性细胞组成,因此胚性细胞能否正常发育是影响胚发育及无核的重要因素。【前人研究进展】目前在萝卜[2]、百合(DC. Fisch)[3]、龙眼()[4]、落叶松()[5]及柑橘()[1]中均证实miR171介导SCL6转录因子调控植物胚性细胞发育;同时发现杏(L.)PsmiR171-是调控杏核发育的重要miRNA[6]。这些研究均表明,miR171介导SCL转录因子调控胚性细胞发育,而miR171家族是功能高度保守的家族[6],因此可推断vvi-miR171s有可能调控影响葡萄胚性细胞发育,进而影响种子发育。2002年在拟南芥中分离得到AtmiR171,并确定、和为其靶基因[3]。SCL6是GRAS家族SCL亚家族的成员,是植物中特有的转录因子,其功能具有多样性,可参与调控体细胞胚发生[7]、根组织发育[8]、重金属胁迫[9]、光周期的调节[10]、节间形成[11]、叶绿素合成[12]、叶片形成[13]、干旱胁迫[14]及花芽分化[15]等多种生长发育进程。目前,miRBase数据库中已收录了38种植物的miR171家族(http://www.mirbase.org/),但不同物种间家族成员数量不同,大豆的miR171家族成员数量多达21个[16],而在拟南芥[15]中仅发现3个,在酿酒葡萄‘黑比诺’中鉴定出8个成员(http:// www.mirbase.org/),说明miR171家族成员既具有保守性,也具有一定的特异性。据报道,GA可调控miR171-进而影响植物生命活动,拟南芥miR171-调节光照条件下叶绿素合成和叶片生长受GA - DELLA信号途径调控;而龙眼早期体胚发生是基于DlmiR171b对GA3的响应,从而介导表达进而调控细胞形态建成[4]。虽然目前在龙眼[4]和百合[3]等物种的研究中,均已证明某个miR171家族成员可靶向SCL转录因子调控胚性细胞发育。【本研究切入点】尚未阐明miR171家族不同成员是共同还是独立介导靶基因参与胚性细胞发育调控,更不清楚miR171家族能否应答赤霉素介导靶基因参与葡萄种子发育的调控。【拟解决的关键问题】拟通过研究vvi-miR171s应答GA3介导靶基因的作用机制,明确它们在赤霉素诱导葡萄无核过程中的作用,从表观遗传学角度阐释赤霉素介导葡萄无核的分子机制。
1 材料与方法
1.1 材料及其处理
以优质的5年生二倍体葡萄‘魏可’为试验材料,于2017年5月2日(花前10 d)用50 mg·L-1GA3浸蘸花序30 s,以清水处理为对照,在花后5 d(5 DAF)、10 d(10 DAF)、20 d(20 DAF)、30 d(20 DAF)和45 d(20 DAF)分别采集果实,并将种子(对照组)/种子区(GA3处理后的无核果实中与对照组种子相对应的组织部位)分离后液氮速冻,存于-80℃。
1.2 vvi-miR171s成熟体序列克隆及前体鉴定
在miRBase数据库(http://www.mirbase.org/)搜索并下载葡萄miR171家族(vvi-miR171s)的成熟体序列。根据miRBase中下载的vvi-miR171s成熟体序列,设计特异引物,利用miR-RACE技术[17]在‘魏可’葡萄果实组织中克隆及测序鉴定其精确序列。
1.3 vvi-miR171s靶基因预测
运用PSRNA Target(http://plantgrn.noble.org/v1_ psRNATarget/),对已鉴定的vvi-miR171s成熟体序列进行靶基因预测。预测时选择User-submitted small RNAs/preloaded transcripts一栏,并将最大期望(maximum expectation)设置为3.0,其他参数为默认设置。
1.4 vvi-miR171s及其靶基因的生物信息学分析
分别使用MEGA5.0软件的邻接法(Neighbor-Joining)、Gene Structure Display Server(GSDS)(http://gsds.cbi.pku.edu.cn/index.php)、SMART网站(http://smart.embl-heidelberg.de/)及MEME网站(http://meme.nbcr.net/meme/)进行系统进化树、基因序列结构、保守结构域及模序分析。启动子序列在CRIBI数据库(http://genomes.cribi.unipd.it/gb2/ gbrowse/public/vitis_vinifera_v2/)和Grape Genome Browser(http://www.genoscope.cns.fr/cgi-bin/ggb/vitis/ 12X/gbrowse/vitis/)中查询,取起始密码子(ATG)上游1 500 bp的序列或vvi-miR171s上游1 500 bp序列作为该基因的预测启动子,并在NEW PLACE网站(https://www.dna.affrc.go.jp/ PLACE/? action=newplace/)进行启动子序列的顺式作用元件分析。
1.5 检测靶裂解位点及3′末端裂解频率
用加ploy(A)尾巴的mRNA反转录合成的cDNA作为PPM-RACE【(Poly(A) polymerase-mediated 3′ RACE)poly(A)聚合酶介导3′RACE RNA】反应的模板,通用引物Gene RACE 3′为:5′-ATTCTAGAG GCCGAGGCGGCCGACATG-3′。用加接头的mRNA反转录成的cDNA作为RLM-RACE【(RNA ligase- mediated 5′ RACE)连接酶介导5′ RACE】反应的模板,通用引物Gene RACE 5′为:5′-GGACACTGACATG GACTGAAGGAGTA-3′。详细步骤参考Wang等[18]的方法。
1.6 数据处理与分析
使用Excel 2017进行数据整理,SPSS 19.0进行相关性分析。
2 结果
2.1 GA3处理后不同发育时期葡萄种子的发育状态
从图1可以看出,GA3使果实纵向拉长。5 DAF对照组和GA3处理组的果实胚珠均正常发育,无明显差异。10 DAF时,GA3处理的果实胚珠与5 DAF的几乎无差别,无明显地生长,而对照组的果实胚珠已经明显大于处理组。20 DAF、30 DAF和45 DAF均观察到对照组的果实胚珠正常发育,在45 DAF已形成坚硬的种子;而GA3处理组在45 DAF果实中种子区仅存一条细线。因此,GA3在诱导葡萄无核过程中强烈地抑制了葡萄种子的发育。
2.2 vvi-miR171s成熟体序列克隆及其序列分析
2.2.1 序列克隆及其序列比对 不同于miRBase数据库中鉴定出10个miR171家族成员,仅在‘魏可’葡萄中鉴定出vvi-miR171a/b/e/f/g/h 6个成员(图2-a),且与miRBase数据库中‘黑比诺’品种的同源序列完全一致(图2-b),表明其序列在葡萄属植物中具有高度的保守性。
2.2.2 vvi-miR171s成熟体序列进化树分析 为了解miR171s在植物中的进化特性,对比了包括葡萄在内的5个物种39条miR171s的成熟体序列,包括葡萄6条、拟南芥3条、苹果()14条、柑橘9条、桃()7条。利用MEGA 5.0构建上述植物miR171s成熟体系统发育树(图3),结果发现这5个物种的miR171s成员进化树被分为3大主枝:其中第二主枝仅有ppe-miR171e一个成员,第三主枝为crt-miR171b-5p和mdm- miR171f-5p,其余成员均在第一主枝。而在第一主枝上的vvi-miR171g和mdm-miR171i与其他成员距离较远,分别位于第二、三分枝,vvi-miR171a、vvi-miR171e、vvi-miR171f、vvi-miR171h和vvi-miR171b集中在第一主枝的第一分枝上,且该分枝的每一小枝均包含5个物种。因此,vvi-miR171a、vvi-miR171e、vvi-miR171f、vvi-miR171h和vvi-miR171b 5个成员亲缘关系较近,与vvi-miR171g的亲缘关系较远。同时发现miR171s在进化速率上有较大差异,而在同一植物中不同成员也呈现出差异性。

图1 GA3对果实种子发育的影响
2.3 vvi-miR171s的靶基因预测及其序列匹配程度分析
基于已鉴定的vvi-miR171a/b/c/d/e/f序列,利用PSRNA Target为其预测到3条靶基因,分别为、、(表1)。其中,vvi-miR171e与靶基因的错配率最低为0,vvi-miR171a、vvi-miR171b和vvi-miR171h的错配率均为0.5,vvi-miR171f的错配率为1.0,vvi-miR171g的错配率最高为4。这表明、和可能为vvi-miR171s的潜在靶基因,且vvi-miR171s不同成员对靶基因的作用强度可能存在差异。

图3 miR171s成熟体的系统发育树(圆点标识为vvi-miR171s)
2.4 vvi-miR171s的靶基因验证及其靶裂解位点分析
植物中miRNAs主要通过裂解靶基因进而调控一系列生长发育进程,因此鉴定miRNAs对靶基因的裂解作用是验证miRNAs靶基因的最有效途径。利用5′-RLM-RACE检测到vvi-miR171s对、和的裂解产物,同时发现有2个裂解位点,分别位于第9位和第12位,裂解频率分别为7/20、10/20;有3个裂解位点,分别位于第9位、第12位和第13位,裂解频率分别为13/25、11/25和1/25;有2个裂解位点,分别位于第9位和12位,裂解频率分别为3/20和20/23。这3条靶基因的裂解位点均具有位于miRNA的5′端第9位和第10位碱基之间所匹配的靶点,这些结果不仅证实了这3条基因是miR171s的真实靶基因,而且也表明它们裂解作用位点的保守性。

图4 vvi-miR171s对靶基因的裂解位点示意图
2.5 vvi-miR171s靶基因的染色体分布及基因结构分析
为了进一步了解SCL家族的基因结构及功能区域,对其进行了染色体定位(图5-a)、构建进化树(图5-b)、基因结构(图5-c)、motif(图5-d)、domain分析(图5-e),发现6个miR171s成员及3个靶基因分别定位在9条不同的染色体上。为了研究SCL家族成员间的进化关系,利用邻接法构建了及其他物种中同源基因蛋白序列的进化树(图5-b)。进化树显示,5个物种的15个SCLs基因共分为两个主枝,其中所有SCL6和SCL22分布在第一分枝上,而所有SCL15聚集在第二主枝上,这说明在进化过程中SCL6和SCL22较为相似;VvSCL6/22与桃、樱桃()和苹果中的同源基因亲缘关系较近,而VvSCL15则与拟南芥、桃、樱桃和苹果中的同源基因进化距离相同,这说明SCL家族的不同成员在进化过程中具有特异性。仅中包含1个内含子区,其他成员的基因序列中只包含UTR区和外显子区。同时发现与其亲缘关系较近的、和的外显子区域长度较为一致,明显长于亲缘关系较远的拟南芥同源基因;而5个的外显子区长度也基本相同。结合motif和domain分析发现,这15个SCL家族成员motif类型及排列顺序较为保守,且均具有GRAS结构域,因此可推测该家族功能较为保守,并在motif分析中找到15个SCL蛋白中该结构域对应的保守基序,分别为motif 5/6/8/9/10(图5-f)。
2.6 vvi-miR171s启动子的顺式作用元件分析
在vvi-miR171s及其靶基因的启动子中,富含5类顺式作用元件分别是激素、光信号、低温和干旱胁迫、组织特异性和昼夜周期节律相关作用元件(图6-a)。对比6个miR171s成员的作用元件发现,数量上,vvi-miR171h最多,而vvi-miR171g的元件数量最少;类型上,除vvi-miR171a/g含有昼夜节律元件外,其余成员的启动子均仅含有其余4类作用元件,其中激素、逆境和组织特异性相关响应元件占总数量的比重较大,而光响应元件数量较少。与vvi-miR171s类似,靶基因中仅启动子中发现1个昼夜节律相关元件,光响应元件数量最少,其他3种类型的元件数量较多。同时发现miR171s及靶基因启动子均含有大量与胚乳和花粉发育有关的顺式作用元件,这些调控元件说明vvi-miR171s及其靶基因不仅对光、低温和干旱胁迫等外界环境信号做出响应,而且还能应答激素,可能参与葡萄无核果实的生长发育。
为进一步认识vvi-miR171s及其靶基因在激素信号途径中的作用,对激素相关顺式作用元件进行分类(图6-b)。所有启动子均含有激素相关响应元件,主要包括GA、SA、生长素(Auxin,IAA)、脱落酸(Abscisic acid,ABA)、细胞分裂素(Cytokinin,CTK)及乙烯(Ethylene,ET)等激素响应元件,表明它们可应答多种激素信号进而参与调控葡萄生长发育。其中,GA、SA和IAA相关元件在9个基因启动子中均存在,而ABA响应元件在vvi-miR171a/g和中未发现,CTK相关顺式作用元件仅在中发现,ET元件则在vvi-miR171e/h和中未发现。另外,在众多激素元件中,GA和SA作用元件的数量明显多于其他激素,而GA和SA是葡萄种子发育的关键激素,特别是与无核葡萄果实的发育密切相关[19-20],表明miR171s及靶基因可能通过响应激素信号参与无核葡萄的发育过程。
2.7 vvi-miR171s及靶基因在葡萄种子发育不同时期的时空表达特征
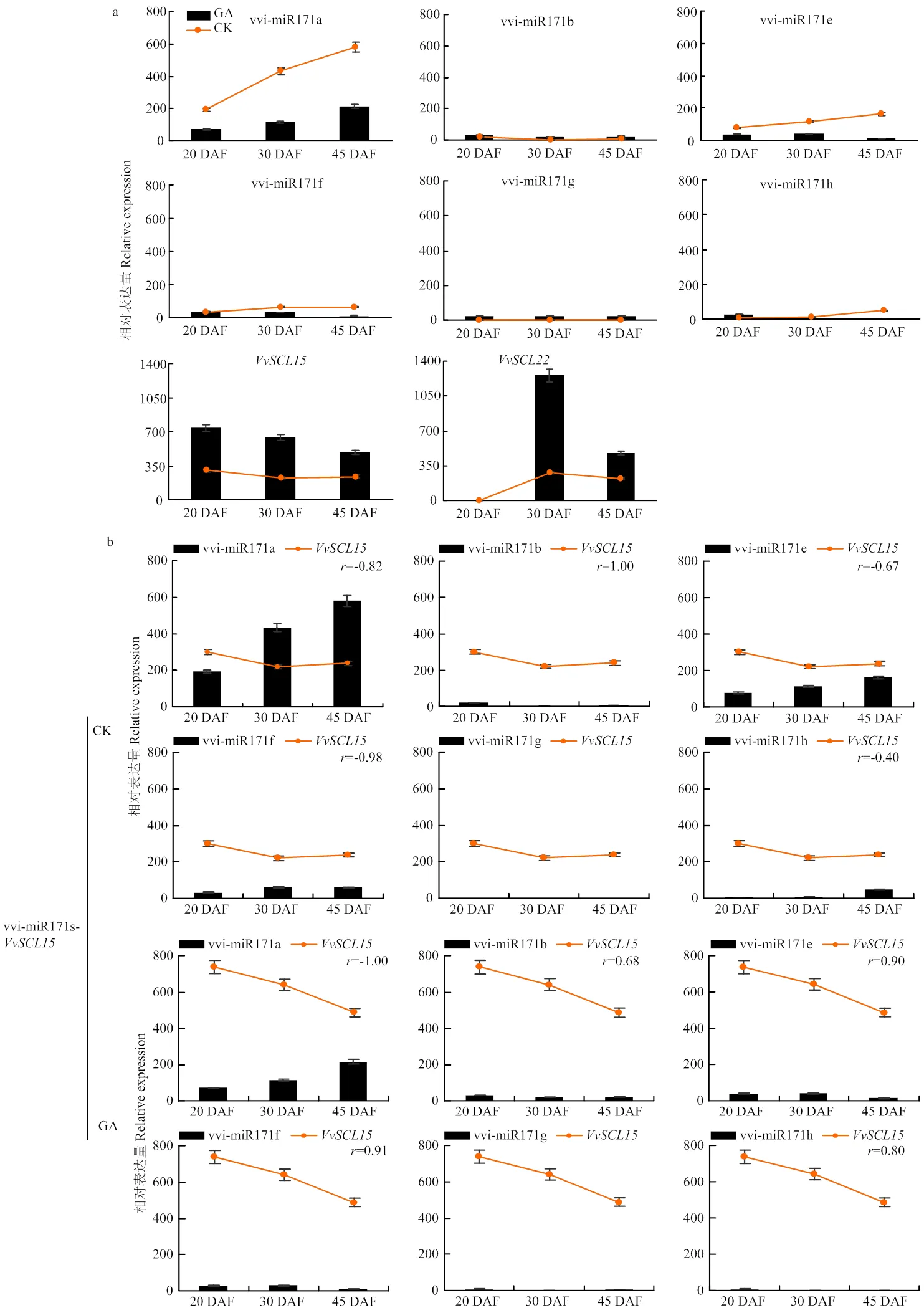
2.7.1 在葡萄种子不同发育时期时空表达分析 如图7所示,根据表达量水平可将vvi-miR171s分为3类,vvi-miR171a为最高,其次是vvi-miR171b/e/f,vvi-miR171g几乎不表达,未检测到vvi-miR171g;靶基因中仅检测到/,未检测到。/呈现完全相反的表达模式,在20 DAF检测到最高水平,在30 DAF最低;而则在20 DAF未检测到表达量,而在30 DAF最高。vvi-miR171s中除vvi-miR171b在20 DAF检测到最高水平外,其他成员均在45 DAF检测到最高水平。vvi-miR171s与/相关性分析发现(图7),vvi-miR171a/f-(=-0.82/-0.98)、vvi-miR171b-(=-1.00)具有显著负相关性,而vvi-miR171b-(=1.00)和vvi-miR171a/f-则为显著正相关(=0.83/0.98),这一结果也为vvi-miR171s成员以不同的途径和作用方式调节靶基因进而调控种子发育提供了证据;vvi-miR171a/f-、vvi-miR171b-在葡萄种子发育过程中均存在负调控关系,结合表达水平认为vvi-miR171a-为调控种子发育过程的主要成员。
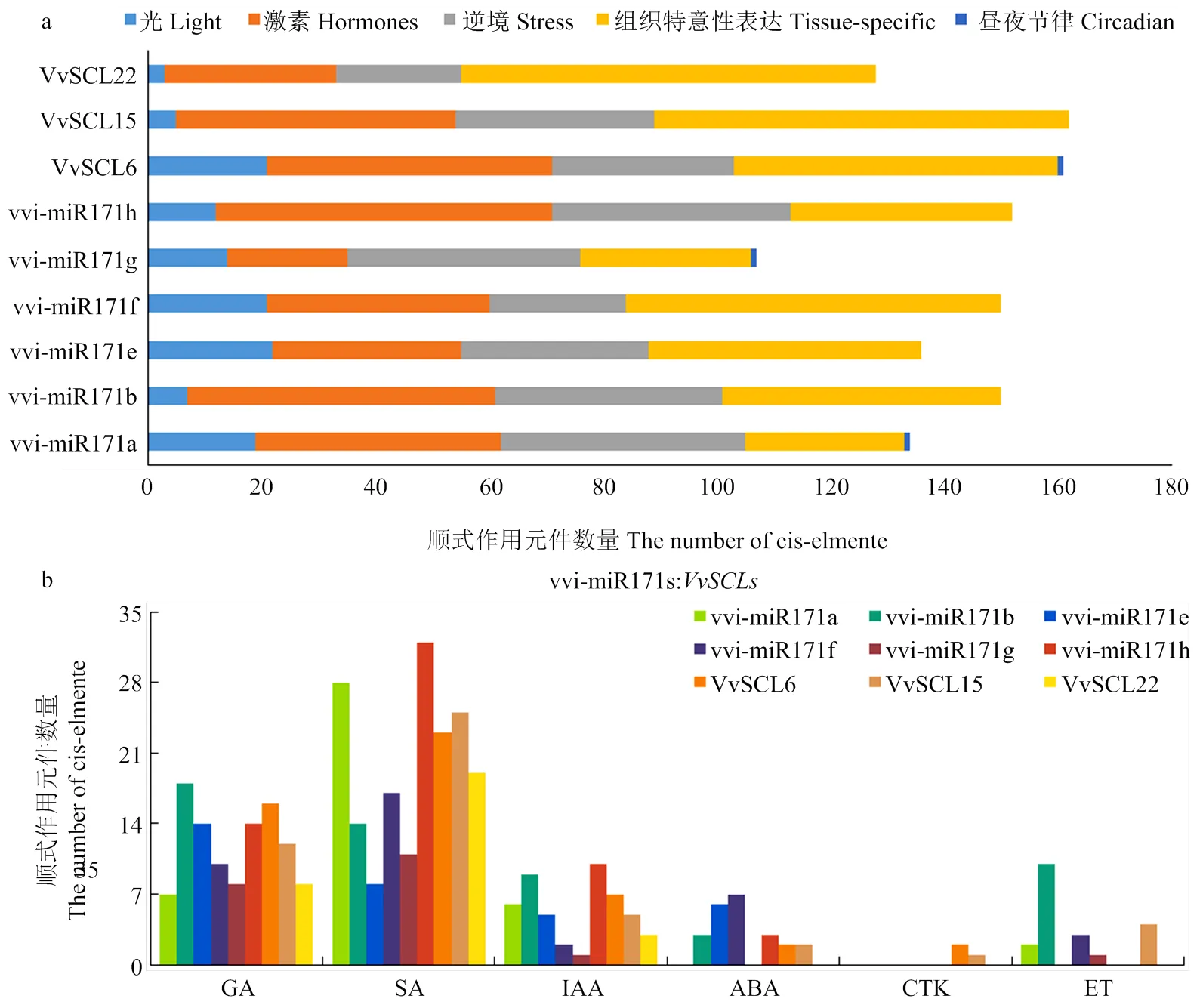
a:vvi-miR171s启动子中各类响应元件的数量;b:响应不同激素的作用元件数量
2.7.2 vvi-miR171s及靶基因应答GA3的模式分析 为进一步认识葡萄vvi-miR171s及其靶基因在种子区对外源GA3的时空响应模式,利用对照和GA3处理的种子区样品cDNA为模板,进行定量分析。图7-a为对照与GA3处理后的葡萄种子区vvi-miR171s及/的表达水平。GA3处理后种子区vvi-miR171s响应GA3的模式可分为2类,vvi- miR171a/e/f/h的表达量显著下调,而vvi-miR171b/g则在GA3处理后上调;与CK相比,的表达量均显著升高。同时相关性分析表明(图7-b、c),GA3处理后,vvi-miR171a-(=-1.00)、vvi- miR171b/g/h-(=-0.88/-0.97/-0.79)呈显著负相关;而GA3处理后种子区vvi-miR171f-由显著负相关性(=-0.98)变为显著正相关(=0.91),同时vvi-miR171/f/h-(=0.91、0.80)为显著正相关,这表明vvi-miR171s在种子发育过程中以不同的模式响应赤霉素信号。
由于miRNA家族中各个成员的作用可能存在冗余现象,因此利用vvi-miR171s的累计表达量与靶基因的表达量进行分析(图8)。值得注意的是,GA3强烈抑制了种子区vvi-miR171s的表达(图8),其中vvi-miR171a(图7-b)的下调程度远超过其他成员,表明在葡萄种子区,vvi-miR171a可能是该家族中应答GA3的主要成员。同时发现,GA3显著上调种子区的和2,且GA3处理后,vvi-miR171a-呈负调控关系,因而推测vvi-miR171a负调控是vvi-miR171s响应赤霉素信号调控种子发育的主要途径。
3 讨论
据报道,不同物种中miR171s的功能不完全一致。在拟南芥[15]、大麦[13]和烟草[21]中发现miR171c调控腋生分生组织形成;落叶松LxmiR171在胚性细胞中过表达后,可促进诱导胚性愈伤组织[22],同时LxmiR171a/b和LxmiR171c还可以调控原胚和体胚诱导阶段[23];百合Lpu-miR171a[24]在胚性愈伤组织阶段表达量最高,而Lpu-miR171b、Lda-miR171a和Lda-miR171c的表达量均在鱼雷形胚阶段出现最高值;甜橙miR171在胚性愈伤组织中有高表达,主要在柑橘细胞胚性诱导和保持过程中发挥作用[1];vvi-miR171s对葡萄花芽分化具有一定作用[25];利用STTM和CRISPR/Cas9技术,发现水稻miR171a参与叶绿素、脯氨酸的合成过程,并对植物生长势和抗旱能力有调节作用[26];SlmiR171b不仅在番茄花粉萌发过程中起负调控作用,也参与了番茄的呼吸跃变过程,并负调控果实中葡萄糖、果糖和番茄红素等营养物质的积累[27];而Lin等[6]发现杏(L.)PsmiR171-与种子发育相关,且不同成员在种子不同发育时期发挥重要作用。
本研究的靶裂解位点结果表明,、和均为vvi-miR171s的靶基因,但对不同时期种子区cDNA进行定量检测,未检测到的表达量,这可能是由于表达量很弱或具有组织、发育时期特异性。与葡萄中的其他microRNAs类似[28-29],miR171s也具有时空特异的表达模式。本研究发现,在葡萄硬核期种子区vvi-miR171a相比其他成员表达量较高,vvi-miR171a可能作为主要成员影响种子发育,说明葡萄miR171s的功能可能与拟南芥、落叶松和柑橘中的存在差异性,葡萄中极可能存在特异性的调控途径。同时,类似于拟南芥AtmiR171c[22,30]及百合Lpu-miRl71a/b[24],葡萄vvi-miR171a与靶基因之间也存在明显的负调控的关系。本研究利用3′-PPM-RACE和5′-RLM-RACE技术验证了vvi-miR171a/b/e/f/g/h对的裂解作用,这与miR171s在拟南芥[31]、落叶松[5]和龙眼[4]中通过对和的转录产物切割而进行调控的结论一致,验证了这3条靶基因的真实性;同时确定了的裂解位点均位于miR171a/b/e/f/g/h的5′端第9位和第10位碱基之间所匹配的靶点上,与拟南芥[31]中miR171靶基因的裂解位点一致,说明miR171s对靶基因裂解作用位点的保守性。
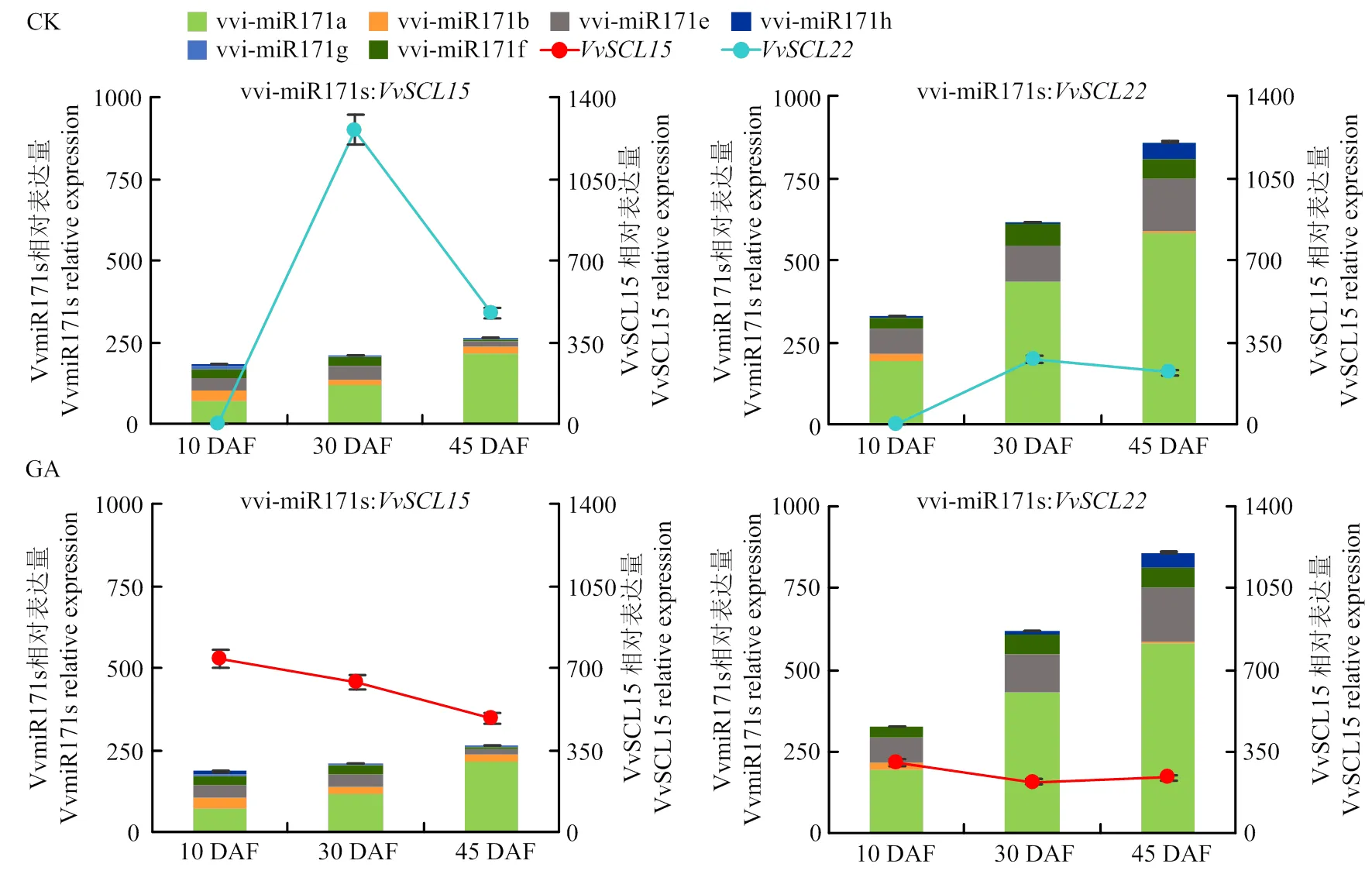
图8 种子/种子区中vvi-miR171s累计表达量与靶基因的时空表达分析、作用模式及应答GA3的模式分析
研究发现miR171及靶基因可参与GA信号转导,GA3可上调龙眼miR171b和的表达进而影响胚性细胞的发育;Ma等[12]发现miR171-SCL受拟南芥GA-DELLA信号途径调节,在叶绿素合成中起关键调控作用,这些研究均证明miR171-SCL可能通过对GA信号的响应调控植物生长进程。与龙眼[4]不同,GA3显著下调了葡萄种子区vvi-miR171a的表达,而上调了和;同时,vvi-miR171a与存在负调控关系,结合GA3高效诱导葡萄无核的效果,及vvi-miR171a可能是miR171s在硬核期调控种子发育的主要成员,推测赤霉素信号通过下调vvi-miR171a影响靶基因的表达,最终影响葡萄种子的发育。本研究初步鉴定了vvi-miR171s-可能是vvi-miR171s调控靶基因,响应赤霉素信号参与葡萄种子发育的主要途径,这将为进一步研究vvi-miR171s及靶基因响应赤霉素信号调控葡萄种子发育的分子机制研究奠定重要基础。
4 结论
在‘魏可’葡萄中鉴定到vvi-miR171a/b/e/f/g/h 6个成员,并利用3′-PPM-RACE和5′-RLM-RACE技术验证了它们对3个靶基因的裂解作用及裂解位点。通过对vvi-miR171s和在葡萄种子中的时空表达模式分析,发现vvi-miR171a-可能作为主要的调控途径响应赤霉素信号并参与调控葡萄种子发育。
[1] WU X M, LIU M Y, GE X X, XU Q, GUO W W. Stage and tissue-specific modulation of ten conserved miRNAs and their targets during somatic embryogenesis of Valencia sweet orange. Planta, 2011, 233: 495-505.
[2] ZHAI L L, XU L, WANG Y, HUANG D Q, YU R G, LIMERA C, GONG Y Q, LIU L W. Genome-Wide Identification of Embryogenesis- Associated microRNAs in Radish (L.) by High- Throughput Sequencing. Plant Molecular Biology Reporter, 2014, 32(4): 900-915.
[3] 贾娜娜. 百合体胚发生过程中miR171及其靶基因的鉴定与表达分析[D].沈阳: 沈阳农业大学, 2016.
JIA N N. Identification and expression analysis of miR171 and target geneduring somatic embryogenesis in[D]. Shenyang: Shenyang Agricultural University, 2016. (in Chinese)
[4] 苏立遥, 张帅, 陈旭,徐小萍, 赖钟雄, 林玉玲. miR171家族成员分子特性及miR171b调控靶标在龙眼体胚发生早期的表达分析. 果树学报, 2018, 35(11): 1324-1334.
SU L Y, ZHANG S, CHEN X, XU X P, LAI Z X, LIN Y L. Molecular characteristics of miR171 family members and analysis of the expression pattern of mi R171b regulatory targets during early somatic embryogenesis in longan. Journal of Fruit Science, 2018, 35(11): 1324-1334. (in Chinese)
[5] LI W F, ZHANG S G, HAN S Y, WU T, ZHANG J H, QI L W. The post-transcriptional regulation ofby miR171 during maintenance of embryogenic potential in(Lamb.) Carr. Tree Genetics & Genomes, 2014, 10: 223-229.
[6] NIU J, WANG J, AN J Y, LIU L L, LIN Z X, WANG R, WANG L B, SHI L L, LIN S Z. Integrated mRNA and miRNA transcriptome reveal a cross-talk between developing response and hormone signaling for the seed kernels of. Scientific Reports, 2016, 6: 35675.
[7] LI H Y, ZHANG J, YANG Y, JIA N N, WANG C X, SUN, H M. miR171 and its target genecontribute to embryogenic callus induction and torpedo-shaped embryo formation during somatic embryogenesis in two lily species. Plant Cell Tissue and Organ Culture, 2017, 130: 591-600.
[8] DI Laurenzio L, WYSOCKA-DILLER J, MALAMY J E, PYSH L, HELARIUTTA Y, FRESHOUR G, HAHN M G, FELDMANN K A, BENFEY P N. The SCARECROW gene regulates an asymmetric cell division that is essential or generating the radial organization of theroot. Cell, 1996, 86(3): 423-431.
[9] ZHOU Z S, HUANG S Q, YANG Z M. Bioinformatic identification and expression analysis of new microRNAs from. Biochemical & Biophysical Research Communications, 2008, 374(3): 538-542.
[10] SIRE C, MORENO A B, GARCIASHAPA M, LOPEZ-MOYA J J, SAN Segundo B. Diurnal oscillation in the accumulation ofmicroRNAs, miR167, miR168, miR171 and miR398. Febs Letters, 2009, 583(6): 1039-1044.
[11] STERNES P R, MOYLE R L. Deep sequencing reveals divergent expression patterns within the small RNA transcriptomes of cultured and vegetative tissues of sugarcane. Plant Molecular Biology Reporter, 2014, 33(4): 931-951.
[12] MA Z X, HU X P, CAI W J, HUANG W H, ZHOU X, LUO Q, YANG H Q, WANG J W, HUANG J R.miR171-targeted Scarecrow-like proteins bind to GT-elements and mediate gibberellin-regulated chlorophyll biosynthesis under light conditions. PLOS Genetics, 2014, 10(8): e1004519.
[13] CURABA J, TALBOT M, LI Z Y, HELLIWELL C. Over-expression of microRNA171 affects phase transitions and floral meristem determinancy in barley. BMC Plant Biology, 2013, 13: 6.
[14] HWANG E W, SHIN S J, YU B K, BYUN M O, KWON H B. miR171 family members are involved in drought response in., 2011, 54(1): 43-48.
[15] WANG L, MAI Y X, ZHANG Y C, LUO Q, YANG H Q. MicroRNA171c-Targeted,, andgenes regulate shoot branching in. Molecular plant, 2010, 3(5): 794-806.
[16] 王艳芳, 赵彦宏, 张萍, 周瑞莲. 大豆miR-171基因家族的进化与功能分析. 植物遗传资源学报. 2015, 16(5): 1089-1092.
WANG Y F, ZHAO Y H, ZHANG P, ZHOU R L. Molecular evolution and targets prediction of gma-miR171 gene family in Soybean. Journal of Plant Genetic Resources, 2015, 16(5): 1089-1092. (in Chinese)
[17] WANG C, WANG X C, KIBET N K, SONG C N, ZHANG C Q, LI X Y, HAN J, FANG J G. Deep sequencing of grape flower and berry Short RNA libraries for the discovery of new microRNAs and verification of the precise sequence of grape microRNAs preserved in miRBase. Physiologia Plantarum, 2011, 143(1): 64-81.
[18] Wang C, Han J, Korir N K, WANG X C, LIU H, LI X Y, LENG X P, FANG J G. Characterization of target mRNAs for grapevine microRNAs with an integrated strategy of modified RLM-RACE, newly developed PPM-RACE and qPCRs. Journal of Plant Physiology, 2013, 170(10): 943-957.
[19] CHENG C X, JIAO C, SINGER S D, GAO M, XU X Z, ZHOU Y M, LI Z, FEI Z J, WANG Y J, WANG X P. Gibberellin-induced changes in the transcriptome of grapevine (×) cv. Kyoho flowers. BMC Genomics, 2015, 16: 128.
[20] CHENG C X, XU X Z, SINGER S D, Li J, ZHANG H J, GAO M, WANG L, SONG J Y, WANG X P. Effect of GA3treatment on seed development and seed-related gene expression in grape. PLoS ONE, 2013, 8(11): e80044.
[21] 张力, 沙爱华. 烟草microRNA171c的功能分析. 植物科学学报, 2016, 34(5): 775-780.
ZHANG L, SHA A H. Functional analysis of microRNA171c in tobacco. Plant Science Journal, 2016, 34(5): 775-780. (in Chinese)
[22] ZHANG S G, ZHOU J, HAN S Y, YANG W H, LI W F, WEI H L, LI X M, QI L W. Four abiotic stress-induced miRNA families differentially regulated in the embryogenic and non-embryogenic callus tissues of. Biochemical and Biophysical Research Communications, 2010, 398: 355-360.
[23] ZHANG J H, ZHANG S G, HAN S Y, WU T, LI X M, LI F W, QI L W. Genome-wide identification of microRNAs in larch and stage-specific modulation of 11 conserved microRNAs and their targets during somatic embryogenesis. Planta, 2012, 236: 647-657.
[24] 王京. 百合体胚发生过程中pre-miR171及eTM171表达分析与载体构建[D]. 沈阳: 沈阳农业大学, 2018.
WANG J. Expression analysis of pre-miR171 and eTM171 during somatic embyogenesis and vector construction in[D]. Shenyang: Shenyang Agricultural University, 2018.
[25] 孙欣. 葡萄microRNA164和microRNA171调控其靶基因的功能分析[D]. 南京: 南京农业大学, 2015.
Sun X. Identification and interaction of grapevine micoRNA164 and micoRNA171 and their targets [D]. 2015, Nanjing: Nanjing Agricultural University, 2015. (in Chinese)
[26] 黄格格. 利用CRISPR/Cas9技术研究miR171a及靶基因功能[D]. 海口: 海南大学, 2019.
Huang G G. Study on the functions of miR171a and its target genes by using CRISPR/Cas9 [D]. Haikou: Hainan University, 2019. (in Chinese)
[27] 潘燕婷. miR171b 在番茄生长发育中的功能研究[D]. 浙江: 浙江大学, 2020.
PAN Y T. The role of miR171b in the regulation of growth and development in tomato [D]. Zhejiang: Zhejiang University, 2020. (in Chinese)
[28] 王文然, 王晨, 解振强, 贾海锋, 汤崴, 崔梦杰, 房经贵. VvmiR397a及其靶基因在葡萄果实发育中的作用分析. 园艺学报, 2018, 45(8): 18-32.
WANG W R, WANG C, XIE Z Q, JIA H F, TANG W, CUI M J, FANG J G. Function analysis of VvmiR397a and its target genesin grape berry development. Acta Horticulturae Sinica, 2018, 45(8): 1441-1455. (in Chinese)
[29] 白云赫, 王文然, 董天宇, 管乐, 宿子文, 贾海锋, 房经贵, 王晨. vvi-miR160s介导应答赤霉素调控葡萄种子的发育. 中国农业科学, 2020, 53(9): 1890-1903.
BAI Y H, WANG W R, DONG T Y, GUAN L, SU Z W, JIA H F, FANG J G, WANG C. vvi-miR160s in mediatingresponse to gibberellin regulation of grape seed development. Scientia Agricultura Sinica, 2020, 53(9): 1890-1903. (in Chinese)
[30] 曾友竞, 林玉玲, 崔彤彤, 陈晓慧, 徐小萍, 张梓浩, 程春振, 陈裕坤, 赖钟雄. 龙眼miR171家族进化特性及其表达分析. 西北植物学报, 2017, 37(2): 258-265.
ZENG Y J, LIN Y L, CUI T T, CHEN X H, XU X P, ZHANG Z H, CHENG C Z, CHEN Y K, LAI Z X. Evolutionary charaterization and expression of miR171 family inLour. Acta Botanica Boreali-Occidentalia Sinica, 2017, 37(2): 258-265. (in Chinese)
[31] LLAVE C, XIE Z X, KASSCHAAU K D, CARRINGTON J C. Cleavage of scarecrow-like mRNA targets directed by a class ofmiRNA. Science, 2002, 297: 2053-2056.
Function Analysis of GA3Mediate miR171s and Its Target Genesin Grape Seed Development
WANG WenRan1, XIE ZhenQiang2, ZHUGE YaXian1, BAI YunHe1, GUAN Le1, WU WeiMin3, ZHANG PeiAn1, ZHENG Ting1, FANG JingGui1, WANG Chen1
1College of Horticulture, Nanjing Agricultural University, Nanjing 210095;2Jiangsu Vocational College of Agriculture and Forestry, Zhenjiang 212499, Jiangsu;3Insititute of Horticulture, Jiangsu Academy of Agricultural Sciences, Nanjing 210014
【】The main objective of the present research was to identify the vvi-miR171s and their target genesfrom grapevine genome, and to confirm the role of vvi-miR171s andVvSCL6/15/22 in grape seed development process and their expression pattern in response to exogenous GA. 【】The mature sequences of vvi-miR171a/b/e/f/g/h were validated by miR-RACE technique from grape (L.) cv ‘Wink’. The potential target genes of vvi-miR171s were predicted by PsRNATarget software, and the chromosome localization, phylogenetic, gene structure, conserved domain and motifs analysis were performed by bioinformatics tools. The potential functions of vvi-miR171s and their target genes were predicted by promoters-elements analysis. RLM-RACE and PPM-RACE verified the cleavage roles of three target genes by vvi-miR171a/b/e/f/g/h, and the qRT-PCR method was used to detect their temporal expression patterns in grape seed and seedless grape induced by exogenous GA3.】Six vvi-miR171s mature sequences in grape were determined and cloned, which were consistent of the sequences of miRBase. Based on this result, the target genes of them, including,andwere prophesied.The target genes bioinformatics analysis showed evolutionary genetic distance betweengenes and homologous genes in apple (), peach (), and cherry ()is relatively closed, whilehad the same genetic distance from the homologous genes in, apple, peach and cherry. Meanwhile, the gene structure ofwas similar to their close genes in phylogenetic tree. All SCL protein sequences had GRAS domain, both element types, arrangement order of motifs. And 5 same motifs for GRAS domain were identified, indicating that the SCL protein structure was relatively conservative, with the conserved functionality. Subsequently, an array of gibberellin and Salicylic acid responsive-acting elements were identified in vvi-miR171s and target genespromoter sequences, indicating their possible involvement in the regulation of GA-responsive and SA-responsive grape growth and development. Moreover, the qRT-PCR analysis revealed that the expression of vvi-miR171a was significantly repressed by GA3in seed, while its target geneshowed reciprocal expression pattern, particularly. There was a strong negative correlation between vvi-miR171a -VvSCL15 in the grape seed of CK and seed region of GAtreatment. Taken together, vvi-miR171a might the major member of vvi-miR171s in response to GA, additionally, it might involve in the grape berry seed development through negative regulating of VvSCL15gene developmental in response to GA3. 【】vvi-miR171a/b/e/f/g/h were identified from grapevine ‘Wink’, and all of members could cleave three target genes. The vvi-miR171a-might be the major member in response to gibberellin and involve in regulating grape seed development.
grape; vvi-miR171s;; seed development; gibberellin

10.3864/j.issn.0578-1752.2021.02.011
2020-04-27;
2020-05-27
国家自然科学基金(31972373)、江苏省自然科学基金(BK20181318)、句容市农业课题(JRNW[2018]03号)
王文然,E-mail:2017104029@njau.edu.cn。通信作者王晨,E-mail:wangchen@njau.edu.cn
(责任编辑 赵伶俐)
