利用CRISPR/Cas9技术研究玉米ZmFKF1在开花过程中的作用
2021-03-08杨敏胥华伟王翠玲杨护魏岳荣
杨敏,胥华伟,王翠玲,杨护,魏岳荣
利用CRISPR/Cas9技术研究玉米在开花过程中的作用
杨敏1,胥华伟2,王翠玲2,杨护1,魏岳荣1
1广东省农业科学院果树研究所/农业部南亚热带果树生物学与遗传资源利用重点实验室/广东省热带亚热带果树研究重点实验室,广州 510640;2河南科技大学农学院,河南洛阳 471000
【】是多种植物开花途径中发挥重要作用的关键基因。为研究玉米功能,利用CRISPR/Cas9基因编辑技术将进行定点编辑,获得编辑突变体。同时以此为材料,通过表型分析及关键开花基因的表达量变化分析,明确在玉米开花途径中的作用,为玉米分子育种及遗传改良提供理论依据。以玉米B104为材料,克隆,通过序列比对明确的基因结构。以为靶标基因,根据CRISPR/Cas9技术原理设计靶点,将设计的靶点序列在玉米参考基因组中进行比对分析,排除非特异性靶位点,最终筛选出在第1外显子上的靶位点ZmFKF1-T1,构建CRISPR/Cas9基因编辑表达载体。利用农杆菌介导法,转化玉米B104幼胚,通过抗性筛选获得抗性愈伤组织,之后诱导出芽和生根,获得T0代编辑阳性植株,并利用特异引物进行验证。利用靶位点扩增测序法,明确T1代编辑植株在预期靶标位点是否发生突变及突变的类型,筛选获得定点突变纯合株系。获得这些材料后,以野生型B104为对照,统计和分析开花表型。同时,利用实时荧光定量PCR技术检测上述材料中与玉米开花途径相关的关键基因的表达量变化,对表型进行进一步的验证。在玉米的第1外显子上设计靶点构建基因编辑表达载体,通过遗传转化获得的转基因株系实现了对的定点突变,共获得T0代编辑阳性株系18株,在预期靶标位点上发生突变的有6株,2种不同的突变类型:单碱基插入和多碱基缺失。通过开花时间统计和分析,与野生型B104相较,3个T2代编辑纯合突变体的开花时间延迟,显著(<0.05)晚于B104。进一步对这些材料中的开花关键基因、和进行表达量检测,发现突变体中这些基因的表达明显(<0.05)低于野生型B104,与晚花表型相符。可利用CRISPR-Cas9技术对进行定点编辑获得基因编辑突变体,且突变体开花时间明显延迟。
CRISPR/Cas9;基因编辑;玉米;;开花时间
0 引言
【研究意义】玉米是中国重要的粮食和饲料作物,由于育种种质局限这一问题,使其在优良品种选育、产量提高等方面面临巨大挑战。开花是植物生长过程的关键阶段,也是热带玉米引种温带种植迫切需要解决的关键问题[1]。因此,研究玉米开花途径关键基因的功能对于玉米分子育种、遗传改良具有重要意义。【前人研究进展】F-box蛋白FKF1(FLAVINBINDING KELCH REPEAT F-BOX1)属于ZTLs蓝光受体家族,是一个包含PAS(LOV)域,F-box motif和Kelch重复结构域的蛋白[2-3]。已有研究表明,在多种植物开花过程中发挥重要作用。在模式植物拟南芥中,通过FKF1-GI-CDF1---CO-FT途径发挥作用,即:FKF1的LOV结构域与GI互作形成复合体,其稳定性受蓝光调控;CDF1(CYCLING DOF FACTOR 1)是的转录抑制子,直接与的启动子结合,阻碍的表达,抑制开花;FKF1和GI都能与CDF1互作,使CDF1在泛素-蛋白酶体系统的作用下降解,从而促进表达[4-6]。在长日照条件下,和表达同步,能形成足够的FKF1-GI复合物,在白天即能激活的转录,CO蛋白的稳定性依赖于光,在光的存在下,傍晚时CO的积累即可达到峰值[7-8],作用于下游开花基因(),促进开花[9-10]。另外,FKF1也可不依赖于与GI互作,通过LOV域与CO互作,使CO保持稳定。即在拟南芥中,可以通过多种途径调控表达,促进表达,进而促进开花[11]。而在短日照条件下,和不能同时表达,形成的FKF1-GI复合物较少,直到午夜转录本的积累才达到较高水平,但此时没有光,处于较低水平,下游的表达也处于较低水平[3],因而不能促进开花。在短日照植物水稻中,可通过下调和上调的表达,进而上调的表达,再继续上调/,促进开花[12]。与拟南芥和水稻相比,玉米中克隆的开花基因相对较少,且大多数候选基因都是从同源分析获得。在玉米第10染色体的10.04区域、第9染色体的9.05区域和第4染色体的4.09区域存在与开花时间或是光周期相关的主效QTL[13-19]。目前,第10染色体10.04区域内的能够极大地缩短开花时间,有效减弱玉米的光周期敏感性[20-21]。赵淑靓[22]初步确定第9染色体9.05区域参与开花的候选基因。而第4染色体的4.09区域的相关研究还未见报道,其中,是位于该区域内可能与玉米开花相关的重要候选基因。之前,刘玲[23]开展了玉米光周期途径中蓝光响应节律基因的分子进化与关联分析研究,但并未对的具体作用进行深入研究。新近发展起来的基因编辑技术CRISPR/Cas9系统为基因功能的研究提供了更为便捷有效的途径。CRISPR/Cas9是继TALLEN、ZFN之后的一种新型高效定点编辑基因的新技术,近年来已成为广大科研工作者试图进行深入研究基因功能的技术热点。目前,CRISPR/Cas9系统已在拟南芥[24]、水稻[25]、小麦[26]、棉花[27]、番茄[28]、杨树[29]、苹果[30]、西瓜[31]、葡萄[32]、芥蓝[33]和柑橘[34]等植物上成功应用。在玉米中,Feng等[35]利用已知功能的证明CRISPR/Cas9系统可用于玉米的基因功能研究。【本研究切入点】是多种植物开花途径中发挥重要作用的关键基因,但在玉米开花途径中的作用还未见报道。【拟解决的关键问题】本研究以玉米B104幼胚为受体材料,以为靶点,利用CRISPR/Cas9技术进行定点编辑,并对获得的纯合突变体进行表型分析和相关基因表达检测验证,明确的生物学功能。以期为玉米分子育种及遗传改良提供理论依据。
1 材料与方法
1.1 植物材料、主要载体及菌株
以玉米B104幼胚为受体材料,利用农杆菌介导的遗传转化法进行遗传转化。试验材料采用常规种植与田间管理。sgRNA中间载体pYLgRNA-U6b、CRISPR/Cas9双元载体由华南农业大学刘耀光院士惠赠,试验所用引物及测序由生工生物工程(上海)股份有限公司完成。Ⅰ购置于NEB公司,T4 DNA ligase和PrimeSTAR GXL DNA Polymerase购置于宝生物工程(大连)有限公司、KOD FX购置于东洋纺(上海)生物科技有限公司,其余分子试剂主要购置于天根生化科技(北京)有限公司和生工生物工程(上海)股份有限公司。大肠杆菌感受态细胞DH5α购自天根生化科技(北京)有限公司;农杆菌感受态细胞EHA105购自北京华越洋生物有限公司。
1.2 ZmFKF1的克隆、蛋白功能域预测、进化树分析、靶点选择和基因编辑载体的构建
参考Maize GDB(https://www.maizegdb.org/)网站玉米B104的序列,利用Primer Premier 5.0设计引物ZmFKF1-F/ZmFKF1-R(表1),以B104的cDNA为模板进行编码序列CDS的克隆,PCR产物经1.5%琼脂糖凝胶电泳检测。将PCR产物回收后送生工生物工程(上海)股份有限公司进行测序,通过序列比对明确的结构。同时从NCBI下载ZmFKF1氨基酸序列,在SMART(http://smart.embl.de/)网站在线预测ZmFKF1的功能域。另外,为研究在进化过程中与其他物种间的亲缘关系,从NCBI下载水稻()、高粱()、拟南芥()、小米()、小麦()、大麦()、甘蔗()、大豆()8个物种的FKF1氨基酸序列,利用MEGA7.0软件对这8个物种及玉米FKF1进行氨基酸序列比对,绘制分子进化树。
根据CRISPR/Cas9系统识别原型间隔序列毗邻基序(protospacer adjacent motif,PAM)上游约20个核苷酸序列的特点,结合在线网站CRISPR-GE(http://skl.scau.edu.cn/)进行靶位点选择。将设计的靶点序列在玉米参考基因组中比对分析,排除非特异性靶位点,最终筛选出在第1外显子上的一个靶位点,序列为:5′-AGGTGGACGCGGAGCCGG GA-3′,该序列无潜在脱靶位点,将其命名ZmFKF1- T1,PAM序列为5′-TGG-3′。参照曾栋昌等[36]方法构建单靶点编辑表达载体pYLCRISPR/Cas9- ZmFKF1-T1:(1)构建含ZmFKF1-T1靶点的pYLgRNA- U6b载体:将接头引物(FKF-U6b-F/FKF- U6b-R)(表1)变性后冷却至室温完成退火,然后经一步酶切连接反应将其接连到pYLgRNA-U6b载体上,并以此为模板进行包含2个反应的第一轮PCR扩增:反应1以U-F/FKF-U6b-R(表1)为引物,反应2以FKF-U6b-F/gR-R(表1)为引物进行扩增。继续以这两个PCR扩增产物为模板,用特异引物Pps-R/ Pgs-L(表1)进行扩增,获得含有ZmFKF1-T1靶点的sgRNA表达盒ZmFKF1-T1-sgRNA;(2)将ZmFKF1- T1-sgRNA表达盒克隆到pYLCRISPR/Cas9载体上:将PCR扩增获得的ZmFKF1-T1-sgRNA表达盒与pYLCRISPR/Cas9质粒混合,用Ⅰ酶、T4 DNA ligase在PCR仪中进行边酶切边连接反应;(3)转化及测序验证:将上述连接产物转化至DH5α 感受态细胞中,挑取单菌落进行菌落PCR阳性检测(引物SP-L1/SP-R,表1),并将PCR阳性菌液进行测序验证,获得编辑表达载体pYLCRISPR/ Cas9-ZmFKF1-T1。之后提取质粒,将其导入农杆菌感受态细胞EHA105中,获得的农杆菌用于后续玉米幼胚转化。
1.3 玉米B104幼胚遗传转化及抗性植株获得
将活化后的农杆菌菌液过夜培养,调节菌液OD600为0.3—0.4。取授粉后9—12 d的玉米穗,去掉外苞叶,在70%的乙醇中灭菌1 min,然后放在超净台上吹干。用手术刀切除玉米籽粒胚乳的2/3,剥取幼胚(共取200粒幼胚,分2次转化)。在22°C黑暗条件下,将未成熟的玉米胚与农杆菌共培养3 d,之后置于28℃,黑暗条件下进行2轮抗性筛选,首先在含有头孢霉素(250 mg·L-1)和PPT(5 mg·L-1)进行第一轮抗性筛选14 d;其次头孢浓度不变,提高PPT浓度到10 mg·L-1进行第二轮抗性筛选14 d,获得抗性愈伤组织,之后诱导出芽,待到小苗长出2—3片叶,并且有主茎时,将其转移到生根培养基中生根,获得抗性再生苗,在6—7叶期用0.125%(V/V)草铵膦涂抹筛选,除去敏感性植株,获得抗性好的再生株系。
1.4 转基因植株的分子检测
分别以B104野生型和抗性再生株系叶片为材料,利用CTAB法提取T0代植株的DNA,使用特异引物Cas9-F/Cas9-R(表1)进行载体特异片段扩增,能扩增出目的片段的植株即为T0代转基因阳性植株。筛选得到的T0代突变株自交结实后得到T1代种子,将T1代种子播种成苗移栽后提取叶片基因组DNA,跨ZmFKF1-T1靶点设计靶点检测引物ZmCri-F/ZmCri-R(表1),其中,ZmCri-F位于靶点上游197 bp(DNA序列),ZmCri-R位于靶点下游216 bp(DNA序列),使用该引物进行PCR扩增、测序及比对分析,确定编辑突变体的突变类型,并筛选纯合突变单株。对上述筛选的纯合突变体自交加代,获得T2代编辑突变体进行表型分析。
1.5 转基因植株的表型分析及开花相关基因的表达量检测
花期性状统计与分析:采用随机分区设计种植3个编辑纯合突变体和野生型B104对照,3个重复,每个小区中各基因型株系种5行,每行6株。从每个转基因株系和野生型中随机选取10株,对抽雄、吐丝及散粉时间进行表型分析和统计,取平均值,并进行方差和值分析。抽雄时间表示播种后至雄穗尖端露出约2 cm的天数;散粉时间表示播种至雄穗开始散粉的天数;吐丝时间表示播种至植株花丝从苞叶突出的天数。
玉米开花关键基因的检测:分别以编辑纯合突变体、B104玉米材料的cDNA为模板,以玉米为内参基因,利用实时荧光定量PCR检测不同基因型材料中开花相关重要基因的表达情况,包括[37]、[38]和[39],引物见表1。试验重复3次。利用2-ΔΔCT方法计算基因相对表达量。
2 结果
2.1 ZmFKF1的克隆与pYLCRISPR/Cas9-ZmFKF1表达载体的构建
以玉米B104的cDNA为模板,以ZmFKF1-F/ ZmFKF1-R为引物,PCR扩增、测序获得的cDNA序列,序列大小为1 857 bp。参考MaizeGDB上的序列,通过DNA与cDNA序列比对发现,含有1个内含子和2个外显子,长度分别为940、256和1 601 bp(图1-A)。通过比对ZmFKF1和其他8个物种FKF1的氨基酸序列结果表明,ZmFKF1与高粱、甘蔗中的FKF1亲缘关系最近(图1-B)。
根据CRISPR/Cas9技术原理,结合的cDNA序列,利用在线网站CRISPR-GE(http://skl. scau.edu.cn/)进行靶位点选择,最终在第1外显子上筛选到一个靶位点ZmFKF1-T1。然后利用酶切连接、PCR扩增等方法将ZmFKF1-T1与sgRNA表达盒连接,再将连接成功的ZmFKF1-T1-sgRNA表达盒组装到pYLCRISPR/Cas9表达载体上(图1-C)。组装好的ZmFKF1-T1-sgRNA表达盒由OsU6b启动子驱动,Cas9由Ubi启动子驱动(图1-C)。
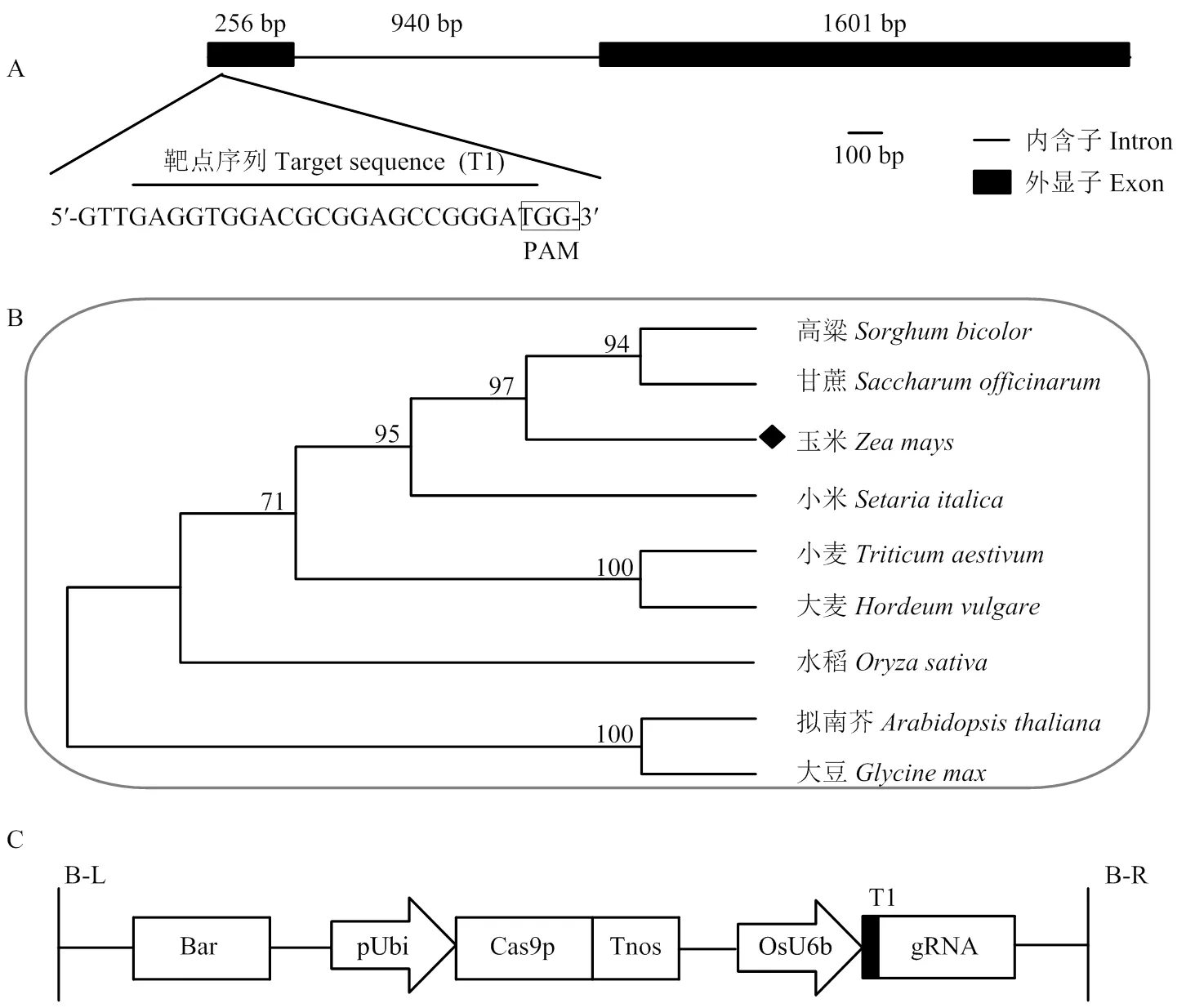
A:靶位点在ZmFKF1中的位置;B:9个物种的FKF1分子进化树分析;C:pYLCRISPR/Cas9-ZmFKF1-T1质粒T-DNA区示意图
2.2 玉米B104幼胚遗传转化
分离获得未成熟B104幼胚,将分离好的幼胚与携带pYLCRISPR/Cas9-ZmFKF1-T1表达载体的农杆菌进行共培养3 d(图2-A),之后转入抗性培养基进行抗性筛选,获得初生抗性愈伤组织(图2-B),提高筛选试剂PPT浓度,进一步进行诱导筛选,获得抗性愈伤组织(图2-C),之后诱导出芽和生根(图2-D和图2-E),获得抗性再生苗,待生长至6—7叶期,用草铵膦涂抹叶片,除去敏感性株系,共获得18个抗性植株。

A:幼胚;B:初生愈伤组织;C:抗性愈伤组织;D:再生抗性芽;E:再生抗性苗
2.3 ZmFKF1编辑植株的分子鉴定
提取18株抗性植株及野生型B104叶片基因组DNA,利用载体特异引物Cas9-F及Cas9-R进行PCR扩增,结果显示,野生型未扩增出目的片段,18株抗性植株都可以扩增出目的条带。共选取了200粒幼胚进行遗传转化,所以阳性率为9.00%(图3-A)。
收获T0代种子大田种植,18个独立株系各种植15株,分别提取叶片DNA,使用靶位点上下游特异引物ZmCri-F/ZmCri-R进行PCR扩增,对扩增产物进行测序。结果表明,在18个独立株系中有6个在靶点位置发生不同类型的编辑,包括单碱基插入和多碱基缺失2种类型,编辑效率为33.33%,同时获得了3个纯合突变体(表2和图3)。
2.4 ZmFKF1编辑突变体的氨基酸序列预测分析
为了更好地说明6个在靶点位置发生编辑的突变体对靶标基因编码蛋白的影响,对编辑突变体的氨基酸序列进行了预测分析。首先,对玉米ZmFKF1功能域进行了预测(图4-A),ZmFKF1包含1个PAS(LOV)域,由第37—107位的氨基酸编码;1个F-box域,由第205—253位的氨基酸编码;4个Klech重复结构域,分别由第300—351、352—405、415—466、517—566位氨基酸编码。同时对C1-6、C2-12、C3-3、C5-7、C6-2和C7-1突变体编码蛋白的氨基酸序列进行预测比对,发现C1-6、C3-3、C6-2和C7-1都在PAS(LOV)域起始位置前后发生了氨基酸突变或缺失;而C2-12和C5-7在该功能域发生氨基酸突变的同时还由于移码突变造成了氨基酸编码提前终止(图4-B)。
2.5 ZmFKF1编辑突变体的表型分析及开花相关基因的检测
为检测编辑后对玉米开花的影响,选择3个T2代编辑纯合株系C2-12、C6-2和C7-1进行大田种植、花期表型记录和分析。结果显示,野生型B104的抽雄时间、散粉时间和吐丝时间分别为62.70、67.87和70.53 d;C2-12分别为63.43、69.13和72.57 d;C6-2分别为64.07、69.37和71.90 d;C7-1分别为63.96、69.04和72.33 d。即在开花的3个时期,C2-12、C6-2和C7-1都比野生型B104晚,初步确定正调控玉米的开花过程(图5和表3)。
为了验证这一结果,又检测了C2-12、C6-2和C7-1中玉米开花关键基因、和的表达情况(图5-C),在3个基因编辑突变体中,、和的表达量明显下调(<0.05),在C2-12中分别下调4.7、3.4和1.7倍,在C6-2中分别下调1.6、2.1和1.5倍,在C7-1中分别下调1.5、1.6和1.6倍。说明发生基因编辑之后,下游基因、和的表达量均下调,这一结果与C2-12、C6-2和C7-1开花延迟的表型相吻合。

表2 ZmFKF1编辑突变体突变类型分析
加粗字体表示PAM序列,下划线处表示插入的碱基,+表示碱基插入,-表示碱基缺失,---表示野生型,无变化
The bold font represented PAM sequence. The underlined base indicated the insertion base. + represented base insertion, - indicated base deletion, --- represented wild type (B104)
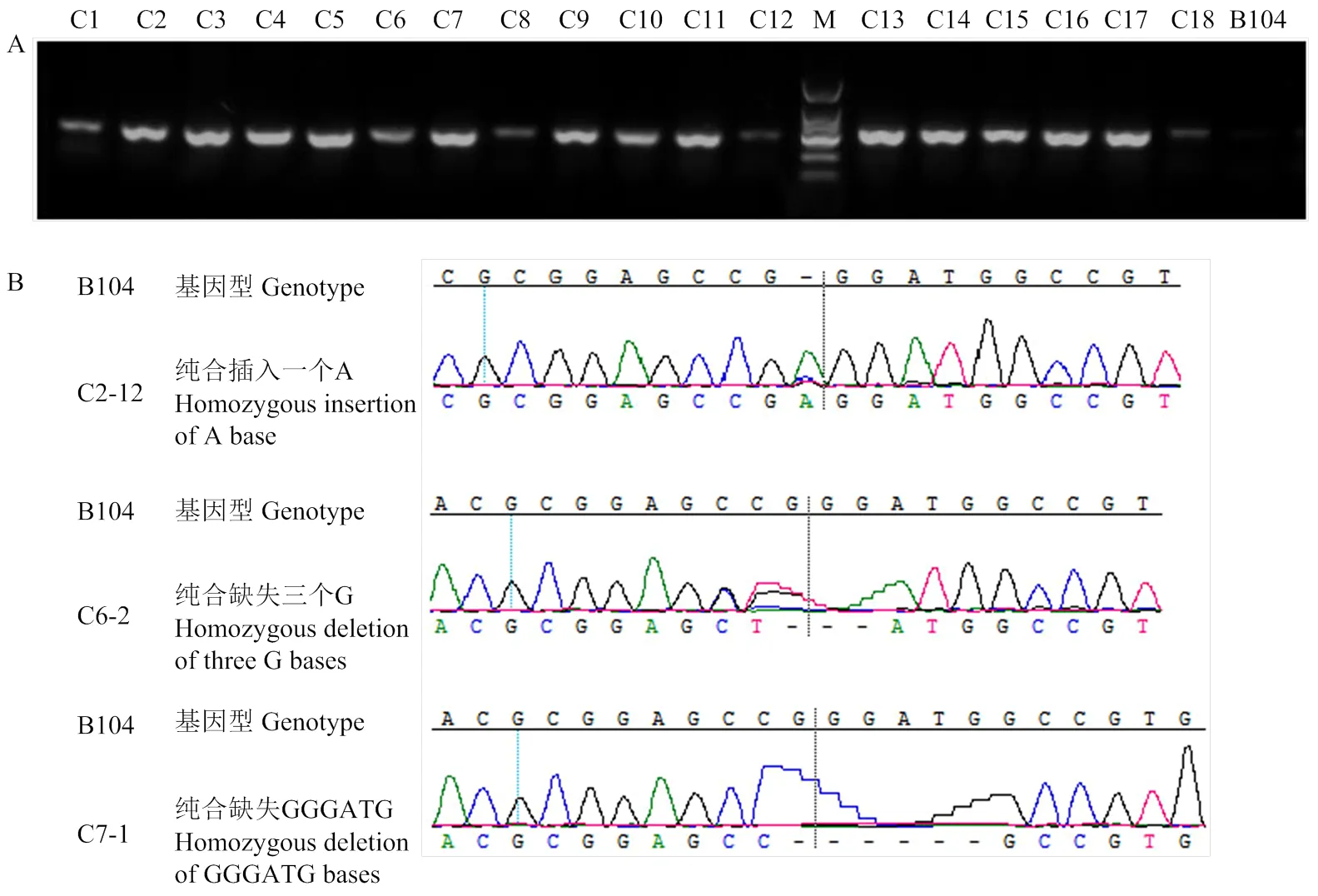
A:ZmFKF1编辑植株的阳性鉴定;B:ZmFKF1编辑纯合体测序结果
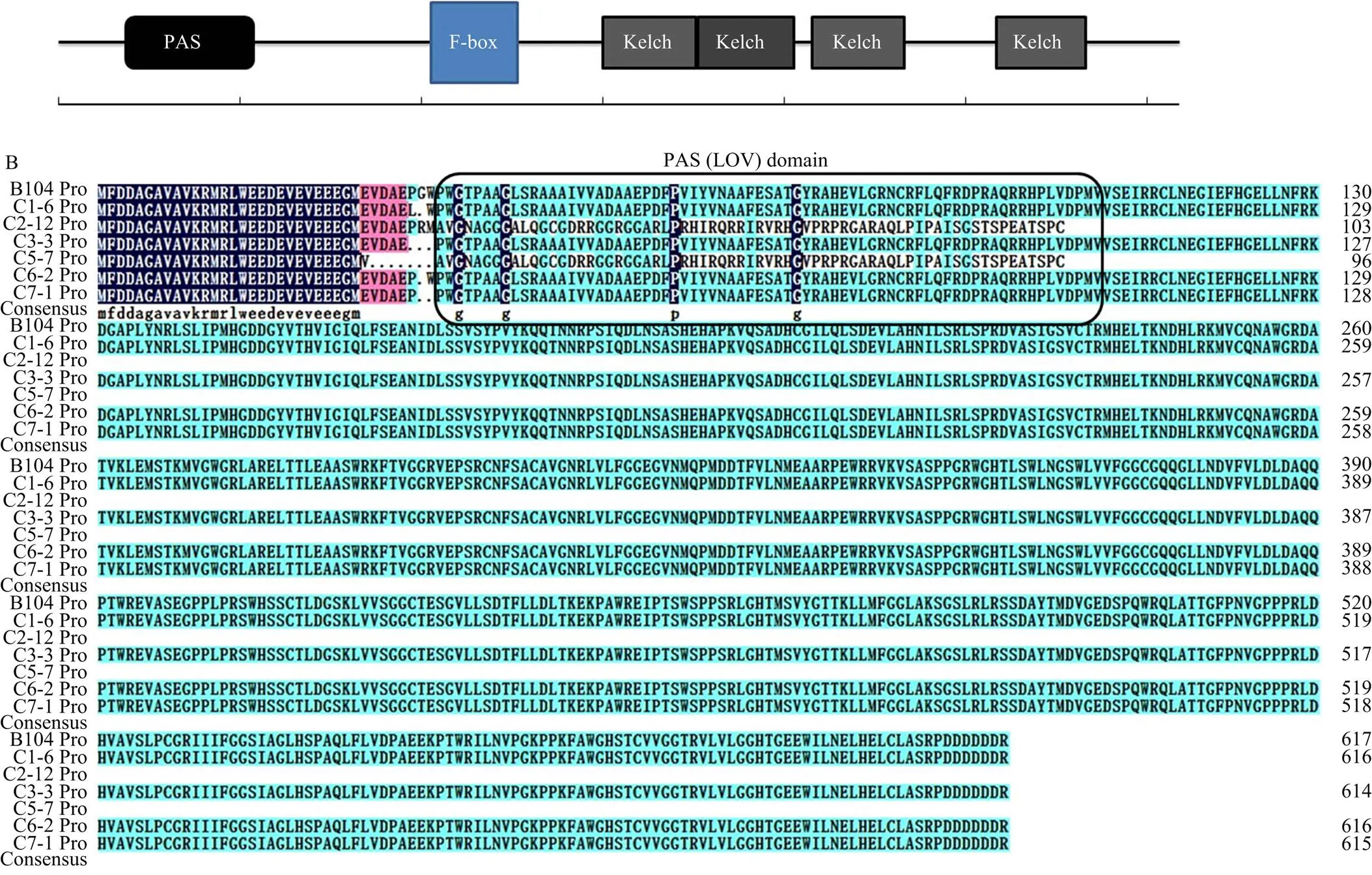
A:ZmFKF1蛋白结构域预测结果;B:ZmFKF1编辑突变体的氨基酸序列预测分析及比对结果
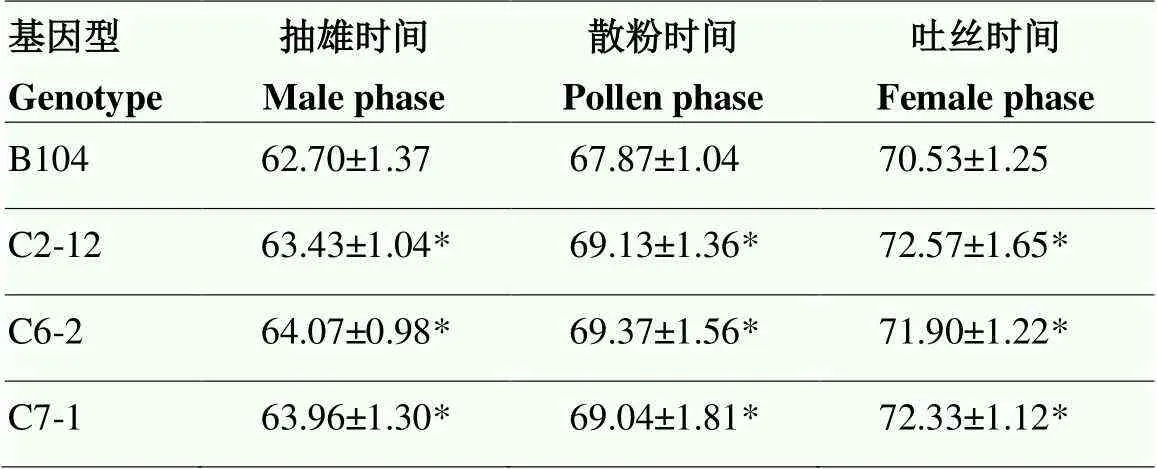
表3 不同基因型植株玉米抽雄时间、吐丝时间和散粉时间的统计
采用随机分区设计种植3个编辑纯合突变体和野生型B104对照,3个重复,每个小区中各基因型株系种5行,每行6株。随机选取各基因型株系0株进行表型统计分析,取平均值,并进行方差和值分析。*表示在0.05水平差异显著
Threegene editing homozygous mutants and wild type B104 were planted in a random area design, with 3 replicates. There were 5 lines of each genotype in each plot, with 6 plants in each row. 10 strains of each genotype were randomly selected for phenotypic statistical analysis. The average value was taken, and the variance andvalue were analyzed. * indicate significant difference at 0.05 level
3 讨论
玉米开花是由多基因控制的,表现数量性状的遗传特点。近年来,随着DNA分子标记技术的发展,数量性状基因定位工作发展很快。研究者们在不同的环境、利用不同的材料、不同的世代群体在玉米第10染色体的10.04区域、第9染色体的9.05区域和第4染色体的4.09区域检测到了开花时间或是光周期主效QTL的存在[13-19],说明在这些区域很可能包含重要的反应因子,与玉米开花性状紧密相关。目前,第10染色体10.04区域是玉米光周期敏感性课题普遍关注的热点,已有研究表明在这一区域内含有参与光周期途径的重要基因,该基因能够极大地缩短开花时间,有效减弱玉米的光周期敏感性[20-21]。赵淑靓[22]开展了对第9染色体9.05区域主效QTL位点的精细定位及候选基因克隆,确定了该区域的重要候选基因。而第4染色体4.09区域的研究工作还未见报道,即位于该区域内,编码一个与拟南芥FKF1同源的蛋白。已有研究表明该基因在拟南芥和水稻的开花过程中发挥重要作用[3,12],暗示可能在玉米开花途径中发挥作用。
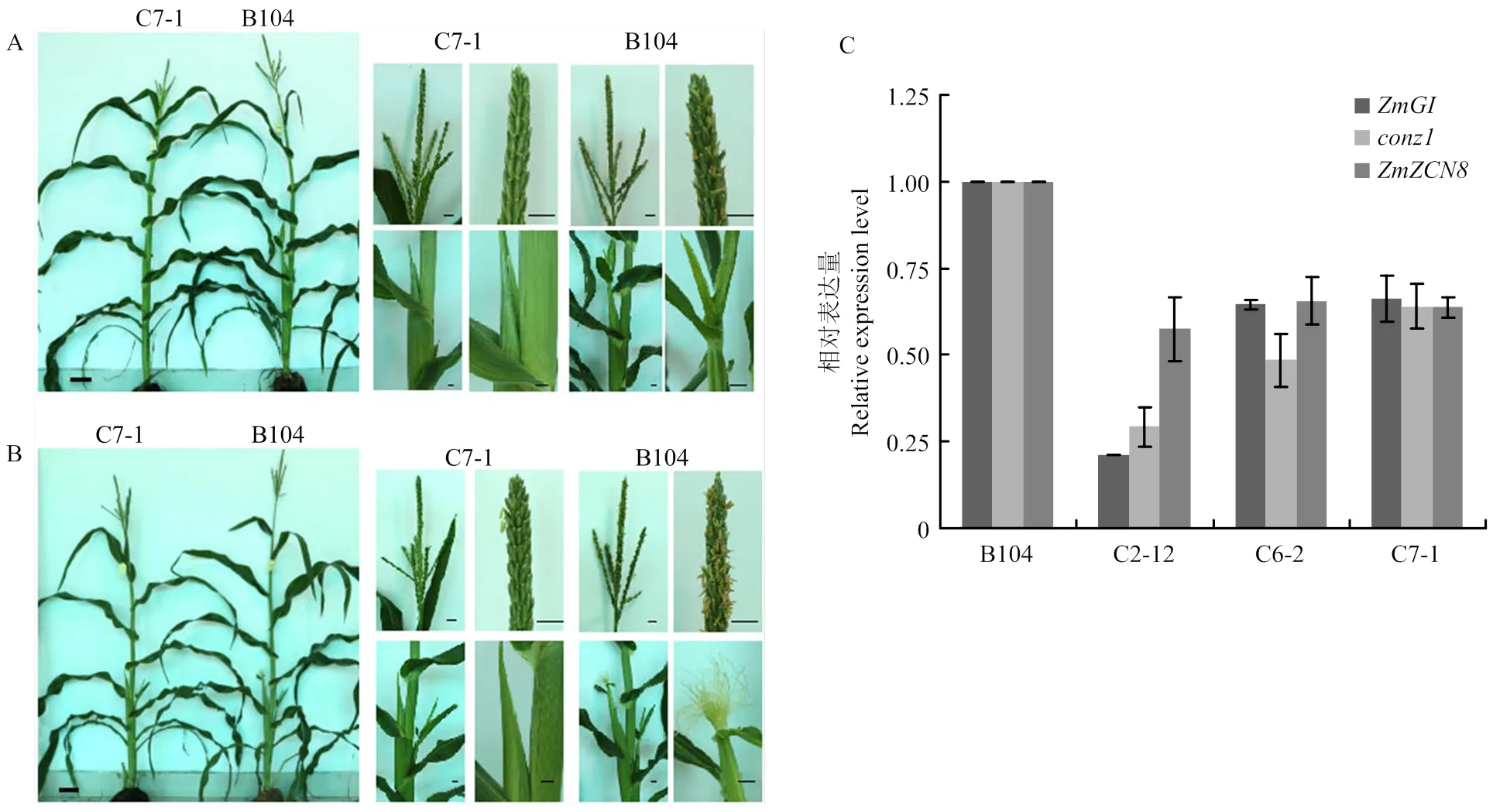
A:C7-1和B104在播种后67 d的表型;B:C7-1和B104在播种后70 d的表型;C:不同基因型材料中开花关键基因的表达情况,其中ZmUbi为内参基因,利用2-ΔΔCT方法计算基因相对表达量结果
CRISPR/Cas9技术是目前最前沿的基因编辑技术,具有载体构建简便易行,靶点选择广泛,打靶效率高等优势,该系统在植物中已有深入研究,但在玉米中的应用还不多。本研究以B104为材料,利用CRISPR/Cas9技术对可能在玉米开花途径中发挥重要作用的进行定点编辑,以B104幼胚为受体材料,获得了18株抗性再生苗,阳性率为9.00%,18株阳性苗中有6个在靶标位点编辑的株系,包括2种不同突变类型,编辑效率为33.33%,阳性效率与编辑效率与Feng等[35]报道的基本一致。之前的研究表明CRISPR/Cas9编辑效率与环境温度和利用不同的特异启动子有关[35,40]。从对的编辑效率结果来看本研究使用的遗传转化条件和启动子的选择都是合理且有效的,实现了对玉米的定点编辑。
在获得玉米的定点编辑突变体后,本研究对获得的6个在靶点位置发生编辑的突变体进行了氨基酸序列预测分析,并与野生型B104ZmFKF1氨基酸序列进行了比对,发现所有的突变体都在靠近ZmFKF1的PAS(LOV)功能域的位置发生了移码突变或氨基酸缺失,其中C2-12和C5-7在靶点位置的编辑还导致了几乎整个PAS区域的氨基酸突变及提前终止,而FKF1蛋白的PAS域被作为信号传感器结构域,在蛋白功能发挥及蛋白互作中发挥重要作用[3-5]。本研究选择3个编辑纯合株系C2-12、C6-2和C7-1进行表型分析,既有ZmFKF1 PAS域提前终止也有氨基酸突变的编辑类型,发现这3个纯合体都表现出一定的晚花表型,虽然表型相对较弱。表型相对较弱可能与玉米开花受多基因调控,表现数量性状遗传的特点有关。同时,进一步检测了在玉米开花途径中发挥重要作用,且与拟南芥、和同源的3个关键基因、和的表达情况,发现这些基因在3个突变体中明显下调,与开花延迟的表型相符,进一步确定在玉米开花途径中发挥作用。通过对3个纯合突变体的表型分析,发现相较C6-2和C7-1突变体,C2-12突变体中关键开花基因的表达量变化更为显著,这可能与C2-12在靶点位置的编辑导致了PAS几乎整个区域的氨基酸突变及提前终止,从而使完全不能发挥作用有关。即的不同编辑方式可能造成了突变体表型强弱的不同。后续,笔者将以现有的其他几种基因编辑突变体为材料,继续筛选更多不同突变类型的纯合系,解析不同位点突变对玉米开花时间的影响。
在长日照模式植物拟南芥中,受生物节律钟的调控,FKF1蛋白可被蓝光激活,与GI在光周期开花过程中通过FKF1-GI-CDF1---CO-FT这条途径调控开花[3-11]。而在短日植物水稻中,OsFKF1也被发现并深入研究,有意思的是,OsFKF1可能是一种自主的开花激活因子,可以不依赖于光周期促进开花,其作用机制为:通过下调和上调的表达,进而上调表达,再通过上调/促进水稻的开花,但是并不影响(拟南芥的同源基因)和(拟南芥的同源基因)的表达,即:在水稻中,是通过-/这条途径调控开花,而不是通过与拟南芥相似的这条经典途径。尽管在水稻中,也发现OsFKF1能与OsGI互作,但是激活的是(在拟南芥中不存在同源基因),而不是与拟南芥同源的,这说明水稻OsFKF1与OsGI的互作与拟南芥作用机制不同,应该另有它用[12]。同时,从水稻OsFKF1的作用途径来看,该基因所调控的、和在拟南芥中都是找不到同源基因的。这些结果说明,在短日照植物水稻和长日照植物拟南芥中的调控方式不同。而本文通过基因编辑玉米发现,突变后,在广州短日照地区是晚花的,那么这些突变体在北方长日照地区是否也具有晚花表型,是否依赖光周期发挥作用,这些都是后续关注和研究的方向。进一步在突变体中检测到、和的表达量下调,说明与这些玉米开花关键基因紧密相关,但是通过与拟南芥类似的信号通路调控开花,还是有其独特的调控方式,这些详细的作用机制需要继续深入研究,这也是笔者后续研究的主要内容。以上这些问题的逐步解析将为阐明的分子作用机制,丰富玉米开花途径和生产实践提供重要的理论基础。
4 结论
利用CRISPR/Cas9技术对玉米材料B104的进行定点编辑,获得不同类型的基因编辑突变植株,明确了在玉米开花过程中的正调控作用。利用CRISPR/Cas9技术研究玉米基因功能是一条非常高效的途径。
[1] 库丽霞, 孙朝辉, 王翠玲, 张君, 张伟强, 陈彦惠.玉米光周期敏感相关性状发育动态QTL定位. 作物学报, 2010, 36(4): 602-611.
Ku L X, Sun Z H, Wang C L, ZHANG J, ZHANG W Q, CHEN Y H. QTL analysis of the photoperiod sensitivity-related traits at different developmental stages in maize (L.). Acta Agronomica Sinica, 2010, 36(4): 602-611. (in Chinese)
[2] Oil M, Kharshiing E V. Photoreceptor mediated plant growth responses: Implications for photoreceptor engineering toward improved performance in crops. Frontiers in Plant Science, 2017, 8: 1181-1194.
[3] Nelson D C, Lasswell J, Rogg L E, COHEN M A, BARTEL B. FKF1, a clock-controlled gene that regulates the transition to flowering in. Cell, 2000, 101(3): 331-340.
[4] Sawa M, Nusinow D A, Kay SA, IMAIZUMI T. FKF1 and GIGANTEA complex formation is required for day-length measurement in. Science, 2007, 318(5848): 261-265.
[5] Imaizumi T. FKF1 F-Box protein mediates cyclic degradation of a repressor ofin. Science, 2005, 309(5732): 293-297.
[6] Goralogia G S, Liu T, Zhao L, PANIPINTO P M, IMAIZUMI T. CYCLING DOF FACTOR 1 represses transcription through the TOPLESS co-repressor to control photoperiodic flowering in. The Plant Journal, 2017, 92(2): 244-262.
[7] Kobayashi Y, Weigel D. Move on up, it’s time for change- mobile signals controlling photoperiod-dependent flowering. Genes & Development, 2007, 21(19): 2371-2384.
[8] Hwang D Y, Park S, Lee S, IMAIZUMI T, SONG Y H GIGANTEA regulates the timing stabilization of CONSTANS by altering the interaction between FKF1 and ZEITLUPE. Molecular Cells, 2019, 42(10): 693-701.
[9] Corbesier L, Vincent C, Jang S, Fornara, F, Fan Q Z, Searle I, Giakountis A, Farrona S, Gissot L, Turnbull C, Coupland G. FT protein movement contributes to long-distance signaling in floral induction of. Science, 2007, 316: 1030-1033.
[10] D’Aloia M, Bonhomme D, Bouché F, TAMSEDDAKK, ORMENESE S, TORTI S, COUPLAND G, PERILLEUX C. Cytokinin promotes flowering ofvia transcriptional activation of theparalogue. The Plant Journal, 2011, 65(6): 972-979.
[11] Song Y H, Smith R W, To B J, MILLAR A J, IMAIZUMI T. FKF1 conveys timing information for CONSTANS stabilization in photoperiodic flowering. Science, 2012, 336(6084): 1045-1049.
[12] Han S H, Yoo S C, Lee B D, AN G. PAEK N C. Rice FLAVIN-BINDING, KELCH REPEAT, F-BOX1 (OsFKF1) promotes flowering independent of photoperiod. Plant Cell Environ, 2015, 38(12): 2527-2540.
[13] Moutiq R, Ribaut J M, Edmeades G, KRAKOWSKY M D, LEE M. Quantitative trait loci for photoperiod response in maize. Maize Genetics Conference Abstracts, 2002(43): 147.
[14] Wang C L, Cheng F F, Sun Z H, TANG J H, WU L C, KU L X ,CHEN Y H. Genetic analysis of photoperiod sensitivity by molecular markers in a tropical by temperate maize recombinant in bred population. Theoretical and Applied Genetics, 2008, 117: 1129-1139.
[15] WANG X T, WU L J, ZHANG S F, WU L C, KU L X, WEI X M, XIE L L, CHEN Y H. Robust expression and association of ZmCCA1 with circadian rhythms in maize. Plant Cell Report, 2011, 30: 1261-1272.
[16] Chardon F, Virlon B, Moreau L, FALQUE M, JOETS J, DECOUSSET L, MURIGNEUX A, CHARCOSSET A. Genetic architecture of flowering time in maize as inferred from QTL meta-analysis and synteny conservation with the rice genome. Genetics, 2004, 162: 2169-2185.
[17] Coles N D, McMullen M D, Balint-Kurti P J,PRATT R C, HOLLAND J B. Genetic control of photoperiod sensitivity in maize revealed by joint multiple population analysis.Genetics, 2010, 184(3): 799-812.
[18] Ducrocq S, Giauffret C, Madur D, COMBES V, DUMAS F, JOUANNE S, COUBRICHE D, JAMIN P, MOREAU L, CHARCOSSET A.Fine mapping and haplotype structure analysis of a major flowering time quantitative trait locus on maize chromosome 10. Genetics, 2009, 183(4): 1555-1563.
[19] Mu G Q, Liang Y, Zhang Z M, WU Y Q, LIU S J, PENG H, ZHANG S Z, PAN G T. Mapping quantitative trait loci associated with photoperiod sensitivity in maize (L.). Agricultural Sciences in China, 2009, 8(1): 24-30.
[20] Hung H Y, SHANNON L M, TIAN F, BRADBURY P J, HOLLAND J B. ZmCCT and the genetic basis of day-length adaptation underlying the post domestication spread of maize. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(28): 1913-1921.
[21] Yang Q, Li Z, Li W. CACTA-like transposable element in ZmCCT attenuated photoperiod sensitivity and accelerated the post domestication spread of maize. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110: 16969-16974.
[22] 赵淑靓. 玉米伪应答调节基因的克隆与表达分析[D]. 洛阳: 河南科技大学, 2018.
Zhao S J. Gene cloning and expression analysis of() in maize[D]. Luoyang: Henan University of Science and Technology, 2018. (in Chinese)
[23] 刘玲. 玉米光周期途径中蓝光响应节律基因的分子进化与关联分析[D]. 雅安: 四川农业大学, 2014.
Liu L. Molecular evolution and association analysis of, a clock-controlled gene in maize[D]. Yaan: Sichuan Agricultural University, 2014. (in Chinese)
[24] Li J F, Norville J E, Aach J, MCCORMACK M, ZHANG D D, BUSH J, CHURCH G M, SHEEN J. Multiplex and homologous recombination-mediated genome editing inandusing guide RNA and Cas9. Nature Biotechnology, 2013, 31(8): 688-691.
[25] Mao Y F, Zhang H, Xu N F, ZHANG B T, GOU F, ZHU J K. Application of the CRISPR-Cas system for efficient genome engineering in plants. Molecular Plant, 2013, 6(6): 2008-2011.
[26] Wang Y P, Cheng X, Shan Q W, ZHANG Y, LIU J X, GAOC X, QIU J L. Simultaneous editing of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew. Nature Biotechnology, 2014, 32 (9): 947-951.
[27] Li C, Unver T, Zhang B H. A high-efficiency CRISPR/Cas9 system for targeted mutagenesis in cotton (L.). Scientific Reports, 2017, 7: 43902.
[28] Ito Y, Nishizawa-Yokoi A, Endo M, MIKAMI M, TOKI S. CRISPR/Cas9-mediated mutagenesis of thelocus that regulates tomato fruit ripening. Biochemical & Biophysical Research Communications, 2015, 467(1): 76-82.
[29] Zhou X H, Jacobs T B, Xue L J,HARDING S A, TSAI C J. Exploiting SNPs for biallelic CRISPR mutations in the outcrossing woody perennialreveals 4-coumarate: CoA ligase specificity and redundancy. New Phytologist, 2015, 208(2): 298-301.
[30] Malnoy M, Viola R, Jung M H,Koo O J, Kim S, Kim J S, Velasco R, Kanchiswamy C N. DNA-free genetically edited grapevine and apple protoplast using CRISPR/Cas9 ribonucleoproteins. Frontiers in Plant Science, 2016, 7: 1904.
[31] Tian S W, Jiang L J, Gao Q, Zhang J, Zong M, Zhang H Y, Ren Y, Guo S G, Gong G Y, Liu F, Xu Y. Efficient CRISPR/Cas9-based gene knockout in watermelon. Plant Cell Reports, 2017, 36(3): 399-406.
[32] 杨禄山, 郭晔, 胡洋, 文颖强. 利用CRISPR/Cas9 系统敲除葡萄中提高对白粉病的抗性. 园艺学报, 2020, 47(4): 623-634.
Yang L S, Guo Y, Hu Y, Wen Y Q. CRISPR/Cas9-mediated mutagenesis ofresults in enhanced resistance to powdery mildew in grapevine (). Acta Horticulturae Sinica, 2020, 47(4): 623-634. (in Chinese)
[33] 郑爱红, 张芬, 江敏, 袁巧, 江雷雨, 陈清, 汤浩茹, 孙勃. 利用CRISPR/Cas9技术靶向编辑芥蓝园艺学报, 2019, 46(1): 57-64.
Zheng A H, Zhang F, Jiang M, Yuan Q, Jiang L Y, Chen Q, Tang H R, Sun B. Targeted editing ofby CRISPR/Ca9 technology in Chinese kale. Acta Horticulturae Sinica, 2019, 46(1): 57-64. (in Chinese)
[34] 邹修平, 范迪, 彭爱红, 何永睿, 许兰珍, 雷天刚, 姚利晓, 李强, 罗克明, 陈善春. CRISPR/Cas9介导柑橘基因启动子的多位点编辑. 园艺学报, 2019, 46(2): 337-344.
Zou X P, Fan D, Peng A H, He Y R, Xu L Z, Lei T G, Yao L X, Li Q, Luo K M, Chen S C. CRISPR/Cas9-mediated editing of multiple sites in the citruspromoter.Acta Horticulturae Sinica, 2019, 46(2): 337-344. (in Chinese)
[35] Feng C, Yuan J, Wang R, Liu Y, Birchler J A, Han F P. Efficient targeted genome modification in maize using CRISPR/Cas9 system.Journal of Genetics & Genomics, 2016, 43(1): 37-43.
[36] 曾栋昌, 马兴亮, 谢先荣, 祝钦泷, 刘耀光. 植物crispr/cas9多基因编辑载体构建和突变分析的操作方法. 中国科学: 生命科学, 2018, 48(7): 783-794.
Zeng D C, Ma X L, Xie X R, Zhu Q L, Liu Y G. A protocol for CRISPR/Cas9-based multi-gene editing and sequence decoding of mutant sites in plants. Scientia Sinica Vitae, 2018, 48(7): 783-794. (in Chinese)
[37] 王新涛. 玉米光周期敏感基因的克隆和功能分析[D]. 郑州: 河南农业大学, 2009.
Wang X T. Cloning and characterization of photoperiod sensitive genein maize. Zhengzhou: Henan Agricultural University, 2009. (in Chinese)
[38] Miller T A, Muslin E H, Dorweiler J E. A maize CONSTANS-like gene,, exhibits distinct diurnal expression patterns in varied photoperiods. Planta, 2008, 227(6): 1377-1388.
[39] Meng X, Muszynski M G, Danilevskaya O N. The FT-like ZCN8 gene functions as a floral activator and is involved in photoperiod sensitivity in maize. The Plant Cell, 2011, 23(3): 942-960.
[40] Leblanc C, Zhang F, Mendez J, Lozano Y, Chatpar K, Irish V F, Jacob Y. Increased efficiency of targeted mutagenesis by CRISPR/Cas9 in plants using heat stress. The Plant Journal, 2018, 93(2): 377-386.
Using CRISPR/Cas9-mediated Targeted Mutagenesis ofDelayed Flowering Time in Maize
YANG Min1, XU Huawei2, WANG Cuiling2, YANG Hu1, WEI Yuerong1
1Institute of Fruit Tree Research, Guangdong Academy of Agricultural Sciences/ Key Laboratory of South Subtropical Fruit Biology and Genetic Resource Utilization (MOA)/Guangdong Province Key Laboratory of Tropical and Subtropical Fruit Tree Research, Guangzhou 510640;2College of Agriculture, Henan University of Science and Technology, Luoyang 471000, Henan
【】is a key gene that plays an important role in many plant flowering pathways. In order to study the function ofediting mutants were obtained by using CRISPR/cas9 technology. In this study, we used these mutants as materials to clarify the role of【】gene was cloned from B104, and its structure was determined by sequence alignment. The targeted sequences ofwere designed according to the principle of CRISPR/Cas9 technology, then these targeted sequences were compared and analyzed in maize reference genome, and the non-specific target site was excluded. Finally, ZmFKF1-T1 on exon 1 ofwas selected to construct CRISPR/cas9 gene editing expression vector. At the same time, B104 immature embryos were selected as explants to transform by-mediated genetic transformation, the resistant calli were obtained through resistance screening, and then buds and roots were induced. The T0generationgene editing positive plants were obtained and verified by using the specific primers of. The target site amplification and sequencing analysis was used to determine whether the T1transgenic lines had mutation at the expected target site ofand the type of mutation, and screened the homozygous lines ofsite mutation. After obtaining the above materials, the flowering phenotypes of these materials were statistically analyzed with wild type as control. At the same time, qRT-PCR was used to detect the expression of key genes that related to flowering pathway in the above materials and further verify the phenotype.【】The target sequence was designed on exon1 ofto construct gene editing expression vector. The transgenic lines were obtained by genetic transformation realized site directed mutagenesis of.A total of 18 T0generationgene-edited lines were obtained, and 6 of them were generated mutation on the exon1 with two different mutations types, including single base insertion and multiple bases deletion. The phenotype analysis shown that the flowering time of three T2generationhomozygous lines was delayed compared with wild type B104. Furthermore, the expression levels of three related flowering pathway genes (,and) in the mutants were significantly lower than those in the wild type B104 (<0.05), which was consistent with the late flowering phenotype of the mutants. 【】CRISPR/Cas9 technology can be used to editto obtain gene editing mutants, and the flowering time of these mutants is significantly delayed compared to wild type.
CRISPR/Cas9; gene editing; maize;; flowering time

10.3864/j.issn.0578-1752.2021.04.003
2020-07-25;
2020-09-23
国家自然科学基金青年基金(31700252)、河南省高等学校重点科研项目(17A180002)、河南省重点研发与推广专项(科技攻关)(202102110010)
杨敏,E-mail:minyang_0123@126.com。通信作者魏岳荣,E-mail:weid18@163.com
(责任编辑 李莉)
