基于Random Forest的水稻细菌性条斑病识别方法研究
2021-02-01袁培森曹益飞马千里王浩云徐焕良
袁培森 曹益飞 马千里 王浩云 徐焕良
(1.南京农业大学人工智能学院, 南京 210095; 2.南京农业大学工学院, 南京 210031)
0 引言
植物表型是植物在一定环境下可观察的形态特征,在植物保护、育种等领域具有重要应用价值,其研究涉及植物学、数据科学、机器学习等领域[1-2]。水稻细菌性条斑病属于水稻表型研究的重要内容,是一种由稻黄单胞菌致病变种引发的水稻病害[3],实时准确地判定水稻细菌性条斑病的分布区域和危害程度是采取有效防控措施和实现精准喷药的关键。目前,水稻细菌性条斑病常用的诊断方法主要依靠人力在田间观察,这种方法不仅费时费力、难以在大范围内展开,还受观察者自身经验的影响[4],并且细菌性条斑病早期症状并不明显,很容易被忽略。因此,研究既能克服传统观测方法局限性,又能精确、有效诊断早期细菌性条斑病病害的技术迫在眉睫[5]。
高光谱成像是植物病害检测中常用的监测技术,具有无污染、高效等特点[6]。通过光谱中的每个波段记录一个图像,反映每个空间像素点的光谱信息,从而弥补了传统光谱分析与成像技术的缺点[7-8]。研究人员对高光谱成像技术应用于植物病理分析进行了大量的研究。WU等[9]利用高光谱成像技术结合病理分析,通过聚合酶链式反应标记不同条件下收集的稻粒真实感染状态,使用主成分分析分离健康稻谷粒和受感染稻谷粒,并使用随机森林极限学习机模型(RF-ELM)对不同感染程度的健康和感染混合粒进行分类。雷雨等[10]对小麦叶片的高光谱图像进行掩膜处理,利用主成分分析法和最大类间方差法获取小麦叶片的条锈病病斑区域,通过面积比判定小麦的条锈病病害等级。LU等[11]利用远程高光谱遥感监测实时和特定位置的水稻氮、磷和硫等含量。SUN等[12]结合高光谱技术和全基因组关联分析技术对水稻种子的生化特性进行表型分析,结果表明,归一化光谱指数与蛋白质含量具有高度相关性。GHAMISI等[13]根据LiDAR和高光谱数据的空间信息进行建模,通过使用支持向量机或随机森林分类技术获得最终的分类图。张帅堂等[14]提出了基于高光谱成像技术和图像处理技术融合的茶叶病斑识别方法。YEH等[15]基于高光谱成像技术,利用光谱角度映射器检测3个不同感染阶段的草莓叶状炭疽病。郭伟等[16]通过高光谱影像的光谱指数、比值光谱指数及归一化差值光谱指数构建偏最小二乘回归预测模型,反演冬小麦全蚀病病情指数。梁琨等[17]利用连续投影算法和自适应加权算法提取特征波段的光谱图像,识别小麦赤霉病。这些研究表明,高光谱可以用于作物病害检测,并且取得了较好的效果。但基于高光谱成像对细菌性条斑病进行早期识别的研究较少。
张智韬等[18]探讨了分数阶微分联合支持向量机分类-随机森林模型改善高光谱监测荒漠土壤有机质含量的效果,通过筛选敏感的归一化光谱指数,建立不同分数阶微分的随机森林模型,并以不同土质中的最佳模型进行组合,构建的支持向量机分类和随机森林模型能够快速评估荒漠土壤有机质含量。竞霞等[19]利用弗劳恩霍夫谱线三波段判别算法提取冠层日光诱导叶绿素荧光数据,结合对小麦条锈病病情严重度敏感的11种反射率微分光谱指数,基于随机森林算法和BP神经网络算法预测小麦条锈病的病情严重度。BISWAS等[20]利用灰度共生矩阵和随机森林对感染炭疽病、白粉病和霜霉病的葡萄叶片进行分类,并确定其严重程度。目前,基于高光谱影像病害的研究多集中于提取光谱指数,再对病害进行识别和分类,基于光谱本身对病害进行识别的研究较少。
本研究以感染细菌性条斑病的水稻叶片为研究对象,利用随机森林算法基于高光谱曲线本身的光谱信息建立水稻细菌性条斑病早期检测模型,并对特征波段重要性进行筛选,以提高识别性能和效果。
1 试验材料与样本采集
1.1 试验材料
试验水稻栽培品种选用2015年被农业部评为超级稻品种的南梗9108。种植前挑选饱满种粒,浸种、催芽后,于2019年8月17日在南京农业大学植物保护学院温室播种,采用盆栽式,共80杯,每杯6~8颗种子,均匀播种,管理方法相同。水稻出苗后,搬运到室外,在自然条件下生长。试验采用的细菌性条斑病菌种由江苏省农业科学院植物保护研究所提供,菌种为致病性强的B5-16病原菌,水稻细菌性条斑病病原菌培育图如图1所示。
2019年9月21日,水稻苗长到4叶期后,接种细菌性条斑病菌。将每株水稻苗的倒二叶叶片完全伸展,用酒精消毒的双针头棉花注射器均匀沾取准备好的菌液,将针头口按住叶片,轻轻按动,使菌液通过压力渗入到叶片细胞间隙内[21]。每片倒二叶首末两端各接种一次,距离保持在8 cm左右,水稻接种后如图2所示。
1.2 高光谱成像系统
本试验采用的推扫型高光谱成像系统(HSI-VNIR-0001, 上海五铃光电科技有限公司)如图3所示。采集软件为Spectral-image成像软件和HSI Analyzer分析软件。高光谱系统采集的水稻叶片光谱波长范围为373~1 033 nm,共616个波段。选用17 mm的成像镜头,确定物距为27 cm,亮度为200 lx,调节曝光时间8 ms使分辨率达到3 000像素。
1.3 数据采集与校正
水稻细菌性条斑病的潜伏期为3 d,前3 d接种点附近的叶片和健康叶片类似。由预试验及植保专家确定,3~7 d为爆发期,在此期限内,细菌性条斑病会快速蔓延。由于设备与场地限制,试验在病害早期的第3天和第7天进行离体拍摄,以6~8片离体水稻叶片样本为1组放置在载物台上进行成像,水稻叶片随着载物台匀速移动,高光谱成像仪获取叶片每个像素在各个波长处的光谱信息和图像数据。共获得1 961个接种水稻细菌性条斑病病原菌的水稻叶片样本的高光谱数据和400个接种蒸馏水的水稻叶片样本的高光谱数据。
对高光谱仪器进行校正后再进行高光谱曲线采集,通过调节光强、图像清晰度、图像失真度来实现高光谱成像系统的校正。物镜之间的高度、电控载物台的移动速度和曝光时间都会影响图像清晰度。为了减少噪声信息对高光谱曲线的影响,进而提高光谱曲线定性或定量分析模型的稳定性和精度,需要对高光谱曲线进行黑白校正[16],采用HSI Analyzer分析软件进行图像处理。先对采集的光谱图像进行校正,图像校正公式为
(1)
式中R(i)——校正后的图像相对反射率
Ir(i)——试验光谱的原始图像反射率
Iw(i)——白板校正后亮电流光谱反射率
Id(i)——黑板校正后暗电流光谱反射率
用水稻样本感兴趣区域(Region of interest, ROI)中的所有像素点的光谱平均值作为该样本的平均光谱。
1.4 光谱信息提取
通过HSI Analyzer加载水稻样本的高光谱图像,放大水稻叶片的高光谱图像,选择接种条斑病病原菌叶片中心10像素×10像素的圆形感兴趣区域,计算感兴趣区域内所有像素点的平均光谱,作为水稻感染条斑病叶片样本的原始高光谱数据。
2 试验方法
2.1 数据预处理
常见的高光谱数据预处理方法有S-G卷积平滑[10]、多元散射校正[14](Multiple scattering correction, MSC)、标准正态变量变换[15](Standard normal variate,SNV)和一阶导数法[17]。比较以上4种方法对染病水稻数据和健康水稻数据进行平滑处理的效果,得到经过S-G、MSC以及SNV处理后的光谱反射率基本保持了原始光谱曲线的走向,但效果不佳,而经过多元散射校正处理后的光谱曲线反射率保持在0~0.6的区间范围内,曲线集中、轮廓清晰,处理效果较好,预处理后的光谱曲线如图4所示。因此本文选定多元散射校正对数据进行预处理。
2.2 随机森林模型结构及参数
随机森林(Random forest,RF)算法解决了决策树创建不稳定、过拟合等问题,在分类精度上比单株决策树有明显的提高。此外,RF算法对比其他分类算法具有分类速度快和能够处理高维数据等特点,而且对噪声和孤立点不敏感,不存在过拟合等问题[22-23]。
将RF算法用于叶片高光谱数据集,数据集中的每行数据对应616个反射率和标签,共2 361行。为保证试验数据划分的随机性和一致性,将2 361行水稻叶片高光谱数据以8∶2的比例随机划分为训练集和测试集[24],训练集共1 889行水稻叶片高光谱数据,用于模型的建立与优化;测试集共472个水稻叶片高光谱数据,用于检验。
利用RF算法对1 889个水稻叶片高光谱数据进行训练。一行叶片数据对应一个决策树,在单棵决策树进行分裂时,有3种方法可以选择[25-26],分别为开平方、取对数和无处理,本模型选择将波段数开平方取整,即从616个反射率中随机选取24个反射率。用全波段反射率的熵和24个反射率的熵做差,算出24个反射率的信息增益。信息增益最大的反射率为这棵决策树的根部节点,其余反射率按信息增益大小依次排列,作为其他非叶子节点,建成一个分类决策树。按照以上步骤,生成大量的决策树,建立基于RF算法的水稻细菌性条斑病识别模型。在构建RF算法时,有3个影响性能和效率的重要参数[27]:
(1)决策树数量
设ntrees为森林中决策树数量,即评估器数量。这个参数对RF算法的精确性影响是单一的。较多的子树可以让模型有更好的性能,但同时会使模型运行变慢。ntrees达到一定值后,RF算法的精确性往往不再上升或开始波动。对于这个参数,需要在训练难度和算法效果之间取得平衡。
(2)叶节点样本数
随机森林是决策树B的集合{T1(X),T2(X),…,TB(X)},其中X=(X1,X2,…,Xp)为分子描述符或与分子有关的p维向量。决策树B产生输出集合B={T1(X),T2(X),…,TB(X)},其中B(B=1,2,…,b)是第B棵树对一个分子的预测,对所有树的输出进行聚合以产生一个最终预测,在本文水稻细菌性条斑病的识别模型中,是集成的决策树的预测结果。
将水稻叶片的高光谱数据整理成一组含n个训练分子的数据集D={(X1,Y1),(X2,Y2),…,(Xn,Yn)},其中Xi(i=1,2,…,n)是描述符向量,Yi对应的标签是0或1,训练算法步骤如下[28]:①从随机水稻叶片训练数据集D中抽取一个随机样本,即随机抽样,替换n个训练分子。②对于每个bootstrap样本,在每个节点上,随机选择随机特征数mtry,在子集中确定最佳分割位置使树长到最大尺寸(即不可能进一步分裂,停止生长)。③重复步骤①、②,直到(足够大的数量)决策树B长至足够大,此时叶节点的样本数为nestimators。
(3)候选特征子集
在决策树生长过程中,所有的随机特征数都在每个节点上测试其分裂性能,而RF算法只测试随机特征数mtry。由于mtry通常非常小(软件中的默认值是分类描述符数量的平方根),因此运行速度非常快。为了获得具有最佳预测强度的模型复杂度,一般算法是通过交叉验证对单个决策树进行修剪,该过程计算量较大。但是,随机森林不作任何修改。所以在水稻叶片的高光谱数据量非常大的情况下,RF算法可以比单个决策树在更少的时间内被训练。
根据RF算法原理[29],本试验取mtry=p、在所有描述符中选择每个节点的最佳分割点时,RF算法与Bagging相同。
2.3 光谱波段选择
水稻叶片的高光谱波长范围为373.79~1 033.59 nm,共616个波段,不是每一个波段对随机森林分类模型都能造成影响,对于重要性较小或者冗余波段,即使受到噪声干扰,对分类结果也没有影响。从616个光谱波段中筛选出对分类结果影响较大的光谱波段,用重要波段分类模型代替全波段分类模型,以此来简化随机森林分类模型,减少计算量。本文模型中对波段重要性的排序步骤如下[26]:①遍历616个波段,利用袋外数据进行预测,将每个波段的预测误差记为{E1,E2,…,Em}。②对袋外数据的某一自变量值进行干扰,再对袋外数据进行预测,将每个波段的预测误差记为{E11,E12,…,E1m}。③干扰后的预测误差与干扰前的预测误差取差值,对m个差值取平均值,将每个差值与平均值比较,其相差越大则该波段的重要程度越高。
2.4 水稻叶片光谱特性分析
选取经过MSC预处理的健康和染病水稻叶片的高光谱数据,对两种不同叶片的光谱曲线进行分析。水稻叶片样本高光谱曲线如图6所示。
在616个全波段水稻叶片高光谱数据中,对本文方法识别水稻细菌性条斑病有重要影响的波段是12个,其中10个均匀分布在第56号波长(429.27 nm)和第69号波长(442.55 nm)之间,即蓝光波长(并不在上述范围内)附近。
剩下2个波长为第301号波长(688.15 nm)和第302号波长(689.24 nm),验证了图6中水稻叶片样本的健康高光谱数据与染病数据对比分析,因为在第275号波长(659.99 nm)附近,健康水稻叶片的光谱反射率到达谷值,与同波段染病的水稻叶片光谱反射率相比,差异明显。
这12个波长均不在绿光波长(550 nm)附近。其他的101个波长也鲜有在绿光波长(550 nm)附近。
因为接种水稻细菌性条斑病病原菌的水稻叶片在发病前期,其细胞内部含有大量的叶绿素,在阳光的照射下呈墨绿色水渍斑点,与健康水稻叶片没有太大区别。在绿光波长(550 nm)附近,染病水稻叶片的光谱反射率与健康水稻叶片的光谱反射率也重合,充分说明了在绿光波长(550 nm)附近没有重要光谱的原因。
在蓝光波长(450 nm)附近,由于蓝光可以被水稻叶片中叶黄素吸收,转给叶绿素进行光合作用,促进水稻生长,导致大量水稻代谢产物堆积在蓝光附近,所以重要光谱基本出现在蓝光波段附近。
3 试验与结果分析
3.1 试验环境与评价指标
试验平台为Windows10系统,16 GB内存,256 GB SSD,1TB HD,Intel QuadCore i7-8700, 4.2 GHz。采用scikit-learn 2.2和Python 3.7编写算法,高光谱数据采用HSI Analyzer和Matlab 7.1软件完成处理。
将预测正确的健康样本数量记为TP,将预测错误的健康样本数量记为TN,将预测正确的染病样本数量记为FP,将预测错误的染病样本数量记为FN[28-29]。采用精确率P、召回率R、综合评价指标F1值、准确率A作为评价指标。
3.2 波段选择结果
RF算法筛选出的水稻叶片的高光谱波长中一共有113个波长对基于随机森林的水稻细菌性条斑病的分类模型有相关性影响。图7为波长重要程度相关性分析示意图。
由图7可知,排序第1的波长是443.58 nm,其重要性分值为0.046 40。重要程度排序第1的波长(443.58 nm,第70号波长)到第11的波长(432.33 nm,第59号波长)对RF算法的重要性依次缓慢递减。排序第12波长(659.99 nm,第275号波长)到第13波长(845.68 nm,第445号波长)对RF算法的重要性迅速下降。之后缓慢下降,此时的光谱波长的重要性已经很低,对RF算法分类水稻细菌性条斑病的影响不大。
从113个对本文模型有影响的波长中选取影响程度最高的前12个光谱波长,波长重要性排序见表1。
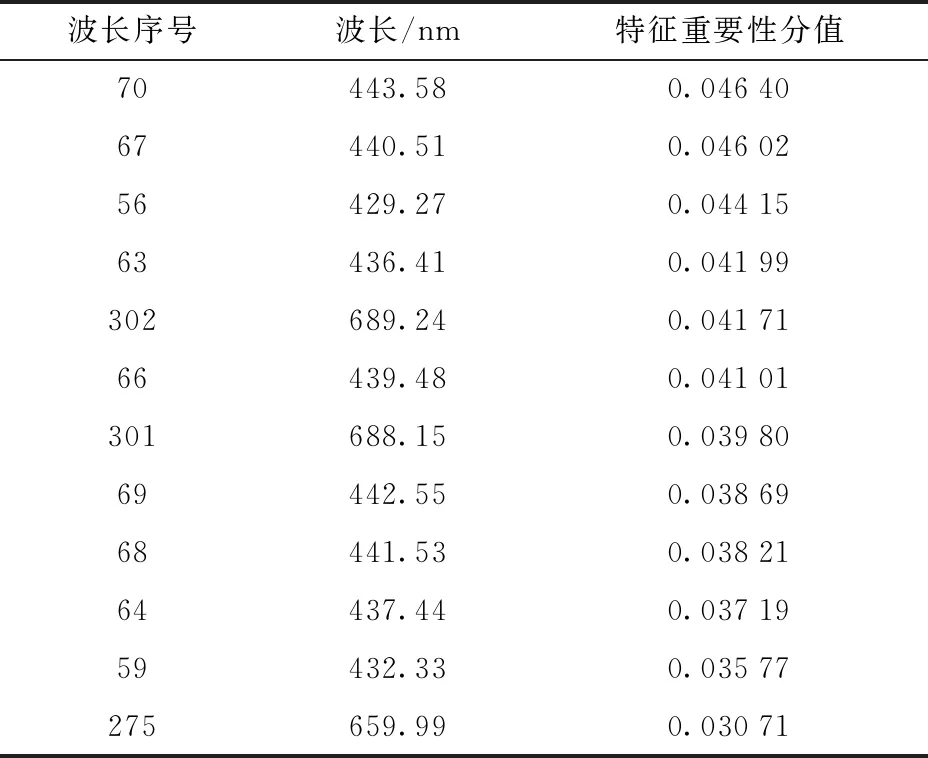
表1 波长重要性排序Tab.1 Band importance ranking
3.3 不同方法对比
试验选取逻辑回归(Logistic regression, LR)、朴素贝叶斯(Naive Bayes, NB)、决策树(Decision tree, DT)、k最近邻(k-nearest neighbor, KNN)、支持向量分类机(Support vector classifier, SVC)和(Gradient boosting decision tree,GBDT)算法进行对比试验,相关算法参数均为scikit-learn 2.2默认参数。
通过对比分析发现朴素贝叶斯准确率最低,只有74.27%(表2)。因为朴素贝叶斯模型在给定输出类别的情况下,假设属性之间相互独立,这个假设在实际应用中往往不成立,当属性个数比较多或者属性之间相关性较大时,分类效果较差。相对逻辑回归算法,RF算法的准确率提高了2.32个百分点,相对朴素贝叶斯算法,RF算法的准确率提高了20.97个百分点,相对决策树算法,RF算法的准确率提高了1.94个百分点,相对KNN算法,RF算法的准确率提高了3.96个百分点,相对SVC,RF算法的准确率提高了3.66个百分点,相对GBT,RF算法的准确率提高了2.02个百分点。
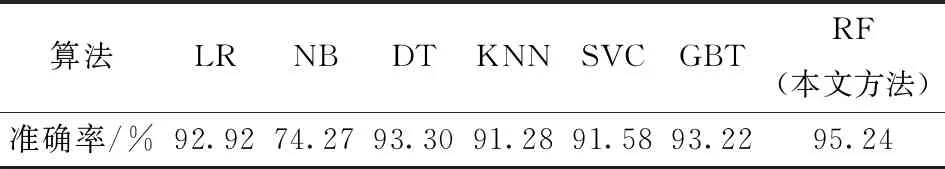
表2 不同分类算法的准确率Tab.2 Accuracy of different classification algorithm
综上所述,基于随即森林的水稻细菌性条斑病识别方法的分类效果最优。
3.4 RF算法结果分析
将427个测试水稻叶片样本数据输入训练好的水稻细菌性条斑病识别算法中,健康样本标签为0,染病样本标签为1。
本文方法对全波段的分类结果的精确率为97.63%,召回率为95.15%,F1值为96.37%,准确率为95.17%。本文中的水稻叶片数据集不均衡,染病叶片远远高于健康叶片数量,F1值较高,从侧面反映本文方法的精确性和稳定性较好。
使用随机森林算法筛选水稻叶片高光谱重要光谱波段,提高模型的预测性能,将用于分类的波段数量从616个减少到12个,数量下降了98.05%,大大简化了模型结构。
本文方法对12个波长分类的识别精确率为94.66%,召回率为99.55%,F1值为97.04%,准确率为94.32%。与全波段分类结果相比,精确率减少了2.97个百分点,召回率增加了4.4个百分点,F1值增加了0.67个百分点,准确率减少了0.85个百分点。虽然准确率有所下降,但是模型结构更加精简,计算复杂度下降,模型精度基本保持不变。
4 结论
(1)通过对水稻叶片全波段光谱曲线分析可得,水稻叶片光谱曲线总体呈上升趋势,患病叶片光谱曲线在590 nm附近,达到高峰;之后开始缓慢波动,在650 nm附近开始下降,在680 nm附近到达谷值,染病水稻叶片的光谱反射率与健康水稻叶片有明显差异;随后迅速上升,在750 nm附近趋于平缓。在蓝光波长(450 nm)和红光波长(664 nm)附近,染病水稻叶片的光谱反射率与健康水稻叶片的光谱反射率差距较大。
(2) RF算法的分类准确率最高,为95.24%,相对LR、NB、DT、KNN、SVC和GBT算法,准确率分别提高了2.32、20.97、1.94、3.96、3.66、2.02个百分点。
(3)采用RF算法对基于全波段和基于12个重要波长的分类结果进行比较。基于12个重要波长的识别精确率为94.66%、召回率为99.55%、F1值为97.04%、准确率为94.32%,与全波段分类结果相比,精确率减少了2.97个百分点,召回率增加了4.4个百分点,F1值增加了0.67个百分点,准确率减少了0.85个百分点。虽然准确率有所下降,但是模型结构更加精简、计算复杂度下降,模型精度基本保持不变。
(4)采用MSC对光谱曲线进行预处理,利用RF算法对细菌性条斑病高光谱影像识别的准确率均高于94%,可以实现对细菌性条斑病的快速识别。
