全基因组拷贝数变异测序检测胎儿生长受限染色体异常的诊断价值 *
2021-01-29罗小金郭岩芸黄和明韦升市刘金星陈婧曹宪振欧德标白江涛
罗小金,郭岩芸,黄和明,韦升市,刘金星,陈婧,曹宪振,欧德标,白江涛
(1.深圳市龙岗区妇幼保健院中心实验室,广东深圳518172; 2.深圳市龙岗区人民医院妇产科,广东深圳518172)
在我国,胎儿生长受限(fetal growth restriction, FGR)发生率为6%~10%,是导致围产死亡的主要原因之一[1]。导致FGR的因素比较复杂,主要包括胎儿、母体和胎盘因素,在胎儿因素中遗传学的染色体和全基因组拷贝数变异(copy number variations, CNVs)是主要原因之一[2]。以往因传统G显带染色体检测技术的局限性,只能分辨大于10 Mb的染色体异常,部分病例是否因遗传因素导致FGR或导致FGR的具体遗传机制并不明确。近年来,随着二代测序技术(next generation sequencing, NGS)在临床上的广泛应用,研究表明,基于NGS的全基因组拷贝数变异测序(copy number variation sequencing, CNV-Seq)相较于传统核型分析、多重连接依赖性探针扩增(MLPA)、荧光原位杂交(FISH)及染色体微阵列分析(CMA)等技术具有高分辨率,高准确性和高基因组覆盖率等优势,可以检出100 kb以上的染色体拷贝数变异情况[3]。本研究通过对138例FGR胎儿同时进行G显带核型分析和CNV-Seq检测,评估基于NGS的低深度全基因组CNV-Seq在明确产前诊断FGR染色体异常的临床价值。
1 资料与方法
1.1研究对象 收集2018年4月至2020年10月深圳市龙岗区妇幼保健院因超声诊断为FGR且进行侵入性产前诊断分析的138例胎儿,均行G显带核型分析和CNV-Seq检测。孕妇年龄(29±4)岁,初产妇94例,经产妇44例,胎儿诊断为FGR时的孕周为(24±3)周,行侵入性产前诊断时的孕周为(25±3)周。FGR诊断标准:综合末次月经、月经史及早孕期B超确定孕龄,利用Hadlock公式计算胎儿体重,低于同孕周正常胎儿平均体重的第10百分位数定义为FGR。排除明确由母体妊娠期高血压疾病、母体感染及母体营养因素异常等原因引起的FGR。本研究经过龙岗区妇幼保健院医学伦理委员会审核批准(No.LGFYYXLL-028),各研究对象及家属均知情同意。
1.2主要仪器及试剂 DNA提取试剂盒(德国Qiagen公司),T4 DNA聚合酶、KlennowDNA聚合酶和T4多聚核苷酸激酶(美国ABI公司),寡核苷酸试剂盒(美国Illumina公司),PCR扩增产物纯化试剂盒(美国Beckman Coulter公司),吉姆萨染液(珠海贝索公司),羊水培养基(广州和能生物公司)。PCR扩增仪(美国Thermo Fisher公司),2100 Bio-analyzer 生化分析仪(美国Agilent公司),HiSeq2000高通量测序仪(美国Illumina公司),CDS-5染色体分散仪(美国Thermotron公司),LeicaGSL120全自动核型扫描分析系统(德国Lecica公司)。
1.3CNV-Seq检测 无菌条件下穿刺取10 mL羊水,1 600 r/min离心10 min,去上清液,按照DNA提取试剂盒说明书提取DNA备用,用超声波打断、末端修复、加polyA尾和接头,加入引物PCR,构建DNA文库,采用Agilent2100生物分析仪和荧光定量PCR对文库进行定量质控。采用HiSeq2000测序平台按50SE单末端10 M数据量进行测序,CNV-Seq检测要求每例DNA量≥10 ng。通过Genome Analyzer Ⅱx分析软件对比将每个测序标签匹配到对应染色体,进行标准化Z值分析,并进行染色体CNV的判定,检测范围为>100 kb的CNVs。检出的CNVs与人类基因组拷贝数变异数据库(Database of Chromosomal Imbalance and Phenotype in Human using Ensembl Resourses, DECIPHER)、人类基因组拷贝数变异多态性数据库(Database of Genomic Variants, DGV)和在线人类孟德尔遗传数据库(Online Mendelian Inheritance in Man, OMIM)进行匹配比对分析。根据美国医学遗传学会(American College of Medical Genetics, ACMG)指南[4],CNVs判读分类为致病性/可能致病性临床意义不明拷贝数(VOUS),可能良性/良性。
1.4核型分析 无菌条件下穿刺取15~20 mL羊水,1 600 r/min离心10 min,去上清液,常规方法[5]进行细胞接种培养、收获、制片及G或N显带染色体核型分析,使用LeicaGSL120全自动核型扫描分析系统扫描玻片上的100个分裂相,挑选长短适中、分散良好核型计数20个,随机分析5个,嵌合体及异常核型加倍计数至100个。核型诊断标准依据人类细胞遗传学国际命名体系(ISCN2016)[6]判读。
1.5短串联重复序列(STR)检测 按照ABI 3130测序仪及Identifier Plus试剂盒说明书对羊水和外周血标本D3S818、D21S11、D18S51、D5S818、D13S317、D7S820、D16S539、D8S1179、PENTAE、AMELOGENIN等15个STR基因座进行检测,应用GeneMapper V3.2自动分析软件进行STR基因分型,平行分析以检测是否存在母体组织污染。
1.6统计学分析 采用SPSS 20.0软件进行统计学分析,计数资料组间率的比较采用卡方检验或Fisher精确概率法,以P<0.05为差异有统计学意义。
2 结果
2.1CNV-Seq与G显带核型分析对>10 Mb染色体异常的检出结果 138例FGR病例中G显带核型分析检出12例>10 Mb染色体异常(8.7%,12/138),包括3例数目异常(分别为2例21三体和1例45,X)、9例结构异常(分别为2例平衡易位、4例缺失或重复和1例新衍生染色体),还检出1例低比例嵌合体。CNV-Seq检出9例>10 Mb染色体异常(6.5%,9/138),2例平衡易位(分别为病例11、12)和1例(病例10)低比例嵌合体(表1)。

表1 核型异常的FGR病例中CNV-Seq检测结果分析
2.2CNV-Seq和G显带核型分析对<10 Mb染色体CNVs的检出结果 138例FGR病例中G显带核型分析检出1例(病例18)<10 Mb染色体异常。CNV-Seq检测出11例<10 Mb染色体CNVs(8.0%,11/138),其中致病性CNVs 8例(5.8%,8/138)(病例13~20),包括2例Williams-Beuren综合征、2例16p微缺失/微重复综合征、1例Miller-Dieker综合征、1例Wolf-Hirschhorn综合征、1例3q29缺失综合征,1例2号染色体父源单亲二倍体(uniparental disomy, UPD),均涉及生长发育迟缓、智力障碍或超声结构异常等相应的综合征表型。另外检出3例VOUS(2.2%,3/138)(病例21~23),主要包括2例微缺失和1例微重复(涉及片段0.2~0.5 Mb),所含致病基因包括ERCC8、NDUFAF2、FUT3、FUT6、ADAMTS2、GRM6、WWOX(表2)。
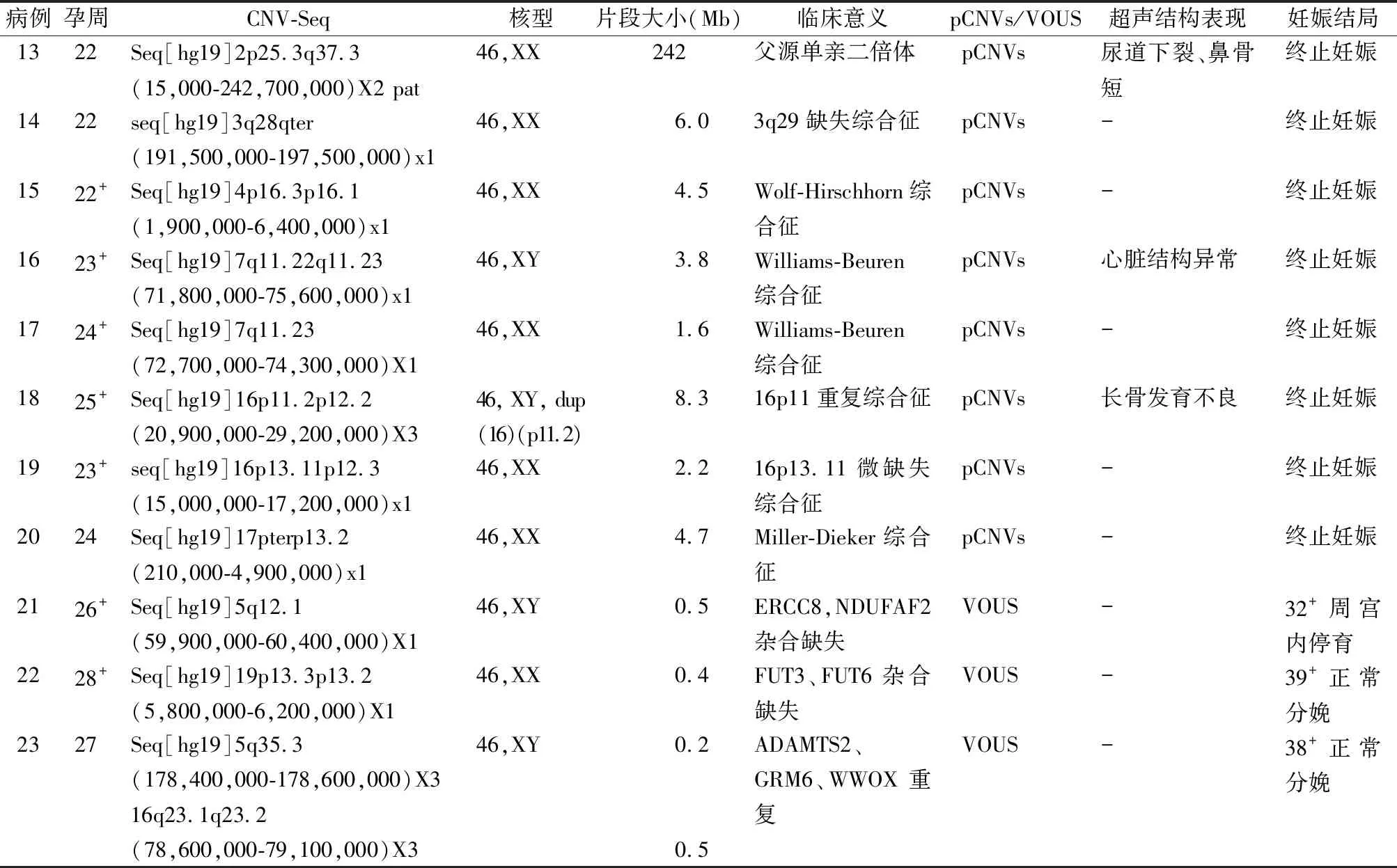
表2 核型正常FGR病例中异常CNV-Seq检测结果分析
2.3CNV-Seq与G显带核型分析对FGR检测结果的对比 138例FGR标本中采用G显带核型分析检出染色体异常13例(9.4%,13/138),采用CNV-Seq检出20例(14.5%,20/138)。两种方法对非整倍体异常和>10 Mb的CNVs的检出率均为4.3%(6/138),差异无统计学意义(χ2=0.000,P>0.05)。对<10 Mb的CNVs的检出率分别为0.7%(1/138)和3.5%(12/138),差异有统计学意义(χ2=9.768,P<0.05)。G显带核型分析检出平衡易位/嵌合体3例(2.2%),CNV-Seq未能检出上述病例。见表3。

表3 核型分析和CNV-Seq对138例FGR病例的检测结果对比分析[n(%)]
3 讨论
FGR的发病涉及多种因素,传统观点认为5%~20%的FGR由胎儿染色体异常所导致[7]。研究显示,239例FGR胎儿同时行核型分析和CMA技术可检出染色体异常37例(15.5%),证实核型分析结合CMA可显著提高FGR染色体异常检出率[8]。相较于传统核型分析,本研究使用的CNV-Seq技术具有高通量、高分辨率和高敏感性等优势,相较于全基因组高通量测序具有深度低、耗时短和成本低等优势。本研究中,采用G显带核型分析结合CNV-Seq技术共检出染色体异常16.7%(23/138),此外,采用CNV-Seq技术额外检出7.2%(10/138)染色体CNVs,证实CNV-Seq应用于产前检测染色体CNVs与CMA具有同等检出功效。相较于CMA技术,CNV-Seq具有全基因组均匀覆盖的优点[9]。本研究中2例平衡易位CNV-Seq未能有效检出,原因在于目前CNV-Seq测序法为单端测序,无法检出不涉及基因剂量改变的染色体异常。理论上平衡易位不存在遗传物质增减,但不排除断裂点破坏关键基因表达导致胚胎停育。本研究中通过G显带核型分析检出1例(病例12)X与6号染色体平衡易位,其断裂点位于Xp22的矮小同源盒(short stature homeobox containing,SHOX)基因区域,SHOX基因缺陷与身材矮小关系密切,属于单倍剂量敏感性基因,其杂合缺失或变异可造成Leri-Weill软骨生成障碍或特发性身材矮小[10]。
研究显示,在核型正常的FGR或合并其他超声异常的FGR病例中,基因芯片能额外检出8%的染色体CNVs[11]。本研究在核型正常的125例FGR胎儿中,采用CNV-Seq技术额外检出7.2%(10/138)小于10 Mb染色体CNVs。致病性CNVs因基因剂量改变、基因断裂及位置效应等方式干扰基因编码蛋白的功能,可导致相应的FGR及其他临床特征[12]。本研究检出的6例致病性CNVs中除1例单亲二倍体外,其他5例均具有明确的综合征临床表型。有文献资料显示,Williams-Beuren综合征、Wolf-Hirschhorn综合征、Miller-Dieker综合征和16p11重复综合征在胎儿时期主要表现为生长发育迟滞、多器官畸形等临床表型,胎儿FGR是其产前超声较为明显的指征[13]。本研究显示CNV-seq在产前可以检出传统G显带核型无法检出的与FGR相关的微缺失微重复综合征,可有效明确其遗传学致病机制。
本研究中的病例18为新发突变的VOUS,区域内含ERCC8和NDUFAF2致病基因,但该胚胎停育是否上述基因杂合缺失导致,还需进一步的功能实验验证。病例19~20的VOUS为遗传自双亲之一,虽缺失/重复区域内含致病基因,但均足月分娩表型正常婴儿。现行CNVs分类中,VOUS的评分标准(-0.89~0.89)比较宽泛,VOUS的检出容易对实验室技术人员和遗传咨询医师造成极大困扰。产前检出VOUS后,父母行CNV-Seq检测有助于结果解释和明确遗传学病因。通常认为VOUS遗传自双亲之一时,其临床意义相对较小,VOUS为新发突变时则反之,但无论是遗传还是新发,均可因外显率和表现度的差异导致临床表型不一致[14]。
本研究还发现,在138例FGR病例中,G显带核型分析和CNV-Seq对染色体非整倍体异常和>10 Mb的CNVs检出率基本一致,通过对比分析,差异无统计学意义(χ2=0.000,P>0.05)。而对于<10 Mb的微缺失/微重复综合征的检出,CNV-Seq相较于G显带核型分析具有显著优势,CNV-Seq能额外检出10例(7.2%,10/138)与FGR相关的小片段致病性CNVs。然而,CNV-Seq也存在技术局限性,例如不能检出平衡易位和倒位、低比例嵌合、点突变等[15]。作为新兴的诊疗技术,CNV-Seq在临床应用仍存在颇多挑战,尚有较多检测的CNVs无法作出合理的临床解释[16]。
综上所述,本研究通过与传统的G显带核型分析对比分析,明确了基于NGS技术的CNV-Seq除可准确检出产前FGR常见的涉及大片段CNVs的染色体非整倍体和结构异常外,还能有效检出10 Mb以下的致病性CNVs及其导致的相关微缺失和微重复综合征,能更系统地揭示FGR的遗传学病因,科学指导孕妇妊娠选择。
