仿刺参SARM基因的克隆、表达和功能研究
2021-01-28孙红娟关晓燕蒋经伟周遵春
孙红娟,陈 仲,贺 迪,高 杉,王 摆,关晓燕,蒋经伟,董 颖,周遵春
(1.辽宁省海洋水产科学研究院,辽宁省海洋水产分子生物学重点试验室,辽宁 大连 116023; 2.凌海市达莲海珍品养殖有限责任公司,辽宁 锦州 121209 )
仿刺参(Apostichopusjaponicus)属棘皮动物门、海参纲、仿刺参属,代表着原始后口动物,在进化和发育生物学研究中占据重要地位[1]。仿刺参生活在潮间带泥沙底质区域,周围环境中存在大量的病原体,这表明机体已经进化形成一套有效的免疫系统。无脊椎动物依赖于先天性免疫系统参与免疫反应,主要通过细胞表面的模式识别受体(PRR)特异地识别病原相关分子模式(PAMP),如蛋白质、糖类、脂质、核酸和这些生物大分子的衍生物[2]。Toll样受体(TLR)是一个在进化上高度保守的免疫受体家族,属于Ⅰ型跨膜蛋白,主要由胞外区、跨膜区和胞质区组成[3]。Toll样受体胞质区是一个与白细胞介素-1(IL-1)受体家族具有高度同源性的TLR和IL-1受体结构区域(TIR),能够募集含有TIR结构的接头蛋白参与信号传导。目前已经发现了5种接头蛋白,包括髓样分化因子(MyD88)、类MyD88接头(MAL)、诱导干扰素β且含有TIR区的接头(TRIF)、TRIF相关接头分子(TRAM)和含有Sterile α和armadillo基序的蛋白(SARM),表明机体在受到病原体刺激后会产生与特定接头蛋白相对应的免疫应答[4]。根据接头蛋白的不同可以将TLR信号通路分为MyD88依赖途径和MyD88非依赖途径。
SARM是最新被发现的TIR结构蛋白,位于MyD88非依赖信号传导途径。该接头蛋白从线虫到哺乳动物中均为高度保守,基因序列包含2个SAM结构域,1个C端的TIR结构域和N端的ARM重复结构域[5]。线虫和文昌鱼的相关研究表明,SARM参与了免疫应答信号通路以及神经发育的过程[6-8]。
目前已经报道的仿刺参Toll样受体信号通路相关基因有Toll、TLR3、MyD88、TRAF6、TRAF3、IRAK4、P105和Rel[9-13],尚未见MyD88非依赖途径接头蛋白的相关报道。笔者通过克隆仿刺参SARM基因,分析其结构序列和进化地位,探索其在仿刺参免疫应答和生长发育过程中的表达模式,为研究Toll样受体信号通路的进化及其在发育和免疫方面的调控机制提供科学依据。
1 材料与方法
1.1 试验材料
试验用仿刺参体质量(6.4±1.1) g,取自辽宁省海洋水产科学研究院引育种中心。试验之前暂养一周,水温16~18 ℃,pH 8.3~8.5,盐度29。试验采用灿烂弧菌(Vibriosplendidus)、假交替单胞菌(Pseudoalteromonasnigrifacien)、希瓦氏菌(Shewanellabaltica)和蜡样芽孢杆菌(Bacilluscereus)4种病原菌进行免疫刺激试验。各菌株在2216E培养基中,温度28 ℃,150 r/min的条件下培养。当密度达到108cfu/mL时,进行刺激试验。
1.2 试验方法
1.2.1 基因全长克隆
参照动物组织RNA提取试剂盒(TIANGEN)说明书进行仿刺参体腔细胞RNA提取。对总RNA的浓度和完整性进行检测,合格后用于后续试验。利用Primer 5.0设计RACE特异引物(GSP)(表1)。试验使用SMARTer 5′/3′RACE试剂盒(Clontech)对仿刺参SARM基因5′端进行克隆,其反应体系见表2。

表1 仿刺参SARM的 RACE引物
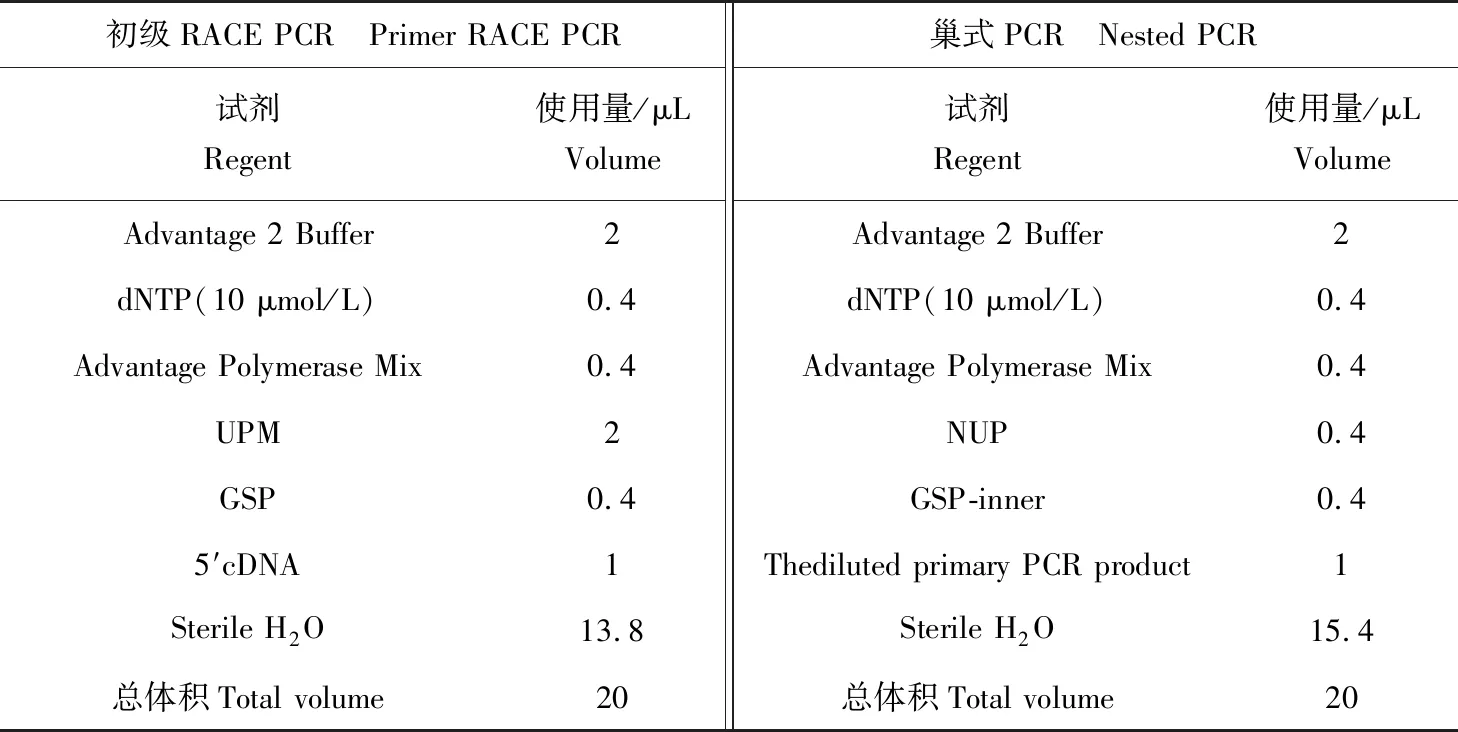
表2 仿刺参SARM的5′RACE PCR反应体系
反应程序为:94 ℃ 5 min;94 ℃ 30 s,72 ℃ 3 min,5个循环;94 ℃ 30 s,70 ℃ 30 s,72 ℃ 3 min,5个循环;94 ℃ 30 s,68 ℃ 30 s,72 ℃ 3 min,30个循环;72 ℃ 10 min。当上述反应没有产生特异性条带时,可以采用巢式PCR,用1 μL稀释过的初级PCR 产物代替用于RACE的cDNAs。反应程序和初级RACE PCR的相同。
试验使用3′-Full Race PrimeScripTMRTase试剂盒(Takara)对仿刺参SARM基因的3′端进行克隆,首先使用已设计好的Outer Primer克隆其反应体系(表3)。反应条件为:94 ℃ 3 min;94 ℃ 30 s,58 ℃ 30 s,72 ℃ 2 min,30个循环;72 ℃ 10 min。可根据实际情况适当地提高或降低退火温度(37~65 ℃)。在设置退火温度时,最好使温度维持在37~65 ℃。从延伸时间温度来看,以1 min/kb为宜。当上述反应没有产生特异性条带时,可以采用Inner Primer进行二次克隆(表3)。反应条件:94 ℃ 3 min;94 ℃ 30 s,61 ℃ 30 s,72 ℃ 2 min,32个循环;72 ℃ 10 min。

表3 仿刺参SARM的3′RACE PCR反应体系
1.2.2 组织和发育样品
试验所研究的组织包括:管足、体壁、肠道、呼吸树和体腔细胞。取15头健康仿刺参,每5头为1组,分为3组,将每组中同一组织的混合样放入1个含有RNA保存液的离心管中。4 ℃条件下保存12 h,后置于-80 ℃超低温冰箱,用于组织表达分布研究。获取仿刺参不同发育时期(受精卵、囊胚期、原肠期、小耳、中耳、大耳、樽型、五触手和稚参)样品,保存方法同上。
1.2.3 免疫刺激试验
将300头健康的仿刺参平均分为5组:对照组与4个菌刺激组。采用灭菌注射器对仿刺参进行体腔注射,对照组每头仿刺参注射100 μL灭菌海水,试验组每头仿刺参注射100 μL菌液。试验采用的注射剂量参考仿刺参体腔细胞中免疫相关酶的应答变化试验[14]。注射4、24、48、72 h和96 h后解剖获取体腔液,每组每个时间点随机选取15头,并随机分成3份,样品混合后在4 ℃,3000 r/min的条件下离心10 min,收集沉淀的体腔细胞。提取总RNA后使用PrimeScriptTMRT Master Mix (Perfect Real Time)试剂盒进行反转录,加5×PrimeScript RT Master Mix 4 μL,反转录RNA的总量不超过900 ng,添加RNase Free dH2O至20 μL。反应条件为:37 ℃ 5 min,85 ℃ 5 s。经过反转录后获得的cDNA样品保存在-20 ℃,用于qRT-PCR试验。
荧光定量PCR使用TB GreenTMPremix Ex TaqTM(TliRNaseH Plus)试剂盒,以Cytb作为内参基因[15],具体信息见表4。反应体系:ROX Reference Dye Ⅱ (50×) 0.4 μL,cDNA模板1 μL,TB Green Premix Ex Taq(Tli RNase H Plus) (2×Conc.)10 μL,正反引物各0.8 μL,dH2O 7 μL。qRT-PCR反应程序:95 ℃ 30 s;95 ℃ 10 s,55 ℃ 40 s,72 ℃ 25 s,40个循环。每个反应重复3次。

表4 仿刺参SARM荧光定量引物信息
1.2.4 数据分析
采用美国国立生物技术信息中心(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)进行开放阅读框预测;采用在线工具Expert Protein Analysis Systerm(http://www.expasy.org/tools/)进行核酸、氨基酸的翻译,亲水性等分析;采用蛋白二级结构预测及注释平台(http://smart.embl-heidelberg.de/)进行结构域分析;采用多重序列比对(http://www.ebi.ac.uk/Tools/msa/clustalw2/)进行序列相似性分析;氨基酸序列全长进行Clustal W对比后采用MEGA 4.0软件来构建邻接进化树, Bootstrap值设为1000次重复。根据qRT-PCR所获取的Ct值,使用软件REST 384 v.2 (Relative Expression Software Tool,REST)进行分析,试验组和对照组之间发生的表达变化采用随机配对检验分析法计算得到[16]。相对表达量选取平均值±标准误差的方式表示。当P<0.05时,表明具有显著差异。
2 结果与分析
2.1 仿刺参SARM基因的序列分析
通过RACE技术,获得仿刺参SARM基因已知部分序列的3′和5′端的序列,拼接得到cDNA,全长为3874 bp,命名为AjSARM,GenBank登录号MN906447。AjSARM基因的5′非编码区131 bp,3′非编码区1331 bp,开放阅读框为2412 bp(图1)。将完整的开放阅读框进行翻译,结果显示,AjSARM基因编码803个氨基酸,预测分子量为89.97 ku,理论等电点为7.76,不存在跨膜结构。BLASTX比对后发现,AjSARM基因与棘冠海星(Acanthasterplanci)SARM基因序列最为相似,核酸序列相似度为50%。

图1 AjSARM基因的cDNA序列和预测的氨基酸序列全长Fig.1 Full-length cDNA sequences and deduced amino acid sequences of AjSARM gene
应用SMART软件对AjSARM蛋白进行二级结构预测,AjSARM蛋白由2个SAM结构域以及1个C端的TIR结构域组成(图2a)。通过Swiss Model对AjSARM蛋白的三级结构进行预测(图2b)。

图2 AjSARM蛋白的二级结构(a)和三级结构(b)示意Fig.2 Schematic diagram of the secondary structure (a) and tertiary structure of AjSARM(b) 图2a中的数值表示AjSARM蛋白的氨基酸序列长度. The values in Fig.2a indicate the length of amino acids in AjSARM.
2.2 仿刺参SARM蛋白序列的进化分析
为了对AjSARM蛋白序列进行进化分析,将处于不同进化地位的10个物种的氨基酸序列全长(表5)进行Clustal W对比后使用MEGA 4.0版本中的邻接方法构建AjSARM蛋白的系统发育树,Bootstrap值设为1000次重复。研究结果显示,AjSARM蛋白与棘冠海星、紫色球海胆的亲缘关系最近(图3)。
2.3 SARM基因在仿刺参不同组织和不同发育时期的表达模式
实时荧光定量PCR试验测得SARM基因在仿刺参不同组织和不同发育时期的表达结果(图4)。以管足为对照,AjSARM基因在体腔细胞中的表达量最高,其次是体壁、肠道、管足和呼吸树。以稚参为对照,其他8个发育时期AjSARM基因的表达均上调。从小耳时期到五触手时期,AjSARM基因的表达量较稳定,均高于前3个时期。其中AjSARM基因在五触手时期表达量最高,在稚参期表达量最低。

表5 不同物种SARM氨基酸序列

图3 AjSARM蛋白序列的系统进化树Fig.3 Phylogenetic tree of AjSARM 使用MEGA 4.0软件构建AjSARM蛋白序列的邻接系统发育树,Bootstrap值设为1000次重复;物种名缩写及登录号参考表5. Neighbor-joining tree of AjSARM is constructed by the MEGA version 4.0; the value of bootstrap is 1000 replicates; abbreviations and the accession numbers of SARM sequences are listed in Tab.5.

图4 SARM基因在仿刺参不同组织和不同发育时期的表达模式Fig.4 Expression of SARM in different tissues and developmental stages in sea cucumber A. japonicus a.SARM在不同组织中的分布,以管足(1×)为对照;b.SARM在不同发育阶段的分布,以稚参(1×)为对照;*表示与对照组相比差异显著(P<0.05). a.the distribution of AjSARM in different tissues, the expression level of AjSARM in tube feet as control (1×); b.the distribution of AjSARM at developmental stages, the expression level of AjSARM at juvenile stage as control (1×); * represent significant difference at the level of P<0.05 from the control.
2.4 不同病原菌刺激后仿刺参SARM基因的表达变化
通过4种病原菌对仿刺参进行免疫刺激,采用实时荧光定量方法检测体腔细胞中AjSARM基因在刺激后4、24、48 h和96 h的表达变化情况。在灿烂弧菌刺激组,AjSARM基因在4 h和24 h都下调,48 h上调后72 h又显著下调,直到96 h才显著上调。在假交替单胞菌组,AjSARM基因表达水平在注射后4、24 h和48 h均下调,到72 h表达水平开始上调,96 h表达水平显著上调7倍。在希瓦氏菌组,AjSARM基因的表达变化情况和灿烂弧菌组相似,在4、24 h和72 h均处于下调表达,在48 h和96 h表达水平上调。在蜡样芽孢杆菌组,AjSARM基因在4 h表达水平下调,随后在24 h和48 h表达水平上调,在72 h表达水平小幅度下调后在96 h表达水平显著上调。
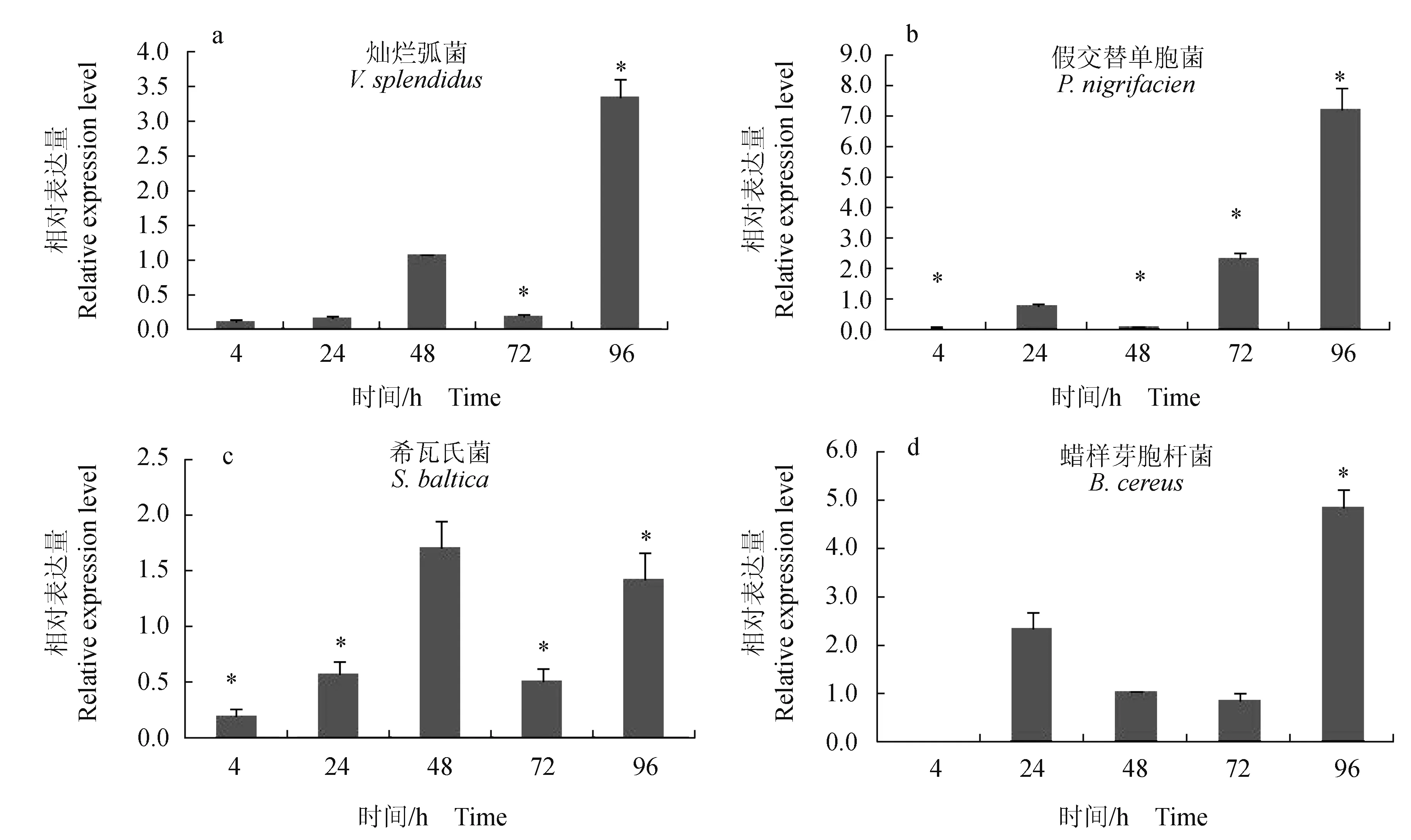
图5 仿刺参体腔细胞中AjSARM基因在4种病原菌刺激条件下的表达变化Fig.5 Relative expression levels of AjSARM in coelomocytes of sea cucumber A. japonicus challenged by four pathogens 试验采用4种病原菌进行体腔注射,剂量为100 μL/头;AjSARM的相对表达量以Cytb的表达水平作为对照;柱形图代表相对表达量的平均值±标准误差,*表示差异显著(P<0.05). Four different pathogens are conducted by coelomic injection for each animal with 100 μL;relative expressions of AjSARM are normalized to the expression of Cytb; the histogram indicates mean expression level of 3 tested pools±SE; * represents significant difference at the level of P<0.05 from the control.
3 讨 论
仿刺参是我国重要的水产养殖经济品种,随着养殖规模的不断扩大,病害的发生成为限制其发展的重要因素。近年来对仿刺参免疫病害相关研究已经成为热点,包括基因的克隆[9-13,17-20]、转录组测序[21-24]和miRNA-mRNA相互作用[25-31]等研究。仿刺参TLR信号通路MyD88依赖途径的研究比较深入,对于MyD88非依赖途径的研究相对较少。在哺乳动物中,MyD88依赖途径通过快速激活NF-κB和MAPK产生炎症细胞因子发挥免疫功能,MyD88非依赖途径通过诱导IFN-β,缓慢激活NF-κB和MAPK产生炎症细胞因子发挥免疫功能[32]。在MyD88非依赖途径中,TLR3和TLR4可以识别细菌或病毒所产生的PAMPs,通过TRAM或TRIF以及SARM的共同作用将信号传递给下游分子。在仿刺参转录组测序结果中未发现TRAM和TRIF的基因片段,但是鉴定到了SARM基因的部分片段,这一接头分子的发现将为仿刺参MyD88非依赖途径的研究奠定基础。
3.1 仿刺参SARM基因的序列特征
SARM基因具有高度的保守性,从节肢动物到哺乳动物中均普遍存在[33-34]。笔者采用RACE法克隆获得AjSARM基因的cDNA全长为3874 bp,开放阅读框为2412 bp,编码803个氨基酸。二级结构由2个SAM结构域以及1个C端的TIR结构域组成,这和典型的SARM基因结构域相比缺少了N端的ARM区。对紫色球海胆的SARM氨基酸序列进行保守结构域预测发现也缺少ARM区,但是在棘冠海星中却含有ARM重复序列。秀丽隐杆线虫SARM基因的同源物TIR-1的N末端含有一段冗余序列,然而在文昌鱼和人类中未出现,推测这段冗余序列在进化过程中消失了[35]。仿刺参SARM基因的结构差异是否会影响其功能,笔者通过荧光定量PCR技术对AjSARM基因在仿刺参不同发育阶段和不同病原菌刺激后的表达模式进行了探索,为解析AjSARM基因的功能奠定基础。
3.2 仿刺参SARM基因的功能分析
研究发现,秀丽隐杆线虫SARM(TIR-1)基因参与了嗅觉神经的发育过程[6]。本试验采用实时荧光定量PCR检测AjSARM基因在仿刺参不同发育阶段的表达模式,发现AjSARM基因从受精卵开始到原肠胚期均为低表达,从小耳期开始至五触手时期都是高表达。Bishop等[36]使用免疫学手段检测了黑海参(Holothuriaatra)幼体的神经发生,发现黑海参神经起源于耳状幼体的原肠胚顶端。AjSARM基因正是从仿刺参小耳期开始出现高表达的,推测该基因参与了仿刺参神经发育的过程。
SARM基因不仅参与了秀丽隐杆线虫神经元发育过程,还能有效地参与免疫应答[6-7]。在组织分布试验结果中发现,AjSARM基因在体腔细胞中的表达量最高。体腔细胞作为仿刺参重要的免疫效应器,在应对不同病原体刺激时能产生特异性的免疫应答,因此试验选取体腔细胞来检测AjSARM基因如何应对4种病原菌的刺激。在灿烂弧菌、假交替单胞菌和希瓦氏菌刺激组,AjSARM基因在4 h和24 h均为被抑制表达,然而在蜡样芽孢杆菌组,24 h表达上调。除了希瓦氏菌组外,其他3个组的AjSARM基因在96 h表达量达到最高。AjSARM基因在应对不同病原菌刺激后不同时间点所表现出的动态表达模式表明它参与了仿刺参体腔细胞免疫应答的过程。
4 结 论
SARM基因作为MyD88非依赖途径的接头分子,在Toll样受体信号传导过程中发挥着重要作用。采用RACE法对仿刺参SARM基因进行全长克隆,获得cDNA全长为3874 bp,开放阅读框为2412 bp,编码803个氨基酸。对氨基酸序列进行保守结构域预测,发现存在TIR保守结构域,但是和其他物种的SARM蛋白结构存在一定的差异。笔者在mRNA层面对AjSARM基因的功能进行了初步探索。采用实时荧光定量PCR方法对AjSARM基因在不同发育阶段和不同病原菌刺激后的表达模式进行分析,试验结果表明,AjSARM基因参与了仿刺参的发育和免疫应答的过程。本试验不仅探索了仿刺参MyD88非依赖途径接头分子SARM的功能,丰富了仿刺参Toll样受体信号通路研究,同时为棘皮动物免疫系统进化研究提供了科学依据。
