拟南芥全基因组精细插入及缺失分子标记
2021-01-09施亚磊于静芳
施亚磊,于静芳,吴 珂,周 俊*
(1. 华南师范大学生物光子学研究院,激光生命科学教育部重点实验室,广州 510631;2. 华南师范大学生物光子学研究院,广东省激光生命科学重点实验室,广州 510631;3. 河南大学生命科学学院,开封 475004)
拟南芥(Arabidopsis thaliana)作为模式生物已有将近50年的历史,被广泛应用于植物生物学的遗传发育研究。拟南芥是二倍体,含有5对染色体,大约27 655个蛋白编码基因[1]。目前,基于正向遗传学(forward genetics)的突变体筛选仍然是遗传研究中十分重要的手段,通过自发突变或人工诱变获得感兴趣的表型性状改变,然后找到这些特定性状变化所对应的突变基因,并揭示其生物学功能。由此可见,鉴定异常表型的基因突变是解决植物生物学问题的关键一步,然而该过程通常是困难并且费时的。目前,在拟南芥的研究中已经发展出很多方法来鉴定突变基因。其中,图位克隆是较常用的方法,它依赖于染色体上位置已知的遗传或分子标记[2-3]。分子标记多指DNA标记,是能够鉴定区别个体或物种差异的染色体上的DNA序列,其源自于不同类型的突变,如插入、缺失、替换、重构或随机重复DNA的复制错误等[4]。分子标记种类很多,如简单序列长度多态性(simple sequence length polymorphism, SSLP)、扩增片段长度多态性(amplified fragment length polymorphism, AFLP)、酶切扩增多态性(cleaved amplified polymorphisms, CAPS)、限制性片段长度多态性(restriction fragment length polymorphism, RFLP)、随机扩增多态性 DNA(random amplified polymorphic DNA,RAPD)、简单重复序列(simple sequence repeat, SSR)和单核苷酸多态性(single nucleotide polymorphism,SNP)等[5-8]。可靠分子标记的开发将极大地促进拟南芥的图位克隆。
插入或缺失(insertion/deletion, INDEL)标记是常用的基于聚合酶链式反应(polymerase chain reaction,PCR)的分子标记。PCR产物通过电泳分析,可以直接获得差异信息和连锁数据[3]。目前,许多INDEL标记已经被开发和报道,并收集在TAIR(Arabidopsis information resource)数据库中。然而,一些已报道的分子标记只能在特定的PCR或电泳条件下工作,或者因不同拟南芥生态型的扩增产物差异太小而无法区分[9]。此外,这些可利用的标记并非均匀地分布在染色体上,通常只有那些与已报道研究的突变体紧密连锁的分子标记才被提交到了TAIR数据库,使得好用的标记数量非常有限,不利于基因定位和连锁分析[10]。随着测序技术的快速发展和测序成本的大幅降低,越来越多的生态型的基因组序列被公布,这使得从全基因组层面开发分子标记成为可能[11]。因此,通过比较基因组学的方法,建立一个精细的、方便使用的分子标记非常有助于拟南芥的图位克隆。
为了寻找尽可能多的分子标记以提高图位克隆效率,本研究进行了哥伦比亚(Columbia, Col)和兰兹伯格(Landsberg erecta,Ler)的全基因组序列比对分析,找出了2个基因组具有差异序列的保守区域。从插入或缺失染色体物理位置中,筛选得到2 321个INDEL标记。此外,我们提供了对应每个分子标记位点的至少一对引物序列(共计4 764对)。本工作能够极大地方便相关研究者找到合适的分子标记,提高拟南芥突变体的图位克隆效率。
1 材料与方法
1.1 植物材料与培养
植物培养方法与之前文献[12-14]所述一致,拟南芥野生型Col、Ler种子经含0.05% Triton X-100的75%乙醇震荡消毒10 min,随后于超净台中用100%乙醇置换75%乙醇,并将种子转移到灭菌滤纸上,干燥15 min后,撒种于1/2MS固体培养基上。培养板置于4℃冰箱中黑暗处理2 d,然后放置于人工培养箱培养5 d,光周期为16 h(光)/8 h(暗),昼夜温度为23℃(昼)/21℃(夜)。
1.2 参考基因组的获取
从 NCBI(national center for biotechnology information)数据库获得拟南芥Col和Ler参考基因组。Col基因组下载地址:https://www.ncbi.nlm.nih.gov/assembly/GCF_000001735.4;Ler基因组下载地址:https://www.ncbi.nlm.nih.gov/assembly/GCA_001651475.1。
1.3 基于基因组比较的分子标记开发
使用MUMmer3.23软件对Col和Ler的基因组进行比较,鉴定全基因组中具有INDEL差异的保守区域。取出INDEL在Col参考基因组的上游和下游各150 bp序列,通过Primer3批量设计引物(条件:引物长度为23个碱基,GC含量约43%,Tm值约53℃,引物对应产物片段长度为80~150 bp)。使用MFEprimer2软件对引物的结合位点数量、引物3'端稳定性、退火温度、GC含量等进行检测。
1.4 DNA提取
DNA提取采用TPS方法[15]。取一片4周拟南芥叶片于 1.5 mL EP 管中,加入 200 μL TPS 缓冲液(100 mmol/L Tris-HCl,pH 8.0 ;10 mmol/L乙二胺四乙酸钠,pH 8.0;1 mol/L氯化钾),电动研磨均匀后,于60℃放置20 min,随后 12 000 r/min离心 10 min。取上清,加入等体积的预冷异丙醇,12 000 r/min离心10 min后弃上清,沉淀用70%酒精悬浮,12 000 r/min再次离心10 min后弃去上清,干燥1 d,获得DNA样品。
1.5 PCR扩增和琼脂糖凝胶电泳
从拟南芥全基因组中选取23个均匀分布在5条染色体上的候选标记合成引物,并进行PCR扩增和琼脂糖凝胶电泳[16]。PCR 体系(20 μL):10 μL 2×Green Taq Mix(Vazyme);2 μL 引物 ;1 μL DNA 模板 ;7 μL H2O。PCR 扩增条件如下 :94℃预变性 3 min ;94℃变性30 s;53℃复性30 s,72℃延伸30 s,35 个循环;最后72℃延伸5 min。琼脂糖凝胶电泳条件:在120 V电场强度下,经4%琼脂糖凝胶电泳30 min后,在紫外光下观察拍照。
2 结果与分析
2.1 Col和Ler全基因组INDEL标记开发和统计
为了开发Col和Ler全基因组的INDEL标记,我们先使用MUMmer3软件将Col的基因组和Ler的基因组进行比对,找出含有INDEL的保守序列片段,再使用Primer3软件进行引物设计,通过MFEprimer2软件对引物进行验证。表1为各个染色体上的INDEL及分子标记的统计。从表1可知,基因组中平均每0.1 Mbp染色体长度大约有2个分子标记,平均每4到5个基因就有1个分子标记。图1展示的是全基因组的INDEL(≥6 bp)及分子标记的长度分布。我们可以看到,随着分子标记差异长度的增加,INDEL及分子标记的数量呈递减趋势。
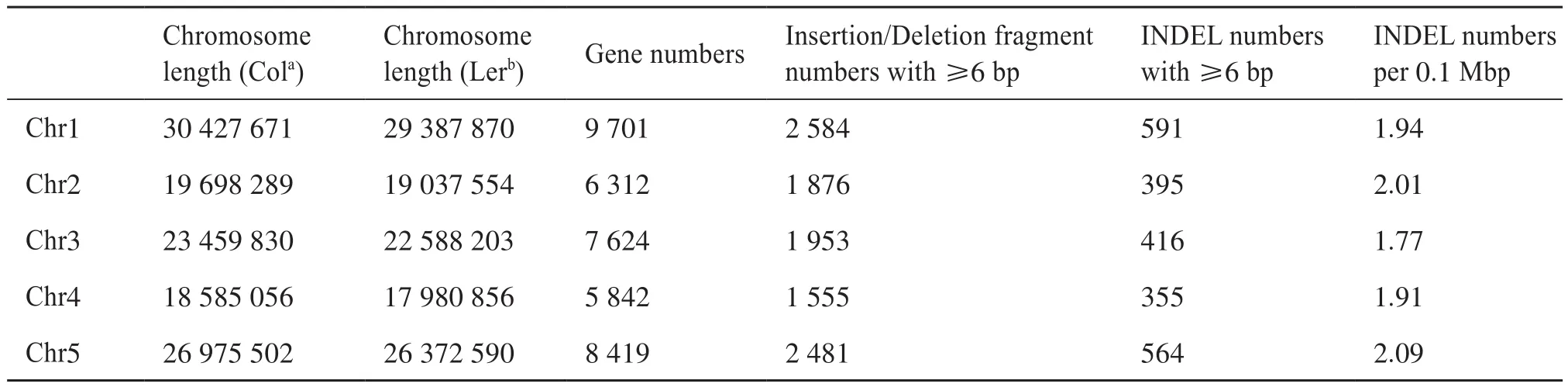
表1 Col和Ler之间INDEL数量的鉴定Tab. 1 Identification of INDEL numbers between Col and Ler
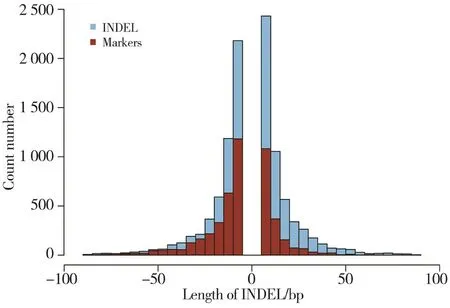
图1 Col和Ler基因组INDEL(≥6 bp)及分子标记的长度分布Fig. 1 Length distribution of INDEL and putative markers between Col and LerINDEL长度不小于6 bp,蓝色代表INDEL,红色为分子标记。随着长度增加,INDEL及分子标记的数量呈递减趋势。Col基因组为参考序列。The length of INDEL (blue) is no less than 6 bp, the molecular markers are labeled whth red. As the length increases, the number of INDEL and molecular markers decreases. The reference genome is from Col.
2.2 INDEL标记在各个染色体上精细分布
从10 449个不小于6个碱基插入或缺失染色体的物理位置中,我们筛选得到2 321个INDEL标记。为了更好地展示INDEL标记在各个染色体上的分布,我们将基因、INDEL和分子标记按照每50 kb的区间进行统计。图2最外侧为染色体的具体起始位置,基因(橙色)、INDEL(蓝色)和分子标记(深灰色)依次排开。分子标记数目最多的是第一号染色体,有591个,而分子标记密度最大的是第五号染色体,平均每0.1 Mbp的染色体物理长度有2.09个(表1和图2)。同样,从图3可以看到第一号染色体的15 Mbp处、第三号染色体14 Mbp处和第五号染色体的14 Mbp处的基因、INDEL和分子标记数量都较其他区域少,应为着丝粒的区段。
2.3 随机分子标记的验证分析

图2 INDEL及分子标记在Col和Ler全基因组上的分布Fig. 2 Distribution pattern of INDEL and markers of Col against Ler最外侧为染色体。从第2圈向内,依次为基因(橙色)、INDEL(≥6 bp,蓝色)和分子标记(深灰色),每50 kb为1个统计单位。The outermost part represents Col chromosome. From the second circle inward, there are genes (orange), INDEL (≥6 bp, blue) and molecular markers(dark gray) in order, with a statistical unit of every 50 kb.
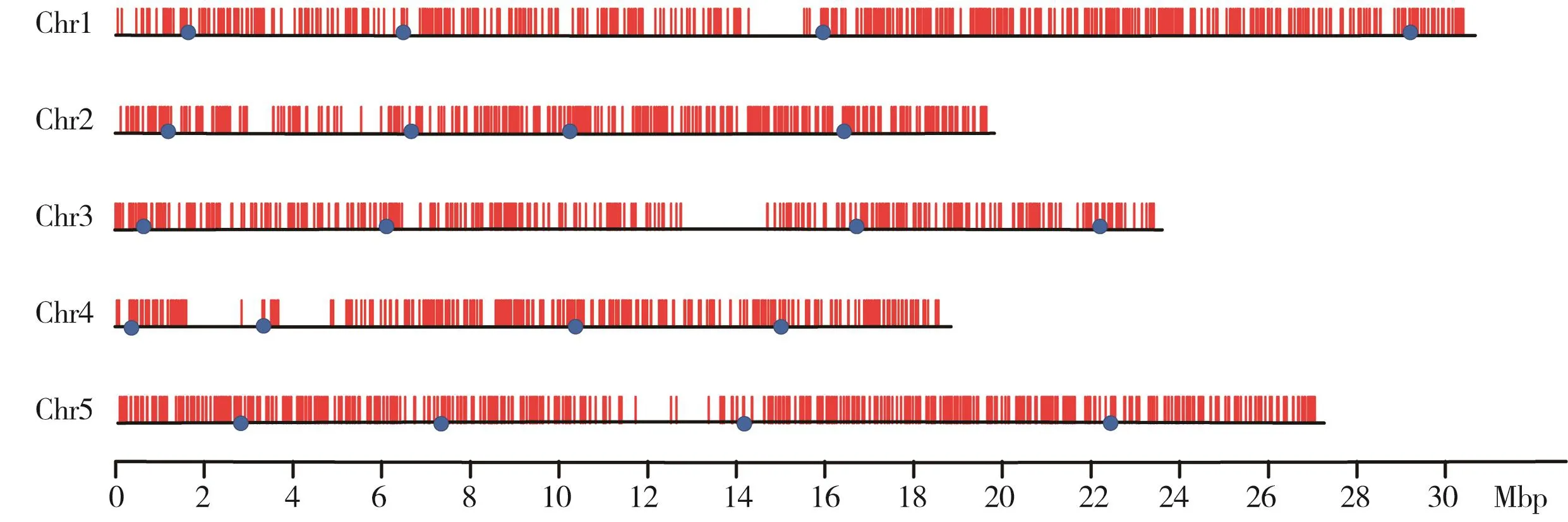
图3 设计的分子标记在染色体上的分布Fig. 3 The distribution of designed molecular markers on chromosomes红色竖线表示每个分子标记在染色体上的位置,蓝色圆点表示为验证的初定位分子标记位点。The red vertical bars indicate the position of each molecular marker on the chromosome, and the blue dots indicate the site of veri fied molecular marker used to first-pass mapping.
针对这2 321个INDEL标记,我们共设计了4 764对引物,使得每个位点有一对或多对引物可供选择。详细引物序列及其染色体的物理位置可从At_InDel_Marker[23](https://github.com/zhoujun1988/AtMarker/blob/master/At_InDel_Marker)获取。在此基础上,我们选择了均匀分布在拟南芥5条染色体的20个分子标记(表2),并通过PCR和琼脂糖凝胶电泳对其特异性进行了验证。图4结果显示,这些引物能够在统一的反应体系和扩增条件下获得稳定的结果。Col与Ler混合样品能够扩增出明显差异条带,使得该套分子标记可直接应用于突变基因的初步定位分析。
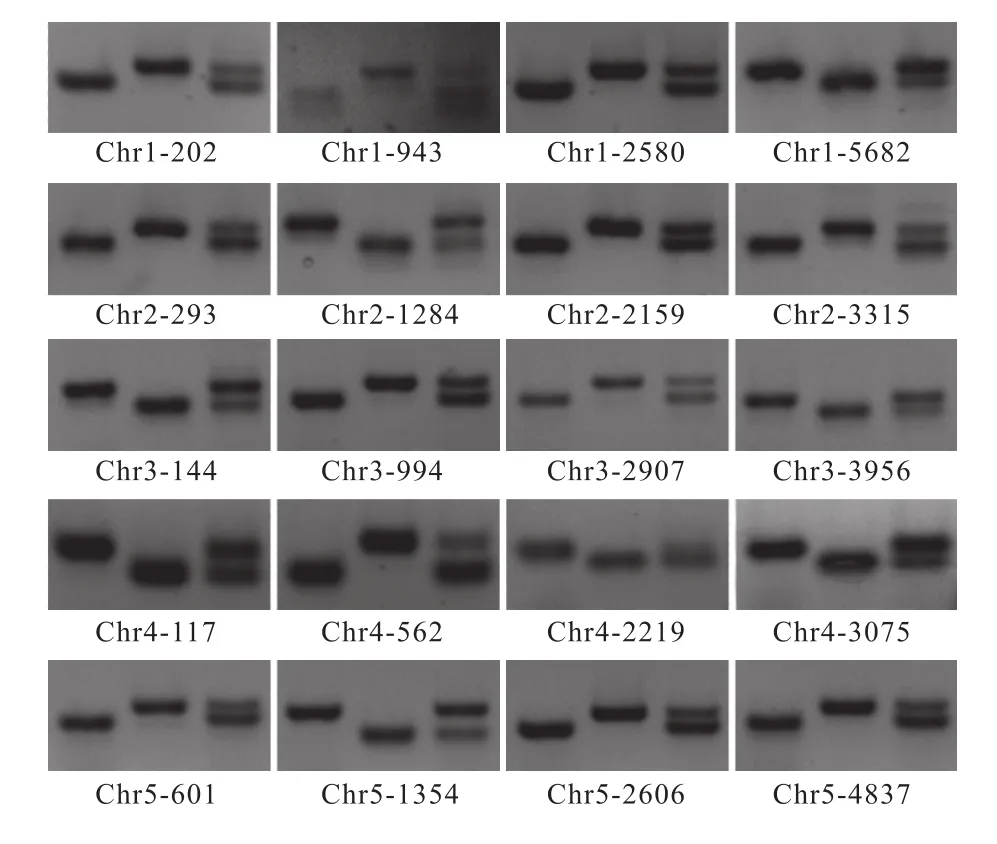
图4 PCR验证用于鉴定突变位点的初定位分子标记Fig. 4 Verified the molecular markers used to first-pass mapping by PCR3个泳道的DNA模板依次来自于Col、Ler及Col与Ler混合样品。DNA templates for the three lanes were obtained from the Col, Ler, Col and Ler mixed samples.
3 讨论
图位克隆是鉴定基因突变位点的经典方法,其实施离不开可靠的遗传标记或分子标记。Col和Ler是拟南芥最常用的2个生态型。基于这2种生态型间的多态性,本研究利用比较基因组学,筛选得到应用于拟南芥基因图位克隆的2 321个新的INDEL标记,并通过PCR验证了随机均匀分布在5条染色体的20个分子标记,后者可直接应用于基因的快速初定位。本工作提供的精细分子标记,能够极大地方便研究者找到合适的候选标记,提高图位克隆作图效率。
经过几十年的发展,DNA分子标记已经从最初的基于印迹反应的RFLP,到基于PCR的SSLP、AFLP、SSR和INDEL,再到基于测序技术的SNP[17-18]。早期的RFLP、AFLP等虽然都对物种鉴定、进化分析和基因图位克隆等有巨大的帮助和促进,但由于各自的不足(如试验条件要求高、操作复杂、或者不是共显性等),这些分子标记的应用还是受到了极大的限制[19-20]。SNP的分子标记数量最多,却因为鉴定需要特殊的条件(如酶切或者测序),使其应用成本较高[23]。SSR和INDEL都可以通过PCR和凝胶电泳来使用[21]。SSR也属于INDEL的范畴,然而,INDEL存在数量更大、对基因组的覆盖更广、应用更便捷高效的优势。
在众多分子标记中,PCR检测多态性(如CAPS、SSLP、AFLP、RAPD)因操作相对简单、结果直观可靠成为标记首选[3,22]。针对拟南芥而言,在TAIR数据库中,由于大多数分子标记并非同时开发,研究者需要事先对试验条件进行优化,一些分子标记往往不能使用相同的PCR反应体系和扩增条件,使得操作变得繁琐。本研究针对新开发的2 321个INDEL标记,设计了至少1对或多对(共计4 764对)可供选择的引物(At_InDel_Marker[23]和表2)。重要的是,我们初步测试的20个分布于5条染色体的分子标记可采用相同的PCR条件得到稳定的结果(图4),这大大提高了工作效率。通过该研究能够方便研究者找到合适的分子标记,在常见的仪器条件和较低的试验成本下可快速进行突变体基因定位。
