山羊内源性鼻内肿瘤病毒enENTV-FJ1 株全基因组测序及生物信息学分析
2020-12-30江锦秀张靖鹏林裕胜毛坤明黄丽丽胡奇林
江锦秀,张靖鹏,林裕胜,毛坤明,游 伟,黄丽丽,胡奇林*
(1.福建省农业科学院畜牧兽医研究所,福建 福州 350013;2.福清市畜牧兽医技术服务中心,福建 福州 350300;3.福建师范大学,福建 福州 350108)
内源性逆转录病毒(Endogenous retrovirus,ER⁃Vs)广泛存在于真核生物中,是在生物进化过程中整合到宿主基因组DNA 中的,并在大多数真核生物种系中以孟德尔遗传方式垂直遗传给后代,与致病性外源性逆转录病毒具有较高的相似性[1-2]。分子病毒学研究显示ERVs 具有限制外源性逆转录病毒感染的作用,比如内源性绵羊肺腺瘤病毒(Endogenous jaagsiekte sheep retrovirus,enJSRV)可以通过受体竞争干扰外源性绵羊肺腺瘤病毒(Jaagsiekte sheep retro⁃virus,JSRV)的侵入,还可以通过干扰外源性JSRV的复制来抵御外源性病毒对宿主的侵袭[3-4]。在绵羊、山羊和大多数野生山羊属基因组中均存在15 拷贝~20 拷贝与外源性逆转录病毒核酸序列有很高相似性的ERVs[4]。典型的ERVs 是具有gag、pro、pol、env 4 个相互重叠或分离的0RF。ERVs 在母羊生殖道高效表达,具有局部免疫调节和防御外源性病毒感染作用。山羊地方性鼻内肿瘤病毒(Enzootic nasal tumor virus of goats,ENTV)属于逆转录病毒科正逆转录病毒亚科β 逆转录病毒属B/D 型逆转录病毒,是引起山羊地方性鼻内肿瘤(Enzootic nasal tumor,ENT)的病原[5]。山羊体内同样具有与ENTV-2 型(ENTV-2)对应的内源性鼻内肿瘤病毒[4](Endogenous enzootic nasal tumor virus,enENTV)。Ortín A 等报道ENTV-2 在患ENT 的山羊体内分布广泛,在病羊的扁桃体、淋巴结、外周血单核细胞、脾脏、肺、骨髓中均有分布,但在其肾脏中却无该病毒[6],表明从患病山羊肾脏中提取enENTV DNA 可以免受外源ENTV-2 的干扰。本研究从患ENT 山羊肾组织中提取总DNA,经PCR 鉴定获得了一株enENTV,并对其进行了全基因组及相关基因序列克隆、测序及序列分析,为鉴定内外源山羊鼻内肿瘤病毒提供序列信息,为研究ENTV-2 的致瘤机制奠定了基础。
1 材料与方法
1.1 实验动物和主要试剂1只患ENT病山羊(根据流行病学、临床症状、剖检病变和RT-PCR 检测结果诊断为ENT 病)来自于福清市,取其肾脏经研磨后提取DNA,经RT-PCR[7]检测ENTV-2 为阴性;GoScript Enzyme Mix、OligodT(Promega)、pMD19-T 载 体、PrimeSTAR Max DNA Polymerase、E. coli DH5α 感受态细胞、DL5000 DNA Marker 均购自宝日医生物技术有限公司;Ezup 柱式动物基因组DNA 抽提试剂盒、IPTG、X-gal购自生工生物工程(上海)股份有限公司;
1.2 enENTV-FJ1 株基因组各片段的PCR 扩增和测序参考enENTV-CHN1 株(KU258881.1)基因组序列,利用Primer 6.0 软件设计用于分段PCR 扩增病毒全基因组序列的引物,由福州铂尚生物技术有限公司合成(表1)。取病羊肾脏研磨后的上清利用Ezup 柱式动物基因组DNA 抽提试剂盒提取总DNA,以其为模板,PCR 分段扩增enENTV 的4 个基因片段。PCR 体系模板上样量为4 ng/μL,扩增程序:98 ℃10 s,55 ℃15 s,72 ℃30 s,共35 循环;72 ℃10 min。PCR 产物回收纯化后分别克隆至pMD19-T 载体中,构建重组质粒,经PCR鉴定正确后,由福州铂尚生物技术有限公司测序鉴定。

表1 enENTV 全基因组扩增引物序列Table 1 Whole genome amplification primer sequence of enENTV
1.3 enENTV-FJ1 株全基因组序列同源性及进化关系分析利用DNAStar7.0 软件中的SeqMan 软件对分段扩增的4 个片段的序列拼接得到全基因组序列,命名为enENTV-FJ1。利用MegAlign 软件对enENTV-FJ1株、enENTV、ENTV-2、ENTV-1、enJSRV和JSRV等参考株相应基因进行同源性分析,利用MEGA 7.0 软件构建enENTV-FJ1株全基因组系统进化树,分析en⁃ENTV-FJ1株的遗传进化关系。
1.4 enENTV-FJ1 株的5'LTR、gag、env 序列同源性及进化关系分析利用MegAlign 软件对enENTVFJ1株的5'LTR基因、gag和env基因及其编码的氨基酸序列与enENTV、ENTV-2、ENTV-1、enJSRV和JSRV等相关病毒参考株的gag基因和env基因及其编码的氨基酸序列进行同源性分析;利用MEGA 7.0 软件构建5'LTR、gag 基因、env 基因系统进化树(enENTVCHN1~enENTV-CHN6 6 个参考株的env 基因均无完整的ORF),并分析该株病毒的进化关系。
1.5 enENTV-FJ1 株5'LTR、gag、env 区酶切位点及gag、env 序列差异性分析利用NEBcutter V2.0对enENTV-FJ1 株、enENTV-CHN1~enENTV-CHN6株及ENTV-2 的5'LTR 区基因序列进行酶切位点分析。利用MegAlign 软件对enENTV-FJ1 株的5'LTR、gag 和env 基因及其编码的氨基酸序列与enENTV、ENTV-2、ENTV-1、enJSRV 和JSRV 等相关病毒参考株的相应基因序列及其编码的氨基酸序列进行差异性分析。
2 结果与讨论
2.1 enENTV-FJ1 株基因组各片段的PCR 扩增和测序结果以患ENT病羊的肾脏提取的DNA为模板,采用enENTV 分段扩增引物经RT-PCR 方法扩增病毒全基因组的4 个片段。结果显示,扩增出与预期大小相符的4个片断目的条带(图1)。测序结果显示,各目的基因片段分别为2 267 bp、2 137 bp、2 461 bp、1 806 bp。表明,正确扩增了enENTV-FJ1株全基因组各基因片段。
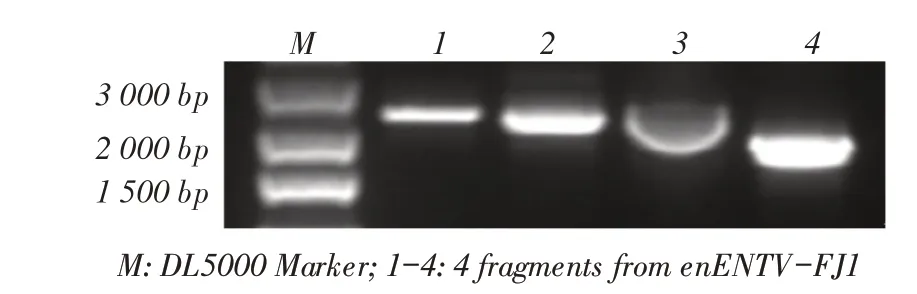
图1 enENTV-FJ1 株全基因组各片段的PCR 扩增结果Fig.1 Fragment amplification results of enENTV-FJ1 whole genome by PCR
2.2 enENTV-FJ1 株全基因组序列同源性及进化关系分析结果enENTV-FJ1 株各片段基因测序结果利用SeqMan 软件拼接出全基因组序列。将序列上传至NCBI,获得序列登录号MN564749。enENTV-2FJ1基因组序列长度为7 923 bp,gag 基因位于569 bp~2 428 bp,编码619 个氨基酸,pro 基因位于2 320 bp~3 189 bp,编码289 个氨基酸,pol 基因位于3 435 bp~5 780 bp,编码781 个氨基酸。env 基因位于5 656 bp~7 512 bp,编码618 个氨基酸。全基因组序列的同源性分析结果显示, enENTV- FJ1 株与enENTVCHN1~enENTV-CHN6 6 个参考株的同源性为95.7%~97.3%,亲缘关系最近;与ENTV-2的同源性为87.0%~93.2%;与ENTV-1 的同源性为87.9%~88.0%;与en⁃JSRV 的同源性为90.5%~90.9%;与JSRV 的同源性为88.2%~88.7%;基于全基因组的进化树结果显示,en⁃ENTV-FJ1 株 与enENTV-CHN1~enENTV-CHN2、en⁃ENTV-CHN4~enENTV-CHN6等5株病毒处于同一进化分支,与enENTV-CHN3株处于一个小的进化分支(图2)。上述结果表明,enENTV-FJ1株与enENTV属于同种病毒。目前,enENTV 全基因组序列只有enENTVCHN1~enENTV-CHN6 等6 条,来 自 于 王 彬 等 从 患ENA 山羊肾脏中测得的enENTV 全基因组序列,该6株病毒的全基因组序列与从山羊分离的ENTV-2 等病毒株的亲缘关系较近,而与来自绵羊的ERVs(enJS⁃RV)全基因组亲缘关系较远,与本研究结果相符[7]。
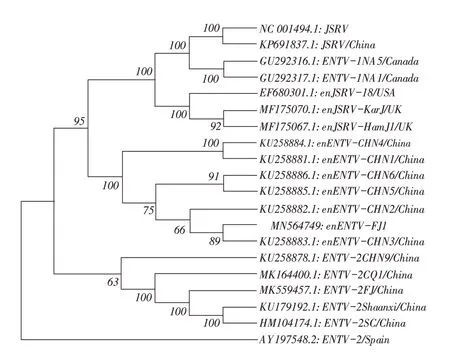
图2 基于全基因组序列构建的enENTV-FJ1 株系统进化树Fig.2 Phylogenetic tree based on whole genome sequences of enENTV-FJ1 strain
2.3 enENTV-FJ1 株的5'LTR、gag、env 序列同源性及进化关系分析结果将enENTV-FJ1株与各参考株的5'LTR、gag 基因、env 基因及其编码氨基酸序列进行同源性分析。结果显示,enENTV-FJ1的5'LTR基因序列、gag基因及其编码氨基酸序列与enENTV参考株相应序列的同源性比其它参考株高,分别为92.8%~99.1%、92.6%~97.5%、92.2%~97.7%;enENTV-FJ1株env 基因及其编码氨基酸序列与enJSRV 参考株相应序列的同源性比其它参考株高(enENTV 参考株的env 基因无完整的ORF),分别为93.3%~93.8%、94.3%~94.8%。构建的5'LTR 进化树结果显示,en⁃ENTV-FJ1 株与enENTV-CHN2 株处于同一进化分支(图3A);gag 基因的进化树结果显示,enENTV-FJ1株与ENTV-2CHN9 株处于一个小的进化分支,与en⁃ENTV-CHN6 株同处于一个较大的进化分支(图3B);env基因的进化树结果显示,enENTV-FJ1株与ENTV-2/Spain株处于同一进化分支(图3C)。表明,enENTVFJ1的5'LTR、gag基因、env基因与来自山羊的ENTV-2病毒株亲缘关系更近。
2.4 enENTV-FJ1 株的5'LTR 区酶切位点及gag、env 序列差异性分析结果对enENTV-FJ1 株、en⁃ENTV-CHN1~enENTV-CHN6 株5'LTR 区以及ENTV-2参考株的5'LTR 区进行酶切位点分析,结果显示,所有ENTV-2 参考株的5'LTR 区均有Hpy188Ⅰ、NlaIV、MowⅠ这3 个酶切位点,而7 个enENTV 株的5'LTR 区均无这3 个酶切位点,这是否为内源enENTV 与外源ENTV-2的区别,需要今后分析更多内源enENTV全基因组序列来验证。

图3 enENTV-FJ1 株5'LTR(A)、gag(B)、env(C)基因系统进化树Fig.3 Phylogenetic tree based on 5'LTR(A), gag(B) and env(C) gene sequences of enENTV-FJ1 strain
enENTV 及enJSRV 的gag 基因与外源ENTV-2 以及JSRV 的gag 基因比较最主要的差异位于366 bp~401 bp(有12个核苷酸的插入),与外源ENTV-1的gag基因比较最主要的差异位于366 bp~401 bp(有8个核苷酸的插入)。与enJSRV、外源ENTV-2、ENTV-1 及JSRV 的gag 基因比较,enENTV-FJ1 株的gag 基因在697 bp~705bp 有9 个核苷酸的插入。2019 年测序的ENTV-2CQ 株在gag 基因有2 处多个核苷酸插入,这一序列特征与山羊源ERVs(XM018046415)的gag基因相同而区别于其它ENTV-2 病毒株,表明ENTV-2CQ 株可能是一株古老病毒,ENTV-2 与ERVs 有着复杂的进化关系[8],本研究结果显示,enENTV-FJ1株的gag 基因在这两处也存在多个核苷酸插入。王小玉等研究显示,与外源性ENTV gag 基因最显著的区别在于,内源性ERVs gag 基因的783 bp~803 bp,编码7 个多聚脯氨酸“PPPPPPP”,而在ENTV 的对应区域则发生了缺失突变和点突变,未编码连续的7 个脯氨酸,这可能是影响gag 功能的主要区域[9]。本研究结果显示,enENTV 及enJSRV 等内源性病毒株与ENTV-2、ENTV-1、JSRV 等外源性病毒株的gag 基因编码的氨基酸序列最主要区别在aa117~aa135(脯氨酸富集区,有2~4 个不连续氨基酸插入)和aa174~aa239(存在不连续的氨基酸位点变异);与enJSRV、外源JSRV 和ENTV 相比,enENTVFJ1 株的gag 基因编码的氨基酸序列在aa229~aa232处插入3 个脯氨酸(aa229~aa236 为7 个连续的脯氨酸),与王小玉等研究结果相符;这2 个区间的多肽序列是否可以作为区别内外源性ENTV 和JSRV 的多肽序列从而建立免疫学检测方法,需要进一步的试验验证。
enENTV及enJSRV的env基因末端与外源ENTV-2以及ENTV-1 的env 基因相比有12 个核苷酸的的缺失,是这些内源病毒与外源ENTV 的区别位点,这为建立ENTV-2特异性检测方法提供了靶点。
与相应病毒参考株的env 基因编码的氨基酸序列相比,enENTV-FJ1 株和ENTV-2 株的氨基酸序列在aa1~aa7 处插入了7 个氨基酸(MFVFFRR)。与外源性病毒株相比,enENTV-FJ1 株的跨膜蛋白(Transmembrane protein,TM)胞质尾区无YXXM 基序。有报道称外源JSRV 在env 基因编码蛋白的胞质尾区存在YXXM 基序,而内源性enJSRV 则无此基序,YXXM 基序是区别内外源病毒的可靠分子标记,是外源性JSRV env基因引起成纤维细胞转化的因素[10-11]。外源性ENTV-2SC、ENTV-2Shaanxi、ENTV-1NA1、ENTV-1NA3、ENTV-2CQ1 等参考株env 基因编码蛋白的胞质尾区均存在YXXM 基序[12-14],表明YXXM 基序也可能是区别内外源ENTV 的分子标记。enENTV 与外源性ENTV 全基因组序列的差别主要在5'LTR区、gag基因和env基因,enJSRV和JSRV的全基因组序列高变区也集中在5'LTR 区、gag 基因和env 基因[15],本研究结果与之相似。
enENTV-FJ1 株在aa510~aa515 存在LDLLQL 保守基序,而这在许多反转录病毒中已经证实与引起宿主的免疫抑制有关,表明LDLLQL 基序也可能是ENTV-2 引起宿主免疫抑制的原因之一[6,16]。
本研究从1 只患ENT 山羊的肾脏中获得1 株全长为7 923 bp 的enENTV 全基因组序列,补充了山羊enENTV 全基因组序列资源。但由于RT-PCR 技术的局限性,所测得的enENTV-FJ1 株的5'LTR 和3'LTR基因序列可能不完整,后续将利用5'RACE 和3'RACE等技术对enENTV-FJ1 株基因组末端序列扩增并测序,以保证其序列的完整性。本研究为研究ENTV-2的致瘤机制奠定了基础。
