基于细胞图卷积的组织病理图像分类研究
2020-12-26崔浩阳张敬谊
崔浩阳,丁 偕,张敬谊
万达信息股份有限公司 数据智能部,上海201112
1 引言
随着深度学习模型、优化算法以及高性能GPU 的出现,使得组织病理学中的计算机辅助诊断研究得到了快速发展。具体而言是利用深度卷积神经网络(CNN)实现对组织病理切片图像的细胞分割、腺体分割、癌症分级、突变鉴定以及预测癌症复发等任务[1-2]。CNN 用于各种任务并取得一定表现主要取决于从图像数据中自动挖掘高维特征的能力,并不依赖于人工创建的组织学特征。Campanella 等[3]将前列腺穿刺切片作为数据集,进行训练并测试CNN 模型鉴别前列腺病灶良恶性。2018年Coudray等[4]利用TCGA数据库中有关肺癌的样本,训练测试CNN模型鉴别腺癌、鳞癌以及识别基因突变的准确率,取得了肺腺癌、鳞癌AUC 为0.97,基因突变的AUC为0.85的结果。此外,在2015年11月至2016 年11 月举行的CAMELYON16 大赛[5],进一步推动了深度卷积网络在病理图像识别领域的发展。但由于病理医生的诊断依据主要是根据病理图像中异常细胞的形态、结构以及空间分布等因素给出最终的病理分析结果。而传统CNN处理的源数据是具有像素值的结构化二维阵列图像,这种结构化的二维阵列数据形式难以表达组织病理学中细胞之间以及腺体之间的关系和组织学特征,也忽略了微观细胞之间的空间关系,因此可能会丢失提高模型性能的一些关键特征信息。
本文引入基于细胞的图结构,利用细胞之间的拓扑结构对组织病理图像进行几何结构建模[6],使模型学习到细胞本身特征的同时,还学习到细胞之间的空间关系特征。提出了图卷积神经网络的病理图像癌症分类方法,通过提取病理图中微观细胞作为图结构中的节点,把细胞之间的空间关系视为图结构中的边。为了准确获得节点的特征信息,本文应用了细胞分割网络,利用分割出的细胞提取其外观特征。为了使节点融合多尺度信息,引入了GraphSAGE模块作为图卷积模块,由于组织学图像中的细胞数量众多,构建出的细胞图结构会存在大量的冗余节点,因此,本文将分层池化和全局池化结合,最终获得具有代表性的节点特征作为图结构的表达。
2 相关工作
图神经网络的表示法最早由Gori 等人[7]在2005 年提出,并在2009年由Scarselli等人[8]进行了进一步阐述,这些早期的研究通过迭代的方式,利用循环神经结构传播邻居信息,直到达到一个稳定的不动点,来学习节点的表示,在此过程中计算代价大。在受到CNN 在计算机视觉领域的启发后,为了减少计算量,利用卷积的概念重新定义了卷积算子,提出了多种图卷积神经网络(GCN)[9],其中主要分为基于频谱的GCN[10-12]和基于空域的GCN[8,13]。频谱的图卷积网络方法一般处理整个图结构,难以并行处理和扩展,基于空域的图卷积方法主要通过聚集节点信息直接在图上进行卷积,并结合采样策略,能够减少计算复杂度。Hamilton[14]提出的Graph-SAGE 方法引入了节点特征聚合函数和小批量训练策略提高了模型的泛化能力。为了进一步学习更好的图结构中的层次特征并降低计算复杂度,已经提出了很多不同的图池化方法,其中常见的方法:简单的在最后一层中将所有节点特征求和;引入一个连接到图中所有节点的虚拟节点以求平均值;使用深度学习框架聚合节点特征;也有将CNN 中架构的通道堆叠的方法应用到所有节点特征中。
GCN 在病理图像应用方面,Lu 等[15]提出一种图神经网络的新型模型,将每个从数字化病理全切片(Whole-Slide Images,WSI)中裁剪的图块作为图结构中的节点,以此构建整张WSI 的图结构,从而避免丢失图块与图块之间的视觉信息,但是该方法并未涉及细胞级图结构的构建。考虑到内存的限制,常规技术依赖于切图块的方法进行CNN训练,但由于癌变区域会聚集在一起,这种方法通常会忽略每个图块之间的空间关系,因此Konda 等[16]提出在病理图像中构建节点关系,将每个图块作为图结构中的一个节点,并添加一些人工特征,以此突出GCN 在医学数据分析的潜力。Zhang[17]等人尝试在宫颈癌细胞分割的工作中应用GCN,提出一种全卷积网络和图方法相结合的分割方法,需要通过掩码构造图像的图结构,并以动态编程的方式获取图结构中最优的全局路径,实现过程复杂。目前GCN 在病理图像的应用通常是将整个图块作为一个节点以构建整个WSI的图结构,因此丢失了微观细胞之间的空间关系特征。本文将细胞图结构与图神经网络结合,利用细胞之间的拓扑关系对病理图像的几何结构进行建模,使得C-GCN 不但能学习到细胞本身的特征,还学习到细胞之间空间关系特征,从而增强模型分析判别的能力。
3 方法
基于细胞图卷积的组织病理图像分类方法主要分为三部分:利用细胞分割网络获得细胞掩码;利用细胞掩码获取细胞的颜色、形状、空间结构以及外观特征构建细胞图结构;利用C-GCN 进行图网络训练。技术路线图如图1所示。

图1 C-GCN总体流程
3.1 细胞分割网络
准确地分割细胞是生成可靠节点特征的关键,因此,本文优选高性能的Hover-Net[18]作为细胞分割网络,其能准确地分割出每个细胞的边界,Hover-Net 利用细胞像素的垂直和水平距离内编码信息等到它们的质心,之后利用距离分类聚集的核,预测出最后的分割结果。本文利用Hover-Net获得的掩码提取细胞形状和外观特征,选取17 个具有代表性的特征描述子,分别为:平均核强度、前/背景平均差异、细胞强度标准偏差、细胞强度偏度、细胞强度的平均熵、灰度共生矩阵(GLCM)的差异、灰度共生矩阵(GLCM)的同质性、灰度共生矩阵(GLCM)的能量、灰度共生矩阵(GLCM)的角二阶矩(ASM)、偏心率、面积、轴的最大值、轴的最小值、周长、稳定性、方向以及质心坐标。
3.2 构建图结构
为了能够使用GCN,必须将病理图像转为图形的表示。一个图的定义为G=(V,E),V为节点的集合,E为边的集合,对于每个节点i,具有d维的节点特征xi∈Rd,可用矩阵表示XN×D,其中N表示节点数,D表示特征向量的维度。ei,j表示节点i和节点j的边,其属于集合E。
本文以分割出来的细胞作为节点,相邻细胞之间的欧几里得距离作为边的属性,以此构建细胞图结构。以欧几里得距离作为相邻细胞之间分配边的依据,其距离单位为像素,本文采用的实验数据均在20倍率下提取,结合实际观测经验和计算便捷,选择距离小于100像素作为分配边的阈值,能够较好表达病理图像中细胞之间的关系,不会出现过于稠密或稀疏的细胞图结构,并将这个数值作为超参数,在其他实验中可根据具体情况进行调整。图结构的构造分为以下两步:
(1)分割细胞,结合细胞掩码提取细胞的形状和外观特征,总共计算出17个代表性特征,这些特征用于形成节点特征矩阵。
(2)在细胞图结构中,将图结构中的边定义为两个细胞之间的相互作用,本文假设欧式距离较小的细胞更可能相互作用,因此在两个细胞之间的距离固定,则在它们之间分配一条边,此外,为了能够使节点融合到更多邻接节点的信息,本文将欧式距离小于100像素的两个细胞之间均分配一条边。
3.3 图网络结构
细胞图结构构建完成后,组织病理图像的癌症分类任务即可视为对图的分类问题。GCN通过节点迭代聚合学习节点特征的表示,在网络的隐藏层中计算新的节点特征向量[12],整个图的表示可以通过汇集学习到的所有新节点特征获得。本文采用Hamilton[14]提出具有聚合特征的GraphSAGE 图卷积模块,对于一个给定的节点,它将第k层的节点v特征的输出表示为k-1 层的节点v特征与第k层节点v的所有邻接节点特征聚合的拼接。

其中,表示在第k层节点v的所有邻接节点的特征聚合,表示在k层节点v的特征,表示在k-1层节点v的特征,(,∀u∈N(u))表示在k-1 层中节点v的邻接节点u的特征向量,W 表示参数矩阵。
图的节点特征计算公式可简化为:

其中,V 为节点特征矩阵,A 是邻接矩阵,SAGEConv是GraphSAGE的卷积运算符。为了提高模型的泛化能力,本文引入图池化模块,将图池化过程分为两个部分,在每次GraphSAGE 之后进行一次分层池化,减少一定数量的节点,该过程的作用与CNN中的池化作用类似,它能够增加模型的鲁棒性并能够提取具有一般性的节点特征。在分层池化之后再进行一次全局池化,将整个图中的所有节点特征进行最大值池化和平均值池化,并将其拼接形成一维特征向量,图卷积中分层池化的具体工作流程如图2所示。

图2 分层池化示意图
从图2中看出,分层池化引入了额外的训练参数投影向量p,节点特征矩阵Xl与p 矩阵相乘后得到y,该过程是估计每个节点到投影向量的分数,可选择分数最大的k个节点,再按照节点的索引idx获得其在原Xl中的节点特征矩阵X͂并与经过激活函数Relu激活后的͂进行点乘,从而获得进攻池化后的节点特征矩阵Xl+1,其对应新的邻接矩阵Al+1则按照索引idx在原邻接矩阵Al中获得。
全局池化在分层池化之后,将最大值池化和平均值池化进行拼接,作为全连接层的输入,如图3所示。

图3 全局池化示意图
本文的图卷积网络结构如图4 所示,其中,V 代表节点矩阵,N为节点数量,F为每个节点的特征维度,A 代表邻接矩阵,FC表示全连接层。
4 实验与讨论
4.1 数据集
Camelyon16 数据集[19]来源乳腺癌患者的前哨淋巴结,是数字化病理全切片图像(WSI)。由Radboud UMC和UMC Utrecht两个机构提供(https://camelyon16.grand-challenge.org/Data/)。它是由400 张完整的淋巴结病理图像组成,分为270 张用于训练和130 张用于测试,包含医生标注的轮廓信息。本文使用170个淋巴结病理图像作为训练集,其中100个正常病理图像和70个癌变病理图像;另外再使用100 个病理图像作为测试集,其中60 个正常病理图像和40 个癌变病理图像。使用CNN 模型进行训练时,将病理图像进行一系列预处理操作后,裁剪为256×256 大小的图像块作为输入。在使用本文提出的C-GCN 模型进行训练时,将病理图像裁剪1 792×1 792 大小的图像块作为输入。病理图像裁剪流程如图5所示,其中病理图像的具体预处理操作可参考文献[20]。表1为在Camelyon16数据集上进行验证实验时,训练集与测试集的样本数量分布情况。

图4 图网络结构图

表1 Camelyon16数据集的图像块数量
结直肠癌(CRC)数据集[16,21]由300 张非重叠图像组成(https://warwick.ac.uk/fac/sci/dcs/research/tia/data/extended_crc_grading/),尺寸大小为4 548×7 548,在20倍率下提取,并根据腺体分化程度,病理学专家将图像分别标记为正常组织、低等级肿瘤和高等级肿瘤。由于数据量比较少,本文通过翻转、旋转、平移等简单的数据增强方法,扩充了280 多张图像。为进行公平的比较,将数据集分为三部分进行交叉验证[21]。由于受内存的限制,CNN 是无法直接对4 548×7 548 大小的高分率图像进行训练,但是在高分辨率图像转换为图结构后,可利用GCN模型对其进行训练,因此,本文将对4 548×7 548大小的图像直接进行训练和三分类预测,以验证本文方法的有效性。其中正常、低等级、高等级三个类别在训练集和测试集的样本数量分布如表2所示。
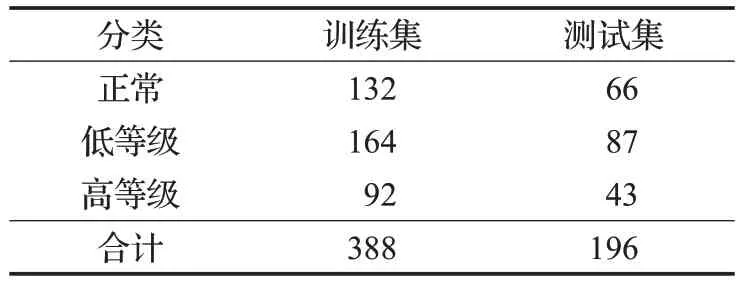
表2 CRC数据集的每个类的数量
4.2 实验
利用PyTorch Germetric(PyG)库[22]实现算法的验证,在训练期间,使用Adam优化函数,学习率为5E-4,权重衰减率为1E-4。之后构建Camelyon16和CRC两个数据集对应的细胞图结构数据集,为C-GCN 提供训练和测试数据。图6 和图7 分别为Camelyon16 数据集和CRC 数据集转换为细胞图结构后,将其可视化的部分细胞掩码和细胞图结构。

图6 Camelyon16的细胞图结构可视化

图5 病理图像处理流程示意图

图7 CRC的细胞图结构可视化
为证明C-GCN 的有效性,本文在Camelyon16 和CRC两个数据集上分别做两组实验。
第一组实验:在Camelyon16上,本文针对正常病理图像和肿瘤病理图像的二分类任务评估六种著名的CNN模型并与C-GCN的分类准确率进行对比。其中六种CNN 分 别 是GoogLeNet[23]、AlexNet[24]、VGG16[25]、ResNet50[26]、MobileNet[27]、Xception[28]。
从表3中可以看出,与CNN模型相比,C-GCN在组织病理图像的癌症分类任务中也能够获得较高的分类准确率。
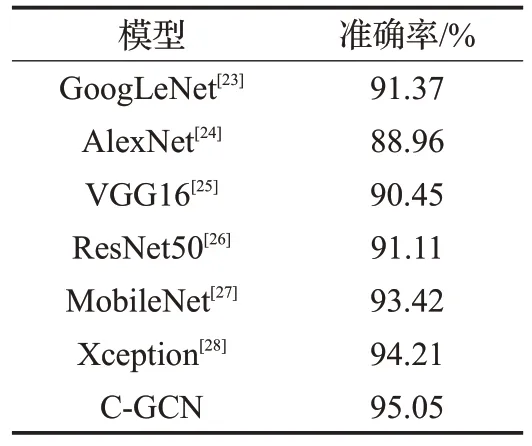
表3 Camelyon16数据上不同模型的评估结果
第二组实验:在CRC 数据集上采用两个分类进行准确率、精确率、召回率和F1 值评估,分别为癌症和高等级,在癌症分类中,将低等级和高等级均视为阳性类别,而在高等级分类中,将正常和低等级均视为阴性类别。准确率指标表示真实的阳性和真实的阴性之和占总数的百分比。表4为癌症分类和高等级分类的混淆矩阵。

表4 预测结果的混淆矩阵(N=196)
表5 中,C-GCN 模型直接对4 548×7 548 的高分辨率病理图像进行三分类训练,从评估指标来看,也获得了比较高的准确率,并且癌症和高等级的召回率很高,说明了本文方法能够准确地将癌变病理图像进行区分,虽然高等级分类的精确率比较低,只有0.70,这是由于在高等级分类中,将低等级分化的癌变也视为了阴性类别,所以在高等级分类中,误将某些倾向于高等级分化的低等级癌变图像也预测为高等级,从而导致了在高等级分类的精确率较低。从图8 的AUC-ROC 曲线来看,癌症与高等级的AUC 值均为0.98,进一步证明了模型在分类预测中具有较高的稳定性和鲁棒性。
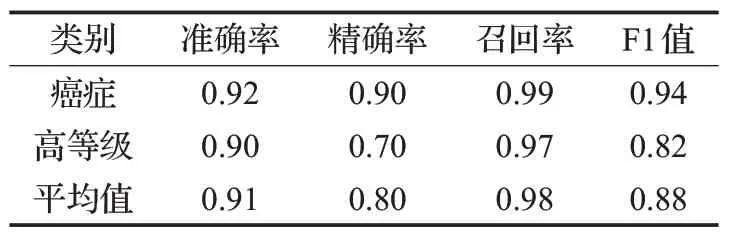
表5 在CRC数据上使用不同的指标评估C-GCN

图8 ROC曲线注意癌症类别包括低等级和高等级
5 结束语
本文提出了基于C-GCN的组织病理图像分类预测的方法。该方法将高分辨率组织病理图像中的细胞作为图结构的节点,并将细胞之间的欧氏距离作为边,以此构建细胞图结构。这种方法在提取高分辨率组织病理图像的微观结构和宏观结构方面表现优异。此外,本文在图卷积中引入了GraphSAGE 模块和图池化模块,不仅有效地聚合了细胞形态和微观结构信息,也减少了计算冗余。并在Camelyon16和CRC两个数据集进行充分验证,证明了本文方法在病理图像分类任务中的优势。但是生成的图结构非常稠密,其中可能会存在大量的噪声节点,这些噪声节点一方面会影响最终的分类准确率,另一方面也增加了网络的计算量,因此需要减少噪声节点对模型的影响,以构建稀疏的图结构,进一步提升图网络的分类效果。
