2株广西猪源H9N2亚型流感病毒遗传进化及生物学特性分析
2020-12-11孔子荣李三木何奇松曾咏芳杨可妍冯淑萍孙翔翔熊毅颜健华
孔子荣 李三木 何奇松 曾咏芳 杨可妍 冯淑萍 孙翔翔 熊毅 颜健华


摘要:【目的】明确广西猪源H9N2亚型流感病毒的遗传特征及分子生物学特性(抗原性、耐药性和致病性),为广西猪源H9N2亚型流感病毒的防控提供科学依据。【方法】以分离自广西百色地区的2株猪源H9N2亚型流感病毒(SW/GX/P2/2011株和SW/GX/P3/2011株)为研究对象,运用RT-PCR扩增其全基因组的8个基因片段(HA、NA、NP、M、NS、PB1、PB2和PA基因),经克隆测序后进行核苷酸序列及氨基酸位点分析。【结果】2株广西猪源H9N2亚型流感病毒的核苷酸序列开放阅读框(ORF)分别是PB2:2280 bp、PB1:2274 bp、PA:2151 bp、HA:1683 bp、NP:1497 bp、NA:1410 bp、M:982 bp和NS:838 bp。2株广西猪源H9N2亚型流感病毒全基因组仅M基因核苷酸序列与猪源H9N2亚型流感病毒的相似性较高,而HA、NA、NP、NS、PA、PB2和PB1基因核苷酸序列均与禽源H9N2亚型流感病毒的相似性较高,在基因型分类上属于G57基因型,为我国广泛流行的H9N2亚型基因型。2株广西猪源H9N2亚型流感病毒的HA蛋白发生R180Q、T213A、D216E、M224L、N285S和V287T突变,NA蛋白抗原决定簇S331V、W403S和Q431K也发生突变;NA蛋白在N2亚型的耐药性关键位点119E、151D、292R、276E和294N位点未发生突变,但在NA蛋白抗原区存在S331V、K367E和Q432K突變,且M2氨基酸位点发生S31N突变。2株广西猪源H9N2亚型流感病毒HA蛋白连接HA1和HA2的氨基酸均为RSSR↓GLF;HA蛋白存在1个因P315S突变而新增的潜在糖基化位点(NCS);NA蛋白未缺失NA潜在糖基化位点,也未出现NA蛋白颈部杆状结构63~65 aa缺失现象,在NA潜在糖基化位点中69、86、146、200和234 aa处非常保守,但发生W402S突变。【结论】从广西百色分离获得的2株猪源H9N2亚型流感病毒(SW/GX/P2/2011株和SW/GX/P3/2011株)虽为低致病力毒株,但在流感病毒基因重组过程中发挥重要作用,且其致病性有增强趋势,已对金刚烷胺类药物产生耐药性。因此,应加强广西地区哺乳动物H9N2亚型流感病毒的监控,并警惕其跨种间传播。
关键词: 猪流感病毒;H9N2亚型;基因重组;抗原性;耐药性;致病性
中图分类号: S852.659.5 文献标志码: A 文章编号:2095-1191(2020)09-2304-07
Genetic evolution and biological characteristics analysis of two strains of H9N2 subtype influenza virus from swine in Guangxi
KONG Zi-rong1, LI San-mu2,HE Qi-song3, ZENG Yong-fang1, YANG Ke-yan1,
FENG Shu-ping3, SUN Xiang-xiang1, XIONG Yi3, YAN Jian-hua2*
(1College of Animal Science and Technology, Guangxi University, Nanning 530004, China; 2Medical College,Guangxi University, Nanning 530004, China; 3Guangxi Center for Animal Disease Prevention and Control, Nanning 530001,China)
Abstract:【Objective】To understand the genetic and molecular biological characteristics(antigenicity, drug resistance and pathogenicity) of pig H9N2 subtype influenza virus from swine in Guangxi and to provide scientific basis for the prevention and control of swine H9N2 influenza virus in Guangxi. 【Method】 Two strains of pig H9N2 subtype influenza virus from swine isolated from Baise in Guangxi(SW/GX/P2/2011 and SW/GX/P3/2011) were used as materials, eight segments(HA, NA, NP, M, NS, PB1, PB2 and PA) of both virus strains whole genome were amplified by RT-PCR. After cloning and sequencing, the nucleotide sequence and amino acid sites were analyzed. 【Result】Nucleotide sequence open reading frame(ORF) of two strains of H9N2 influenza virus isolated from pigs were PB2:2280 bp, PB1:2274 bp, PA:2151 bp, HA:1683 bp, NP:1497 bp, NA:1410 bp, M:982 bp and NS:838 bp, respectively. In genomes of the two isolated strains,only the nucleotide sequence of M gene of the two isolates had high similarity with H9N2 subtype influenza virus of swine origin, while the nucleotide sequences of HA,NA,NP,NS,PA,PB2 and PB1 genes had high similarity with H9N2 subtype influenza virus of avian origin, and belonged to G57 genotype in genotype classification, which was the H9N2 subtype genotype widely prevalent in China. Mutations of R180Q, T213A, D216E, M224I, N285S and V287T occurred in HA protein of the two isolated strains, and the NA protein antigenic determinants S331V, W403S and Q431K were also mutated. In the drug resistance locus analysis, the isolated virus strains did not mutate at the 119E, 151D, 292R, 276E and 294N sites of the NA protein in the N2 subtype resistance,but S331V, K367E and Q432K mutations were found in the antigen region of NA protein,and the M2 amino acid site produced S31N mutations.The amino acids connec-ting HA1 and HA2 of the two isolates were RSSR↓GLF. A new potential glycosylation site(NCS) existed in HA protein due to P315S mutation. There was no deletion of NA potential glycosylation site and 63-65 aa deletion in the neck rod structure of NA protein. Among the potential glycosylation sites of NA,69,86,146,200 and 234 aa were very conservative, but W402S mutation occurred. 【Conclusion】The two H9N2 influenza viruses(SW/GX/P2/2011 and SW/ GX/P3/2011) isolated from Baise, Guangxi are low virulence strains, but they play an important role in the gene recombination of influenza viruses, and their pathogenicity tends to increase. They have developed amantadine resistance. Therefore, it is necessary to strengthen the surveillance of H9N2 subtype influenza virus in mammals in Guangxi, and to guard against its cross species transmission.
Key words: swine influenza virus; H9N2 subtype; genetic recombination; antigenicity; drug resistance; pathogenicity
Foundation item: National Natural Science Foundation of China(31660713); Guangxi Natural Science Foundation(2015GXNSFEA139002)
0 引言
【研究意义】猪流感病毒(Swine influenza virus,SIV)隶属于正黏病毒科(Orthomyxoviridae)A型流感病毒属,主要引发猪群发生呼吸道疾病(汪琪等,2017;李鸽等,2019)。猪是流感病毒进化过程中的混合器,能将禽源、猪源和人源的不同亚型流感病毒基因进行重组(李海燕等,2004;兰德松等,2018;汪琪等,2018)。H9N2亚型是一种极易跨越种间屏障的流感病毒亚型(Butt et al.,2005),若在猪群中与其他亚型流感病毒不断重组或重配,极有可能产生感染人类的新型流感病毒。因此,加强猪源H9N2亚型流感病毒流行监测对整个流感病毒防控体系的建设和完善具有重要意义。【前人研究进展】李海燕等(2004)研究发现,我国猪群中存在H9N2亚型SIV,且证实猪只充当禽源、猪源和人源流感病毒重组的混合器。H9N2亚型流感病毒能为H5N1亚型、H7N9亚型和H5N6亚型等感染人类的流感病毒提供内部基因,是一个极其危险的信号(Gu et al.,2014)。陈建新等(2011)研究表明,H9N2亚型SIV不仅能感染小鼠,还在小鼠体内表现出较强的致病性,与HA蛋白裂解位点显示的低致病性不一致,其原因可能是流感病毒在哺乳动物体内与在家禽体内的致病机理不一致。伍和明等(2012)对广西猪源H9N2亚型流感病毒全基因组序列进行分析,结果发现分离病毒株具备较强的跨种属传播能力。殷斌等(2015)对1株山东H9N2亚型SIV进行遗传进化分析,发现该毒株是禽流感病毒(Avian influenza virus,AIV)向猪体适应的毒株。刘晓敏等(2017)成功建立了禽源H9N2亚型SIV反向遗传操作技术平台,为后续开展SIV的基因功能研究打下基础。孙王杨吉等(2019)对1株浙江H9N2亚型SIV进行遗传进化和致病性分析,结果表明猪可能是AIV获得感染哺乳动物能力的过渡宿主。Sun等(2019)于2017—2018年从华南地区的家禽中分离获得8株H9N2亚型流感病毒,通过系统发育进化树及生物学特性分析,发现H9N2亚型流感病毒向哺乳动物传播的能力不断增强。此外,Samir等(2019)通过对埃及的家养鸡群进行样品采集,并选取5种亚型毒株进行测序及HA基因进化分析,结果发现HA基因与亚洲G1系的亲缘关系较近,且各基因片段在不断进化或重组。【本研究切入点】至今,有关广西地区猪源H9N2亚型流感病毒的研究报道较少,尤其在遗传特征研究及生物学特性分析方面更少。【拟解决的关键问题】对分离自广西百色地区的2株猪源H9N2亚型流感病毒进行遗传进化分析,并通过关键位点分析了解其抗原性、耐药性及致病性,为广西猪源H9N2亚型流感病毒的防控提供科学依据。
1 材料与方法
1. 1 试验材料
猪源H9N2亚型流感病毒SW/GX/P2/2011株和SW/GX/P3/2011株均分离自广西百色市乐业县送检的病猪组织样品,由广西动物疫病预防控制中心分离鉴定并保存。SPF鸡胚购自北京梅里亚维通实验动物技术有限公司;病毒核酸抽提、胶回收和质粒提取试剂盒及DL2000 DNA Marker等购自天根生化科技(北京)有限公司;M-MLV反转录酶、抑制降解酶(RRI)、Taq DNA聚合酶、pMD18-T载体及大肠杆菌DH5α感受态细胞等购自宝生物工程(大连)有限公司。
1. 2 引物设计与合成
参考Hoffmann等(2001)扩增A型流感病毒基因组的引物设计原则,利用Primer 5.0设计8对引物用于扩增广西猪源H9N2亚型流感病毒基因组各片段(表1)。所有引物均委托宝生物工程(大连)有限公司合成。
1. 3 RT-PCR扩增
以接种SPF鸡胚增殖获得的病毒尿囊液抽提病毒总RNA,并以此为模板进行RT-PCR扩增。RT-PCR反应体系25.0 μL:2×Step Bufffer 10.0 μL,PrimeScript 1 Step Enzyme Mix 1.0 μL,上、下游引物各0.5 μL,RNA模板5.0 μL, RNase Free dH2O補足至25.0 μL。扩增程序:42 ℃反转录1 h;94 ℃预变性5 min;94 ℃ 40 s,56 ℃ 40 s,72 ℃ 60 s,进行30个循环;72 ℃延伸10 min。扩增产物采用1.5%琼脂糖凝胶电泳进行检测。
1. 4 目的条带回收与克隆
按琼脂糖凝胶DNA回收试剂盒说明进行DNA目的片段回收纯化,将回收纯化获得的DNA目的片段与pMD18-T载体连接,经转化和涂板后挑斑进行重组质粒鉴定,阳性重组质粒送至宝生物工程(大连)有限公司测序。
1. 5 病毒基因组测序分析
利用SeqMan进行序列拼接以获得完整的病毒基因组序列,再使用MEGA 5.0绘制毒株遗传进化树并分析相关功能位点。
2 结果与分析
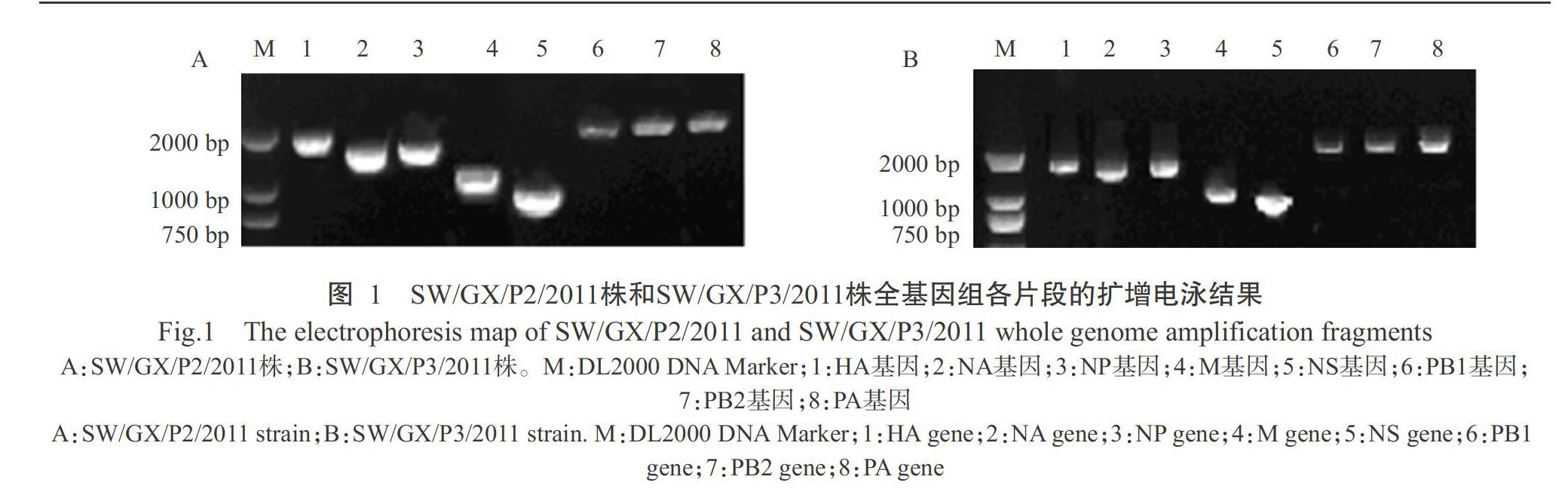
2. 1 猪源H9N2亚型流感病毒各基因片段扩增结果
利用设计的特异性引物进行RT-PCR扩增,电泳检测获得的目的条带分别为HA、NA、NP、M、NS、PB1、PB2和PA基因(图1),与预期结果一致,说明广西猪源H9N2亚型流感病毒SW/GX/P2/2011株和SW/GX/P3/2011株全基因组各片段均扩增成功。
2. 2 猪源H9N2亚型流感病毒基因核苷酸序列比对及遗传进化分析结果
广西猪源H9N2亚型流感病毒SW/GX/P2/2011株和SW/GX/P3/2011株经基因克隆测序得到的核苷酸序列开放阅读框(ORF)分别是PB2:2280 bp、PB1:2274 bp、PA:2151 bp、HA:1683 bp、NP:1497 bp、NA:1410 bp、M:982 bp和NS:838 bp。使用NCBI中的BLAST进行比对分析,获得与2株广西猪源H9N2亚型流感病毒各基因片段核苷酸序列相似性最高的参考毒株(表2)。利用MEGA 5.0对SW/GX/P2/2011株和SW/GX/P3/2011株的各基因片段进行遗传进化分析,其中,基于HA基因核苷酸序列的遗传进化分析显示这2株猪源H9N2亚型流感病毒与A/duck/Hong Kong/Y280/97株属于同一分支(图2),为BJ/94系中的Y280亚系;基于NA基因核苷酸序列的遗传进化分析显示这2株猪源H9N2亚型流感病毒与A/chicken/Hong Kong/G9/97株属于同一分支,为BJ/94系中的G9/97亚系;基于NS基因核苷酸序列的遗传进化分析显示这2株猪源H9N2亚型流感病毒与A/chicken/Shanghai/F/98株属于同一分支,为BJ/94系中的F/98亚系;基于PB1、PA和NP基因核苷酸序列的遗传进化分析均显示这2株猪源H9N2亚型流感病毒属于F/98亚系;基于PB2基因核苷酸序列的遗传进化分析显示这2株猪源H9N2亚型流感病毒与A/duck/Shantou/2102/2000株属于同一分支,属于DK1系;基于M基因核苷酸序列的遗传进化分析显示这2株猪源H9N2亚型流感病毒与A/quail/Hong Kong/G1/97株属同一分支,属G1/97系。由于SW/GX/P2/2011株和SW/GX/P3/2011株的基因组成与目前广泛流行的G57基因型(图3)相一致,故确定广西猪源H9N2亚型流感病毒为G57基因型。
2. 3 猪源H9N2亚型流感病毒表面蛋白抗原性分析结果
SW/GX/P2/2011株和SW/GX/P3/2011株与参考株DK/HK/Y280/97、DK/HK/Y439/97和QA/HK/G1/97的HA抗原表位比对分析结果显示,7个抗原区在147~153 aa、162~167 aa和267~273 aa等3个抗原位区上保守,除此之外有7个抗原位点发生变异,分别是117~181 aa区的R180Q、195~205 aa区的T/E198A、211~227 aa区的T213A、D216E和M/V224L及284~289 aa区的N285S和V287T。以疫苗株SH/F/98、CK/HK/G9/97和QA/HK/G1/97为参考,进行猪源H9N2亚型流感病毒NA抗原性位点比对分析,结果发现在抗原决定簇发生S331V、W403S和Q431K突变,且在红细胞结合位点发现K367E、D399G、W403S和Q432K存在明显变异。NA酶活性位点分析结果表明,2株猪源H9N2亚型流感病毒在NA蛋白基质结合位点(118R、224R、243D、277E、350K和425E)上与参考毒株无差异。由此推测,SW/GX/P2/2011株和SW/GX/P3/2011株的表面蛋白抗原性已发生改变。
2. 4 猪源H9N2亚型流感病毒耐药性分析结果
通过对比N2亚型抗神经氨酸酶抑制剂NA蛋白酶活性位点氨基酸序列,结果显示SW/GX/P2/2011株和SW/GX/P3/2011株在119E、151D、292R、276E和294N位点均未发生突变,说明广西猪源H9N2亚型流感病毒对神经氨酸酶抑制剂不具耐药性;但在NA蛋白抗原区发生S331V、K367E和Q432K突变,而这些突变可能会影响病毒的耐药性;M2氨基酸位点分析发现其氨基酸序列发生S31N突变,说明广西猪源H9N2亚型流感病毒对金刚烷胺类药物耐药。综上所述,SW/GX/P2/2011株和SW/GX/P3/2011株已对金刚烷胺类药物产生耐药性,但对神经氨酸酶抑制剂依然敏感。
2. 5 猪源H9N2亚型流感病毒致病性关键位点分析结果
SW/GX/P2/2011株和SW/GX/P3/2011株HA蛋白连接HA1和HA2的氨基酸序列均为RSSR↓GLF,仅存在2个碱性氨基酸残基,说明这2株猪源H9N2亚型流感病毒为低致病性毒株;HA蛋白有8个保守的潜在糖基化位点,但发生P315S突变导致在313 aa处新增1个糖基化位点(NCS);NA蛋白共有8个保守的潜在糖基化位点,未发生因NA潜在糖基化位点缺失而增强病毒致病性的情况;与参考毒株CK/SH/F/98相比,发现这2株猪源H9N2亚型流感病毒NA蛋白颈部杆状结构在63~65 aa处并未发生缺失(表3)。在对病毒致病性产生影响的NA潜在糖基化位点中,69、86、146、200和234 aa处非常保守,但发生W402S突变,可能对神经氨酸酶活性产生影响;在内部基因中,与致病力有关的氨基酸位点与参考毒株相比均较保守,如PB2蛋白的627E和701D(陆家海等,2007),PB1蛋白的436Y、622G和709V,PA蛋白的315F和515T,NP蛋白的184K和319N。可見,SW/GX/P2/2011株和SW/GX/P3/2011株为低致病性H9N2亚型流感病毒,但存在毒力变强的趋势。
3 讨论
H9N2亚型SIV致死率较低因而在实际生产中常被忽视,但从整个养殖业的发展及公共卫生意义角度出发,SIV尤其是H9N2亚型SIV的监测和防控意义重大。本研究结果表明,SW/GX/P2/2011株和SW/GX/P3/2011株全基因组仅M基因核苷酸序列与猪源H9N2亚型流感病毒的相似性较高,而HA、NA、NP、NS、PA、PB2和PB1基因核苷酸序列均与禽源H9N2亚型流感病毒的相似性较高,故推测这2株猪源H9N2亚流感病毒应是由禽源H9N2亚型流感病毒感染猪群进化产生的宿主适应性变异毒株,但以现有数据不能推断其产生来源。这2株猪源H9N2亚型流感病毒在基因型分类上属于G57基因型,为我国广泛流行的H9N2亚型基因型,且与我国已报道的H7N9、H5N2和H5N1亚型流感病毒具有相似内部基因组成,说明SW/GX/P2/2011株和SW/GX/P3/2011株具备为多种亚型流感病毒提供内部基因的可能,与孙王杨吉等(2019)认为禽源H9N2亚型流感病毒未发生突变即能感染猪群的观点一致,进一步佐证猪作为流感病毒进化混合器的观点。
劉红旗,程坚,彭大新,贾立军,张如宽,刘秀梵. 2002. 我国部分地区H9亚型禽流感病毒血凝素基因序列比较与遗传发生关系分析[J]. 微生物学报,42(3):288-297. [Liu H Q,Cheng J,Peng D X,Jia L J,Zhang R K,Liu X F. 2002. Comparison of sequences of the hemagglutinin gene and phylogenetical analysis of H9 subtype avian influenza virus isolated from some regions in China[J]. Acta Microbiologica Sinica,42(3):288-297.]
刘金华,史为民,吴清民,郭玉璞. 2004. 鸡源H9N2亚型流行性感冒病毒神经氨酸酶基因序列分析[J]. 病毒学报,20(3):237-241. [Liu J H,Shi W M,Wu Q M,Guo Y P. 2004. Sequence analyses of neuraminidase genes of H9N2
influenza viruses isolated from chickens[J]. Chinese Jour-
nal of Virology,20(3):237-241.]
刘晓敏,汪琪,杨海明,宫晓倩,阮宝阳,张鹏,单同领,童武,童光志,于海. 2017. 禽源H9N2亚型猪流感病毒反向遗传操作技术平台的建立[J]. 中国动物传染病学报,25(2):29-34. [Liu X M,Wang Q,Yang H M,Gong X Q,Ruan B Y,Zhang P,Shan T L,Tong W,Tong G Z,Yu H. 2017. Establishment of reverse genetics system of avian-like H9N2 subtype swine influenza virus[J]. Chinese Journal of Animal Infectious Diseases,25(2):29-34.]
陆家海,张定梅,王国玲. 2007. 高致病性禽流感H5N1病毒研究进展[J]. 国际病毒学杂志,14(3):65-68. [Lu J H,Zhang D M,Wang G L. 2007. Advances in the study of highly pathogenic avian influenza H5N1 virus[J]. International Journal of Virology,14(3):65-68.]
孙王杨吉,刘源,刘子拓,范佳文,石火英. 2019. 一株H9N2亚型猪流感病毒的遗传进化和致病性分析[J]. 中国预防兽医学报,41(5):449-454. [Sun W Y J,Liu Y,Liu Z T,Fan J W,Shi H Y. 2019. Genetic characterization and pathogenicity analysis of swine influenza H9N2 subtype virus[J]. Chinese Journal of Preventive Veterinary Medicine,41(5):449-454.]
汪琪,刘晓敏,杨海明,王帅勇,单同领,童武,李国新,童光志,于海. 2017. 三源重组H1N1亚型猪流感病毒反向遗传操作平台的建立[J]. 中国动物传染病学报,25(3):28-33. [Wang Q,Liu X M,Yang H M,Wang S Y,Shan T L,Tong W,Li G X,Tong G Z,Yu H. 2017. Establishment of reverse genetics system of triple-reassortant H1N1 subtype swine influenza virus[J]. Chinese Journal of Animal Infectious Diseases,25(3):28-33.]
汪琪,杨海明,汪秀会,刘晓敏,王帅勇,姚云,朱世强,单同领,童武,周艳君,李国新,郑浩,高飞,姜一峰,童光志,于海. 2018. 欧洲类禽H1N1亚型猪流感病毒反向遗传操作平台的建立[J]. 中国动物传染病学报,26(3):23-27. [Wang Q,Yang H M,Wang X H,Liu X M,Wang S Y,Yao Y,Zhu S Q,Shan T L,Tong W,Zhou Y J,Li G X,Zheng H,Gao F,Jiang Y F,Tong G Z,Yu H. 2018. Establishment of reverse genetics system of european avian-like H1N1 subtype swine influenza virus[J]. Chinese Journal of Animal Infectious Diseases,26(3):23-27.]
伍和明,韦达有,易春华,徐贤坤,何奇松,孙翔翔,蒋家霞,付薇,熊毅. 2012. 广西猪源H9N2亚型流感病毒全基因组序列分析[J]. 南方农业学报,43(3):389-392. [Wu H M,Wei D Y,Yi C H,Xu X K,He Q S,Sun X X,Jiang J X,Fu W,Xiong Y. 2012. Complete genome sequence analysis of H9N2 subtype influenza virus from swine in Guangxi[J]. Journal of Southern Agriculture,43(3):389-392.]
殷斌,孙文博,杜以军,郭立辉,丛晓燕,李俊,时建立,隋超,陈蕾,陈智,万仁忠,吴家强. 2015. 山东H9N2亚型猪流感病毒的分离鉴定及遗传进化分析[J]. 南方农业学报,46(9):1691-1697. [Yin B,Sun W B,Du Y J,Guo L H,Cong X Y,Li J,Shi J L,Sui C,Chen L,Chen Z,Wan R Z,Wu J Q. 2015. Isolation,identification and genetic evolution analysis of H9N2 subtype swine influenza virus in Shangdong Province[J]. Journal of Southern Agriculture,46(9):1691-1697.]
Baigent S J,McCauley J W. 2001. Glycosylation of haemagglutinin and stalk-length of neuraminidase combine to regulate the growth of avian influenza viruses in tissue culture[J]. Virus Research,79(1-2):177-185.
Butt K M,Smith G J D,Chen H L,Zhang L J,Leung Y H C,Xu K M,Lim W,Webster R G,Yuen K Y,Peiris J S M,Guan Y. 2005. Human infection with an avian H9N2 influenza avirus in Hong Kong in 2003[J]. Journal of Clini-cal Microbiology,43(11):5760-5767.
Gu M,Chen H Z,Li Q H,Huang J Q,Zhao M J,Gu X B,Jiang K J,Wang X Q,Peng D X,Liu X F. 2014. Enzoo-tic genotype S of H9N2 avian influenza viruses donates internal genes to emerging zoonotic influenza viruses in China[J]. Veterinary Microbiology,174(3-4):309-315.
Hoffmann E,Stech J,Guan Y,Webster R G,Perez D R. 2001. Universal primer set for the full-length amplification of all influenza A viruses[J]. Archives of Virology,146(12):2275-2289.
Horimoto T,Kawaoka Y. 2001. Pandemic threat posed by avian influenza A viruses[J]. Clinical Microbiology Reviews,14(1):129-149.
Pinto L H,Holsinger L J,Lamb R A. 1992. Influenza virus M2 protein has ion channel activity[J]. Cell,69(3):517-528.
Samir A,Adel A,Arafa A,Sultan H,Ahmed H A H. 2019. Molecular pathogenic and host range determinants of rea-ssortant Egyptian low pathogenic avian influenza H9N2 viruses from backyard chicken[J]. International Journal of Veterinary Science and Medicine,7(1):10-19.
Sun H L,Lin J T,Liu Z T,Yu Y N,Wu M H,Li S,Liu Y,Feng Y L,Wu Y Q,Li M L,Jiao P R,Luo K J,Liao M. 2019. Genetic,molecular,and pathogenic characterization of the H9N2 avian influenza viruses currently circulating in South China[J]. Viruses,11(11):1040.
Sun X X,Xu X K,Liu Q,Liang D J,Li C Y,He Q S,Jiang J X,Cui Y M,Li J,Zheng L F,Guo J G,Xiong Y,Yan J H. 2013. Evidence of avian-like H9N2 influenza A virus among dogs in Guangxi,China[J]. Infection,Genetics and Evolution,20:471-475.
(責任编辑 兰宗宝)
