lncRNA HOTAIR和H19 SNPs与胃癌及EB病毒相关胃癌遗传易感性的关系*
2020-12-03卫美蓉王笑峰罗兵
卫美蓉, 王笑峰, 罗兵△
lncRNA和SNPs与胃癌及EB病毒相关胃癌遗传易感性的关系*
卫美蓉1, 王笑峰2, 罗兵2△
(1西京学院医学院,陕西 西安 710123;2青岛大学医学院基础医学院微生物学教研室,山东 青岛 266021)
探讨长链非编码RNA (lncRNA)及基因多态性与胃癌和EB病毒相关胃癌(EBVaGC)易感性的关系。研究对象选择EB病毒阴性胃癌(EBVnGC)组织65例、EBVaGC组织50例及115例健康人外周血标本,并从中提取其DNA备用。选择rs3024270和rs3741219,以及rs4759314和rs874945共4个基因多态性位点,利用Taq-Man MGB等位基因分型试剂盒对各单核苷酸多态性(SNP)位点基因进行分型,统计分析实验结果。及的SNPs在全部标本中均符合Hardy-Weinberg平衡。(1)rs3024270位点的SNP在胃癌组与对照组中的差异有统计学意义(<0.05);其中G等位基因为保护基因,个体携带G等位基因可显著降低罹患胃癌的风险(<0.01);(2)rs4759314位点在胃癌组与对照组中基因型和等位基因分布频率均有显著差异(<0.05);基因型为GG的人群,胃癌的发病风险显著增加(<0.05);且胃癌的风险基因可能是G等位基因,携带G等位基因人群罹患胃癌的风险显著增加(<0.05);(3)rs3741219及rs874945位点的各基因型及等位基因频率在两组间无显著差异(>0.05);(4)rs4759314位点G等位基因在EBVaGC组的分布高于EBVnGC组,而EBVaGC和EBVnGC组间其他SNPs的分布差异均无统计学意义(>0.05)。rs3024270和rs4759314位点的SNPs与胃癌的发病风险有关,但与EBVaGC的发病风险之间未观察到明确的关联性。
胃癌;长链非编码RNA;基因;基因;EB病毒;单核苷酸多态性;易感性
据2018年WHO统计显示,全球范围内胃癌的发病率及死亡率分别居于第5位及第3位,也位居我国消化道恶性肿瘤之首,因此对人类的健康产生了极其严重的影响[1-2]。在胃癌中, EB病毒(Epstein-Barr virus, EBV)相关胃癌(EBV-associated gastric carcinoma, EBVaGC)仅占10%,然而其绝对数却是较高的[3]。胃癌的形成过程是复杂多变的,其中主要受到环境和遗传两方面因素的共同作用,同时也存在高发病率及高死亡率的情况,因此加强对胃癌病因及易感基因的研究,对今后胃癌高危人群的预防及胃癌患者的早期诊断及治疗具有重要的意义[4]。生物功能众多的长链非编码RNA(long noncoding RNA, lncRNA)通过调控不同层面基因表达,参与众多肿瘤的形成[5-10]。lncRNA H19和HOTAIR的差异性表达均参与了胃癌的发生发展[11-12]。单核苷酸多态性(single nucleotide polymorphism, SNP)是常见的遗传变异[13]。近年来,研究发现lncRNA的SNPs也可导致lncRNA的表达和功能异常,继而在肿瘤的易感性和干预肿瘤发展及预后机制中发挥重要作用[14]。然而其与胃癌及EBVaGC易感性的相关研究,目前却少见报道。本研究检测EBVaGC和EBV阴性胃癌(EBV-negative gastric carcinoma, EBVnGC)患者中和的4个SNP位点,以探讨这些基因多态性与胃癌及EBVaGC易感性的关系。
材料和方法
1 研究对象
胃癌组:共有115例胃癌组织(新鲜组织和石蜡包埋组织)病理标本,均来自青岛大学医学院附属医院。纳入病例均属于山东地区汉族人群,为2017年1月~2019年10月由病理科确诊的胃癌患者。利用原位杂交技术将其分为EBVaGC 50例和EBVnGC 65例,具体方法为检测胃癌组织中的EBER1 (EBV编码的小分子非多聚腺苷酸)的转录情况。提取所有胃癌组织的DNA。所有胃癌病例均在一般情况及病理学特征等方面无显著差异。
对照组:于同所医院门诊召集115例健康体检者。所有入选人群均签署知情同意书,且由研究者收集其各项病例资料及一般资料,并进行差异分析,结果为>0.05。纳入标准:排除肿瘤与自身免疫性疾病及各种其他感染性疾病的健康山东汉族人群。同时采集每名体检者静脉血5mL,保存于抗凝试管中,以备随后提取DNA之用。
2 方法
2.1SNP的筛选根据HapMap数据库所提供的数据,对基因多态性进行筛选,并利用Haploview 4.2软件分析数据。设置连锁不平衡值2>0.8,最小等位基因频率>5%,同时采用F-SNP软件对入选SNP位点的预测功能进行分析,最终选取了4个SNPs,分别为rs3024270和rs3741219,以及rs4759314和rs874945。
2.2组织细胞DNA提取及基因多态性检测利用QIAamp DNA FFPE试剂盒(Qiagen),且根据操作说明书对完整性及纯度较高的标本进行DNA提取。随后在利用TaqMan MGB试剂盒及实时荧光定量PCR仪(Applied Biosystems)对所选择标本进行分型。分型结果通过SDS 2.4软件分析。整个实验操作在独立双盲的原则下由2位研究人员完成,且每次实验均设立阴性对照。实验后期根据随机方式选择已测样本的10%进行重复检测验证,以保证结果的真实性。所用的引物及探针序列见表1。

表1 H19及HOTAIR SNPs编码基因的引物序列及探针序列
F: forward; R: reverse.
3 统计学处理
利用SPSS 18.0进行统计分析。采用2检验分析各SNPs在各组的基因型和等位基因频率分布差异,通过值、OR值及95% CI对结果进行描述。
结果
1 Hardy-Weinberg平衡检验
对H19及HOTAIR SNPs位点的分布情况进行Hardy-Weinberg平衡检验,2检验结果为>0.05,即表明本实验所选胃癌病例及健康对照标本均具有群体代表性。
2 H19 rs3024270和rs3741219基因多态性检测分析结果
rs3024270和rs3741219在胃癌组及对照组中的基因多态性检测分析结果详见表2。其中胃癌组与对照组中rs3024270位点的基因型和等位基因分布频率间差异有统计学意义(<0.05)。与胃癌组相比,对照组携带GC和GG基因型的个体显著增多(2=9.787,=0.002, OR=0.405, 95% CI: 0.228~0.717;2=4.783,=0.029, OR=0.408, 95% CI: 0.181~0.922); G等位基因在对照组的分布高于胃癌组(2=9.131,0.003, OR=0.552, 95% CI: 0.375~0.813); G等位基因为保护基因,携带G等位基因可显著降低罹患胃癌的风险(2=11.084,=0.001, OR=0.406, 95% CI: 0.237~0.693)。而rs3741219位点基因多态性在胃癌组与对照组之间分布差异无统计学意义(>0.05)。
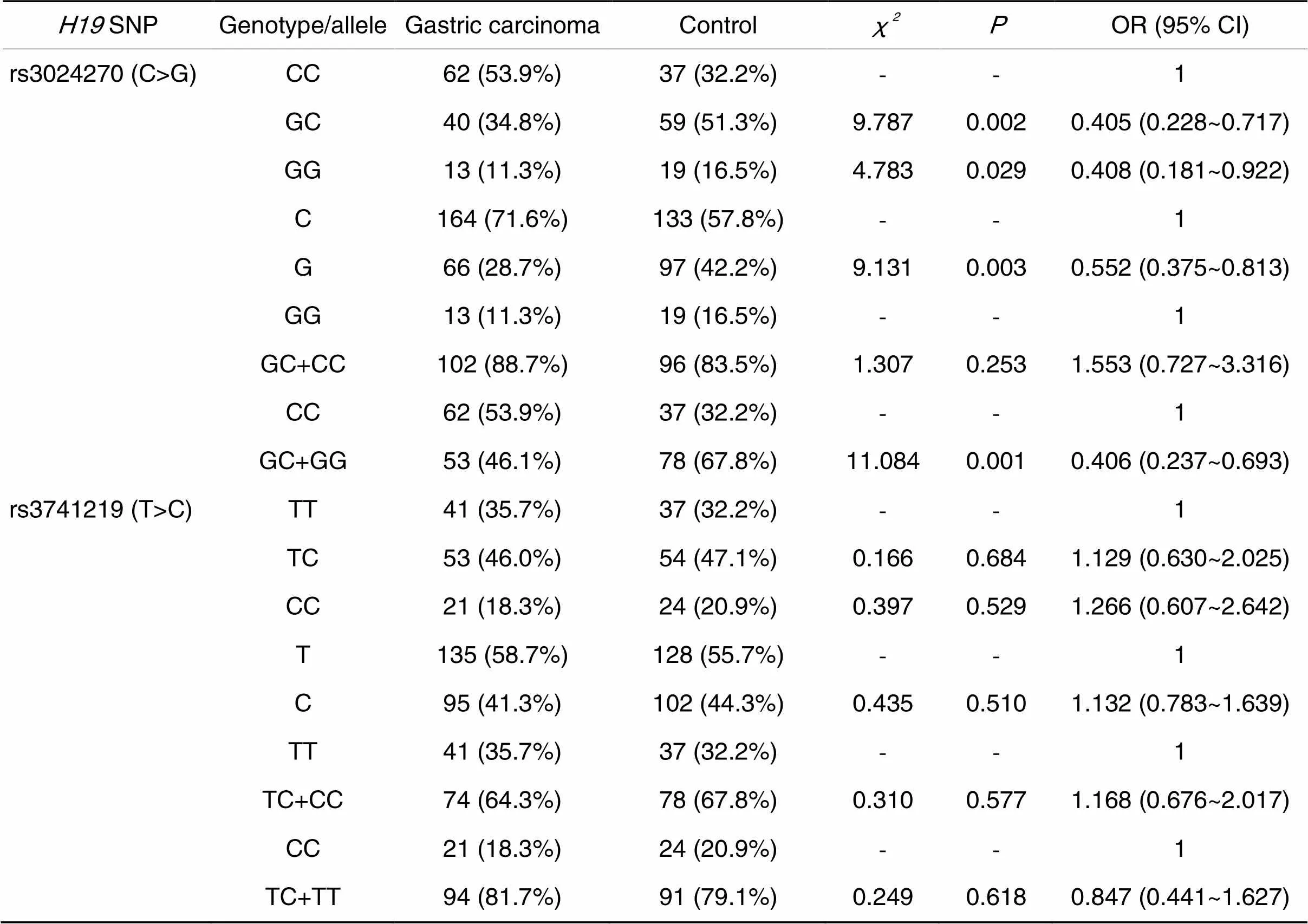
表2 胃癌组与对照组H19 rs3024270和rs3741219基因多态性比较
rs3024270和rs3741219位点基因多态性在EBVaGC和EBVnGC两组中的分布结果详见表3。对各基因型、等位基因频率及携带等位基因频率在两组中的差异进行统计,均显示差异无统计学意义(>0.05)。
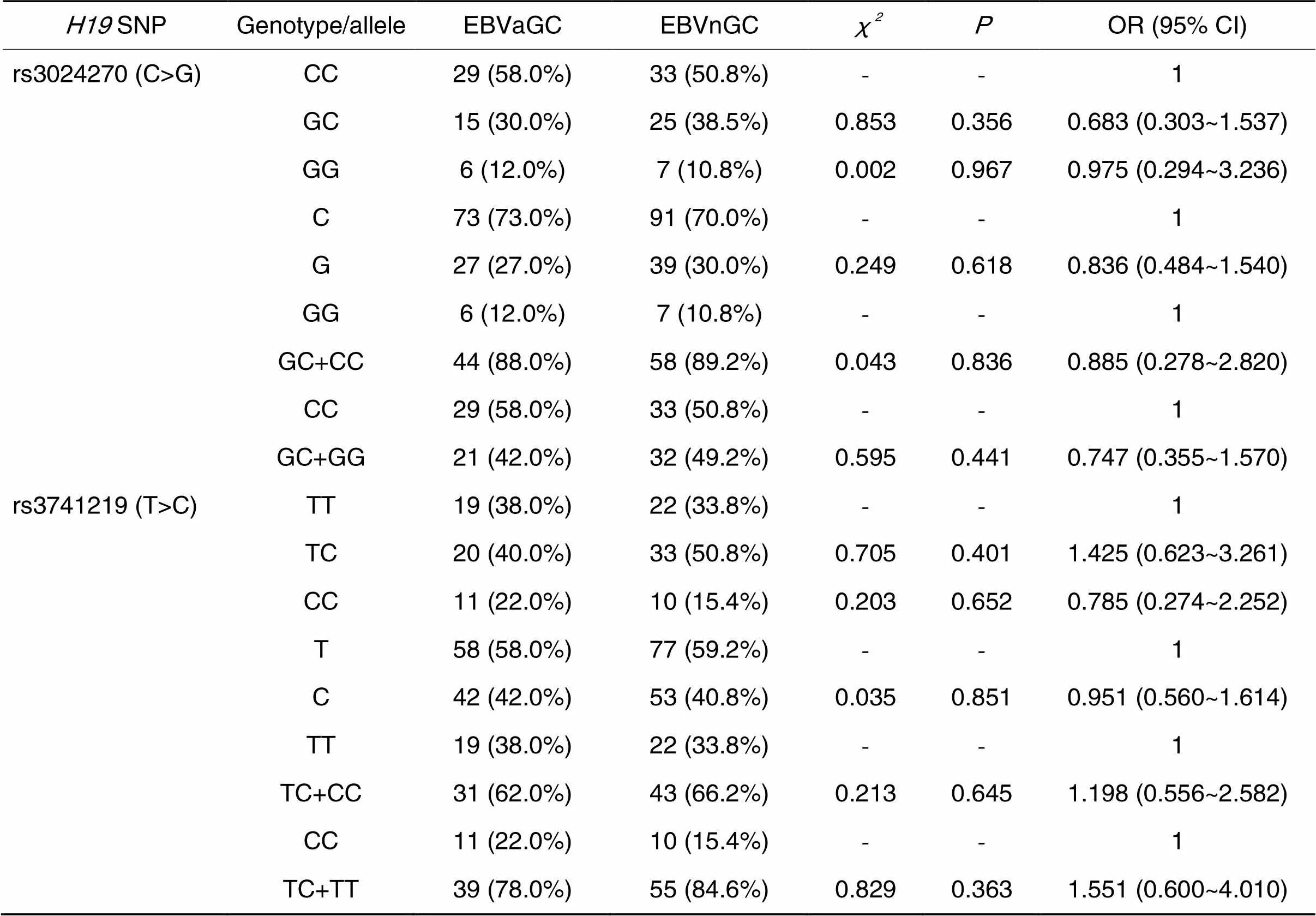
表3 EBVaGC组与EBVnGC组H19 rs3024270和rs3741219基因多态性比较
3 HOTAIR rs4759314和rs874945基因多态性检测分析结果
rs4759314和rs874945基因多态性在胃癌组及对照组中的检测分析结果见表4。其中rs4759314位点在胃癌组与对照组中基因型和等位基因分布频率均有显著差异(<0.05)。携带GG基因型可显著增加胃癌的发病风险(2=5.085,=0.024);胃癌组G等位基因的频率明显高于对照组(2=9.813,0.002, OR=1.858, 95% CI: 1.259~2.744),表明胃癌的风险基因可能是G等位基因,携带G等位基因人群罹患胃癌的风险增加(2=5.343,=0.021, OR=1.887, 95% CI: 1.098~3.241)。而rs874945位点的基因多态性在两组间分布的差异则无统计学意义(>0.05)。
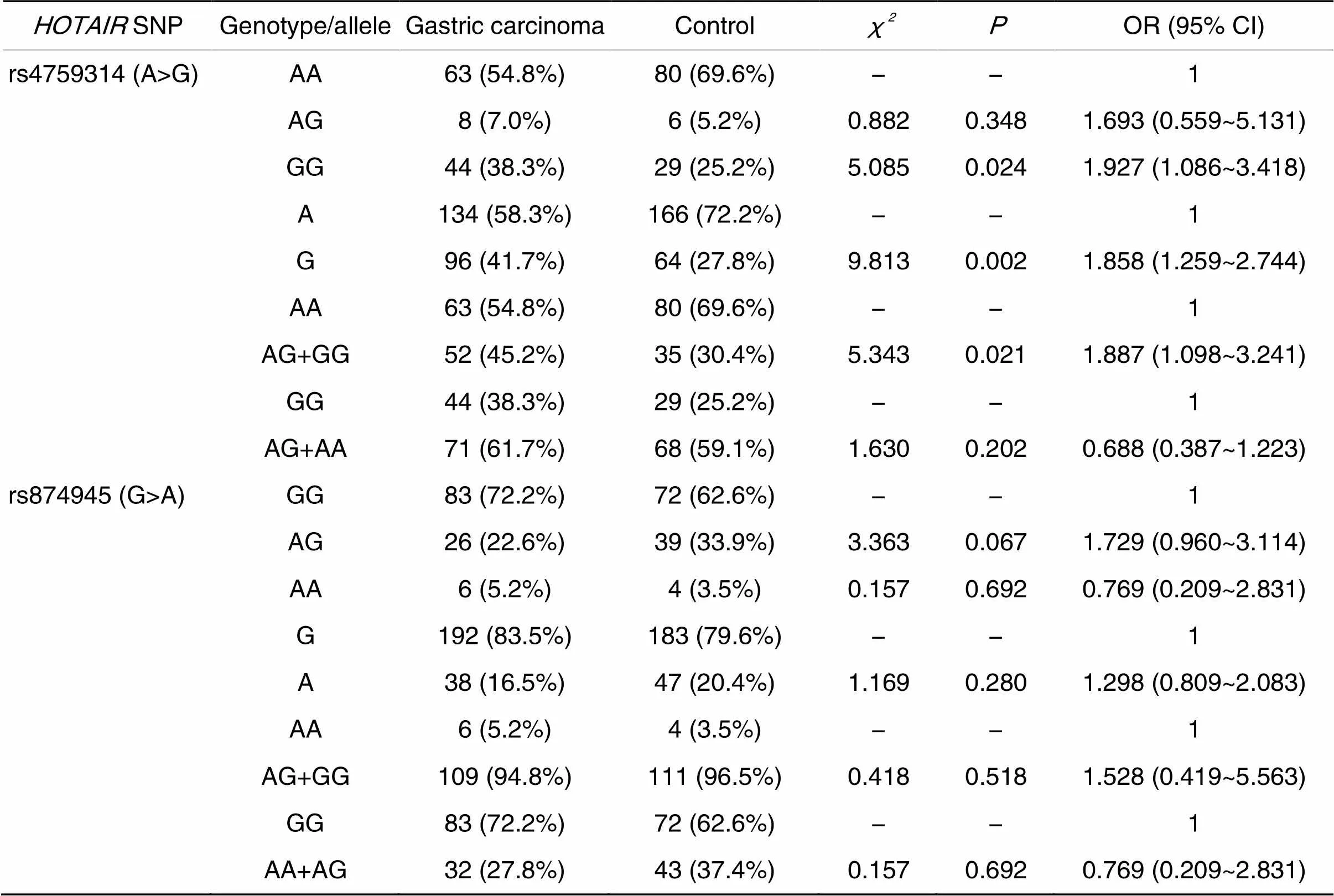
表4 胃癌组与对照组HOTAIR rs4759314和rs874945基因多态性比较
rs4759314和rs874945基因多态性在EBVaGC和EBVnGC组中的检测分析结果见表5。其中rs4759314位点等位基因频率在两组中的分布情况有显著差异(2=6.240,=0.012, OR=1.966, 95% CI: 1.154~3.351)。而其余各基因型及等位基因频率在两组中分布的差异均无统计学意义(>0.05)。
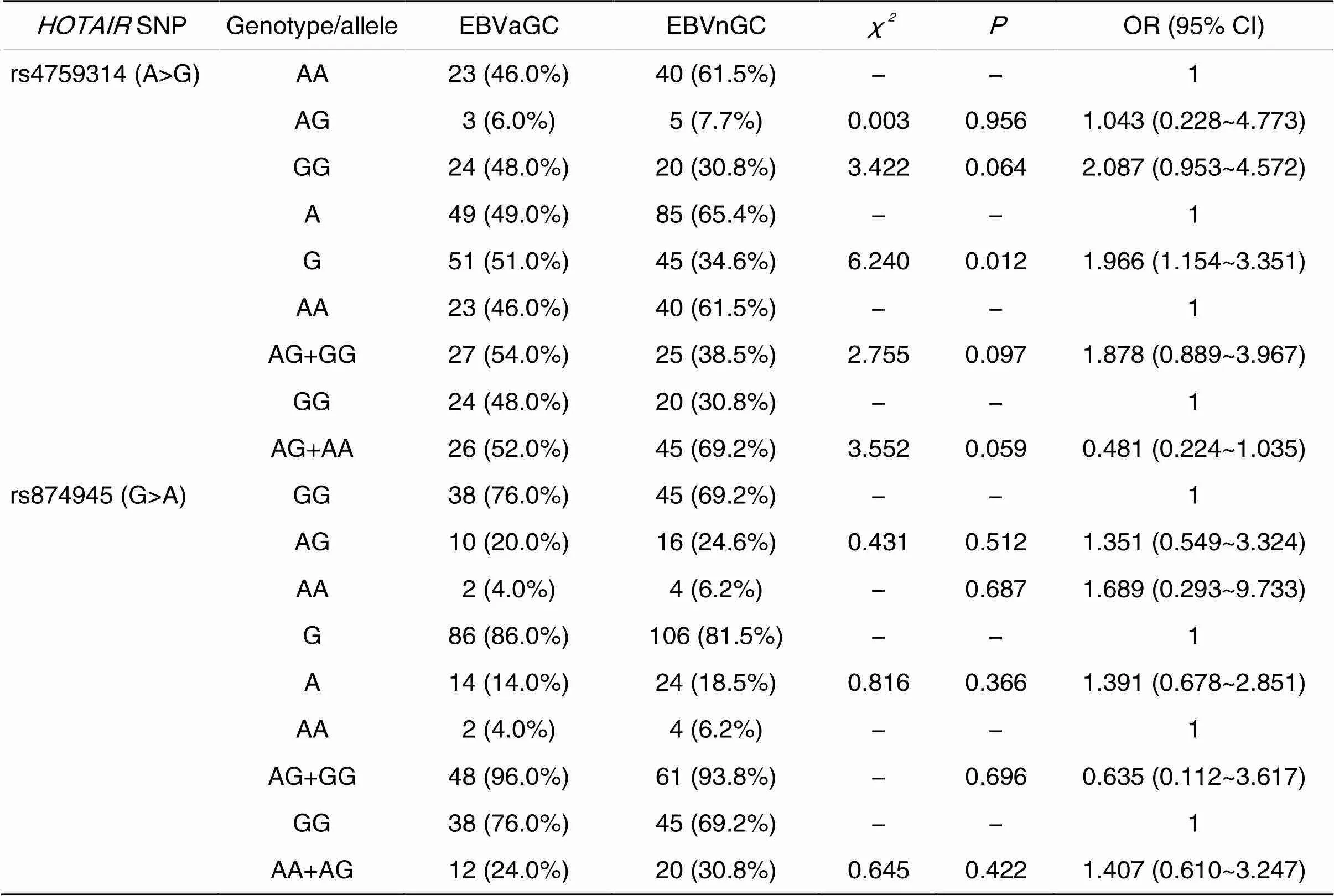
表5 EBVaGC组与EBVnGC组HOTAIR rs4759314和rs874945基因多态性比较
讨论
大量研究结果显示,环境危险因素与遗传因素是诱发胃癌的主要因素,且过程是极其复杂的,如基因暴露于恶性环境中,长期受到不良刺激会引起DNA损伤而突变,癌基因的激活,抑癌基因失活。如果个体携带某肿瘤包括胃癌的易感基因,则其患某肿瘤的风险显著提高。lncRNA在细胞正常的发育、增殖和凋亡等生理过程中,通过不同的调控方式,发挥着抑癌或促癌因子的作用,已成为近年研究的热点。在人类基因组的遗传变异中, SNP的变异率超过了1%,是常见的遗传变异,且lncRNA的SNPs也具有与蛋白编码基因SNPs相似的功能,在参与调节基因表达及疾病的易感性中发挥重要作用。目前全基因组关联性研究(genome-wide association study, GWAS)已经发现了一些与胃癌易感性基因密切相关的SNP[15-20]。然而由于GWAS分析的统计标准过分严格,造成一定程度遗传度缺失情况,因此探索更多胃癌相关SNPs,并深入研究其功能及致癌机制,仍然是当前胃癌研究的主要任务之一。
lncRNA H19是目前研究最早且与肿瘤的关系被人们密切关注的lncRNA。研究显示, H19可通过作用于miR-675而间接抑制CALN1的表达或者通过直接上调ISM1的表达,最终在胃癌的发生、发展、侵袭、迁移等过程中发挥重要的影响[21-22]。近期研究也显示SNPs与肿瘤的发病风险同样密切。Xia等[23]研究SNPs (rs3741219和rs217727)与乳腺癌风险之间的关联,在分层分析中发现, rs217727位点在怀孕次数大于2次的女性人群中,携带CT和TT基因型的个体患乳腺癌的风险显著降低。Yang等[24]研究SNPs与中国汉族人群胃癌易感性之间的关联,结果发现rs217727和rs2839698位点SNPs与胃癌的易感性有关,其中CT和TT基因携带者胃癌的发病风险显著增加。本研究观察rs3024270和rs3741219位点SNPs与胃癌易感性的关系,结果发现在rs3024270位点携带GC和GG基因型可显著降低罹患胃癌的风险。这一结果提示G等位基因可能为胃癌的保护基因,携带G等位基因可显著降低罹患胃癌的风险(=0.001)。而rs3741219位点的研究结果却与以往不同,并未发现其与胃癌的发病风险有关。出现这种差异的原因可能是实验选取样本量的大小及存在一定选择性偏倚。以上结果也提示,基因多态性与肿瘤易感性的关系受不同种族、不同地区、不同肿瘤及不同环境等多方面影响。
HOTAIR是反义非编码RNA,是当前备受关注的参与基因组修饰的lncRNA之一。HOTAIR在多种肿瘤中存在差异表达,并且对不同肿瘤的发生发展及预后产生一定影响[25-27]。进一步研究表明, HOTAIR可与蛋白质多梳复合抑制物相结合,形成复合物,从而改变其在全基因组中的定位,并诱导染色体组蛋白H3的第27位氨基酸(赖氨酸)发生甲基化,最终对肿瘤的增殖和转移发挥作用[5]。Gupta[26]等的研究表明, HOTAIR在胃癌组织中的表达量显著高于癌旁组织。HOTAIR高表达可能会影响胃癌多方面的临床特征,如肿瘤分期、侵袭转移、预后等。Guo等[27]在研究贲门癌组织中的表达情况时还观察到该基因的SNP与其高表达之间存在基因型特异效应。SNPs与肿瘤易感性的相关研究结果也显示rs920778位点的SNP与食道癌发病风险显著相关[28]。本研究表明,rs4759314的多态性与罹患胃癌的风险有关,其中携带GG基因型的人群可显著增加胃癌的发病风险。这预示着胃癌的风险基因可能是G等位基因,携带G等位基因人群罹患胃癌的风险增加。而rs874945位点的SNP与胃癌易感性之间确未发现相关性。出现这一结论的原因,其一,可能是因为rs4759314虽位于基因的内含子区,然而其具有启动子区的生物学效应,为高风险位点,且该位点的SNP也与周围基因的表型有关;其二,可能跟本研究所存在的不足有关,如未能考虑环境因素及幽门螺杆菌感染情况对与SNP在胃癌发病过程中的影响。后续工作中将考虑众多因素,设置多种对照,进一步完善此研究。
EBV相关肿瘤的致瘤机制是:通过抑制机体免疫系统对病毒转化细胞的清除作用,从而实现EBV感染的免疫逃逸。在EBVaGC形成过程中, EBV可通过诱导宿主基因CpG岛区的异常甲基化,使得宿主细胞的蛋白质编码基因出现异常表达,或干扰microRNA的表达,进而影响宿主细胞基因的表达,从而促进EBVaGC的形成[8-9,29]。EBVaGC致癌机制的深入研究还显示, EBV感染可通过启动子的高甲基化来调控lncRNA H19的表达水平,进而参与EBV相关肿瘤的发生发展。本研究观察lncRNA与的SNPs与EBVaGC易感性的关系,结果显示rs4759314等位基因频率在两组中的分布情况有差异, G等位基因在EBVaGC组的分布显著高于EBVnGC,而两组其余基因型及等位基因携带者之间则无显著差异,且其他3个SNPs的分布情况在EBVaGC和EBVnGC两组中的差异均无统计学意义。本研究结果尚不能说明lncRNA SNPs与EBVnGC易感性之间存在明显关联。出现当前结果的原因可能是因为EBVaGC发病机制具有独特性和复杂性,且SNPs对lncRNA功能和结构的影响过程是受多因素干扰的复杂过程。同时本实验的样本量有限,后期应该增加样本量,选择多位点进一步深入研究lncRNA SNPs与EBVaGC易感性之间的关系。
综上所述,rs4759314和rs3024270的SNPs与胃癌易感性密切相关。携带风险基因的人群则罹患胃癌的风险增大,相反携带保护基因则其胃癌的易感性降低。这一结论为胃癌易感人群的筛查及疾病防治提供了参考。
[1] Bray F, Ferlay J, Soerjomataram I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(6):394-424.
[2] Yang L, Zheng R, Wang N, et al. Incidence and mortality of stomach cancer in China, 2014[J]. Chin J Cancer Res, 2018, 30(3):291-298.
[3] Fukayama M, Chong JM, Kaizaki Y. Epstein-Barr virus and gastric carcinoma[J]. Gastric Cancer, 1998, 1(2):104-114.
[4] Chen W,Zheng R, Baade PD, et al. Cancer statistics in China, 2015[J]. CA Cancer J Clin, 2016, 66(2):115-132.
[5] Elsayed ET, Salem PE, Darwish AM, et al. Plasma long non-coding RNAas a potential biomarker for gastric cancer[J]. Int J Biol Markers, 2018, 33(4):528-533.
[6] Chen JS, Wang YF, Zhang XQ, et al. H19 serves as a diagnostic biomarker and up-regulation of H19 expression contributes to poor-prognosis in patients with gastric cancer[J]. Neoplasma, 2016, 63(2):223-230.
[7] Cancer genome atlas research network. comprehensive molecular characterization of gastric adenocarcinoma[J]. Nature, 2014, 513(7517):202-209.
[8] Gulley ML. Genomic assays for Epstein-Barr virus-positive gastric adenocarcinoma[J]. Exp Mol Med, 2015, 47:e134.
[9] Nishikawa J, Yoshiyama H, Iizasa H, et al. Epstein-barrvirus in gastric carcinoma[J]. Cancers (Basel), 2014, 6(4):2259-2274.
[10] Zhang K, Luo Z, Zhang Y, et al. Circulating lncRNA H19 in plasma as a novel biomarker For breast cancer[J]. Cancer Biomark, 2016, 17(2):187-194.
[11] Huang C, Cao L, Qiu L, et al. Upregulation of H19 promotes invasion and induces epithelial-to-mesenchymal transition in esophageal cancer[J]. Oncol Lett, 2015, 10(1):291-296.
[12] Dong H, Xu G, Meng W, et al. Long noncoding RNA H19 indicates a poor prognosis of colorectal cancer and promotes tumor growth by recruiting and binding to eIF4A3[J]. Oncotarget, 2016, 7(16):22159-22173.
[13] Wang DG, Fan JB, Siao CJ, et al. Large-scale identification, mapping, and genotyping of single-nucleotide polymorphisms in the human genome[J]. Science, 1998, 280(5366):1077-1082.
[14] 张明娇,吴勇,李珉珉,等. 长链非编码RNAs的生物功能及其与器官发育和恶性肿瘤的关系[J]. 中国病理生理杂志, 2015, 31(11):2107-2112.
Zhang MJ, Wu Y, Li MM, et al. Long noncoding RNAs: biological functions and applications in organ development and malignant tumors[J]. Chin J Pathophysiol, 2015, 31(11):2107-2112.
[15] Sakamoto H, Yoshimura K, Saeki N, et al. Genetic variation in PSCA is associated with susceptibility to diffuse-type gastric cancer[J]. Nat Genet, 2008, 40(6):730-740.
[16] Wang LD, Zhou FY, Li XM, et al. Genome-wide association study of esophageal squamous cell carcinoma in Chinese subjects identifies susceptibility loci at PLCE1 and C20orf54[J]. Nat Genet, 2010, 42(9):759-763.
[17] Shi Y, Hu Z, Wu C, et al. A genome-wide association study identifies new susceptibility loci for non-cardia gastric cancer at 3q13.31 and 5p13.1[J]. Nat Genet, 2011, 43(12):1215-1218.
[18] Hu N, Wang Z, Song X, et al. Genome-wide association study of gastric adenocarcinoma in Asia: a comparison of associations between cardia and non-cardia tumours[J]. Gut, 2016, 65(10):1611-1618.
[19] Mocellin S, Verdi D, Pooley KA, et al. Genetic variation and gastric cancer risk: a field synopsis and meta-analysis[J]. Gut, 2015, 64(8):1209-1219.
[20] Jin G, Ma H, Wu C, et al. Genetic variants at 6p21.1 and 7p15.3 are associated with risk of multiple cancers in Han Chinese[J]. Am J Hum Genet, 2012, 91(5):928-934.
[21] Li H, Yu B, Li J, et al. Overexpression of lncRNA H19 enhances carcinogenesis and metastasis of gastric cancer[J]. Oncotarget, 2014, 5(8):2318-2329.
[22] Niinuma T, Suzuki H, Nojima M, et al. Upregulation of miR-196a and HOTAIR drive malignant character in gastrointestinal stromal tumors[J]. Cancer Res, 2012, 72(5):1126-1136.
[23] Xia Z, Yan R, Duan F, et al. Genetic polymorphisms inlong noncoding RNA H19 are associated with susceptibility to breast cancer in Chinese population[J]. Medicine (Baltimore), 2016, 95(7):e2771.
[24] Yang C, Tang R, Ma X, et al. Tag SNPs in long non-coding RNA H19 contribute to susceptibility to gastric cancer in the Chinese Han population[J]. Oncotarget, 2015, 6(17):15311-15320.
[25] 陈文慧,何晓华,张鼎,等. 长链非编码RNA HOTAIR在接受调强放射治疗鼻咽癌患者中的表达及其临床意义[J]. 中国病理生理杂志, 2019, 35(8):1439-1444.
Chen WH, He XH, Zhang D, et al. High expression of HOTAIR in nasopharyngeal carcinoma patients receiving intensity-modulated radiotherapy and its clinical significance[J]. Chin J Pathophysiol, 2019, 35(8):1439-1444.
[26] Gupta RA, Shah N, Wang KC, et al. Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis[J]. Nature, 2010, 464(7291):1071-1076.
[27] Guo W, Dong Z, Bai Y, et al. Associations between polymorphisms of HOTAIR and risk of gastric cardia adenocarcinoma in a population of north China[J]. Tumour Biol, 2015, 36(4):2845-2854.
[28] Zhang S, Lee DS, Morrissey R, et al. Early or late antibiotic intervention prevents-induced gastric cancer in a mouse model[J]. Cancer Lett, 2014, 355(1):106-112.
[29] Yang CA, Scheibenbogen C, Bauer S, et al. A frequent Toll-like receptor 1 gene polymorphism affects NK and T-cell IFN-γ production and is associated with-induced gastric disease[J]. Helicobacter, 2013, 18(1):13-21.
Relationships between lncRNA/SNPs and genetic susceptibility to gastric carcinoma/EBV-related gastric carcinoma
WEI Mei-rong1, WANG Xiao-feng2, LUO Bing2
(1,,710123,;2,,,,266021,)
To investigate the potential associations between the single nucleotide polymorphisms (SNPs) of long noncoding RNA (lncRNA)and the susceptibility to gastric carcinoma, especially to Epstein-Barr virus (EBV)-associated gastric carcinoma (EBVaGC).Peripheral blood samples from 65 cases of EBV-negative gastric carcinoma (EBVnGC), 50 cases of EBVaGC and 115 cases of healthy people were collected. A total of 4 TagSNPs,rs3024270 and rs3741219, as well asrs4759314 and rs874945, were selected. The Taq-Man MGB allele typing kit was used to detect the genotype of each SNP locus, and the experimental results were statistically analyzed.(1) There were significant differences of both genotypic and allelic frequencies atrs3024270 locus between gastric carcinoma group and control group (<0.05). Individuals carrying the G allele atrs3024270 locus had significantly low risk of gastric carcinoma (<0.01), indicating that the G allele was protective. (2) People with the GG genotype atrs4759314 locus had significantly high risk of gastric carcinoma (<0.05). Carrying the G allele increased the risk of gastric carcinoma, which indicated that the risk gene for gastric carcinoma might be the G allele. (3) No significant difference of the genotypic and allelic frequencies atrs3741219 andrs874945 loci between gastric carcinoma group and control group was observed (>0.05).(4) The G allele frequency atrs4759314 locus in EBVaGC group was significantly higher than that in EBVnGC group. However, no difference of the other 3 SNPs was found between EBVaGC group and EBVnGC group (>0.05).The SNPs atrs3024270 andrs4759314 loci are related to the risk of gastric carcinoma, but not significantly related to the risk of EBVaGC.
Gastric carcinoma; Long noncoding RNA;gene;gene; Epstein-Barr virus; Single nucleotide polymorphism; Susceptibility
R735.2; R730.23
A
10.3969/j.issn.1000-4718.2020.11.015
1000-4718(2020)11-2030-07
2020-03-29
2020-05-13
国家自然科学基金资助项目(No.81571995);西京学院校级科研基金资助项目(No.XJ200101)
Tel: 17602970596; E-mail: luobing212@163.com
(责任编辑:林白霜,罗森)
