黄麻SSR标记与纤维产量性状的相关性
2020-11-27张力岚张列梅牛焕颖祁建民陶爱芬方平平张立武
张力岚 张列梅 牛焕颖 徐 益,2,3 李 玉,2,3 祁建民,2 陶爱芬,2 方平平,2 张立武,2,3,*
黄麻SSR标记与纤维产量性状的相关性
张力岚1,2,3,**张列梅1,**牛焕颖1徐 益1,2,3李 玉1,2,3祁建民1,2陶爱芬1,2方平平1,2张立武1,2,3,*
1福建农林大学农学院/ 作物遗传育种与综合利用教育部重点实验室/ 福建省作物设计育种重点实验室, 福建福州 350002;2福建农林大学农业农村部东南黄红麻实验观测站/ 福建省麻类种质资源共享平台/ 福建省南方经济作物遗传育种与多用途开发国际科技合作基地, 福建福州 350002;3福建农林大学海峡联合研究院基因组与生物技术中心, 福建福州 350002
鉴定出与纤维产量相关性状连锁的SSR标记有助于黄麻纤维产量的分子标记辅助育种。本研究以311份黄麻种质资源为研究材料, 通过2016—2018年表型鉴定, 结合116对SSR引物扩增出397个SSR标记, 利用SPSS软件对SSR标记与纤维产量性状进行相关性分析。结果表明, 311份黄麻种质资源9个纤维产量性状的变异系数范围为13.05%~76.78%, 表现出广泛的遗传变异。农艺性状相关分析表明, 这些性状间存在极显著相关, 其中分枝高和节数的平均相关系数最高(= 0.931), 其次是单株鲜皮重和单株鲜茎重(= 0.781)、单株干皮重与单株鲜皮重(= 0.779)。方差分析表明, 节数、分枝数、株高以及分枝高等性状在不同年份间相对稳定, 广义遗传力较高。进而通过皮尔逊相关法鉴定与纤维产量性状相关的SSR标记。每个性状与这些相关SSR标记逐步回归分析表明, 与纤维产量性状显著相关的标记有6个, 单个SSR标记的贡献率为3.9%~22.5%。这些结果将会加快黄麻设计育种进程。
黄麻; 纤维产量; 农艺性状; SSR; 相关性; 回归分析
黄麻属(L.)有100多个种, 生产上具有栽培价值的有圆果种()和长果种(), 属于一年生韧皮部纤维作物(2=2=14), 是我国重要的经济作物之一。目前, 黄麻主要在东南亚地区种植, 喜欢温暖湿润的气候, 其在农业、工业、制造业等方面发挥了重大作用[1]。目前, 我国已经成为黄麻主要生产国孟加拉国的主要出口市场(http:// www.ccpit.org/)。黄麻可以成为制作纺织品、麻床垫、重金属污水吸附剂、麻制膜等产品的原材料[2]。
黄麻纤维产量等农艺性状属于数量性状, 往往受多基因控制[3-4]。在作物育种中, 越来越多的分子标记应用到农艺性状改良。目前, 不同作物利用较多的分子标记, 主要有SSR (simple sequence repeat)、SNP (single nucleotide polymorphism)等。其中, SSR因成本低、效果好、检测方便等特点得到了广泛应用[5-7]。Satya等[8]在10,041个黄麻基因中开发了12,772个SSR, 并鉴定了与韧皮纤维形成相关基因的SSR标记多态性。这些SSR标记可被用来构建遗传图谱及农艺性状鉴定。鉴定出与农艺性状相关的分子标记, 可通过分子标记辅助选择, 达到性状改良目的。
通过适当的分离群体进行QTL (quantitative trait loci)定位, 或者利用自然群体进行关联分析, 可以鉴定出与作物农艺性状连锁的分子标记。QTL定位在黄麻遗传育种研究中有一定的报道。Tao等[9]利用简化基因组测序手段在黄麻重组自交系群体(F8)中检测到了69,446条SLAFs (specific-locus amplified fragment sequencing)标记, 其中5074条具有多态性, 选择913条多态性标记用于构建遗传图谱, 结合群体表型, 检测到了11个株高QTL。利用自然群体, 在没有足够多标记条件下, 往往无法有效地进行关联分析; 这种情况下, 可利用多元逐步回归鉴定出和农艺性状显著相关的分子标记[10-11]。Lou等[10]对115份不同来源的高羊茅品种的农艺性状和遗传多样性进行了评价, 共检测到51个与农艺性状相关的SSR标记。黄麻的全基因组未被全部绘制[12-13], 限制了分子标记辅助选择在黄麻农艺性状改良上的应用。
目前, 涉及作物农艺性状之间的相关性分析已有较多的报道。如张彬等[14]通过相关分析, 发现不同小麦品系的产量与茎蘖数呈极显著正相关, 有效穗数与产量、茎蘖数呈显著正相关, 穗粒数与千粒重、株高呈负相关。殷冬梅等[15]发现, 花生的主茎高与侧枝长、百果重与百仁重均呈极显著正相关, 而单株结果数与百仁重呈显著负相关。Kumar等[16]对63个黄麻种质资源的25个农艺性状进行了评价, 发现纤维产量与开花期、株高、茎粗和鲜重呈显著正相关。但是, 涉及到利用分子标记与黄麻自然群体农艺性状进行相关性研究的却鲜有报道。
本研究以311份黄麻种质资源作为材料, 利用SSR标记进行多态性分析。先利用皮尔逊相关分析黄麻9个纤维产量性状与SSR标记的相关性。继而利用逐步回归法鉴定与纤维产量性状显著相关的标记。鉴定出和黄麻农艺性状显著相关的SSR标记, 可用于黄麻分子标记辅助育种。
1 材料与方法
1.1 材料
来源不同的311份黄麻种质资源[17]均由福建农林大学麻类遗传育种与综合利用实验室提供。分别于2016年5月1日、2017年4月20日和2018年5月1日在福建省三明市尤溪县福建农林大学洋中科教基地, 按照2行区, 行长为3.5 m, 株行距为1.2 m´0.1 m, 采用单因素随机区组设计种植供试材料。边行种植2行材料作为保护行, 田间管理同大田。
1.2 基因组DNA提取
剪取2 g黄麻幼嫩叶片, 用2% CTAB法提取黄麻基因组DNA。测定DNA浓度并将其浓度稀释到50 ng μL-1, 保存在-20℃的冰箱备用。
1.3 PCR扩增与SSR多态性检测
本研究所用的SSR标记主要来源于实验室前期研究[18-19], 由生工生物工程(上海)股份有限公司合成。PCR扩增采用10 μL的反应体系, 包括DNA模板2μL、10 μmol μL–1上下游引物各0.25 μL、PCR mix buffer和ddH2O各3.75 μL, 最后再加10 μL石蜡油覆盖。反应扩增程序为95℃ 3 min; 95℃ 9 s, 57℃ 30 s, 72℃ 45 s, 9个循环; 95℃ 30 s, 55℃ 30 s, 72℃ 45 s, 34个循环; 72℃ 10 min; 4℃保存。对PCR产物进行聚丙烯酰胺凝胶电泳分离, 并检测SSR标记多态性。将所测得的标记多态性数据以0或1的方式输入电脑保存。
1.4 纤维产量相关性状考察
黄麻纤维产量相关性状考察参考《黄麻种质资源描述规范和数据标准》[20]。在黄麻工艺成熟时, 测量10株左右植株的株高、分枝高、茎粗、鲜皮厚、节数、分枝数、单株鲜茎重、单株鲜皮重、单株干皮重等性状。
1.5 数据统计分析
用SPSS 13.0处理数据。采用皮尔逊相关分析去分析黄麻各纤维产量性状间、黄麻各性状与标记间的相关系数。通过多元线性逐步回归法[21]拟合与黄麻农艺性状具有显著相关性的SSR标记。其中X是各个SSR标记产生的基因型, Y是黄麻各个农艺性状。
2 结果与分析
2.1 黄麻9个纤维产量性状的统计分析
黄麻9个纤维产量性状3年表型的基本统计分析如表1所示。9个性状的变异系数范围是13.05%~76.78%。其中, 节数是黄麻9个性状中变异系数最大的, 其变异系数为55.96%~75.61%, 在不同材料之间表现出丰富的变异; 而变异系数最小的是茎粗与株高, 其变异系数分别为15.83%~17.21%、13.05%~16.71%, 变异范围相对较窄。9个性状的香农-维纳多样性指数(Shannon-Wiener index)变化在1.52~2.62之间。其中, 分枝高、节数、分枝数、单株鲜茎重和单株鲜皮重的遗传多样性指数较大, 说明这些性状在每一级中的分布较均匀。
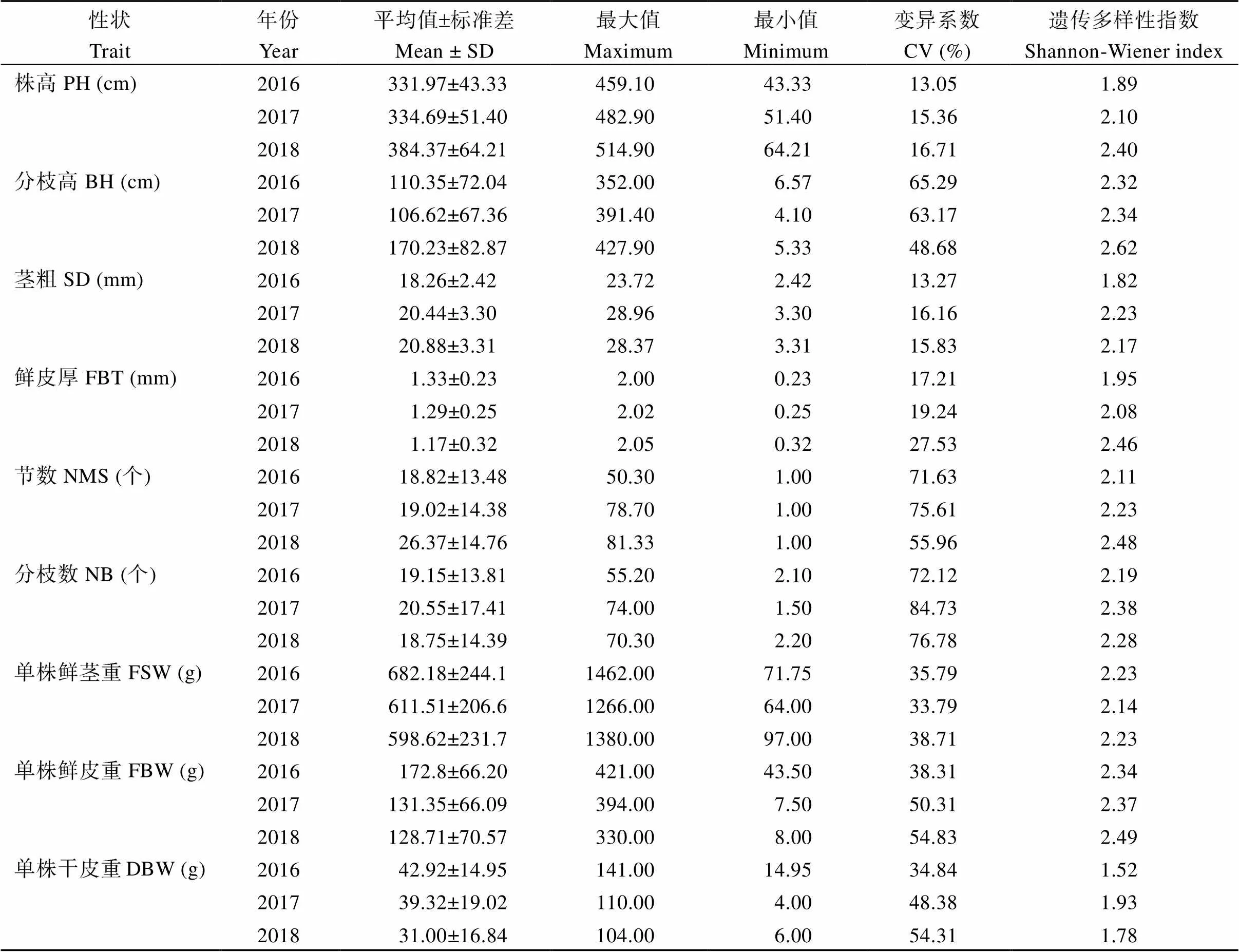
表1 黄麻9个纤维产量相关性状的统计分析
SD: 标准差; CV: 变异系数; PH: 株高; BH: 分枝高; SD: 茎粗; FBT: 鲜皮厚; NMS: 节数; NB: 分枝数; FSW: 单株鲜茎重; FBW: 单株鲜皮重; DBW: 单株干皮重。
SD: standard deviation; CV: coefficient of variation; PH: plant height; BH: branch height; SD: stem diameter; FBT: fresh bark thickness; NMS: nodes of main stem; NB: number of branches; FSW: fresh stem weight per plant; FBW: fresh bark weight per plant; DBW: dry bark weight per plant.
2.2 纤维产量性状的方差分析
以品种和年份为控制因子, 对9个纤维产量性状进行双因素方差分析(表2)。黄麻9个纤维产量性状的广义遗传力变化在0.61至0.90之间。其中, 株高、分枝高、节数和分枝数的遗传力较高, 性状较为稳定, 而鲜皮厚的遗传力最低。这表明, 大多数纤维产量性状的遗传效应在不同年份间是稳定存在的,受外界环境的影响较小, 主要取决于品种本身。
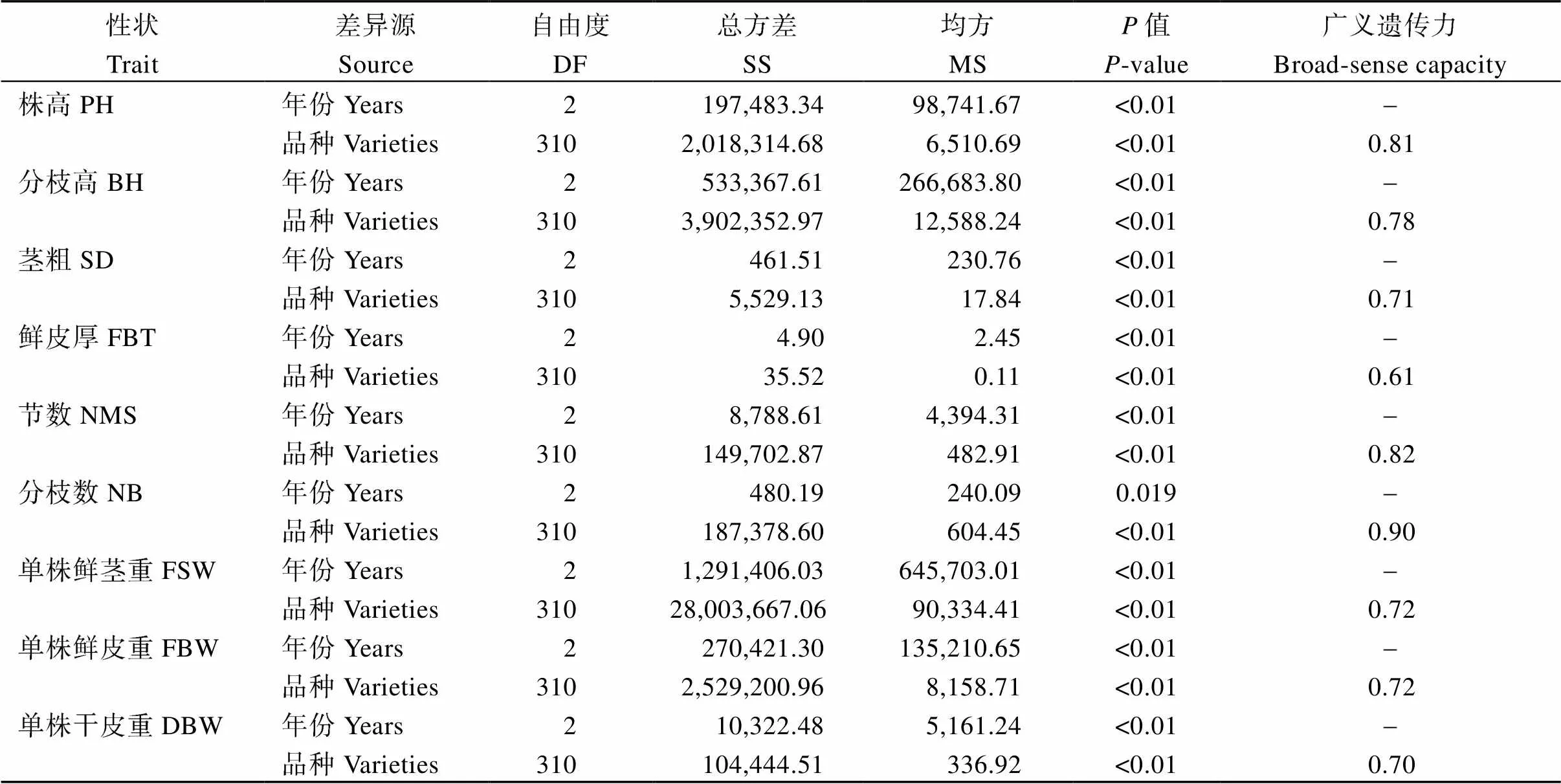
表2 黄麻9个纤维产量性状的方差分析
缩写同表1。Abbreviations are the same as those given in Table 1.
2.3 纤维产量性状的相关分析
不同纤维产量性状的不同年份间相关分析如表3所示。其中, 分枝数(=0.806, 0.725, 0.771)、株高(=0.727, 0.684, 0.741)与节数(=0.727, 0.684, 0.741)在不同年份间的相关系数较高, 均大于0.6。而鲜皮厚(=0.328, 0.177, 0.381)、单株鲜茎重(=0.458, 0.368, 0.370)和单株干皮重(=0.307, 0.267, 0.398)的相关系数较低。结果显示, 节数、分枝数、株高以及分枝高等纤维产量性状在不同年份间相对稳定, 与这些性状具有较高的广义遗传力是一致的。
大多数黄麻纤维产量性状间存在极显著的相关性(表4)。9个性状间相关系数最大的3个依次是0.959 (分枝高与节数, 2016年)、0.883 (单株鲜皮重和单株干皮重, 2017年)、0.816 (单株鲜皮重和单株鲜茎重, 2016年), 达到极显著水平; 且这3组性状在3年中的相关系数均为较大值。而节数与茎粗、鲜皮厚与分枝高、鲜皮厚与节数的相关系数均小于0.2, 表现出不相关。而分枝高与分枝数、节数与分枝数均呈极显著负相关。
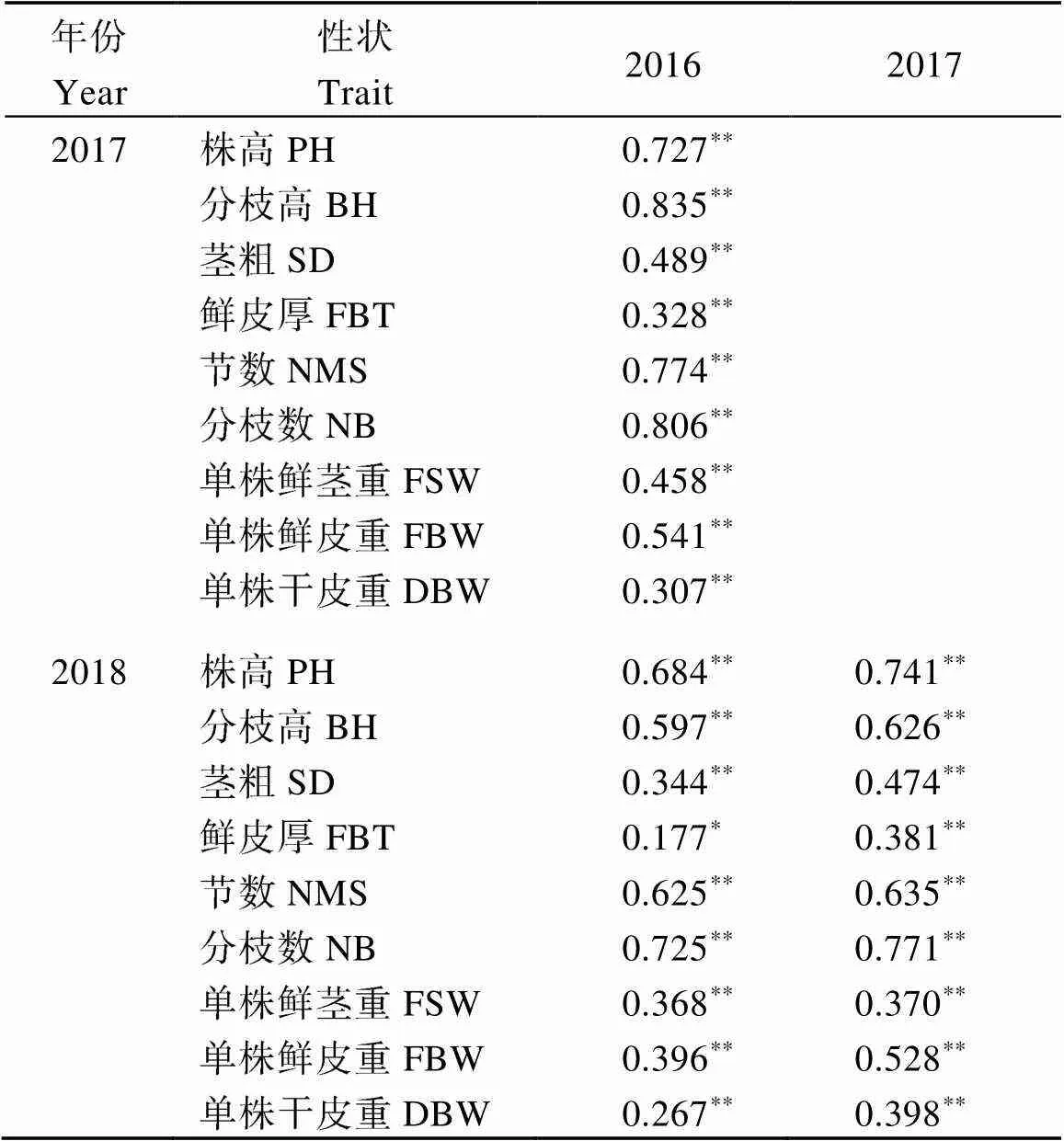
表3 黄麻9个纤维产量相关性状的不同年份间相关分析
*和**分别表示在< 0.05和< 0.01水平显著。缩写同表1。
*and**mean significant differences at< 0.05 and< 0.01, respectively. Abbreviations are the same as those given in Table 1.
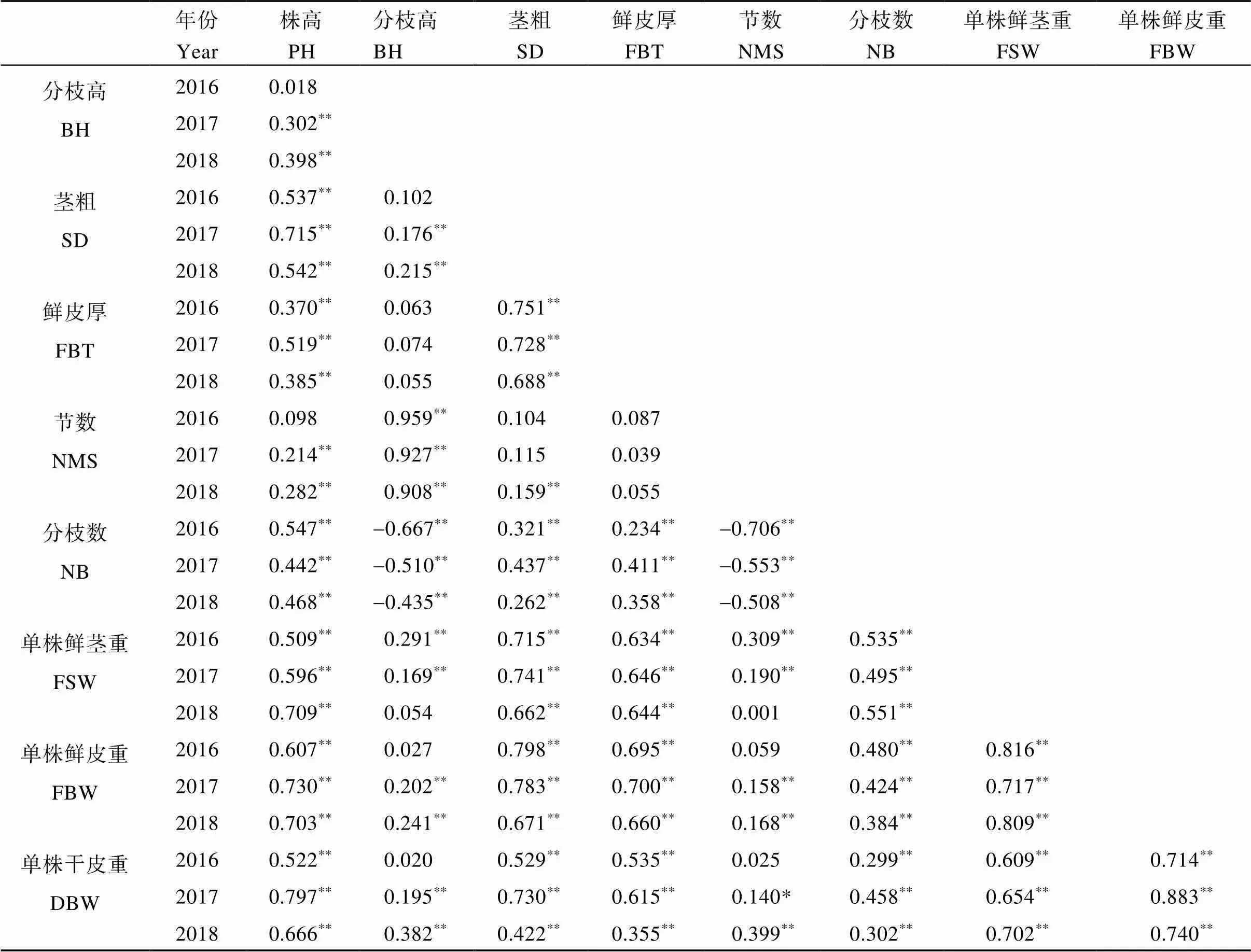
表4 黄麻9个纤维产量性状的相关分析
*和**分别表示在< 0.05和<0.01水平显著。缩写同表1。
*and**mean significant differences at< 0.05 and< 0.01, respectively. Abbreviations are the same as those given in Table 1.
2.4 纤维产量性状与SSR标记的相关性
116对SSR引物对311份种质资源进行多态性分析, 获得397个标记。利用SPSS软件, 对397个SSR标记与9个纤维产量性状进行相关性分析(表5)。结果表明, 单个纤维产量相关性状可以与多个分子标记建立相关性, 说明这些性状是由多个基因位点控制的。与株高、分枝高、茎粗、鲜皮厚、节数、分枝数、单株鲜茎重、单株鲜皮重、单株干皮重相关的SSR标记数分别为37、66、78、64、60、38、24、74、41。其中, 与株高、分枝高、茎粗、鲜皮厚、节数、分枝数、单株鲜茎重、单株鲜皮重、单株干皮重相关性最高的SSR标记分别为CcID102 (=-0.341,≤0.01)、CoSSR179 (=-0.347,≤0.01)、CoSSR227 (=-0.373,≤0.01)、Juph 112 (= 0.459,≤0.01)、CoSSR179 (=-0.333,≤0.01)、Juph059 (= 0.429,≤0.01)、Juph023 (=-0.378,≤0.01)、Juph 112 (= 0.426,≤0.01)、Juph059 (= 0.414,≤0.01)。
由表5可知, 同一标记可以与多个性状建立相关性。标记CcID102与株高(=0.31)、分枝高(=-0.199)、茎粗(=0.166)、鲜皮厚(=0.163)、节数(=-0.141)、分枝数(=0.358)、单株鲜茎重(=0.303)、单株鲜皮重(=0.177)、单株干皮重(=0.245)均呈极显著相关, 推测带标记CcID102标记的黄麻品种, 其与纤维产量正相关的相关性状将会有一定程度的提高, 而节数与分枝数可能会减少。Juph 112与单株干皮重(=0.329)、茎粗(=0.371)、鲜皮厚(=0.459)、分枝数(=0.309)均呈极显著正相关, 预测具有标记Juph 112的黄麻品种, 其此部分纤维产量相关性状将有一定程度的上升。CoSSR227与单株干皮重(=-0.214)、分枝高(=-0.316)、茎粗(=-0.373)、鲜皮厚(=-0.286)、节数(=-0.316)、单株鲜皮重(=-0.325)均呈极显著负相关, 预测携带标记CoSSR227的黄麻品种, 其此部分纤维产量相关性状的数值将会下降。标记CcID167与除分枝高以外的其余8个黄麻性状呈极显著正相关。预测携带CcID167的黄麻品种, 除分枝高以外的其余8个性状将有一定程度的提升。标记CoSSR019与除分枝数以外的其余8个性状存在显著相关, 预计携带此标记的黄麻品种, 其分枝高数值降低, 其余7个性状的数值将上升。
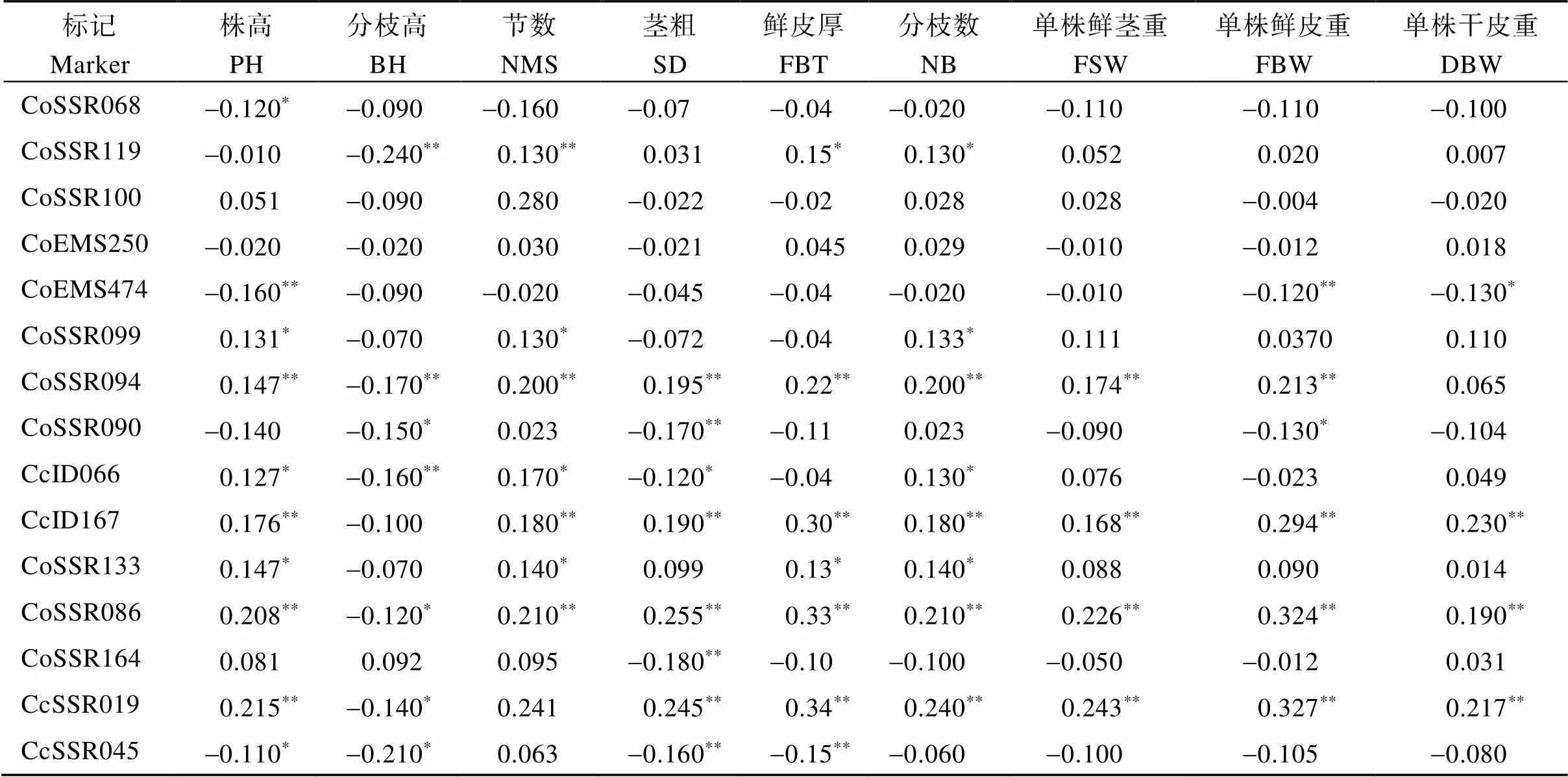
表5 部分SSR标记和9个纤维产量性状间的相关系数
*和**分别表示在< 0.05和< 0.01水平显著。缩写同表1。
*and**mean significant differences at< 0.05 and< 0.01, respectively. Abbreviations are the same as those given in Table 1.
2.5 纤维产量性状与SSR标记的多元线性逐步回归
在分析单个纤维产量性状与SSR标记相关性的基础上, 继而进行这些SSR标记与纤维产量性状的多元线性逐步回归(表6)。结果表明, 与9个黄麻纤维产量性状相关联的SSR标记有6个, 且其贡献率(2)的范围在3.9%~22.5%之间。其中, Juph059对鲜皮厚所解释的变异为22.5%, CoSSR305对单株鲜茎重解释的变异为15.3%, CoSSR015对茎粗所解释的变异为13.1%。此外, Juph059对鲜皮厚的标准回归系数最大, 其数值为0.475, 表明Juph059对此性状存在较大正相关。CoSSR192对单株鲜皮重的标准回归系数为-0.449, 表明CoSSR192对此性状存在较大负相关。CoSSR305对单株鲜茎重的标准回归系数是0.391, 表示CoSSR305对此性状存在较大正相关。
通过多元线性回归法分析, 也发现单个标记可与多个性状相关联。株高和分枝数均与标记CcID102相关联, 其解释变异率分别为19.9%和3.9%, 而株高与分枝数这2个性状本身就呈极显著相关。CoSSR179对分枝高和节数的解释变异率分别为12.1%和11.1%, 2个性状的相关系数是所有相关性中最高的。标记CoSSR192与单株鲜皮重和单株干皮重均相关, 变异解释率分别为20.2%和7.1%, 而这2个性状的平均相关系数大于0.7, 呈极显著正相关。这说明, 这些纤维产量性状之间存在一定的遗传相关。
3 讨论
纤维产量改良是黄麻育种的重要目标。本研究黄麻9个纤维产量性状统计分析显示, 这些数量性状的变异系数变化范围比较大, 体现出这些种质资源符合作为数量性状研究的群体要求, 也可以作为黄麻育种的基础材料。变异系数小于20%的纤维产量性状为株高和茎粗, 说明这2个性状在该种质资源群体中变化范围相对较窄; 而其余纤维产量相关性状的变异系数均大于30%, 表明这些性状在该种质资源群体中存在广泛的变异。
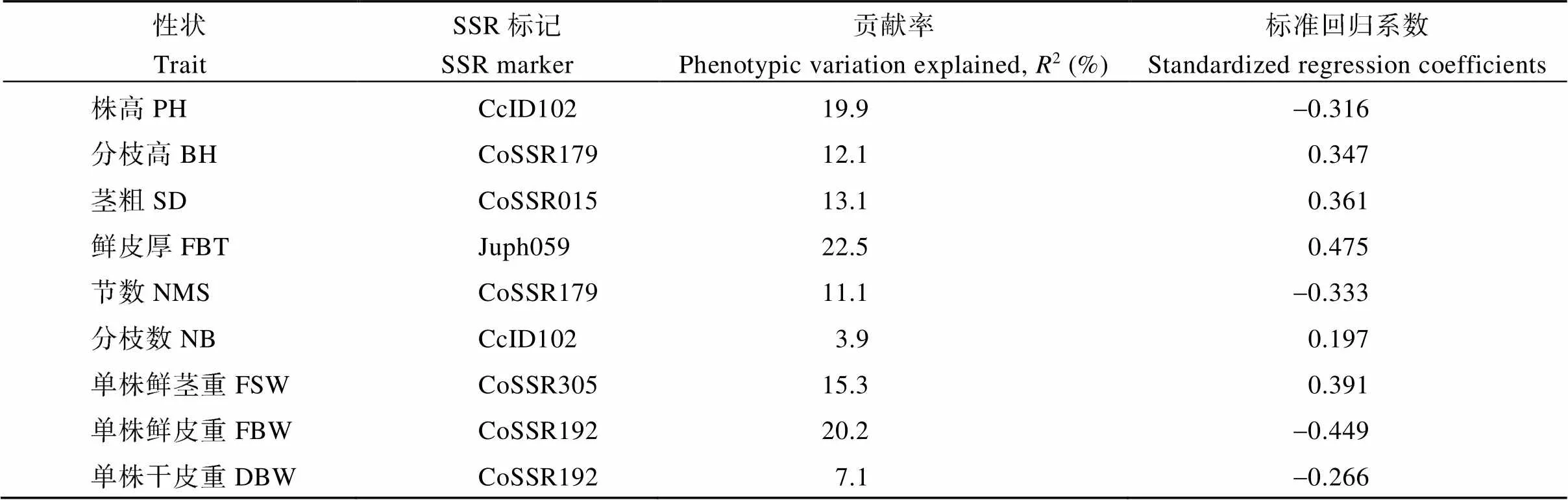
表6 SSR标记与纤维产量相关性状之间的多元逐步回归分析
缩写同表1。Abbreviations are the same as those given in Table 1.
黄麻9个纤维产量性状相关分析反映出大多数性状在不同年间相对稳定, 但同一性状在不同年份间仍然表现出一定差异。比如, 节数在2016和2017年的平均值分别为18.82和19.02, 而在2018年则增长至26.37, 株高、分枝高等性状表现出类似的趋势; 但单株鲜皮重等3个纤维产量性状却表现出减少, 推测与2018年生长后期遭遇台风引起一定倒伏有关。杨洛淼等[22]在连续3年环境下对始穗期、抽穗期、结实率、千粒重和单株粒重进行了统计, 其中2015年的统计结果与其他2年(2013年和2014年)与明显的差异, 推测这可能跟2015年播种移栽时期气温低对苗期的影响有关。同一性状在不同年份环境下表现出一定差异, 可能与不同年份间的气候环境差异有关。
SSR标记与纤维产量性状的相关性结果显示, 一个性状可以与多个SSR标记具有相关性。本研究中, 与株高、分枝高相关的SSR标记数分别为37个和6个。Kraakman等[23]对大麦的农艺性状和AFLP标记进行相关分析, 发现与产量相关的标记共有8个, 与产量稳定性相关的标记有5个。司二静等[24]在大麦中发现, 与株高、芒长和穗粒数相关联的标记均有3个, 与穗长相关联的标记有6个, 与千粒重相关联的标记有4个。这说明一个性状可与多个标记关联, 也表明这些农艺性状往往是由多基因控制的。
一个SSR标记可以与多个性状具有相关性。标记CcID102、CoSSR086、CoSSR094和CoSSR019与黄麻的多个性状同时关联。这种情况在其他作物中也有所体现。Zhao等[25]通过相关分析发现, 标记gpw5179-6D同时与小麦的粒长、粒面积和粒周长相关, gwm583-5D与粒周长和粒长相关。Abbasi等[26]通过104个SSR标记对甜菜的各个性状进行了相关分析, 发现标记FDSB502与甜菜的糖提取系数、糖浆、糖含量和叶片钾含量均相关。Fan等[27]研究表明主要控制粒长和粒重性状, 具有较大的贡献率, 同时对粒宽和粒厚有微效作用, 具有较小的贡献率。Yue等[28]研究认为, 水稻的叶片性状与产量性状间存在显著相关性, 因而主效叶绿素含量的标记可应用于水稻高产的分子标记辅助育种中。这些结果表明, 一个SSR标记与多个性状具有相关性可能是由于这个标记对应的多效性QTL引起的。
本研究的9个数量性状皆属于纤维产量性状, 这些性状间普遍存在较大的表型相关系数。这也体现在与这些性状都相关的SSR标记数。如分枝高与节数之间的相关系数均大于0.9, 则与分枝高和节数均有关联的SSR标记数高达35个, 而株高与分枝高之间的相关系数小于0.4, 而与此2个性状均相关的SSR位点仅有8个。这些结果说明, 黄麻9个纤维产量性状间的表型相关系数较大, 与这些性状都相关的SSR标记数较多, 这从标记层面反映出这些数量性状之间存在较高的遗传相关[29]。性状间高遗传相关可能是由于控制这些相关性状的基因位点相互连锁或某个基因位点的一因多效引起的。
在分析单个纤维产量性状与SSR标记相关性的基础上, 本研究继而利用逐步多元线性回归法, 筛选出与性状显著相关的标记, 可以大大减轻黄麻分子标记辅助育种的工作量。周金超等[30]通过Pearson相关分析得到花生77个与18个农艺性状显著相关SSR标记, 进一步利用逐步多元回归分析得到14个与13个农艺性状关联SSR标记。李丹阳等[31]通过Pearson相关分析发现, 77个分子标记与8个品质性状存在显著相关, 而运用逐步多元回归分析可得到45个与8个品质性状相关的标记, 标记所解释的表型变异范围为8.1%~28.5%之间。本研究通过逐步多元线性回归, 最终获得了6个与性状显著相关的标记, 其中CcID102与黄麻的株高、分枝高均具有显著相关, 贡献率分别为19.9%和12.1%。通过逐步多元线性回归法筛选出的显著相关标记, 可有效地减轻育种工作者的工作量, 从而加快黄麻设计育种的培育进程。
4 结论
黄麻9个纤维产量性状之间普遍存在显著相关。其中, 节数、分枝数、株高以及分枝高等性状相对稳定。皮尔逊相关结合逐步回归分析鉴定出6个与纤维产量性状显著相关的标记, 单个SSR标记的贡献率变化在3.9%至22.5%之间。
[1] Zhang L W, Ibrahim A K, Niyitanga S, Zhang L M, Qi J M. Jute (spp.) Breeding. Advances in Plant Breeding Strategies: Industrial and Food Crops, 2019. pp 85–113.
[2] 粟建光, 戴志刚, 杨泽茂, 唐蜻, 谢冬微, 陈基权, 许英, 徐建堂, 张利国, 龚友才, 宋宪友, 程超华, 邓灿辉. 麻类作物特色资源的创新与利用. 植物遗传资源学报, 2019, 20: 11–19. Su J G, Dai Z G, Yang Z M, Tang Q, Xie D W, Chen J Q, Xu Y, Xu J T, Zhang L G, Gong Y C, Song X Y, Cheng C H, Deng C H. Innovation and utilization of characteristic germplasm for bast fiber crops., 2019, 20: 11–19 (in Chinese with English abstract).
[3] Mondal S, Hadapad A B, Hande P A, Badigannavar A M. Identification of quantitative trait loci for bruchid (Olivier) resistance components in cultivated groundnut (L.)., 2014, 33: 961–973.
[4] Crouzillat D, Phillips W, Fritz P J, Pétiard V. Quantitative trait loci analysis inusing molecular markers. Inheritance of polygenic resistance toin two related cacao populations., 2000, 114: 25–36.
[5] Banerjee S, Das M, Mir R R, Kundu A, Topdar N, Sarkar D, Sinha M K, Balyan H S, Gupta P K. Assessment of genetic diversity and population structure in a selected germplasm collection of 292 jute genotypes by microsatellite (SSR) markers., 2012, 3: 11–25.
[6] Liu S, Liu H, Wu A, Hou Y, An Y, Wei C. Construction of fingerprinting for tea plant () accessions using new genomic SSR markers., 2017, 37: 93.
[7] Dachapak S, Somta P, Poonchaivilaisak S, Yimram T, Srinives P. Genetic diversity and structure of the zombi pea [(L.) A. Rich] gene pool based on SSR marker analysis., 2017, 145: 189–200.
[8] Satya P, Chakraborty A, Jana S, Majumdar S, Karan M, Sarkar D, Datta S, Mitra J, Kar C, Karmakar P, Singh N. Identification of genic SSRs in jute (, Malvaceae) and development of markers for phenylpropanoid biosynthesis genes and regulatory genes., 2017, 136: 784–797.
[9] Tao A, Huang L, Wu G, Afshar R K, Qi J, Xu J, Fang P, Lin L, Zhang L, Lin P. High-density genetic map construction and QTLs identification for plant height in white jute (L.) using specific locus amplified fragment (SLAF) sequencing., 2017, 18: 355.
[10] Lou Y, Hu L, Chen L, Sun X, Yang Y, Liu H, Xu Q. Association analysis of simple sequence repeat (SSR) markers with agronomic traits in tall fescue (Schreb.)., 2015, 10: e0133054.
[11] Prabhu D A, Ray J D, Singh S K, Valerio H V, Smith J R, Purcell L C, King A, Fritschi F B. Genome-wide association analysis of diverse soybean genotypes reveals novel markers for nitrogen traits., 2015, 8: 1–15.
[12] Islam M S, Saito J A, Emdad E M, Ahmed B, Islam M M, Halim A, Hossen Q M, Hossain M Z, Ahmed R, Hossain M S, Kabir S M, Khan M S, Khan M M, Hasan R, Aktar N, Honi U, Islam R, Rashid M M, Wan X, Hou S, Haque T, Azam M S, Moosa M M, Elias S M, Hasan A M, Mahmood N, Shafiuddin M, Shahid S, Shommu N S, Jahan S, Roy S, Chowdhury A, Akhand A I, Nisho G M, Uddin K S, Rabeya T, Hoque S M, Snigdha A R, Mortoza S, Matin S A, Islam M K, Lashkar M Z, Zaman M, Yuryev A, Uddin M K, Rahman M S, Haque M S, Alam M M, Khan H, Alam M. Comparative genomics of two jute species and insight into fibre biogenesis., 2017, 3: 16223.
[13] 姚嘉瑜, 张立武, 赵捷, 徐益, 祁建民, 张列梅. 黄麻全基因组SSR鉴定与特征分析. 作物学报, 2019, 45: 14–21. Yao J Y, Zhang L W, Zhao J, Xu Y, Qi J M, Zhang L M. Evaluation and characteristic analysis of SSRs from the whole genome of jute ()., 2019, 45: 14–21 (in Chinese with English abstract).
[14] 张彬, 李金秀, 王震, 冯浩, 李金榜. 小麦主要农艺性状的相关性及聚类分析. 作物杂志, 2018, (3): 57–60. Zhang B, Li J X, Wang Z, Feng H, Li J B. Correlation and cluster analysis of agronomic traits in wheat lines., 2018, (3): 57–60 (in Chinese with English abstract).
[15] 殷冬梅, 李拴柱, 崔党群. 花生主要农艺性状的相关性及聚类分析. 中国油料作物学报, 2010, 32: 212–216. Yin D M, Li S Z, Cui D Q. Agronomic character and cluster analysis of peanut cultivar., 2010, 32: 212–216 (in Chinese with English abstract).
[16] Ghosh R K, Sreewongchai T, Nakasathien S, Phumichai C. Phenotypic variation and the relationships among jute (species) genotypes using morpho-agronomic traits and multivariate analysis., 2013, 7: 830.
[17] Zhang L W, Yuan M H, Tao A F, Xu J T, Lin L H, Fang P P, Qi J M. Genetic structure and relationship analysis of an association population in jute (spp.) evaluated by SSR markers., 2015, 10: e0128195.
[18] 张立武, 袁民航, 何雄威, 刘星, 方平平, 林荔辉, 陶爱芬, 徐建堂, 祁建民. GenBank数据库中黄麻EST-SSR标记的开发及其通用性评价. 作物学报, 2014, 40: 1213–1219. Zhang L W, Yuan M H, He X W, Liu X, Fang P P, Lin L H, Tao A F, Xu J T, Qi J M. Development and universality evaluation of EST-SSR markers from GenBank in jute., 2014, 40: 1213–1219 (in Chinese with English abstract).
[19] Zhang L L, Gao Z Z, Wan X B, Xu Y, Zhang L M, Tao A F, Fang P P, Qi J M, Zhang L W. Development of novel small indel markers in jute (spp.)., 2017, 10: 169–176.
[20] 粟建光. 黄麻种质资源描述规范和数据标准. 北京: 中国农业出版社, 2005. pp 7–27. Su J G. Descriptors and Data Standard for Jute (L. &L.). Beijing: China Agriculture Press, 2005. pp 7–27 (in Chinese).
[21] Kraakman A T, Wageningen A J, Niks R E, Stam P. Linkage disequilibrium mapping of yield and yield stability in modern spring barley cultivars., 2004, 168: 435–446.
[22] 杨洛淼, 孙健, 赵宏伟, 王敬国, 刘化龙, 邹德堂. 不同年份冷水胁迫下水稻抽穗期和产量性状的QTL分析. 中国农业科学, 2016, 49: 3489–3503. Yang L M, Sun J, Zhao H W, Wang J G, Liu H L, Zou D T. QTL analysis of heading date and yield traits inrice under cold water stress in different years., 2016, 49: 3489–3503 (in Chinese with English abstract).
[23] Karrakman A T, Nikes R E, Berg P M, Stam P, Eeuwijk F A. Linkage disequilibrium mapping of yield and yield stability in modern spring barley cultivars., 2004, 168: 435–446.
[24] 司二静, 张宇, 汪军成, 孟亚雄, 李葆春, 马小乐, 尚勋武, 王化俊. 大麦农艺性状与SSR标记的关联分析. 作物学报, 2015, 41: 1064–1072. Si R J, Zhang Y, Wang J C, Meng Y X, Li B C, Ma X L, Shang X W, Wang H J. Association analysis between SSR markers and agronomic traits in barley., 2015, 41: 1064–1072 (in Chinese with English abstract).
[25] Zhao J L, Wang H W, Zhang X C, Du X Y, Li A F, Kong L R. Association analysis of grain traits with SSR markers betweenand hexaploid wheat (L.)., 2015, 14: 1936–1948.
[26] Abbasi Z, Majidi M M, Arzani A, Rajabi A, Mashayekhi P, Bocianowski J. Association of SSR markers and morpho- physiological traits associated with salinity tolerance in sugar beet (L.)., 2015, 205: 785–797.
[27] Fan C, Xing Y, Mao H, Lu T, Han B, Xu C, Li X, Zhang Q. GS3, a major QTL for grain length and weight and minor QTL for grain width and thickness in rice, encodes a putative transmembrane protein., 2006, 112: 1164–1171.
[28] Yue B, Xue W Y, Luo L J, Xing Y Z. QTL analysis for flag leaf characteristics and their relationships with yield and yield traits in rice., 2006, 33: 824–832.
[29] 祁建民, 卢浩然, 郑云雨, 王英娇. 黄麻数量性状遗传关系分析. 作物学报, 1991, 17: 145–150. Qi J M, Lu H R, Zheng Y Y, Wang Y J. Analysis on the genetic relationship of quantitative characters in jute (L.)., 1991, 17: 145–150 (in Chinese with English abstract).
[30] 周金超, 杨鑫雷, 崔顺立, 侯名语, 陈焕英, 穆国俊, 刘立峰. 花生SSR标记与农艺性状的相关性. 作物学报, 2014, 40: 1197–1204. Zhou J C, Yang X L, Cui S L, Hou M Y, Chen H Y, Mu G J, Liu L F. Correlation between SSR markers and agronomic traits in peanut (L.)., 2014, 40: 1197–1204 (in Chinese with English abstract).
[31] 李丹阳, 刘凯歌, 卢济明, 杨鑫雷, 崔顺立, 穆国俊, 陈焕英, 刘立峰. 花生SSR标记与品质性状的相关分析. 分子植物育种, 2017, 15: 2695–2701.Li D Y, Liu K G, Lu J M, Yang X L, Cui S L, Mu G J, Chen H Y, Liu L F. Correlation analysis of SSR markers and quality traits in peanut (L.)., 2017, 15: 2695–2701 (in Chinese with English abstract).
Correlation between SSR markers and fiber yield related traits in jute (spp.)
ZHANG Li-Lan1,2,3,**, ZHANG Lie-Mei1,**, NIU Huan-Ying1, XU Yi1,2,3, LI Yu1,2,3, QI Jian-Min1,2, TAO Ai-Fen1,2, FANG Ping-Ping1,2, and ZHANG Li-Wu1,2,3,*
1Key Laboratory of Ministry of Education for Genetics, Breeding and Multiple Utilization of Crops / Fujian Key Laboratory for Crop Breeding by Design / College of Agriculture, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian, China;2Experiment Station of Ministry of Agriculture and Rural Affairs for Jute and Kenaf in Southeast China / Fujian Public Platform for Germplasm Resources of Bast Fiber Crops / Fujian International Science and Technology Cooperation Base for Genetics, Breeding and Multiple Utilization Development of Southern Economic Crops, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian, China;3Center for Genomics and Biotechnology, Haixia Institute of Science and Technology, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian, China
Simple sequence repeat (SSR) markers correlated to fiber yield traits would be a beneficial tool for molecular marker-assisted breeding in jute. In this study, 397 SSR markers were screened from 311 jute germplasms by the phenotype identification from 2016 to 2018 and 116 pairs of primers. The correlation analysis between SSR markers and fiber yield related traits by SPSS revealed that the range of the coefficient of variation related to 9 fiber traits was from 13.05% to 76.78%, indicating a comprehensive genetic variation. The correlation analysis of the agronomic traits showed that there were significantly correlations among these traits. The average correlation coefficient between branch height and nodes of main stem was the highest (= 0.931), followed by the correlation coefficients (= 0.781) of fresh bark weight per plant and fresh stem weight per plant and the coefficients (= 0.779) of dry bark weight and fresh bark weight per plant. Analysis of variance (ANOVA) showed that the traits of main stem nodes, number of branches, plant height and branch height were relatively stable in different years with the higher broad heritability capacity. Furthermore, the SSR markers associated with fiber yield related traits were identified by Pearson correlation method. Stepwise regression analysis among each trait associated SSR markers indicated that there were six markers associated significantly with fiber yield related traits, and phenotypic variation explained by each SSR marker varied from 3.9% to 22.5%. These results will accelerate the development of molecular design breeding in jute.
jute; fiber yield; agronomic traits; SSR; correlation; regression analysis
本研究由国家自然科学基金项目(31771369)和国家现代农业产业技术体系建设专项(CARS-19-E06)资助。
This study was supported by the National Natural Science Foundation of China (31771369) and the China Agriculture Research System (CARS-19-E06).
张立武, E-mail: lwzhang@fafu.edu.cn, zhang_liwu@hotmail.com
**同等贡献(Contributed equally to this work)
张力岚, E-mail: 1204549467@qq.com; 张列梅, E-mail: zhangliemei@126.com
2020-02-16;
2020-04-15;
2020-05-11.
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20200511.1342.004.html
10.3724/SP.J.1006.2020.04035
