广西原产和引种荔枝种质资源的遗传多样性分析及核心种质构建
2020-11-09李冬波徐宁秦献泉李鸿莉侯延杰邱宏业张树伟朱建华彭宏祥
李冬波 徐宁 秦献泉 李鸿莉 侯延杰 邱宏业 张树伟 朱建华 彭宏祥


摘要:【目的】利用SSR分子標记对广西荔枝种质资源进行遗传多样性分析并构建核心种质,为广西荔枝种质资源的保存、遗传改良及创新利用提供理论依据。【方法】以广西原产和引种的88份荔枝种质资源为研究对象,利用40对SSR引物对其进行遗传多样性和聚类分析,在此基础上按原始群体50.00%、25.00%和12.50%的取样比例,采用逐步聚类法优先选择性状优良材料的原则构建广西荔枝核心种质。【结果】40对SSR引物从88份荔枝种质资源中共扩增出282条清晰的条带,每对引物扩增条带数2~13条,平均7.05条,其中,多态性条带182条,每对引物扩增的多态性条带数1~13条,平均4.55条,每对引物扩增条带的多态性比率为14.29%~100.00%,平均64.54%。88份荔枝种质资源的遗传相似系数为0.83~0.96,说明其遗传多样性较低;在遗传相似系数0.86处,88份荔枝种质资源可分为七大类群,第Ⅰ~Ⅶ类群分别包含3、12、6、6、1、2和58份种质,其中第Ⅰ类群均为龙荔种质资源,第Ⅱ类群主要为早熟种质,第Ⅳ类群主要为早熟实生种质,表明荔枝种质间的亲缘关系与熟期存在一定的相关性。88份荔枝种质资源在40个SSR位点的平均观测等位基因数(Na)、有效等位基因数(Ne)、Shannon指数(I)和期望杂合度(He)分别为3.2750、2.1137、0.8092和0.5107。核心种质-1、核心种质-2和核心种质-3的种质资源数量分别为44、22和11份,其中,核心种质-2的Ne、I和He 3个遗传多样性参数均最高,分别为2.1774、0.8452和0.5271;仅核心种质-3的Na与原始群体存在极显著差异(P<0.01),3组核心种质的其余遗传多样性参数与原始群体无显著差异(P>0.05),表明核心种质-1和核心种质-2均能充分代表原始群体的遗传多样性,根据核心种质构建的原则,最终选择核心种质-2作为广西荔枝核心种质。【结论】SSR分子标记是一种适用于荔枝资源遗传多样性分析和核心种质构建的理想分子标记。广西荔枝种质资源遗传背景相对较窄,遗传多样性较低,今后应加强荔枝种质资源的引进与利用。
关键词: 荔枝;SSR分子标记;遗传多样性;遗传相似系数;核心种质;聚类分析
中图分类号: S667.102.4 文献标志码: A 文章编号:2095-1191(2020)07-1537-08
Abstract:【Objective】In order to provide a theoretical basis for the preservation,genetic improvement and innovative utilization of litchi(Litchi chinensis Sonn.),the SSR molecular markers were used to analyze the genetic diversity and construction of core collections of litchi in Guangxi. 【Method】In this study, 88 litchi germplasm resources originated and introduced in Guangxi were used as materials and 40 pairs of SSR primer combinations were used to study the genetic diversity and cluster analysis. On the basis of this, the stepwise clustering and traits preferred sampling strategy were used to construct core collections of litchi in Guangxi according to the sampling rates 50.00%, 25.00% and 12.50% of the original germplasm resources. 【Result】The results showed that, a total of 282 clear bands were amplified from 88 litchi germplasm resources by 40 pairs of SSR primers and the bands per pair of primers ranged from 2 to 13 with the mean value of 7.05, and among them 182 bands were polymorphism. The number of polymorphic bands amplified by each pair of pri-mers was 1-13, with the mean value of 4.55. The polymorphism rate of amplified bands per pair of primers was 14.29%-100.00%, with the mean value of 64.54%. Genetic similarities among all germplasm resources ranged from 0.83 to 0.96, which indicated a relatively low genetic diversity.The result of system cluster analysis showed that 88 litchi germplasm resources were divided into 7 groups at 0.86 of the similarities. Groups I to VII contained 3, 12, 6, 6, 1, 2 and 58 germplasm resources, respectively. Germplasm resources in group I were all Dimocarpus confinis resources, germplasm resources in group II were mainly early maturing resources, germplasm resources in group IV were mainly early maturing seedlings. The clustering results showed that there was a certain correlation between the genetic relationship and maturity of litchi.The average observed number of alleles (Na), the average effective number of alleles(Ne), the average Shannons information index(I) and the expected heterozygosity(He) were 3.2750, 2.1137, 0.8092 and 0.5107, respectively. The number of germplasm resources in core collection-1, core collection-2 and core collection-3 were 44, 22 and 11 respectively. The genetic diversity parameters Ne, I, and He of core collection-2 were the highest, which were 2.1774, 0.8452 and 0.5271, respectively. Among all the genetic parameters, only Na of core collection-3 was very significantly different from that of original germplasm resources(P<0.01), and other genetic diversity parameters of the core collections had no significant difference(P>0.05), indicating that both core collection-1 and core collection-2 could stand for the genetic diversity of original germplasm resources. According to the principle of core collection construction, core collection-2 was determined to be the final core collection of litchi in Guangxi. 【Conclusion】SSR molecular markers are ideal molecular markers suitable for analysis of genetic diversity and construction of litchi core collections. The litchi germplasm resources in Guangxi have relatively narrow genetic background and low genetic diversity. Therefore, the introduction and utilization of litchi germplasm resources in Guangxi should be strengthened in the future.
Key words: litchi; SSR molecular markers; genetic diversity; genetic similarity index; core collection; cluster analysis
Foundation item: China Litchi and Longon Industry Technology Research System Construction Program(CARS-33-03); Guangxi Natural Science Foundation(2016GXNSFAA380128);Science and Technology Development Project of Guangxi Academy of Agricultural Sciences(Guinongke 2015YT50; Guinongke 2016ZX06)
0 引言
【研究意义】荔枝(Litchi chinensis Sonn.)为无患子科(Sapindaceae)荔枝属(Litchi)植物,是南亚热带地区重要的水果(董晨等,2019)。广西作为我国第二大荔枝主产区,种质资源丰富,在博白县和浦北县交界的六万大山有大面积的野生荔枝。20世纪80年代吴仁山(1986)编写的《广西荔枝志》记载有荔枝品种(单株)64个。随后通过资源调查筛选到一大批具有優良性状的荔枝种质资源,并选育出钦州红荔、贵妃红、草莓荔、桂荔1号、桂荔2号等品种(朱建华等,2005;李鸿莉等,2017;徐宁等,2017)。广西拥有丰富的荔枝种质资源,可为作物育种和遗传研究提供广阔的遗传基础,但同时给种质资源的保存利用及相关研究带来很大困难。因此,利用分子标记技术开展广西荔枝种质资源遗传多样性分析并构建核心种质,对广西荔枝种质资源的保存、遗传改良及创新利用具有重要意义。【前人研究进展】核心种质是指采用一定方法从整个种质资源中选择一部分材料,以最小的资源数量和遗传重复性,可最大程度地代表整个遗传资源的多样性,从而便于种质保存、评价及利用。目前,构建核心种质主要基于形态标记和分子标记两类,其中,形态标记方法简便,可操作性强,但易受环境影响,误差大;分子标记方法具有遗传信息量大、效率高且不受环境影响等特点,逐渐成为构建核心种质的主要方法(艾叶等,2019)。用于构建核心种质的分子标记主要有SSR、AFLP和RAPD等,其中SSR分子标记具有操作简便、成本低廉的特点,广泛应用于作物遗传多样性分析及核心种质构建(缪黎明等,2016)。至今,利用SSR分子标记构建的果树核心种质主要有桃(李银霞等,2007)、苹果(张春雨等,2009)、葡萄(郭大龙等,2012)和刺梨等(鲁敏等,2017),而基于SSR分子标记进行荔枝遗传多样性的研究也有较多报道。Viruel and Hormaza(2004)开发出12个荔枝SSR分子标记,并用于对21份来自中国、越南、印度、美国、澳大利亚和泰国的荔枝种质资源和4份来自泰国的龙眼种质进行遗传多样性分析,结果表明12个SSR分子标记可将21份荔枝种质资源分为两大类,且从龙眼中扩增出多态性片段,说明SSR分子标记在荔枝及其近缘种的鉴定、遗传多样性分析和种质保存中具有重要的应用价值。向旭等(2010)利用30个EST-SSR分子标记对96份来自广东、广西、福建、海南和云南等地的荔枝种质资源进行遗传多样性分析,结果将这些荔枝种质资源分成八大类群,聚类结果与种质生态类型和植物学性状特征基本相符。刘冰浩(2012)利用10个SSR分子标记对广西荔枝种质资源(32份来自博白江宁的野生荔枝种质、46份来自钦州贵台的半野生荔枝种质及9个栽培品种)进行遗传多样性分析,结果表明,半野生荔枝种质遗传多样性较野生荔枝种质丰富,栽培品种与野生荔枝种质的遗传距离最远,而半野生荔枝种质在遗传物质上更接近野生荔枝种质。李焕苓等(2018)用SSR和InDel分子标记对232份海南荔枝种质资源进行遗传多样性分析,结果表明大多数来自海南的荔枝种质聚在一起,说明海南荔枝种质资源间亲缘关系较近,遗传基础相对较窄。【本研究切入点】广西的荔枝种质资源较丰富,虽然已有针对部分种质资源进行遗传多样性分析的研究报道,但未见对广西荔枝资源进行全面收集、系统评价及遗传多样性分析,并构建核心种质的研究报道。【拟解决的关键问题】利用SSR分子标记对88份荔枝种质资源进行遗传多样性和聚类分析,采用逐步聚类优先法构建荔枝核心种质,为广西荔枝种质的保存、遗传改良及创新利用提供理论依据。
1 材料与方法
1. 1 试验材料
供试的88份荔枝种质资源及其原产地见表1,其中,原产于我国广西58份、广东10份、福建5份、海南4份、云南2份、台湾1份、泰国1份、越南1份、美国3份,另外3份为产于广西的荔枝近缘种龙荔(编号71、72和73),均取自广西农业科学院园艺研究所的荔枝种质圃。
主要试剂:Taq DNA聚合酶、dNTPs和DL2000 DNA Marker购自宝生物工程(大连)有限公司;琼脂糖、丙烯酰胺、甲叉双丙烯酰胺、过硫酸铵、四甲基乙二胺、氢氧化钠、甲醛和硝酸银购自南宁恒因生物科技有限公司。所有化学试剂均为分析纯。
主要仪器设备:PCR仪(Biometra,德国)、紫外分光光度计(Thermo Fisher,美国)、移液器(Eppendorf,德国)、离心机(Eppendorf,德国)和电泳仪(北京六一仪器厂)。
1. 2 样品采集
选取完全展开且无病虫害的嫩叶装入保鲜袋中,将其置于冰盒后迅速带回实验室,置于-20 ℃冰箱保存备用。
1. 3 基因组DNA提取
采用改进的CTAB法(李冬波等,2007)提取荔枝基因组DNA,用1.0%琼脂糖凝胶电泳检测DNA完整性,用紫外分光光度计测定DNA的纯度和浓度,用TE缓冲液稀释至20 ng/μL后放入-20 ℃冰箱保存备用。
1. 4 PCR扩增与电泳检测
SSR引物LMY3~LMY12和GLE1~GLE30(Vi-ruel and Hormaza,2004;孙清明等,2011)委托生工生物工程(上海)股份有限公司合成。PCR反应体系20.0 μL:20 ng/μL基因组DNA 2.5 μL,2.5 mmol/L dNTPs 0.5 μL,10×PCR缓冲液2.0 μL,10 μmol/L上、下游引物各0.5 μL,5 U/μL Taq DNA聚合酶0.2 μL,用ddH2O補足至20.0 μL。扩增程序:94 ℃预变性4 min;94 ℃ 1 min,50 ℃ 1 min,72 ℃ 2 min,进行35个循环;72 ℃延伸10 min,4 ℃保存。PCR产物用8%聚丙烯酰胺凝胶电泳检测(银染),最后数码相机拍照保存。
1. 5 统计分析
SSR是一种共显性分子标记,每个样品的SSR扩增谱带按“有”或“无”赋值“1”和“0”,将其转化为“1,0”矩阵。利用NTSYSpc 2.10进行遗传相似性分析。根据遗传相似系数,采用Dice估测遗传距离,再运用非加权算术平均配对法(UPGMA)进行聚类分析。基于聚类分析结果,按照原始群体50.00%、25.00%和12.50%的取样比例,采用多次聚类优先取样法进行核心种质构建。利用DataTrans 1.0将“1,0”矩阵转换为基因型数据,利用Popgene 1.32计算观测等位基因数(Na)、有效等位基因数(Ne)、Shannon信息指数(I)和期望杂合度(He)等遗传多样性参数,用SPSS 13.0的 t 检测对所构建核心种质与原始群体间差异进行显著性分析。
2 结果与分析
2. 1 SSR分子标记多态性分析结果
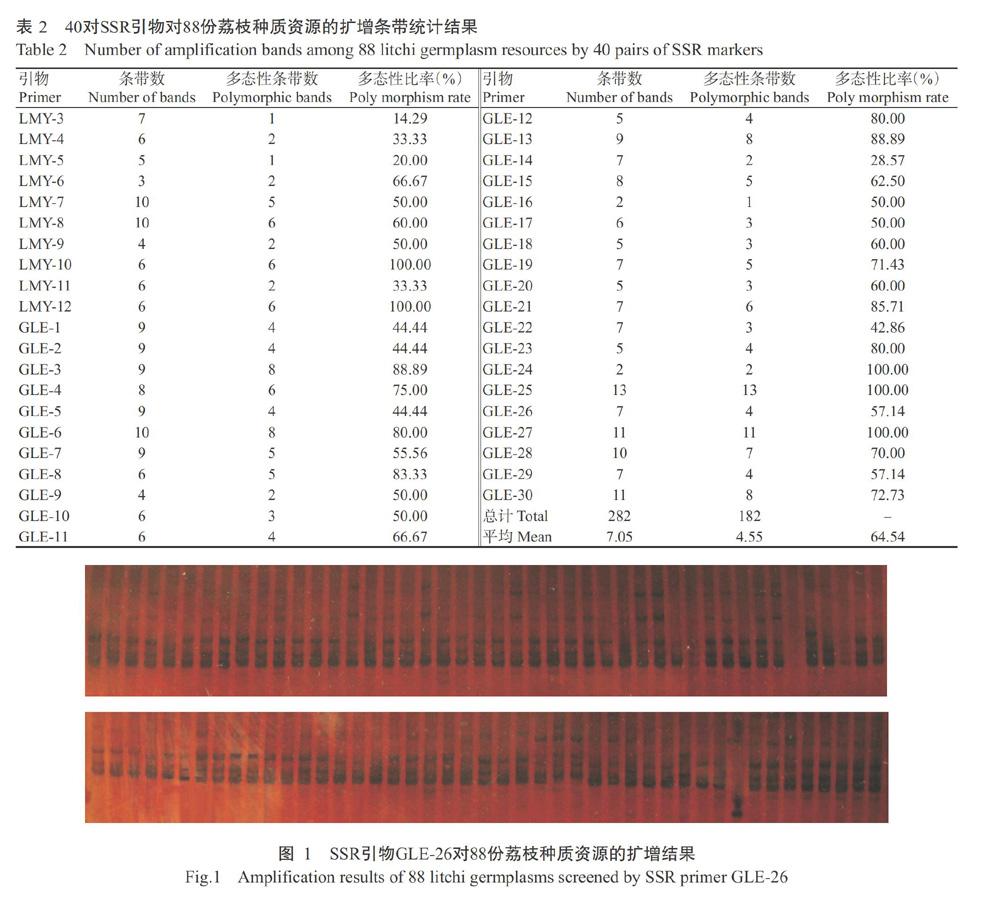
由表2和表3可看出,40对SSR引物从88份荔枝种质资源中共扩增出282条清晰的条带,每对引物扩增条带数2~13条,平均7.05条,其中,多态性条带182条,每对引物扩增的多态性条带数1~13条,平均4.55条,每对引物扩增条带的多态性比率为14.29%~100.00%,平均64.54%。40对SSR引物从88份荔枝种质资源中均扩增出多态性条带,说明选用的SSR引物在荔枝中多态性较高,适合对广西荔枝资源进行遗传多样性分析。SSR引物GLE-26对88份荔枝种质资源的扩增结果如图1所示。
2. 2 广西荔枝种质资源聚类分析结果
由图2可知,88份荔枝种质资源的遗传相似系数为0.83~0.96,说明其遗传多样性较低,其中,3个荔枝近缘种龙荔单株与其他材料遗传相似系数最小,为0.83;实生-4(50)与红灯笼(52)间的遗传相似系数最大,达0.96;在遗传相似系数0.86处,88份荔枝种质资源可分为七大类群,其中第Ⅰ类群包含大菉2号(73)、小董1号(72)和龙虎山2号(71)共3份龙荔种质资源;第Ⅱ类群包含楠西早熟(75)、BC(70)、火烧荔(83)、台妃子(65)、水荔(18)、灵山四月半(30)、桂早荔(26)、立夏红(29)、水东黑叶(15)、兰竹(55)、四月红(13)和三月红(12)共12份种质资源,主要为早熟种质;第Ⅲ类群包含陈紫(5)、99-9(41)、99-6(38)、陀提(6)、99-8(40)和妃子笑(4)共6份种质资源;第Ⅳ类群为普宁当地(78)、德宝早熟(74)、大新3号(77)、靖西1号(76)、褐毛长叶(64)和褐毛大叶(63)共6份种质资源,主要为早熟实生种质;第Ⅴ类群包含江宁野生荔(80)1份种质资源;第Ⅵ类群包含99-7(39)和99-5(37)2份资源;其余58份资源为第Ⅶ类群。综上所述,荔枝的亲缘关系与熟期存在一定的相关性。
2. 3 广西荔枝核心种质构建结果
根据荔枝种质资源聚类分析结果,对88份荔枝种质资源进行分组,采用逐步聚类法优先选择性状优良材料的原则按原始群体50.00%、25.00%和12.50%的比例分别构建3个候选核心种质:核心种质-1(44份)、核心种质-2(22份)和核心种质-3(11份)。其中,核心种质-2中的22份荔枝种质资源聚类分析结果如图3所示。在遗传相似系数0.87处22份荔枝种质资源聚成11组,采用优先选取性状优良材料的原则,在各组内选取四两果(2)、江宁野生荔(80)、草莓荔(24)、陀提(6)、灵山香荔(7)、锦钟(16)、鹅蛋荔(62)、靖西1号(76)、妃子笑(4)、桂早荔(26)和龙虎山2号(71)共11份荔枝种质资源作为核心种质-3的入选材料。
2. 4 广西荔枝核心种质的遗传多样性评价
由表3可看出,88份荔枝种质资源在40个SSR分子标记位点的Na、Ne、I和He平均值分别为3.2750、2.1137、0.8092和0.5107;核心种质-1、核心种质-2和核心种质-3的种质资源数量分别为44、22和11份,平均每个位点Na分别是原始群体的96.95%、93.89%和84.73%,其中,核心种质-2的Ne、I和He 3个遗传多样性参数均最高,分别为2.1774、0.8452和0.5271。对原始群体和3组核心种质的遗传多样性参数进行 t 检测,结果显示,仅核心种质-3的Na与原始群体存在极显著差异(P<0.01),3组核心种质的其余遗传多样性参数与原始群体无显著差异(P>0.05),表明按照原始群体的50.00%和25.00%取样比例构建的核心种质-1和核心种质-2均能充分代表原始群体的遗传多样性。根据核心种质构建原则,最终选择核心种质-2作为广西荔枝核心种质。
3 讨论
目前,有关广西荔枝种质资源的遗传多样分析研究已有报道。彭宏祥等(2006)利用7个AFLP分子标记对27份广西荔枝优稀种质资源进行遗传多样分析,结果显示扩增条带的多态性比例19.51%~45.12%,27份种质可聚为七大类群,遗传相似系数为0.26~0.86。沈庆庆等(2013)利用10个ISSR分子标记将83份桂西南早熟荔枝实生资源聚为三大类群,遗传相似系数为0.64~0.95。本研究利用40个SSR标记对广西荔枝种质资源圃中具有代表性的种质资源进行遗传多样性分析,结果表明,88份荔枝种质资源可分为七大类群,遗传相似系数范围(0.83~0.96)低于向旭等(2010)对96份来自国家种质圃荔枝资源的遗传相似系数范围(0.35~1.00),说明广西荔枝种质资源遗传背景相对较窄,今后应加强荔枝种质资源的引进与利用。
根据前期对荔枝种质资源的性状和来源地调查,结合本研究荔枝种质聚类分析结果可看出,第Ⅰ类群中的3份材料为龙荔种质资源,与其余荔枝资源间亲缘关系较远,第Ⅱ类群中的12份材料主要为早熟品种,第Ⅳ类群中的6份材料主要为早熟实生资源,说明种质间亲缘关系与熟期存在一定的相关性,与向旭等(2010)的研究结论相似。此外,来自云南的褐毛长叶(64)和褐毛大叶(63)与桂西南的早熟荔枝[普宁当地(78)、德宝早熟(74)、大新3号(77)和靖西1号(76)]聚在一起,說明地理位置相近的种质亲缘关系较近,但来自广东、福建、海南等省及国外的荔枝未全部表现出这一聚类趋势,其原因可能是世界各地频繁的引种交流导致种质间遗传背景非常复杂,地理位置相近的种质资源并非总是聚在一起。
构建核心种质的目的是以最少的种质资源数量和遗传重复最大程度地代表原始种质的遗传多样性,取样策略和取样比例是核心种质构建的关键。目前常用取样策略有多次聚类随机取样法、优先取样法和偏离度取样法等3种。其中,优先取样法构建的核心种质更具有代表性,被广泛使用(缪黎明等,2016),主要包括位点优先和性状优先,前者优先选择具有稀有等位变异和等位变异数较多的材料,后者优先选择性状优良的材料(卜海东等,2012;常利芳等,2018)。本研究采用多次聚类性状优先取样法,先利用SSR分子标记对其进行聚类分组,在选取组内遗传距离较近的材料时,结合前期的形态观察优先选择性状优良的材料,在保证核心种质能将原始种质资源的遗传信息较完整保留下来的同时,入选核心种质的材料性状更加突出,据此构建的广西荔枝核心种质具有更好的实用性。本研究按原始群体50.00%、25.00%和12.50%的取样比例构建核心种质-1、核心种质-2和核心种质-3等3个群体,分别保留了原始群体Na的96.95%、93.89%和84.73%,符合核心种质代表原始群体70%~80%以上遗传多样性的标准(李自超等,1999)。通过分析3组核心种质的遗传多样性参数,发现核心种质-2的Ne、I和He 3个遗传多样性参数均最高,且仅核心种质-3的Na与原始群体存在极显著差异,3组核心种质的其余遗传多样性参数与原始群体无显著差异,表明按照原始群体的50.00%和25.00%取样比例构建的核心种质-1和核心种质-2均能充分代表原始群体的遗传多样性,而核心种质-3较核心种质-2减少资源数量的同时,降低了原始群体的遗传多样性,因此核心种质-2更适合作为广西荔枝核心种质。但核心种质的构建是一个动态的过程,今后应深入调查和评价荔枝种质资源,不断补充和完善广西荔枝核心种质。
4 结论
SSR分子标记是一种适用于荔枝资源遗传多样性分析和核心种质构建的理想分子标记。广西荔枝种质资源遗传背景相对较窄,遗传多样性较低,今后应加强荔枝种质资源的引进与利用。
参考文献:
艾叶,陈璐,谢泰祥,陈娟,兰思仁,彭东辉. 2019. 基于SSR荧光标记构建建兰品种核心种质[J]. 园艺学报,46(10):1999-2008. [Ai Y,Chen L,Xie T X,Chen J,Lan S R,Peng D H. 2019. Construction of core collection of Cymbidium ensifolium cultivars based on SSR fluorescent markers[J]. Acta Horticulturae Sinica,46(10):1999-2008.]
卜海东,张冰冰,宋洪伟,梁英海,刘延杰,程显敏,顾广军,刘畅. 2012. 利用SSR结合表型性状构建寒地梨资源核心种质[J]. 园艺学报,39(11):2113-2123. [Bu H D,Zhang B B,Song H W,Liang Y H,Liu Y J,Cheng X M,Gu G J,Liu C. 2012. Construction core collections of pear germplasms in cold region by SSR and phenotypic traits[J]. Acta Horticulturae Sinica,39(11):2113-2123.]
常利芳,白建荣,李锐,张丛卓,张效梅,杨瑞娟. 2018. 基于SSR标记构建甜玉米群体的核心种质[J]. 玉米科学,26(3):40-49. [Chang L F,Bai J R,Li R,Zhang C Z,Zhang X M,Yang R J. 2018. Construction of a core co-llection of sweet corn populations based on SSR markers[J]. Journal of Maize Sciences,26(3):40-49.]
董晨,魏永赞,王弋,郑雪文,李伟才. 2019. 荔枝(Litchi chinesis Sonn.)果实酸性转化酶LcSAI生物信息学和表达分析[J/OL]. 广西植物. http://kns.cnki.net/kcms/detail/45.1134.Q.20191207.0858.004.html. [Dong C,Wei Y Z,Wang Y,Zheng X W,Li W C. 2019. Bioinformatics and expression analysis of acid invertase LcSAI of Litchi chinesis Sonn.[J/OL]. Guihaia. http://kns.cnki.net/kcms/detail/45.1134.Q.20191207.0858.004.html.]
郭大龙,刘崇怀,张君玉,张国海. 2012. 葡萄核心种质的构建[J]. 中国农业科学,45(6):1135-1143. [Guo D L,Liu C H,Zhang J Y,Zhang G H. 2012. Construction of grape core collections[J]. Scientia Agricultura Sinica,45(6):1135-1143.]
李冬波,李江舟,朱建华. 2007. 适宜AFLP分析的龙眼基因组DNA提取方法[J]. 西南农业学报,20(3):478-480. [Li D B,Li J Z,Zhu J H. 2007. A method for genomic DNA preparation of longan suitable to AFLP analysis[J]. Southwest China Journal of Agricultural Sciences,20(3):478-480.]
李鸿莉,彭宏祥,朱建华,秦献泉,徐宁,陆贵锋,李冬波,黄凤珠. 2017. 优质荔枝新品种‘桂荔1号的选育[J]. 果树学报,34(1):125-128. [Li H L,Peng H X,Zhu J H,Qin X Q,Xu N,Lu G F,Li D B,Huang F Z. 2017. Breeding of new high quality litchi cultivar ‘Guili No.1[J]. Journal of Fruit Science,34(1):125-128.]
李焕苓,田婉莹,孙进华,张新春,张蕾,王果,王树军,王家保. 2018. 基于SSR和InDel标记的海南荔枝种质资源遗传多样性分析[J]. 分子植物育种,16(4):1343-1356. [Li H L,Tian W Y,Sun J H,Zhang X C,Zhang L,Wang G,Wang S J,Wang J B. 2018. Analysis of genetic diversity on litchi(Litchi chinensis Sonn.) germplasm resources from Hainan by SSR and InDel markers[J]. Molecular Plant Breeding,16(4):1343-1356.]
李银霞,安丽君,姜全,赵剑波,李天红. 2007. 桃(Prunus persica(L.) Batsch.)品种核心种质的构建与评价[J]. 中国农业大学学报,12(5):22-28. [Li Y X,An L J,Jiang Q,Zhao J B,Li T H. 2007. Establishment and evaluation of the core collection of peach(Prunus persica(L.) Batsch.) cultivars[J]. Journal of China Agricultural University,12(5):22-28.]
李自超,張洪亮,孙传清,王象坤. 1999. 植物遗传资源核心种质研究现状与展望[J]. 中国农业大学学报,4(5):51-62. [Li Z C,Zhang H L,Sun C Q,Wang X K. 1999. Status and prospect of core collection in plant germplasm resource[J]. Journal of China Agricultural University,4(5):51-62.]
刘冰浩. 2008. 广西荔枝野生、半野生资源及栽培品种亲缘关系的研究[D]. 南宁:广西大学. [Liu B H. 2008. Genetic relationship of litchi resources from grow wild,semi-wild and cultivated genotypes in Guangxi Province of China[D]. Nanning:Guangxi University.]
鲁敏,鄢秀芹,白静,张怀山,王道平,安华明. 2017. 基于 EST-SSR标记与果实品质性状的贵州野生刺梨核心种质构建[J]. 园艺学报,44(8):1486-1495. [Lu M,Yan X Q,Bai J,Zhang H S,Wang D P,An H M. 2017. Construction of core collection in wild Rosa roxburghii from Guizhou Province using EST-SSR markers and fruits quality traits[J]. Acta Horticulturae Sinica,44(8):1486-1495.]
缪黎明,王神云,邹明华,李建斌,孔李俊,余小林. 2016. 园艺作物核心种质构建的研究进展[J]. 植物遗传资源学报,17(5):791-800. [Miao L M,Wang S Y,Zou M H,Li J B,Kong L J,Yu X L. 2016. Review of the studies on core collection for horticultural crops[J]. Journal of Plant Genetic Resources,17(5):791-800.]
彭宏祥,曹辉庆,朱建华,廖惠红,黄凤珠,李江舟,梁文. 2006. 利用AFLP分子标记对广西荔枝优稀种质遗传多样性及分类研究[J]. 西南农业学报,19(1):108-111. [Peng H X,Cao H Q,Zhu J H,Liao H H,Huang F Z,Li J Z,Liang W. 2006. Studies on the application of AFLP molecular markers on genetic diversity and classification of good and rare litchi resources in Guangxi[J]. Southwest China Journal of Agricultural Sciences,19(1):108-111.]
沈庆庆,朱建华,彭宏祥,何新华. 2013. 桂西南早熟荔枝实生资源遗传多样性的ISSR分析[J]. 广西植物,33(2):225-228. [Shen Q Q,Zhu J H,Peng H X,He X H. 2013. Genetic diversity of early-maturing seedling litchi resources in southwest Guangxi by ISSR[J]. Guihaia,33(2):225-228.]
孙清明,马文朝,马帅鹏,赵俊生,白丽军,陈洁珍,蔡长河,向旭,欧良喜. 2011. 荔枝EST资源的SSR信息分析及EST-SSR标记开发[J]. 中国农业科学,44(19):4037-4049. [Sun Q M,Ma W C,Ma S P,Zhao J S,Bai L J,Chen J Z,Cai C H,Xiang X,Ou L X. 2011. Characteristics of SSRs derived from ESTs and development of EST-SSR markers in litchi(Litchi chinensis Sonn.)[J]. Scientia Agri-cultura Sinica,44(19):4037-4049.]
吴仁山. 1986. 广西荔枝志[M]. 广州:广东科技出版社:10-45. [Wu R S. 1986. Guangxi litchi annals[M]. Guangzhou:Guangdong Science and Technology Press:10-45.]
向旭,欧良喜,陈厚彬,孙清明,陈洁珍,蔡长河,白丽军,赵俊生. 2010. 中国96个荔枝种质资源的EST-SSR遗传多样性分析[J]. 基因组学与应用生物学,29(6):1082-1092. [Xiang X,Ou L X,Chen H B,Sun Q M,Chen J Z,Cai C H,Bai L J,Zhao J S. 2010. EST-SSR analysis of gene-tic diversity in 96 litchi(Litchi chinensis Sonn.) germplasm resources in China[J]. Genomics and Applied Bio-logy,29(6):1082-1092.]
徐寧,曾世江,朱建华,潘俭,黄济良,秦献泉,彭宏祥,梁宗活,蒙春玲,陈铁,杨杰,谭识良,劳绍贞,谭征,李冬波,陆贵锋,李鸿莉,黄凤珠. 2017. 荔枝新品种‘桂荔2号[J]. 园艺学报,44(5):1011-1012. [Xu N,Zeng S J,Zhu J H,Pan J,Huang J L,Qin X Q, Peng H X,Liang Z H,Meng C L,Chen T,Yang J,Tan S L,Lao S Z,Tan Z,Li D B,Lu G F,Li H L,Huang F Z. 2017. A new litchi cultivar ‘Guili 2[J]. Acta Horticulturae Sinica,44(5):1011-1012.]
张春雨,陈学森,张艳敏,苑兆和,刘遵春,王延龄,林群. 2009. 采用分子标记构建新疆野苹果核心种质的方法[J]. 中国农业科学,42(2):597-604. [Zhang C Y,Chen X S,Zhang Y M,Yuan Z H,Liu Z C,Wang Y L,Lin Q. 2009. A method for constructing core collection of Malus sieversii using molecular markers[J]. Scientia Agricultura Sinica,42(2):597-604.]
朱建华,彭宏祥,潘介春,李江舟. 2005. 广西荔枝种质资源研究及品种选育[J]. 福建果树,(3):11-13. [Zhu J H,Peng H X,Pan J C,Li J Z. 2005. Study on germplasm resour-ces and varieties selection of litchi in Guangxi[J]. Fujian Fruits,(3):11-13.]
Viruel M A,Hormaza J I. 2004. Development,characterization and variability analysis of microsatellites in lychee(Litchi chinensis Sonn,Sapindaceae)[J]. Theoretical and Applied Genetics,108(5):896-902.
(责任编辑 陈 燕)
