鳌肢动物Wnt基因家族的全基因组与进化分析
2020-11-02梁园,周雁,2*
梁 园,周 雁,2*
1.复旦大学生命科学学院遗传工程国家重点实验室,上海 200438
2.中国国家人类基因组中心上海市疾病与健康基因组学重点实验室,上海 201203
Wnt基因编码了一类分泌性的糖蛋白配体,长度通常在350 到400 个氨基酸之间,以具有23~24个保守的半胱氨酸为特征[1-4]。Wnt分子激发的信号通路参与了多种生物过程的调节,在生物体的发育过程中起着至关重要的作用,包括细胞形态的分化、体细胞形态和功能稳态的维持、胚轴形成,组织的再生和免疫应答调节[5,6]。
Wnt基因家族的起源非常古老,且具有多样性。利用已有的基因组信息对Wnt基因家族的系统发育分析结果显示,在刺胞动物和两侧对称动物的共同祖先中可能存在13 个Wnt亚家族[7,8]。其中,在腔肠动物门的海葵(Nematostella vectensis)中发现了12 个Wnt 亚家族[9]。此外,在哺乳动物中还发现了Wnt基因的扩增现象,例如在人类的基因组中共鉴定出了19 个Wnt基因,包括了12 个Wnt亚家族[10]。然而,在节肢动物中,Wnt基因的数量存在着物种间的差异。在昆虫类的果蝇和蜜蜂的基因组中分别发现了7 个Wnt基因亚家族[11],然而在甲壳纲动物蚤状溞(Daphnia pulex)中则鉴定出了12 个Wnt基因亚家族[12]。另外,根据Wnt基因在基因组上分布的位置关系,研究者发现Wnt基因在基因组上存在一定程度的连锁分布现象,形成了具有共线性的基因簇。在刺胞动物和两侧对称动物的共同祖先中可能有2 个具有共线性的Wnt基因簇,其中一个基因簇由Wnt9,-1,-6 和-10连锁分布形成,另一个由Wnt5和Wnt7组成的另一个基因簇,但目前尚不清楚在节肢动物中是否仍然存在这一基因簇[7,11,13]。
迄今为止,在节肢动物门的螯肢动物中,仍然缺乏关于Wnt基因的全基因组的分析研究。不过,近期发表的拼接质量较高的螯肢动物的基因组数据为全面研究螯肢动物中的Wnt基因的特征提供了资源。在本研究中,我们采用生物信息学方法对螯肢动物的Wnt基因序列进行了鉴定和分析,并进一步比较了其数量、结构、基因组分布和系统发育关系,以期为Wnt基因家族在节肢动物乃至后生动物中的和进化过程和功能的进一步研究提供有力的基础。
1 材料与方法
1.1 螯肢动物基因组序列获取
我们收集了公开数据库中的7 种螯肢动物的基因组和1 种的转录组,包括蝎子类(Centurouroides sculpturatus),蜘蛛类(Parasteatoda tepidariorum),蜱螨类(Tetranychus urticae,Dinothrombium tinctorium,Ixodes scapularis),鲎类(Limulus polyphemus,Tachypleus tridentatus)和海蜘蛛类(Pycnogonumsp.)。所收集的7 种螯肢动物的基因组都拼接到了scaffold 或染色体水平。海蜘蛛Pycnogonumsp.的原始RNA 测序数据从NCBI SRA 数据库下载得到,检索号为SRR8745912。
1.2 螯肢动物Wnt 基因家族的核酸序列和氨基酸序列的获取
利用NCBI 数据库中代表性的节肢动物的Wnt基因的氨基酸序列为搜索序列,来搜索螯肢动物基因组和转录组中的的Wnt基因家族成员[11,14,15],首先使用BlastP 和TBlastN 算法[16]搜索螯肢动物预测基因的蛋白序列和基因组序列,e 值阈值选择10-5。然后将获得的序列与Pfam 数据库进行比较,检测是否存在Wnt 保守结构域。接下来使用PF00110 这一Wnt 保守结构域的隐马尔可夫文件,利用HMMER 程序包[17]的HMMsearch 程序在螯肢动物的预测基因中搜索是否存在其它Wnt基因。第三,使用Clustal W[18]对每个物种已搜索到的Wnt基因进行聚类,使用HMMbuild 程序构建物种特异的Wnt 隐马尔可夫文件,然后再一次用HMMsearch 对螯肢动物的预测基因进行搜索,以获得物种特异的Wnt基因家族候选序列。鉴定得到的Wnt 蛋白序列在SMART 中进行比对,以鉴定信号肽和其它保守结构。鉴定出的Wnt 蛋白结构域使用IBS 1.0.3 软件绘制[19]。
1.3 Wnt 蛋白多序列比对和系统发育分析
选择鉴定得到的2 种鲎、1 种蝎子、1 种蜘蛛、1 种螨虫、1 种蜱虫、1 种海蜘蛛,以及已知的1种蜜蜂、1 种甲虫、1 种蚜虫、1 种蜈蚣、1 种水蚤和1 种家鼠的Wnt基因用于进行系统发育分析。首先用Clustal W[18]的BLOSUM 打分矩阵进行多序列比对,然后使用PhyML 3.0[20]采用最大似然法构建系统发育树,使用SH-aLRT 方法对分支的支持度进行评估。
2 结果与分析
2.1 螯肢动物Wnt 基因家族的鉴定
我们在8 种螯肢动物中共鉴定得到87 个Wnt基因家族成员。其中,在中华鲎和美洲鲎中分别发现了15 个和18 个Wnt基因,在蝎子中发现了13 个Wnt基因,在蜘蛛中发现了12 个Wnt基因,在二斑叶螨、肩突硬蜱和天鹅绒螨虫中分别鉴定到了8、7、6 个Wnt基因,在海蜘蛛中发现了8 个Wnt基因。每个基因都在之后的系统发育分析中进一步进行了亚家族分类。在蝎子,蜘蛛和海蜘蛛中,鉴定到的Wnt基因的氨基酸序列均由300 至400 个氨基酸构成。在二斑叶螨、肩突硬蜱、和天鹅绒螨虫中,大多数鉴定得到的Wnt基因的氨基酸序列长度在300 至400 之间,除了在每个物种中分别鉴定出了各2 个相对较短的Wnt 氨基酸序列以及一个较长的Wnt16 序列。在中华鲎和美洲鲎的基因组中,Wnt 氨基酸序列的长度出现了更高的变异度,包含了两条较长的TtWnt1 和TtWnt5c 以及若干较短的Wnt 蛋白序列。
2.2 螯肢动物的Wnt 基因氨基酸序列的保守结构域分析
进一步对螯肢动物的Wnt基因的氨基酸序列特征和结构域进行分析,结果显示,在包括鲎、蝎子、蜘蛛和海蜘蛛在内的物种中的Wnt 氨基酸序列都含有高度保守的Wnt 结构域(见图1a),位于序列的C 末端附近,在这些Wnt 氨基酸序列的N 末端还发现了多种结构序列,包括长度为20~30个氨基酸的信号肽序列、跨膜区序列或低复杂度区域。然而在3 个蜱螨类物种中鉴定到的一部分Wnt基因的氨基酸序列的N 末端则缺少了这些结构域(见图1b)。此外,我们对在蝎子、蜘蛛和海蜘蛛中鉴定到的长度在300 至400 aa 之间的Wnt基因的氨基酸序列做了多序列比对,结果显示这些序列中存在21 个保守的半胱氨酸残基,呈分散分布于序列当中,这与在其它后生生物中鉴定到的Wnt基因家族的氨基酸序列的特征是一致的,这些半胱氨酸残基可能有助于这些Wnt 蛋白质的正确折叠。

图1 螯肢动物Wnt 基因结构Fig.1 The structure of Wnt gene in Chelicerata
2.3 螯肢动物的Wnt 基因在基因组上的连锁分布
为了进一步探索螯肢动物Wnt基因的进化现象,我们比较了其中6 个物种的Wnt基因在基因组上的分布(见图2)。结果发现除了美洲鲎以外,其它螯肢动物都保留了进化上保守的基因簇Wnt9-Wnt1-Wnt6-Wnt10中的一部分,各自伴随着谱系特异性重排或丢失现象。中华鲎基因组中的Wnt6和Wnt1基因位于其3 号染色体上,蝎子和蜘蛛基因组中的Wnt6和Wnt1基因分别位于各一条scaffold 上。此外,在蝎子的另一条scaffold 上还发现了Wnt9-Wnt6-Wnt1的连锁分布,在肩突硬蜱的基因组中的一条scaffold 上发现了相邻的Wnt9-Wnt1基因簇。有趣的是,我们在中华鲎、美洲鲎、蝎子和蜘蛛的基因组中还发现了Wnt5和Wnt7基因簇,分别位于中华鲎的2 条染色体上和美洲鲎的3 条scaffolds 上。除此之外,我们还发现了除了这两个基因簇以外的新的Wnt基因簇。在美洲鲎基因组中,有一串相邻分布的Wnt11-Wnt7基因簇,位于1 条scaffold 上。在蝎子中,发现了相邻分布的的Wnt2和Wnt16基因簇。在蜘蛛基因组中,Wnt4和Wnt11被发现相邻分布于1 条scaffold 上。
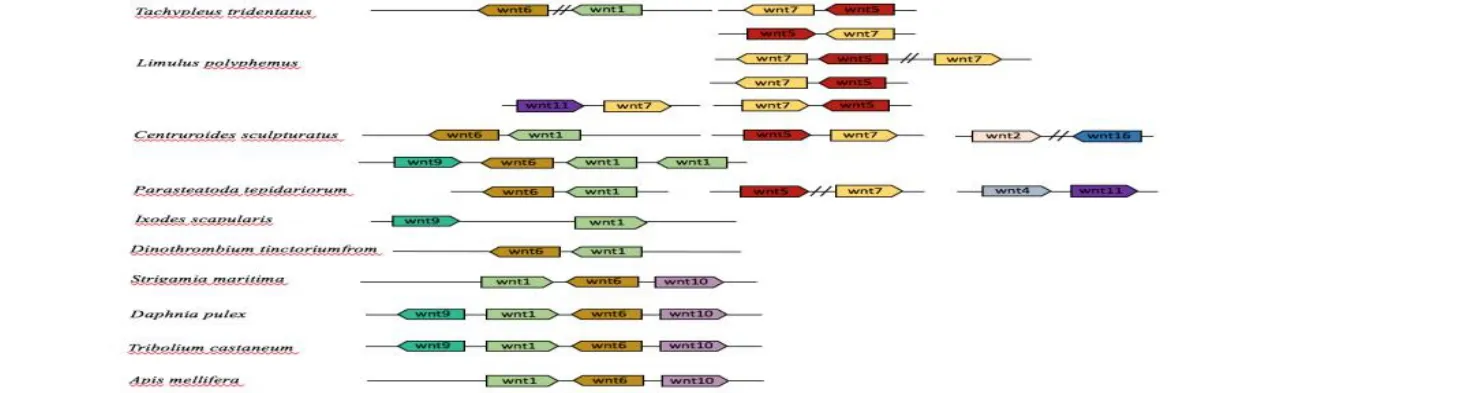
图2 螯肢动物Wnt 基因的连锁分布Fig.2 The conserved Wnt gene cluster in six chelicerates and four other arthropods
2.4 螯肢动物Wnt 基因的系统发育树构建及分析
为了进一步阐明螯肢动物的Wnt基因的系统发育关系,我们利用7 个螯肢动物以及其它节肢动物(包括蜈蚣、水蚤、蜜蜂、蚜虫、以及甲虫)的Wnt基因的氨基酸序列,采用最大似然法构建了Wnt基因家族的系统发生树,采用小鼠的Wnt3基因序列作为外类群。系统发育树显示,螯肢动物的Wnt家族可分为11 个不同的亚家族(见图3)。如在其它节肢动物中所发现的,这些螯肢动物的基因组发生了Wnt3亚家族的缺失。有趣的是,螯肢动物的基因组中还另外缺失了Wnt10亚家族。同时,我们仅在蝎子和肩突硬蜱中鉴定到了Wnt9亚家族序列。与其它类节肢动物基因组大多拥有单拷贝的Wnt基因所不同,螯肢动物的Wnt5,Wnt7和Wnt11亚家族基因表现出具有多拷贝的扩增现象(见图3),这可能暗示了在螯肢动物进化过程的早期出现了基因组的扩增现象。对于每个物种而言,在中华鲎、美洲鲎,蝎子和蜘蛛中分别鉴定出9、10、9、10 个Wnt亚家族。在海蜘蛛基因组中,鉴定到了8 个Wnt亚家族。值得注意的是,在螨虫和蜱虫中共鉴定出6 个和7个Wnt亚家族,与其它螯肢动物相比,表现出了Wnt2和Wnt7的丢失现象,这可能与其进化速率是有关的。此外,从系统发育树上,我们发现Wnt1和Wnt6亚家族作为姐妹类群聚成一支,且具有分数较高的的支持度,这表明这两个亚家族可能是由于串联重复进化而来的。我们还发现Wnt2,Wnt6,Wnt8和Wnt16亚家族的基因都分为了支持度较高的两个分支(螯肢动物分支和非螯肢动物分支),这表明这些基因亚家族在螯肢动物和其它节肢动物之间的进化距离更远。
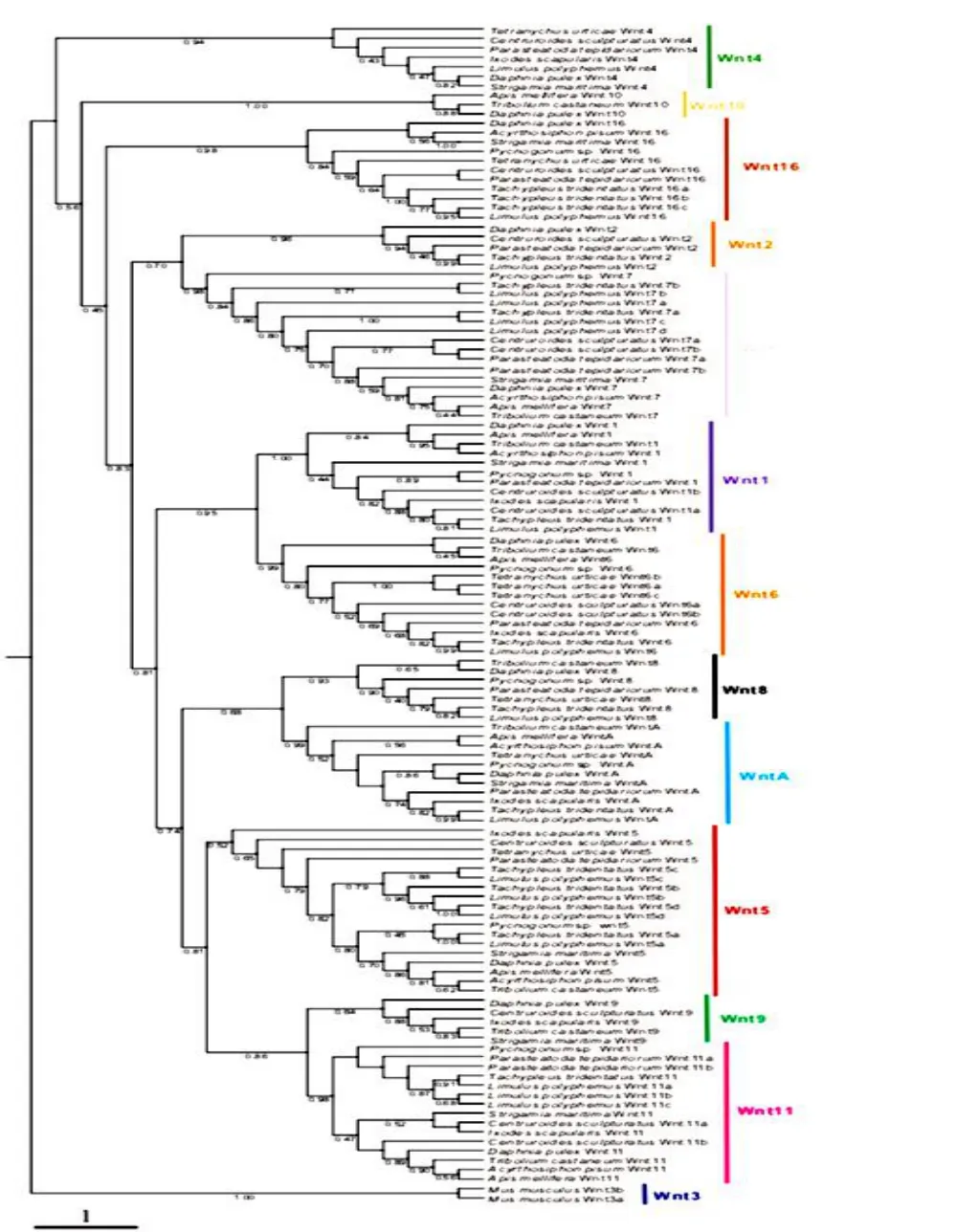
图3 Wnt 基因家族系统发生树Fig.3 The phylogenetic tree of the Wnt families
3 讨论
在本研究中,我们用生物信息学方法全面鉴定了8 种螯肢动物的Wnt基因家族,这可以为进一步理解Wnt基因在后生动物进化过程中的进化模式提供基础。尽管在基因组的测序和拼接过程中会有一定的错误,但是这8 个高深度、高覆盖度、高拼接水平的基因组和转录组资源为我们提供了从全基因组和转录组水平鉴定Wnt基因的条件。从结构分析结果来看,在蝎子、蜘蛛和海蜘蛛中,大多数Wnt 氨基酸序列都包含可识别的信号肽区域,而其它物种中的一些Wnt序列则显然缺乏该基序。考虑到信号肽的生物信息学预测算法仍是有限的,这些序列仍然有包含现有预测软件无法识别的信号肽的可能性,需要进一步的细胞生物学实验来确定这些缺乏信号肽的序列是否为非分泌型Wnt 蛋白。从基因组的分布情况来看,在蝎子中我们发现了2 个位于不同scaffold 上的Wnt6-Wnt1簇,同时分别在两种鲎的不同scaffold 和染色体上的发现了2 个和3 个Wnt5-Wnt7簇,这可能暗示了在鲎的祖先中曾发生了全基因组复制事件。从系统发育分析结果来看,螯肢动物的Wnt基因家族包括了11 个亚家族,丢失了Wnt3和Wnt10亚家族,这表明螯肢动物的共同祖先可能已缺失了这两个亚家族。其它亚家族的丢失是在螯肢动物的进化过程中发生的。此外,在螨虫和蜱虫中鉴定出了数量较少的6 个和7 个Wnt亚家族,这可能是因为它们在整个进化过程中具有更快的进化速度从而导致了大量的Wnt基因丢失。相反,Wnt5,Wnt7和Wnt11亚家族在鲎和蝎子中具有多个基因的拷贝,表现出了较大程度的扩张,而在其它节肢动物(如昆虫和甲壳类动物)中则表现为单拷贝。对这些螯肢动物中扩张的Wnt亚家族的具体功能研究正在进行当中。
4 结论
在本研究中,我们对螯肢动物中的Wnt基因家族做了全基因组的生物信息学鉴定和分析,从螯肢动物的8 个物种中共鉴定出了87 个Wnt基因。我们进一步分析了这些Wnt基因的基因结构,发现这些基因包含高度保守的Wnt结构域、信号肽、跨膜区域或低复杂度区域。对这些Wnt基因在基因组上的分布研究发现,在螯肢动物中的Wnt基因簇主要存在两种连锁分布模式(Wnt9-Wnt6-Wnt1或Wn5-Wnt7),并且还有一些谱系特异性的基因簇。螯肢动物中的Wnt基因家族的系统发育分析结果显示了Wnt5、Wnt7和Wnt11的扩张以及Wnt3和Wnt10的缺失,这些发现为进一步研究螯肢动物中的Wnt基因家族的功能提供了基础。
