基于线粒体ND2 基因的龙头鱼群体遗传多样性分析
2020-10-21蒋艳琳朱文斌杨天燕
蒋艳琳,孟 玮,朱文斌,杨天燕
(1.浙江海洋大学水产学院,浙江舟山 316022;2.浙江海洋大学海洋与渔业研究所,浙江省海洋水产研究所,浙江舟山 316021)
龙头鱼Harpodon nehereus,俗称虾潺、狗母鱼、豆腐鱼等,属灯笼鱼目Myctophiformes,龙头鱼科Harpodontidae,龙头鱼属Harpodon,为近岸暖水性小型经济鱼类,主要栖息于大陆架水域下层,广泛分布于西太平洋及印度洋海域,主要包括日本、韩国及中国沿海海域[1],我国尤以浙江舟山及乐清一带分布较广且产量较高,主要生产季节龙头鱼渔获物所占比例高达70%以上[2]。20 世纪70 年代以来,随着带鱼、大黄鱼、乌贼等主要渔业资源的衰退,龙头鱼等次要的经济种类数量逐渐增加,并以其独特的商业价值与营养价值逐渐被人们挖掘与喜爱[3]。目前国内外关于龙头鱼的研究多集中在外部形态特征[4]、渔业生物学特性[5-7]、资源数量分布[8-10]、摄食习性[11]和食品加工[12-15]等方面,而遗传背景相关研究报道较少,仅见线粒体基因组全序列[16]和分子标记技术开发应用[17-20]。NADH 脱氢酶第二亚基(NADH dehydrogenase subunit 2,ND2)是线粒体电子传递复合体的重要组分,也是线粒体上13 个蛋白质编码基因之一[21],因具有进化速率适中且包含丰富的遗传信息等优点,目前被广泛运用于鱼类遗传关系和系统分类地位的研究[21-24],显示出其良好的分子标记特性。了解和掌握渔业种群结构及其变化规律,是开展资源开发管理的基础和前提,而遗传结构则是种群结构的内在体现,综合反映了种群大小组成、繁育系统、隔离程度及迁移格局的时空[25]。本研究以线粒体ND2 基因为分子标记,对我国5 个地理种群的龙头鱼遗传结构和变异进行了比较分析,了解群体之间的遗传分化和种群历史动态,旨在揭示我国近海龙头鱼种质资源现状,为保护并合理开发利用龙头鱼资源提供参考资料。
1 材料与方法
1.1 样本采集
98 尾实验样本分别于2018年7 月-2019 年4 月间采自于我国沿海5 个地点,分别为海口(HK,18 尾)、南通(NT,22 尾)、青岛(QD,23 尾)、泉州(QZ,14 尾)和湛江(ZJ,21 尾)。采样地点详见图1。剪取适量背部新鲜肌肉组织浸泡于无水乙醇中,置于4 °C 冰箱保存。
1.2 基因组DNA 提取和PCR 扩增
采用传统的Tris 酚-氯仿法[26]进行基因组DNA 的提取,充分洗涤后将自然晾干的DNA 溶解于100 μL 灭菌水中,置于-20 °C 冰箱中保存备用。根据Genbank 数据库中龙头鱼线粒体基因组全序列(Genbank 登录号:JX534239),为保证PCR 扩增和拼接序列的准确性,采用Primer premier 软件[27]设计扩增2 对ND2 基因的引物,送至上海生工生物工程有限公司进行合成,具体引物序列信息如表1 所示。
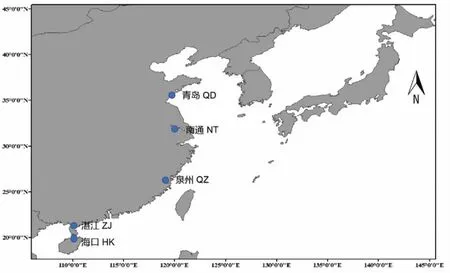
图1 龙头鱼采样图Fig.1 The sampling locations of H.nehereus

表1 扩增ND2 基因引物信息Tab.1 The information of ND2 primers in this study
PCR 反应体系为25 μL,其中包括2.5 μL 10×Buffer,2 μL dNTP,0.25 μL Taq 酶,上下游引物各1 μL,DNA 模板1 μL,最后加入17.25 μL 灭菌的超纯水补足至终体积25 μL。PCR 扩增在Bio-Rad C1000M Thermal Cycler 仪器上进行,扩增条件为:95 °C 预变性5 min,然后95 °C 变性30 s,50 °C (52 °C)退火30 s,72 °C 延伸75 s,共35 个循环,最后72 °C 延伸5 min。扩增产物用1%的琼脂糖凝胶电泳检测其大小和纯度。凝胶呈像系统拍照后,挑选条带亮度较高的PCR 产物送至上海美吉生物有限公司进行双向测序。
1.3 数据分析
应用DNAstar[28]软件包对序列进行编辑、排序和校对,得到所有龙头鱼样品的线粒体ND2 基因全序列。利用DnaSP 6.0[29]软件和Arlequin 3.5[30]确定单倍型数目(number of haplotype)并计算单倍型多样性(haplotype diversity,Hd)、核苷酸多样性(nucleotide diversity,Pi)、平均核苷酸差异数(average number of nucleotide differences,K)。利用Mega 5.0[31]软件计算碱基转换数 (number of transition,Ts) 和碱基颠换数(number of transversion,Tv),并统计碱基组成、变异位点数(variable site)、简约信息位点数(parimony informative sites)和群体间遗传距离。采用邻接法(neighbor-joining,NJ),以Kimura 双参数模型(kimura-2-parameter,K2P)为基础,构建系统发育树,同时利用单倍型最小跨度树来分析单倍型间的遗传关系[32],为保证系统树的可靠性,对数据进行1 000 次自举检测(bootstrap analysis,BP)。
利用Arleqwin 3.5 软件进行Tajima's D 和Fu's Fs 中性检验,并且通过分子方差分析(analysis of Molecular Variance,AMOVA)对遗传变异的分布模式进行分析,计算群体内和群体间遗传分化指数。利用所得数据进行种群动态研究,分析龙头鱼群体是否经历过种群扩张,若经历过种群扩张,运用公式τ=2 ut来计算龙头鱼种群扩张时间[33]。其中τ 代表扩张时间参数(Tau),u 代表进化速率,t 代表自扩张以来所经历的代数,扩张时间T=t×代时;u=μ k,其中μ 代表单位碱基的变异速率,k 代表所分析序列的长度。
2 结果与分析
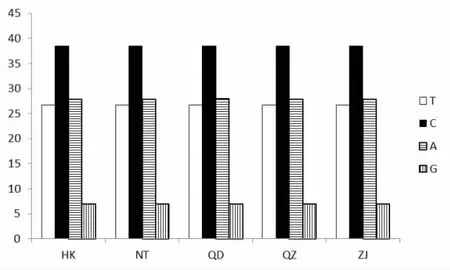
图2 龙头鱼ND2 基因密码子第3 位点碱基组成(%)Fig.2 Nucleotide composition of the 3rd codon in ND2 gene of H.nehereus(%)
2.1 序列特征分析
对5 个群体共98 尾龙头鱼线粒体ND2 基因序列进行比对分析,共获得有效长度为1 046 bp 的龙头鱼ND2 基因序列,未发现碱基的插入与缺失。全部位点中,变异位点(variable sites)19 个,单一可变位点(singleton variable sites)18 个,简约信息位点(parsimony informative sites)1 个。18 个碱基发生了转换,主要发生在A-G 之间;2 处碱基发生颠换,均为A-T;转换数高于颠换数,与线粒体基因组DNA 进化过程中转换频率高于颠换的规律一致[34]。4 种碱基平均含量分别为:T 29.2%、C 34.9%、A 24.4%、G 11.6%。其中G 碱基的含量偏低,表现出明显的反G 偏倚。A+T 的含量为53.6%,略高于G+C 的含量,与大多数脊椎动物线粒体DNA 碱基G+C 含量位于37%~50%之间的情况一致[35]。从密码子碱基组成情况来看,不同群体间也存在较大差异,表现出强烈的偏倚性。第1 密码子中,C、A 碱基使用频率较T、G 高;第2 密码子中,碱基的使用明显偏向T 和C;第3 密码子中C 碱基含量最高,而G 碱基含量显著偏低(图2)。
98 尾个体共定义了19 种单倍型,各单倍型分布情况如表2 所示。其中Hap1 出现频率最高,其次为Hap3,由海口、青岛和湛江3 个群体共享,其余每种单倍型均为一个群体所独有。5 个地理群体中,单倍型类型最多的为湛江群体(8 种),最少的是泉州群体,仅有2 种单倍型(Hap1 和Hap13)。
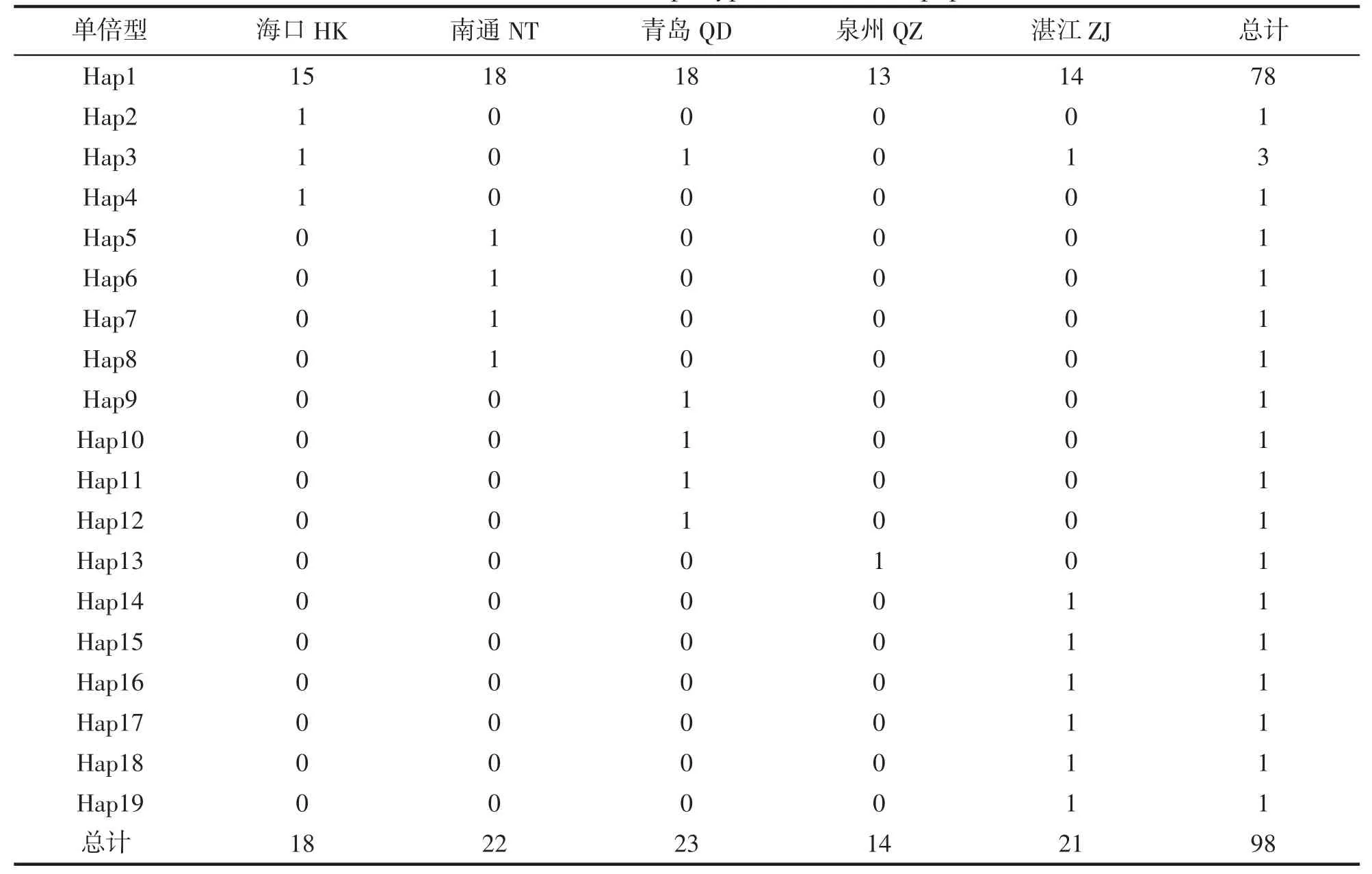
表2 单倍型在不同群体中的分布Tab.2 The distribution of haplotypes in different populations
2.2 遗传多样性与分化研究
单倍型多样性(Hd)和核苷酸多样性(Pi)是评价种群遗传多样性的两个重要依据[36]。本研究中,5 个群体龙头鱼遗传多样性如表3 所示,总体单倍型多样性(Hd)和核苷酸多样性(Pi)分别为0.367 6±0.063 8 和0.000 427±0.000 424。从总体上来看,不同群体间遗传多样性呈现出相同的趋势,由高到低依次为湛江>青岛>南通>海口>泉州,其中湛江群体单倍型多样性和核苷酸多样性均最高,分别为0.566 7±0.128 8 和0.000 723±0.000 618,泉州群体单倍型多样性和核苷酸多样性均最低,分别为0.142 9±0.118 8 和0.000 138±0.000 236。
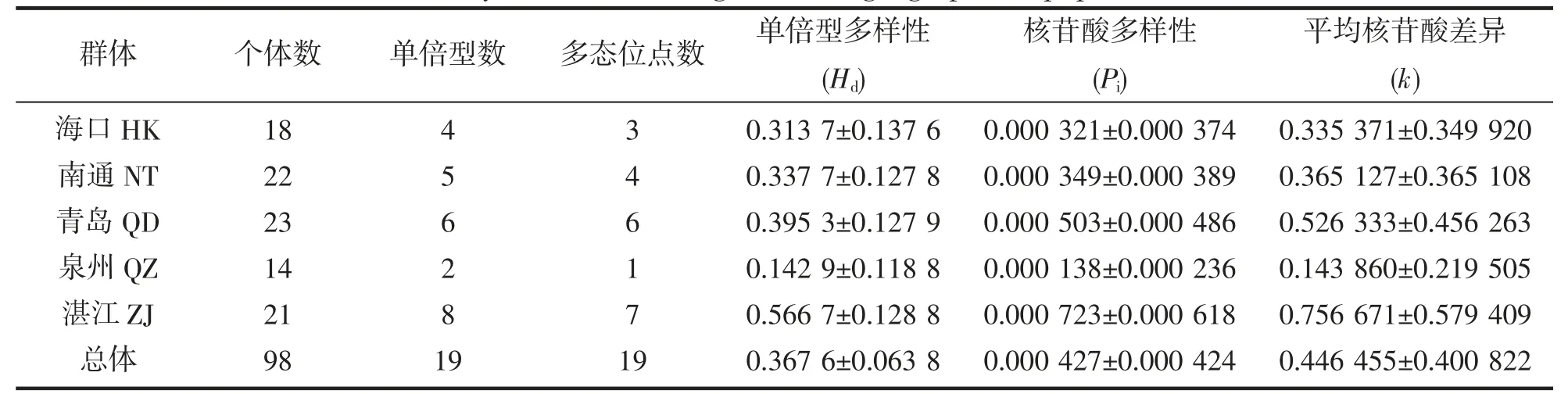
表3 龙头鱼5 个地理群体ND2 基因序列遗传多样性指数Tab.3 Genetic diversity index on ND2 gene for 5 geographical populations of H.nehereus
遗传分化指数FST值是反映各群体间遗传分化程度高低的重要指标,能够衡量群体中是否存在随机交配,进一步揭示群体间的遗传分化情况。Wright 认为:当0<FST<0.05,表明群体间不存在遗传分化;当0.05<FST<0.15,表明群体间存在低度的遗传分化;当0.15<FST<0.25,表明群体间存在中度遗传分化;当FST>0.25 时,表明群体间存在高度遗传分化[37]。利用DnaSP 软件计算5 个地理群体龙头鱼遗传分化值数FST值及对应的P 值详见表4。由表中结果可知,群体间遗传分化系数FST值介于-0.014 46~0.009 29 之间,FST值均小于0.05,且两两群体间P 值不显著,表明龙头鱼群体之间不存在显著遗传分化,可能与群体之间存在一定的基因交流,群体间进行随机交配有关。
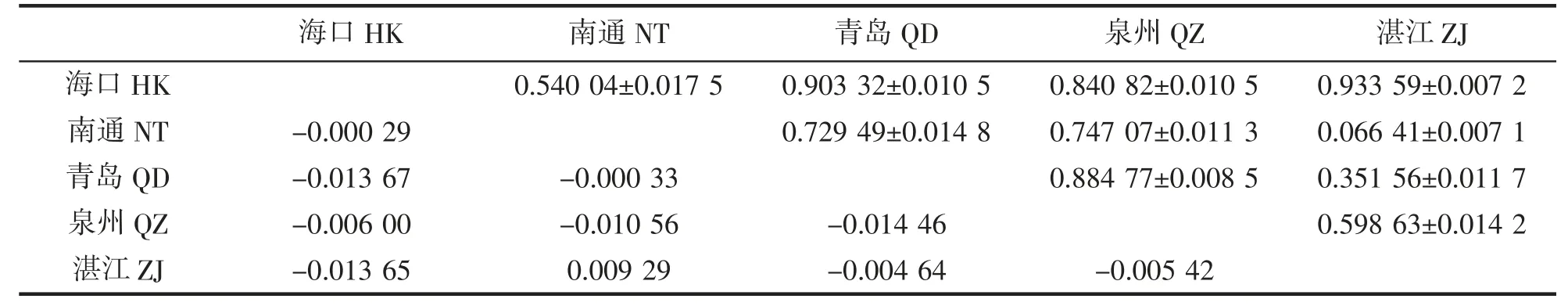
表4 龙头鱼群体间的FST 值(对角线下方)和P 值(对角线上方)Tab.4 Pairwise FST(below diagonal)and associated P(above diagonal)values among populations of H.nehereus
为了解龙头鱼遗传变异的地理分布模式,采用Arlequin 软件对龙头鱼群体进行了AMOVA 分析(表5)。结果显示种群内变异占99.68%,而种群间变异仅为0.32%,表明遗传变异主要来自于种群内,群体之间遗传分化不明显。
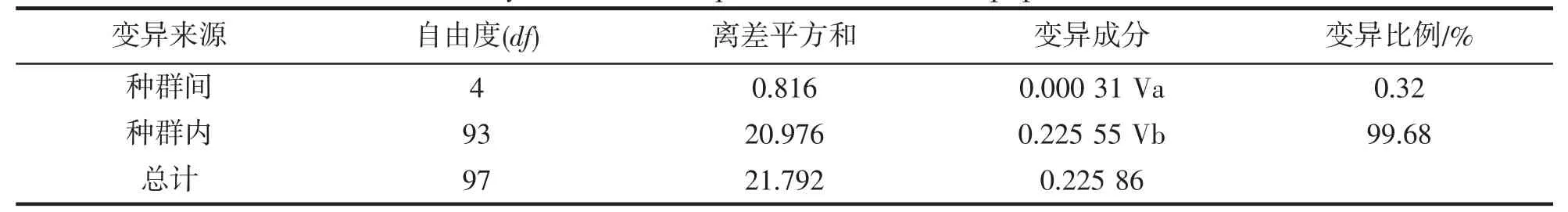
表5 龙头鱼不同种群遗传变异的分子变异分析Tab.5 AMOVA analysis of ND2 sequences in different populations of H.nehereus
2.3 遗传距离和系统发育关系探讨
物种的进化能力及对环境的适应性不但取决于种群内遗传变异,而且依赖于种群结构[38]。利用Mega软件计算不同群体间K2P 平均遗传距离见表6。由表可知,5 个地理群体的龙头鱼遗传距离介于0.000 228~0.000 607 之间,结果表明海口、南通、青岛、泉州及湛江群体间的遗传距离均较小,遗传分化均不明显,不同地理群体的龙头鱼尚未达到种的分化。
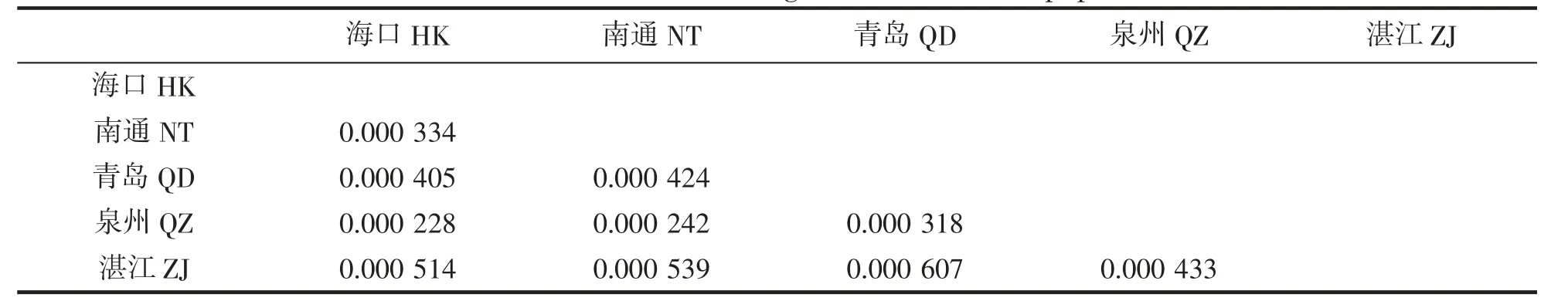
表6 龙头鱼5 个地理群体间遗传距离Tab.6 Genetic distances among five H.nehereus populations
从NCBI 数据库下载近缘物种狗母鱼科Synodontidae、狗母鱼属Synodus 的杂斑狗母鱼Synodus variegatus(GenBank 登录号:AY524977)和蛇鲻属Saurida 的鳄蛇鲻Saurida wanieso(GenBank 登录号:KP027408)作为外群,基于Kimura 双参数模型(kimura 2-parameter),利用NJ 法构建系统发育树。通过100 0 次Bootstrap 统计分析,比较分支之间的支持率,支持率数值越高,则表明该分支的支持率也越高[39]。结果显示龙头鱼19 个单倍型在系统树上聚成一支,未形成明显的地理分化(图3)。
2.4 种群历史动态分析
种群动态反映种群数量在时间和空间上的变化,把握种群动态的规律有利于种群种质资源的合理利用与有效保护,制定切实可行的捕捞计划。本研究采用Tajima's D 与Fu's Fs 检验,预测历史上种群是否发生扩张并进一步估算种群扩张时间。Tajima's D 值与Fu's Fs 值若为负值且达到显著性水平(即P<0.05),则表明该种群可能经历过群体扩张[40]。中性检验结果表明,5 个群体的Tajima's D 值和Fu's Fs 均为负值,且除泉州群体外,其余4 个群体P 值均显著(P<0.05),推测历史上海口、南通、青岛和湛江群体龙头鱼曾经历过种群扩张(表7)。参考ND2 基因2.5%~3.6%每百万年的变异速率[23,41],龙头鱼通常1 龄达到性成熟[1],代时取1,则扩张时间T=t,据此估算出龙头鱼发生种群扩张的时间约在(1.87~1.30)万年前的第四纪更新世晚期。
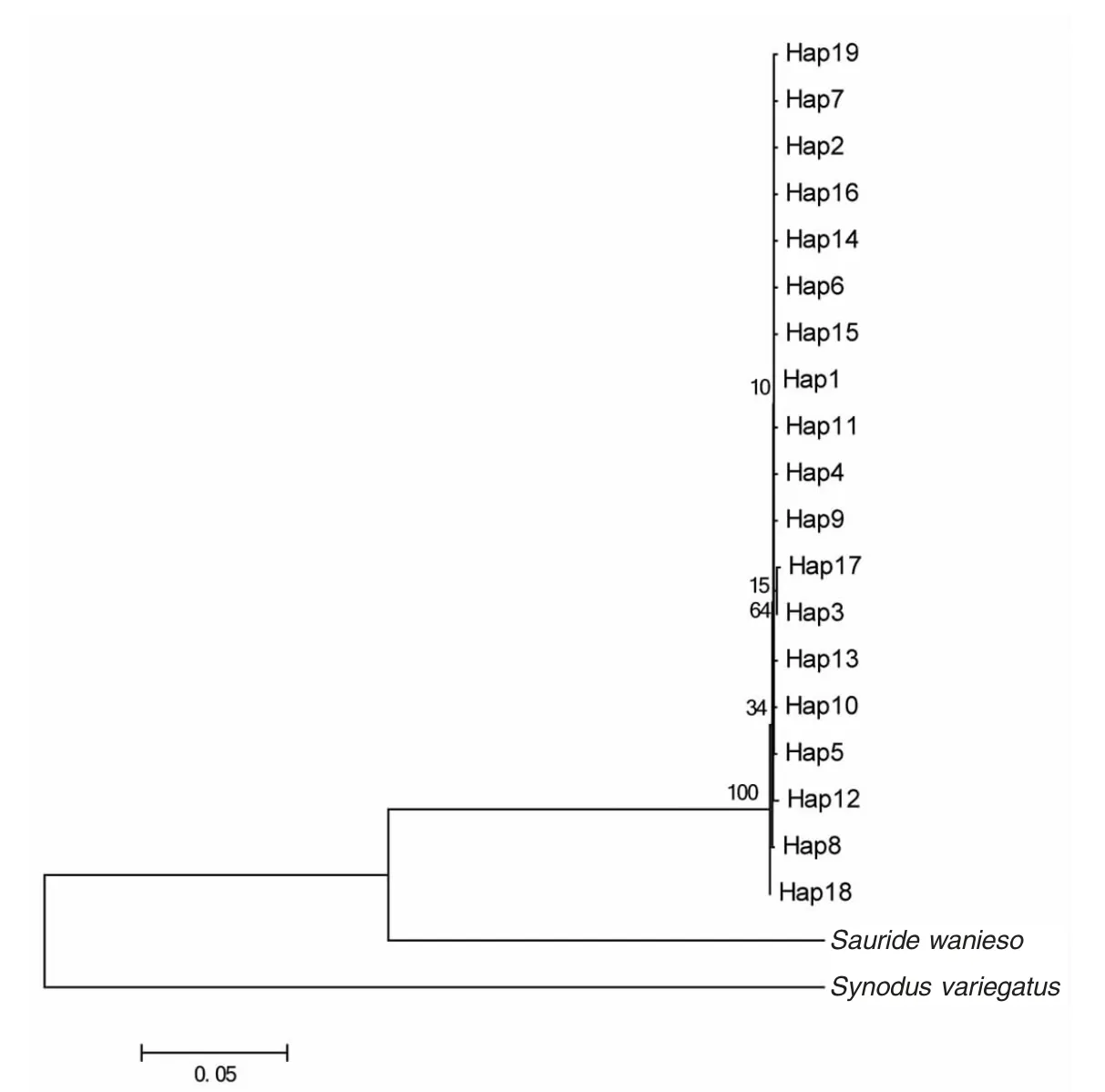
图3 基于线粒体ND2 基因序列构建的单倍型邻接树Fig.3 NJ tree of haplotypes based on mtDNA ND2 sequences
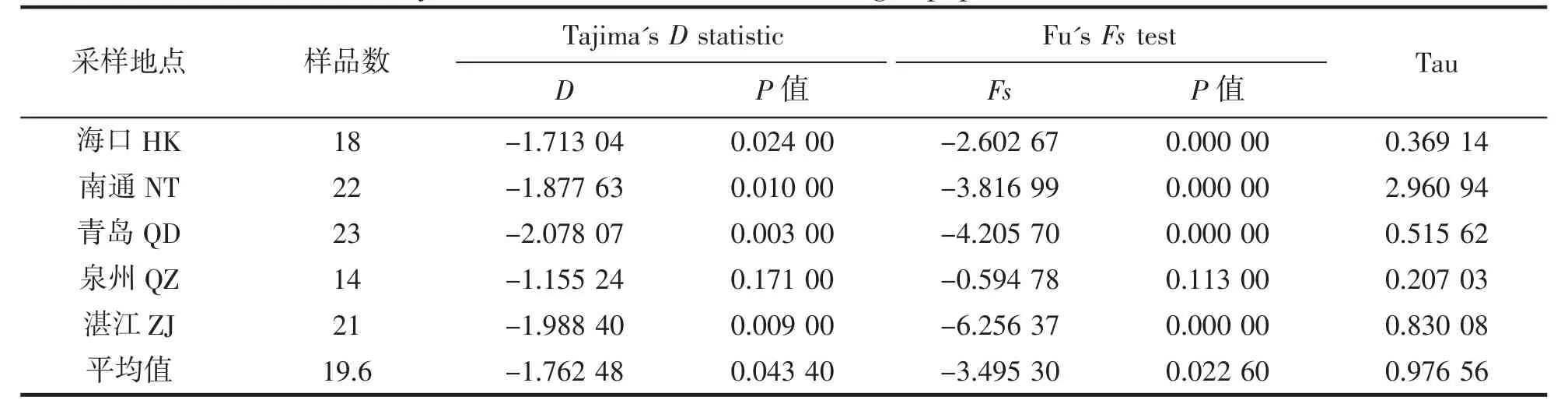
表7 5 个地理群体龙头鱼中性检测结果Tab.7 Tajima's D and Fu' Fs values among 5 populations of H.nehereus
3 讨论
遗传多样性是衡量物种适应环境能力的关键指标,在生物进化过程中起到内在动力的作用,是物种生存与进化的潜在遗传基础。遗传多样性程度越丰富,物种对环境的适应能力越强,生存与进化能力也越强。反之,遗传多样性越贫乏,表明物种对于环境的适应、生存及进化能力都会降低,甚至会威胁到物种的生存[42]。与其他海洋鱼类相比,本研究中龙头鱼呈现出低单倍型多样度、低核苷酸多样度的格局,与郭易佳等[20]采用线粒体Cyt b 基因分析我国近海龙头鱼种群遗传结构获得的结果一致,表明该鱼类遗传多样性整体处于较低水平。此外,龙头鱼的遗传多样性(0.367 6±0.063 8)与核苷酸多样性(0.000 427±0.000 424)相较于香鱼Plecoglossus altivelis (0.708±0.041,0.002 42±0.000 33)[43]和少鳞鱚Sillago japonica (0.945 3±0.015 5,0.009 718±0.005 445)[44]都偏低,一定程度上也反映出龙头鱼较低的遗传多样性。
龙头鱼两两群体间平均遗传距离较小(0.000 228~0.000 607),AMOVA 分析结果也显示遗传变异主要来自于种群内个体间,表明龙头鱼不同地理群体间亲缘关系较近,NJ 聚类树也未形成明显的分支,推测我国沿海龙头鱼可能来自于相同的祖先。从龙头鱼遗传分化情况来看,两两群体间遗传分化系数均为负值,推测可能是漂浮性鱼卵和仔鱼期易受到洋流等因素影响,群体间产生较为频繁的基因交流引起同质化现象,导致遗传分化不显著。
采用Tajima's D 和Fu's Fs 中性检验对ND2 序列进行种群扩张事件检测,结果表明除泉州群体外,其余4 个群体的Tajima's D 值和Fu's Fs 值均为负且P 值小于0.05,历史上这些海域的龙头鱼可能经历过种群扩张事件,估算扩张时间在(1.87~1.30)万年前的第四纪更新世晚期。推测扩张事件与这一时期冰期与间冰期循环出现,海洋地理环境发生变化,从而引起的地理隔离、种群基因交流受到阻碍有关,使得龙头鱼种群出现了分化与扩张[45]。
台湾海峡作为我国重要的海上交通要道,被称为海上交通的“咽喉”,频繁的海上航运与交通运输,对位于台湾海峡西岸的泉州沿岸龙头鱼群体的生存栖息环境产生一定影响。此外,台湾海峡海底蕴藏着丰富的石油资源,泉州周边石油开采和化工企业的生产运营也给当地海域造成了难以避免的危害,上述原因或许与泉州群体龙头鱼遗传多样性水平偏低有一定关系。作为我国近海重要的小型经济鱼类,为实现龙头鱼种质资源的保护与管理,需要根据实际情况制定切实可行的保护方案。首先,从栖息环境方面考虑。应加强对栖息地环境保护,规范海上作业方式,减少海洋环境污染,维持生态平衡,避免种质资源遭受更大的危害[46]。其次,面对日益增加的捕捞压力,可以从捕捞方式手段来考虑。实行捕捞许可证,限制捕捞工具的使用规格,采用合适的捕捞方式;实行休渔期、禁渔期,禁止产卵期捕捞作业,加强对产卵场的保护[41]。另外,也有必要强化人们的法律意识,开展普法宣传教育,加强渔业立法及渔业管理,加大执法力度,实现渔业生产的高效管理[22]。
